Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.57 no.4 Palmira Oct./Dec. 2008
Artículo de revisión
Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia. AA 237. Palmira, Valle del Cauca, Colombia. Autor para correspondencia: jemunozf@palmira.unal.edu.co; acmorilloc@palmira.unal.edu.co; ymorilloc@palmira.unal.edu.co
RESUMEN
Se revisó el uso e importancia, ventajas, desventajas y características de la técnica Microsatélites Amplificados al Azar (RAM) en uchuva Physalis peruviana, mora Rubus spp, guayaba Psidium guajava y heliconias Heliconia spp. En mora se diferenciaron las especies R. glaucus, R. robustus y R. urticifolius, se detectaron duplicados y se encontró alta variabilidad genética en R. glaucus, la especie más importante. En uchuva se encontró alta diversidad y dos accesiones de fruto rojo que se diferenciaron genéticamente de las amarillas y una región geográfica con alta variabilidad. En guayaba los cebadores fueron altamente polimórficos y se encontró alta variabilidad en el Valle del Cauca. En heliconias y especies relacionadas se diferenciaron las familias del orden Zingiberales, algunos subgéneros y variaciones en la especie. La técnica es de bajo costo, utiliza un cebador, no requiere información previa, es altamente polimórfica y diferencia especies en los taxones evaluados.
Palabras claves: Physalis peruviana; Psidium guajava; Rubus spp; Heliconia spp; marcadores moleculares; análisis genéticos.
ABSTRACT
The use and importance, advantages, disadvantages and features of the Random Amplified Microsatellites RAMs technique, were reviewed in Cape gooseberry Physalis peruviana, blackberry Rubus spp, guava Psidium guajava and heliconias Heliconia spp. In blackberry, we differentiated the species R. glaucus, R. robustus y R. urticifolius, detected duplicated accessions and found high genetic diversity in R. glaucus, the most important specie. In cape gooseberry we found high diversity and two red fruit accessions genetically differentiated from the yellow fruit ones and a geographical region with high variability. In guava, primers were highly polymorphic and found high variability in Valle del Cauca region. In Heliconia and related species we differentiated families belonging to Zingiberal order, between some sub genera and variation among specie. The technique has low cost of implementation, use a single primer, do not require previous information, is highly polymorphic and can differentiate species is taxa were technique was evaluated.
Key words: Physalis peruviana; Psidium guajava; Rubus spp; Heliconia spp; molecular markers; genetic analysis..
Entre las técnicas disponibles para estudiar la diversidad genética vegetal se encuentran los RAPD (Random Amplified Polymorphic DNA), AFLP (Amplified Fragment Length Polymorphism), microsatélites y los RAM (Random Amplified Microsatellites). En los últimos diez años los microsatélites han cobrado importancia en estudios que van desde caracterización individual hasta mapeo genómico. Debido al alto nivel de polimorfismo, carácter codominante, facilidad de aplicación y alta reproducibilidad son marcadores ampliamente utilizados y muy informativos. En la identificación de individuos estrechamente relacionados, clones o cultivares se estima que los microsatélites tienen poder de discriminación siete veces mayor que los RAPD (Kraic et al., 1998) y se consideran los marcadores ideales para estudios de mapeo de ligamiento (Hearne et al., 1992; Morgante y Olivieri, 1993) y evolutivos.
En plantas los oligonucleótidos, compuestos por elementos repetitivos del tipo TG y GATA/GACA, detectan polimorfismo cuando se utilizaban como sondas RFLP (Restriction Fragment Length Polymorphism). Los sitios de microsatélites se distribuyen ampliamente con una frecuencia de 1 cada 50.000 pares de bases; se presentan en 34 especies vegetales, y el elemento repetido más común es el dinucleótido AT (Morgante y Olivieri, 1993).
Con marcadores basados en microsatélites se han elaborado mapas genéticos de soya, arroz y trigo (Akkaya et al., 1992; Roder et al., 1995), Pinus spp (Smith y Devey, 1994) y Citrus spp (Ferreira y Grattapaglia, 1998). Se han desarrollado microsatélites para C. sinensis (Novelli et al., 2006), C. limon (Golein et al., 2005) y mandarinas (Koehler-Santos et al., 2003).
Los Microsatélites Amplificados al Azar (RAM) se utilizan en caracterización de diversidad genética en microorganismos, animales y plantas. La revisión describe la técnica RAM, analiza los elementos asociados con la técnica (calidad, cantidad de ADN, condiciones de amplificación, electroforesis, análisis estadístico) y presenta la aplicación de la técnica en mora Rubus spp, Uchuva Physallis peruviana, guayaba Psidium guajava y heliconias Heliconia spp.
Zietkiewicz et al. (1994) propusieron un método para medir la diversidad genética en plantas y animales usando cebadores basados en microsatélites. Esta técnica combina los beneficios de los microsatélites y los RAPD (Random Amplified Polymorphic DNA). Hantula et al. (1996) seleccionaron cuatro primers (GT, ACA, CCA, CGA) con una longitud de 18 bases que incluyen un extremo 5' "degenerado" de tres nucleótidos, el cual sirve de anclaje para asegurar la unión del primer al inicio del microsatélite. Ellos denominaron a esta técnica RAM (Random Amplified Polymorphic DNA).
Los RAM se basan en la reacción en cadena de la polimerasa (PCR), el método es altamente reproducible y permite la detección de polimorfismo en el ADN intra e interespecífico. Los fragmentos de ADN amplificados en la reacción están compuestos de dos microsatélites suficientemente cercanos para amplificar por PCR el área entre ellos (Zietkiewicz et al., 1994).
La base genética de estos marcadores es la misma de los RAPD, es decir, utiliza un solo primer de secuencia arbitraria, por lo cual la secuencia blanco también es desconocida; difiere de los RAPD porque estos tienen menor tamaño del cebador que los RAM. Al igual que los RAPD, los RAM detectan un solo alelo por locus.
El poder de resolución de los marcadores genéticos está determinado por el nivel de polimorfismo que pueden detectar. Esto se ve afectado principalmente por la frecuencia de mutación en los sitios del genoma involucrados. Como el rango evolutivo entre los microsatélites es considerablemente más alto que en la mayoría de los otros tipos de ADN, hay gran probabilidad de hallar polimorfismo mediante los RAM que por otras técnicas, incluyendo los RAPD. La fuente de variabilidad en los fragmentos obtenidos por la técnica RAM no es aún conocida pero hay tres posibilidades (Hantula et al., 1996):
- Las mutaciones en el sitio de unión podrían evitar la amplificación de un fragmento como en los marcadores RAPD. Esto es posible pero puede ser menos probable con los microsatélites porque la variabilidad en estos elementos resulta principalmente de diferencias en el número de repeticiones.
- El resultado de una inserción o supresión entre el fragmento amplificado podría originar amplio polimorfismo o ausencia de productos, dependiendo de la habilidad de amplificación.
- La variabilidad en el número de repeticiones del microsatélite podría determinar el tamaño del polimorfismo
Extracción de ADN
La aplicación de las diferentes técnicas para el análisis del genoma (construcción de librerías, RFLP, PCR, AFLP) depende de la habilidad para obtener ADN puro, con alto rendimiento y buena calidad. En general las técnicas basadas en marcadores microsatélites requieren bajas cantidades de ADN (10 - 100 ng por reacción). Técnicas en las cuales la cantidad y la calidad del ADN son factores determinantes en los procesos de amplificación obligan a la utilización de kits comerciales aumentando costos.
ADN de calidad óptima no ocasiona problemas en la amplificación; por encima e incluso por debajo de los 5 ng se obtienen buenos resultados. Por el contrario, ADN degradado o ligado a contaminantes provenientes de la extracción (fenol, detergentes, etc.) puede inhibir la actividad de la polimerasa. La precipitación con etanol y lavados con etanol al 70% eliminan contaminantes e inhibidores, pero conducen a pérdidas en la concentración inicial del material.
En algunas especies la presencia de polimerasas, ligasas, endonucleasas de restricción y polifenoloxidasas dificulta la obtención de ADN. En guayaba la gran cantidad de sustancias fenólicas provocó la oxidación rápida de los tejidos. Para obtener ADN de buena calidad se utilizó el kit DNeasy plant (QUIAGEN, 2004). (Sanabria et al., 2006).
Cuantificación del ADN
La cantidad de ADN se puede determinar mediante fluorómetro, modelo DYNA QUANT 200 marca Hoefer, que usa como estándar de calibración ADN Lambda a 100 ng/ml. Otra metodología utiliza el ADN del bacteriófago Lambda de concentración conocida y por comparación determina la concentración aproximada de cada muestra.
El rango de concentraciones generalmente debe estar entre 0.01-1 ng para ADN de plásmido o fago y 25-50 ng para DNA genómico en 25 ml. En técnicas basadas en marcadores microsatélites se utilizan bajas concentraciones de ADN molde (10 ng/ml).
Con el método de extracción descrito por Dellaporta et al. (1983) se obtuvieron en mora entre 20 y 100 ng/uL en un volumen final de 80 uL, cantidad suficiente para realizar al menos cuatro reacciones (Morillo et al., 2005).
Condiciones de amplificación
Para este tipo de marcador molecular se utilizan cebadores de secuencias cortas repetidas y conocidas, de una longitud de 18 bases que incluyen un extremo 5' "degenerado", pues son secuencias complementarias a una región del genoma (Tabla 1). Las técnicas basadas en marcadores microsatélites utilizan primers que contienen de 10 a 30 nucleótidos y entre 40-60% en GC (Guanina-Citocina). En algunos casos puede ser importante tener zonas ricas en GC en el extremo 3' del primer, de forma que el alineamiento se favorece en las áreas implicadas en la elongación.

El coctel de amplificación para la reacción de microsatélites RAM se prepara en un volumen final de 25 ml que contiene dNTPs (0.2 mM), 1X PCR Buffer, 1 unidad de Taq Polimerasa, 2 mM de Primer, 10 ng de ADN total y 1.5 mM de MgCl2 y se lleva a volumen con agua HPLC. Aunque las condiciones pueden variar dependiendo de la especie estudiada y de cada situación (Tabla 2). En experiencias recientes se utilizaron con buenos resultados 12.5 ml de volumen final.
En la Reacción en Cadena de la Polimerasa (PCR) algunos factores pueden afectar el proceso de amplificación o multiplicación in vitro del ADN.
Por lo general el Buffer de reacción 10x está formado por 100 mM Tris-HCl (pH entre 8 y 9 a 25°C), 500 mM KCl, 1% de Triton X-100 y 15mM de MgCl2. El Buffer Tris regula el pH de la reacción que afecta la actividad y fidelidad de la enzima Taq polimerasa. Moderadas concentraciones de KCl pueden incrementar la actividad de la enzima de 50% a 60%, la concentración óptima es de 50mM (Binder, 1997; Henegariu, 2000). El uso de aditivos aumenta la especificidad y fidelidad de la PCR.
La adición de betamina, dimetilsulfóxido (DMSO) y formamida es beneficioso cuando el ADN molde posee regiones ricas en GC y forma potentes estructuras secundarias que detienen la polimerasa (Rees et al., 1993). Como el ADN con regiones ricas en GC puede ocasionar ineficiente separación de las cadenas, es benéfico el uso de aditivos, pues la betamina reduce la cantidad de energía requerida para separar las cadenas del ADN molde y DMSO y formamida interfieren en la formación de enlaces de hidrógeno entre ambas cadenas. También se pueden usar detergentes para estabilizar la enzima como el Tween 20, Laureth 12 (0.1%) o Tritón X-100. En otros protocolos se incorporan polietilenglicol (PEG), glicerol, seroalbúmina bovina (BSA) como proteína captadora de iones inhibidores de la Taq polimerasa (Lozada, 2002; Promega, 2005).
El volumen de la reacción es variable, aunque en ocasiones se recomienda emplear volúmenes pequeños con pocas cantidades de ADN (Henegariu, 2000).
La concentración de magnesio afecta el funcionamiento de Taq ADN polimerasa. Los componentes de la reacción tales como el ADN molde, agentes quelantes presentes en la muestra (EDTA o citrato), dNTP y proteínas pueden afectar la cantidad de magnesio libre (Henegariu, 2000; Promega, 2005). La carencia puede inactivar la enzima, y el exceso reduce la fidelidad de la enzima y puede incrementar las uniones inespecíficas (Eckert y Kunkel, 1990). La concentración óptima de la sal se encuentra en el rango de 1.5 a 3.0 mM.
Los dNTP (dATP, dCTP, dGTP, dTTP) se deben añadir en iguales concentraciones, puesto que pueden captar iones Mg2+. El desequilibrio en las concentraciones en sólo un dNTP baja el rendimiento de la reacción y favorece la incorporación errónea de nuevas bases, aumenta la tasa de error de copia. La concentración más utilizada es de 0.2 mM (Brinkmann Instruments, 2004; Henegariu, 2000).
Existen diferentes tipos de ADN polimerasa que replican el ADN. Estas se pueden clasificar en Termolábiles (37°C - 42°C) y termoestables (74°C, resiste entre 40 - 50 segundos a 96°C) (Lozada, 2002). Una de las más utilizadas ha sido la enzima termoestable Taq ADN polimerasa, aislada de la bacteria Termus aquaticus (Taq). Carecen de actividad 3'-> 5' exonucleasa, lo que las hace menos seguras a la hora de comparar las fidelidades. Para garantizar el funcionamiento se recomienda no usar alto número de ciclos, iguales concentraciones de dNTP, disminuir la duración de cada etapa y la concentración de MgCl2 debe oscilar entre 1.5 mM y 3.0 mM (Binder, 1997).
Se recomienda utilizar entre 1 y 1.25 U de enzima en 50ml de reacción. Incrementos en la cantidad y el tiempo de extensión generan la aparición de artefactos debido a la actividad exonucleasa intrínseca 5&39; -> 3&39; (Promega, 2005; Lozada, 2002). En la (Tabla 3) se describe el perfil de amplificación de la técnica.
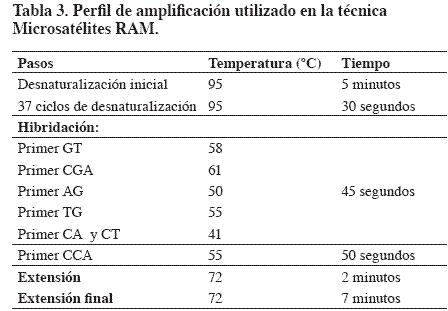
Se utiliza un primer (Figura 1 ) y se amplifican dos secuencias microsatélites; en la cadena B se observan las secuencias microsatélites AC y GT.
Los productos de amplificación se separan por electroforesis en geles de agarosa al 1.2% a 90 voltios durante tres horas, visualizándose en un transiluminador, o en geles de poliacrilamida 19:1 (acrilamida: bisacrilamida) al 8%, teñidos con sales de plata.
Electroforesis
La migración de los fragmentos de ácidos nucleicos (ADN o ARN) en un gel de agarosa sometido a un campo eléctrico depende del voltaje del campo y del tamaño del poro del gel. La separación efectiva de los fragmentos de ADN o ARN (resolución) depende de la masa y de la carga de los fragmentos, en realidad, de la relación carga/masa. Transcurrida la electroforesis, la localización relativa de los fragmentos se determina mediante distintos métodos. La tinción con bromuro de etidio, una sonda fluorescente tras iluminación con luz UV, es un método generalizado, ya que la sonda se intercala entre la doble hélice de ADN y emite luz (Sambrook et al., 1989).
La electroforesis en gel de agarosa del ADN fragmentado es una técnica habitual que separa los fragmentos, los localiza en función del peso molecular por comparación con la movilidad de unos estándares de tamaño conocido, y si fuera preciso, extrae de nuevo el fragmento de interés para otras aplicaciones (amplificación, clonación, secuenciación, transfección, etc.) (Sambrook et al., 1989).
Una de las ventajas de la técnica RAM es que se pueden separar los productos amplificados utilizando geles de agarosa al 1.2% y visualizar en un transiluminador con luz ultravioleta. También se pueden separar en geles de poliacrilamida para obtener más alta resolución y mejor separación de los fragmentos, obteniendo mayor número de bandas por marcador.
El número de bandas del patrón de amplificación obtenido en agarosa es menor que en poliacrilamida. Los geles en agarosa son más fáciles de hacer, suministran buena información y son más baratos. La electroforesis en poliacrilamida requiere más agilidad y experticia, pero permite información más precisa (Sambrook et al., 1989).
El número de bandas polimórficas posiblemente se vea afectada por la técnica de electroforesis utilizada, especialmente por el polímero empleado para la creación de la matriz porosa a través de la cual se separan los fragmentos generados en la amplificación. En el trabajo realizado en mora (Rubus spp) se generaron 43 bandas polimórficas utilizando seis primers RAM en geles de agarosa. En el trabajo de caracterización de guayaba (Psidium guajava) (Sanabria et al., 2006), con estos mismos primers, usando geles de poliacrilamida, se encontraron 74 bandas polimórficas, superior a un trabajo realizado en Psidium spp por Rueda et al. (2006) con marcadores RAPD.
Análisis estadístico
El análisis estadístico consta de diferentes etapas: Análisis de similitud según el coeficiente de Nei-Li, Análisis de correspondencia Múltiple, Análisis Boot-strap, y el Análisis de Varianza Molecular (AMOVA) (Henríquez, 2000).
Similitud en término de marcadores moleculares: Coeficiente de similitud de Nei-Li. Para el estudio del conjunto de individuos, se parte de la definición de similitud de Nei-Li (Nei y Li, 1979) (Leung et al., 1993) también conocida como similitud de Dice (Sneath y Sokal, 1973) cuya fórmula es:
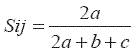
Donde:
Sij= Similitud entre el individuo i y el j.
a= Número de bandas presentes simultáneamente en los individuos i y j.
b= Número de bandas presentes en i y ausentes en j.
c= Número de bandas presentes en j y ausentes en i.
El coeficiente de Dice omite la consideración de pares negativos (0-0) y da doble peso a los pares positivos (1-1) (Sneath y Sokal, 1973), lo que hace útil en términos de similitud del ADN, en que la ausencia compartida de una banda no es necesariamente indicación de similitud entre dos individuos.
Se construye una matriz de variables binarias (cero: ausencia de bandas, uno: presencia de bandas) en la cual las accesiones forman las filas y las columnas las bandas evaluadas en cada uno de ellos.
La matriz de similitud se construye con el programa SIMQUAL del paquete "Numerical Taxonomy System for Personal Computer" (NTSYS-pc versión 1.8).
El Análisis de Correspondencia Múltiple (ACM) se realiza utilizando el paquete estadístico SAS que permite visualizar de manera clara y detallada los datos obtenidos. También facilita la representación multidimensional de un grupo de objetos o individuos con características descriptivas en un espacio determinado (el plano). Esta representación se obtiene con base en la dependencia que existe entre categorías que corresponden a las filas y columnas de una tabla de datos, de manera que la asociación se basa en variables específicas.
Desde el punto de vista operativo el análisis asocia todos los individuos con todas las características con que fueron calificados; es decir, asocia todas las filas con todas las columnas determinando el nivel de proximidad (asociación) (Joseph et al., 1992).
Cuando los dendrogramas se construyen con base en los grados de similitud entre individuos los agrupamientos no son sensibles a los procedimientos estadísticos usuales de computación. Por lo tanto, Felsenstein (1985) propuso usar bootstrapping (Efron, 1979) como camino para obtener la apreciación no paramétrica de los límites de confianza mediante el programa WINBOOT (Yap y Nelson, 1996).
El Análisis de Varianza Molecular (AMOVA) permite establecer la existencia de grupos en las poblaciones. El programa AMOVA-PREP (Excoffier et al., 1992), diseñado para automatizar la preparación de archivos de salida mediante el programa Laurent Excoffier-AMOVA, se emplea para generar matrices de distancia basados en el coeficiente de Dice y para crear archivos de distancia, grupos y población (Henríquez, 2000).
APLICACIONES DE LA TéCNICA RAM
Con los marcadores RAM se estudian poblaciones (Hantula, et al., 1996), se mide diversidad genética en plantas y animales, se muestra la base de la variación de los individuos, se permite seleccionar regiones del ADN, se permite analizar la información que se expresa y la que no se expresa ya que el número de polimorfismos detectables es teóricamente ilimitado (Mahuku et al., 2002).
Además la técnica no requiere estimación exacta de la cantidad del ADN antes de la reacción, la cual es útil cuando se analizan grandes tamaños de muestra. La metodología se ajusta para pequeños laboratorios en términos de equipos y facilidades de costo, no requiere conocimiento previo de secuencias ni uso de isótopos radiactivos (Hantula et al., 1996).
Trabajos sobre caracterización de diversidad genética vegetal sugieren que la técnica RAM es útil para identificar duplicados en bancos de germoplasma, para establecer grupos o asociaciones entre individuos de una población de acuerdo con la especie, localización geográfica, relaciones filogenéticas, entre otras.
En la colección de mora, Rubus spp, de la Universidad Nacional de Colombia, Sede Palmira, que cuenta con accesiones de R. glaucus (31), R. urticifolius (3) y R. robustus (2), Morillo et al. (2005) estudiaron la variabilidad genética utilizando seis marcadores microsatélites RAM (CCA, CGA, AG, CT, TG, y CA). El producto amplificado se observó mediante electroforesis en geles de agarosa al 1.2% a 90 voltios durante tres horas, y se utilizó una cámara CBS Scientific SGE-020-02; se visualizó con luz ultravioleta en un transiluminador.
Los seis primers produjeron 43 bandas polimórficas que reflejaron 74% de polimorfismo con pesos moleculares entre 260 Kb y 1500 Kb. El cebador TG hizo mayor aporte a la variación (Fst: 0.61). El análisis RAM diferenció materiales por especie, lugar de procedencia e identificó materiales similares (UNAPM19 y UNAPM25).
Los coeficientes de Dice y Nei-Li de nivel de similitud de 0.55 diferenciaron la población en seis grupos y 27 genotipos. El grupo formado por R. robustus presentó el valor más bajo de similitud (0.25) debido a características morfológicas o moleculares. La heterocigosidad promedio de la población total fue de 0,31, oscilando entre 0.01 (grupo F R. robustus) y 0.25 (grupo B R. glaucus); el valor de Fst (0,4980) evidenció alta variabilidad genética dentro (52%)y entre grupos (48%), debido posiblemente a migraciones humanas, flujo de genes, tipo de reproducción, polinización, entre otros. Las introducciones procedentes de los departamentos del Valle, Cauca y Nariño hicieron mayor aporte a la variabilidad genética total; el corregimiento de Juntas (Ginebra-Valle) exhibió amplia diversidad en materiales silvestres.
En la colección de uchuva Physalis peruviana L, Bonilla et al. (2008) estudiaron la diversidad genética en 43 introducciones representativas de cinco departamentos de Colombia (Nariño, Valle del Cauca, Cauca, Caldas y Cundinamarca) utilizando la técnica molecular RAM. El análisis se realizó sobre 42 loci polimórficos obtenidos con siete cebadores (GT, CT, CGA, CA, AG, TG y CCA); el tiempo de separación de los productos amplificados fue cuatro horas.
Se generaron 50 bandas en un rango de longitud de 250 a 1.000 pares de bases, incluidas ocho de carácter monomórfico que no se consideraron en el análisis. Los loci polimórficos por cebador variaron entre 1 (CCA) y 10 (GT). El loci polimórfico con un criterio del 99% fue de 97.61%. El análisis de Nei-Li, a nivel de 86% de similitud, diferenció cuatro grupos (A, B, C y D) y 24 haplotipos, los agrupamientos no se relacionaron estrictamente con el origen geográfico.
La heterocigosidad calculada para la población (0.2559) fue considerablemente alta, la cual puede estar asociada con la probable naturaleza alógama de la especie o deberse a introducciones silvestres UNPU048 y UNPU053 (de fruto rojo) de la cuenca del río Guabas (municipio de Ginebra, Valle), genéticamente muy distantes (similitud de 68%). El ACM separó cuatro grupos con coeficiente de similitud de 86%. Las 39 introducciones del grupo 1 pertenecieron a P. peruviana L.; las introducciones de fruto rojo se reunieron en el grupo 2 y las introducciones recolectadas en el departamento del Valle en los grupos 3 y 4.
Sanabria et al. (2006) evaluaron mediante RAM la diversidad y estructura genética en 53 accesiones de Psidium guajava procedentes de nueve transeptos del Valle del Cauca. Los seis primers utilizados (CT, CGA, CA, AG, TG y CGA) generaron 74 bandas polimórficas con pesos moleculares de 100 pb a 700 pb que superaron el número logrado por Rueda et al. (2006) con RAPD. En la investigación los productos de amplificación se separaron por electroforesis en geles poliacrilamida al 7%, a 160 voltios durante una hora. Se utilizó una cámara de DNA Sequencing System. FB-SEQ-3545 de FisherBiotech.
Los cebadores fueron altamente polimórficos (88-100%), presentaron alta heterocigosidad insesgada (He= 0.4025 con TG, 0.4493 con CGA y promedio 0.4386). Comparada con la diversidad media (Hs: 0.1975) en 20 accesiones de P. guajava recolectadas en América (Rueda et al. 2006) puede decirse que existe alta diversidad genética en las accesiones recolectadas en el Valle del Cauca.
El Análisis de Varianza Molecular (AMOVA) reveló que el 64.51% de la varianza total genotipica contabilizó para variaciones dentro de transeptos. El agrupamiento del análisis de clasificación no correspondió a un patrón geográfico, lo cual indica que la alta diseminación de la guayaba se debe tal vez al hombre y a los animales.
El análisis conjunto de las características morfológicas y moleculares suministró una caracterización confiable. La variación total de los descriptores morfológicos y valores moleculares fue explicada en 77.58% mediante componentes principales, en el cual las variables originales alcanzaron valores de comunalidad entre 57.22 – 95.99% en cinco variables sintéticas generadas. El Análisis de Correspondencia Múltiple caracterizó cuatro grupos, en los cuales según el análisis discriminante casi el 100% de las accesiones quedaron debidamente clasificadas.
Cobo (2004) evaluó la diversidad genética de especies de Heliconia spp con cuatro primers (GT, CGA, AG, CA) generando un patrón de 27 bandas. El número de bandas por primer varió entre cinco (CGA y CA) y nueve (GT). Los patrones de amplificación detectaron variación entre los individuos analizados. El análisis molecular confirmó el predominio de las características genéticas.
La técnica RAM discriminó el subgénero o género de los materiales y corroboró la clasificación de Krees et al. (1999); sin embargo, es necesario evaluar otros primers o marcadores moleculares. A nivel de similitud de 0.65 diferenció tres grupos y 13 haplotipos. El primer grupo se diferenció por inflorescencias erectas, características de la sección Heliconia. El segundo grupo lo conformaron las especies del subgénero Grigssia con inflorescencias de tipo pendular; a nivel de similitud de 0.55 se diferenciaron dos grupos marcados que correspondieron a las familias Strelitziaceae y Heliconiaceae.
El análisis de Correspondencia Múltiple separó a los individuos en grupos de acuerdo con las características morfológicas: especies de inflorescencia erecta conformaron un grupo cercano, las de inflorescencia pendular otro y se situaron muy distantes las del género Strelitzia.
Los marcadores RAM (Arcos et al., 2004) establecieron distancias y posibles relaciones entre especies. Según el índice de similitud Dice y upgma (Unweighted Pair Group Mean Arithmetic) se diferenciaron las familias del orden y en general los grupos correspondieron a la propuesta de Krees et al. (1999). También se diferenciaron los subgéneros Heliconia y Griggsia y las especies dentro de los subgéneros, al igual que variaciones dentro de H. latispatha roja y H. latispatha amarilla.
La técnica RAM discriminó los materiales y concordó en lo fundamental con la clasificación de Kress et al. (1999) que sitúa en el subgénero Heliconia, sección Heliconia, las especies H. wagmeriana, H. stricta, H. orthotricha, H. aurea, H. bihai; y en el subgénero Griggsia L. Andersson las especies H. rostrata de la sección Rostratae W.J. Kress, ined. y H. chartacea y H. platystachys en la sección Pendulae W.J. Kress, ined.
La técnica RAM es útil para estudiar diversidad genética vegetal porque no necesita información previa, es altamente polimórfica y de bajo costo.
En la colección de mora se diferenciaron las especies R. glaucus, R. robustus y R. urticifolius, se detectaron duplicados y se identificaron regiones de alta diversidad.
En la colección de uchuva se detectaron duplicados, hubo alta diversidad y se identificaron materiales genéticamente distantes dentro de P. peruviana.
En guayaba la técnica permitió detectar polimorfismo y diversidad alta en el Valle del Cauca.
Se diferenció la familia Heliconiacea de otras familias del orden Zingiberales, se diferenciaron algunas subespecies y se encontró variación intraespecífica.
A la división de investigación DIPAL de la Universidad Nacional de Colombia por la financiación de las investigaciónes. Al grupo de Diversidad Biológica, Herney Darío Vásquez, Martha Liliana Bonilla, Katherine Espinosa, Andrés Mauricio Posso, Alba Lucía Arcos y Gloria Magally Cobo.
1. Akkaya, M.S.; Bhagwat, A.A.; Cregan, P.B. 1992. Length polymorphisms of Simple Sequence Repeat DNA in Soybean. Genetics 132: 1131-1139. [ Links ]
2. Arcos, A. L.; Mondragón, A. J.; Muñoz, J. E., Botero, S. 2004. Colecta y caracterización molecular con marcadores tipo RAM (Microsatélites aleatorios) de heliconias y especies relacionadas, p 346-347. En: Congreso Colombiano de Botánica: Botánica, Diversidad y Cultura, 2, Popayán, Noviembre. Memorias. [ Links ]
3. Binder, A. 1997. Standard PCR Conditions. Disponible en:http://www.uni-graz.at/~binder/thesis/node32.html#SECTION005610000000000000. Acceso: 25-11-2007. [ Links ]
4. Bonilla M. L.; Espinosa K.; Posso A. M.; Vásquez, H.; Muñoz, J. E. 2008. Caracterización molecular de 43 accesiones de uchuva de seis departamentos de Colombia. Acta Agron (Palmira) 57(2): 109-115. [ Links ]
5. Brinkmann Instruments. 2004. PCR optimization. Disponible en: http://www.brinkmann.com/utilities/404.asp [ Links ]
6. Cobo, G. M. 2004. Caracterización morfológica y molecular de materiales comerciales de Heliconia spp. Trabajo de Especialista (Biotecnología), Palmira: Universidad Nacional de Colombia 96 p. [ Links ]
7.Dellaporta S. L.; Wood, J.; Hicks, J. B. 1983. A plant DNA minipreparation: Versión II. Plant. Molec. Biol. Rep. 14: 19-21. [ Links ]
8.Eckert, K.A.; Kunkel, T.A. 1990. High fidelity DNA synthesis by the Thermus aquaticus DNA polymerase. Nucl. Acids Res. 18: 3739- 44. [ Links ]
9. Efron, B. 1979. Bootstrap methods: Another look at the jackknife. Ann Stat. 7 (1): 1-26. [ Links ]
10. Excoffier, L.; Smouse, P.E.; Quattro, J. M. 1992. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data. Genetics 131: 179 -191. [ Links ]
11. Felsenstein, J. 1985. Confidence limits on phylogenies: an approach using the bootstrap. Evolution 39: 783-791. [ Links ]
12.Ferreira, M. E.; Grattapaglia, D. 1998. Introducción al uso de marcadores moleculares en el análisis genético. Brasilia, D.F: Embrapa-Cenargen. 221 p. [ Links ]
13. Golein, A. M.; Koltunow, A.; Talaie, Z.; Zamani Ebadi, A. 2005. Isolation and characterization of microsatellites loci in the lemon (Citrus limon). Mol Ecol Notes 5: 253-255. [ Links ]
14. Hantula, J.; Dusabenyagasani, M.; Hamelin R.C. 1996. Random Amplified Microsatellites (RAMs) a novel method for characterizing genetic variation within fungi. Eur Path. 26: 159-166. [ Links ]
15. Hearne, C. M.; Ghlosh, S.; Todd. J. A. 1992. Microsatellites for linkage analysis of genetic traits. Trend Genet. 8: 288-294. [ Links ]
16. Henegariu, O. 2000. PCR and multiplex PCR: guide and troubleshooting. Disponible en: http://info.med.yale.edu/genetics/ward/tavi/PCR.html. Acceso: 20-02-2008. [ Links ]
17. Henríquez N., M. A. 2000. Diversidad genética de Phaeoisariopsis griseola (Sacc.) Ferraris utilizando marcadores moleculares. Trabajo de grado (Ing. Agr) Palmira: Universidad Nacional de Colombia. 102 p. [ Links ]
18. Joseph, J.D.; Anderson, R.; Tatham ,R.; Black, W. 1992. Multivariate data Analysis with Readings. 3rd ed. USA. 554 p. [ Links ]
19. Koehler – Santos, P.; Cunha, A. L.; Dornelles, L. B. De Freitas. 2003. Characterization of mandarin citrus germplasm from Southern Brazil by morphological and molecular analyses. Pesq. Agropec. Bras. 38 (7): 797-806. [ Links ]
20. Kraic, J.; Sakova, M., Gregova, E. 1998. Comparison of differentiation capability or RAPD and SSR markers in commercial barley (Hordeum vulgare L.) cultivars. Cereal Res Commun. 26: 375-382. [ Links ]
21. Kress W. J.; Betancur, J.; Echeverry, B. 1999. Heliconias: Llamaradas de la selva colombiana. Guía de Campo. Bogotá, Colombia: Uribe Editores. 195 p. [ Links ]
22. Leung, H.; Nelson, R. H.; Leach, J. E. 1993. Population structure of plant pathogenic fungi and bacteria. Adv Plant Pathol 10:157-205. [ Links ]
23. Lozada, M. 2002. Técnica de Reacción en Cadena de la Polimerasa. Disponible en: http://www.monografias.com/trabajos11/tamau/tamau.shtml Acceso: 03-05-2008. [ Links ]
24. Mahuku, G.S.; Henríquez, M. A.; Muñoz, J., Buruchara, R.A. 2002. Molecular markers dispute the existence of the Afro-Andean group of the Bean Angular Leaf Spot pathogen, Phaeoisariopsis griseola. Phytopathology 96 (6): 580-589. [ Links ]
25. Morgante, M.; Olivieri, A.M. 1993. PCR-amplified ?microsatellites as markers in plant genetics. Plant J 3: 175-182. [ Links ]
26. Morillo, A.; Morillo, Y.; Zamorano, A.; Vásquez, H.; Muñoz J. E. 2005. Caracterización molecular con microsatélites aleatorios RAM de la colección de mora Rubus spp, de la Universidad Nacional de Colombia, Sede Palmira. Acta Agron (Palmira) 54 (2): 15-24. [ Links ]
27. Nei, M.; Li, W.H. 1979. Mathematical model for studying genetic variation in terms of restriction endonucleasa. Proc Natl Acad Sci USA 79: 5267 - 5273. [ Links ]
28. Novelli, V.M.; Cristofani M.; Souza A. A, Machado M. A. 2006. Development and characterization of polymorphic microsatellite markers for the sweet orange (Citrus sinensis L. Osbeck). Genet Mol Biol 29 (1): 90-96. [ Links ]
29. Promega. 2005. PCR Core Systems. Instructions for use of products M7660 and M7665. Disponible en:http://www.promega.com/tbs/tb254/tb254.htlm. Acceso: 03-15-2008. [ Links ]
30. Quiagen. 2004. Dneasy plant mini and maxi handbook. Disponible en: http://www.quiagen.com. Acceso: 03-16-2008. [ Links ]
31. Rees, W. A.; Yager, T. D.; Korte, J.; Von Hippel, P. H. 1993. Betaine can eliminate the base pair composition dependence of DNA melting. Biochemistry 32: 137-144. [ Links ]
32. Roder, M.S.; Plaschke, J.; Konig, S.U.; Borner, A.; Sorrells, M.A.;Tanksley, S.D.; Ganal, M.W. 1995. Abundance, variability and chromosomal location of microsatellites in wheat. Mol Gen 246: 327-333. [ Links ]
33. Rueda, A.; Muñoz, J. E.; Saavedra, R.; Palacio, J. D.; Bravo, E. 2006. Caracterización molecular del banco de germoplasma de guayaba Psidium spp del Centro de Investigación de Corpoica, Palmira. Fitotec Colomb, 6 (2): 26-32. [ Links ]
34. Sambrook, J.; Fritsch, E.F.; Maniatis, T. 1989. Gel Electrophoresis of DNA. p3-60. In: Sambrook, J.; Fritsch, E.F.; Maniatis, T. (eds.). Molecular Cloning: a Laboratory Manual. New York: Cold Spring Harbor Laboratory Press. [ Links ]
35. Sanabria, H.; García, M.; Díaz, H.; Muñoz, J.E. 2006. Caracterización molecular con marcadores RAM de árboles nativos de Psidium guajava (guayaba) en el Valle del Cauca. Acta Agron (Palmira) 55 (1): 23-30. [ Links ]
36. Smith, D. N.; Devey, M. E. 1994. Ocurrence and inheritance of microsatellites in Pinus radiata. Genome 37: 977-983. [ Links ]
37. Sneath, P. H. A.; Sokal, R. R. 1973. Numerical Taxonomy: The Principles and Practice of Numerical Classification. San Francisco: W.H. Freeman. 573 p. [ Links ]
38. Yap, I.V.; Nelson, R. J. 1996. Winbooot: A program for performing bootstrap analysis of binary data to determine the confidence limits of UPGMA based dendrograms. Manila, Philippines: IRRI (Discussion Paper Series No 14). [ Links ]
39. Zietkiewicz, E.; Rafalski A.; Labuda D. 1994. Genome fingerprinting by Simple Sequence Repeat (SSR)-anchored Polymerase Chain Reaction Amplification. Chain Reaction Amplification. Genomics 20: 176-183. [ Links ]














