Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.59 no.4 Palmira Oct./Dec. 2010
1Centro Internacional de Agricultura Tropical CIAT, AA 6713, Cali, Valle, Colombia. 2Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia. AA 237. Palmira, Valle del Cauca, Colombia. 3Product Development Manager at Donald Danforth Plant Science Center Autor para correspondencia: Adriana Mercedes álzate adrimeral@hotmail.com.
Usando la técnica de marcadores moleculares, tipo microsatélites, se evaluó la variabilidad genética de 717 genotipos de yuca Manihot esculenta Crantz colectados en fincas de pequeños agricultores de la región Caribe de Colombia, en los departamentos de Atlántico, Magdalena, Córdoba y Sucre. Mediante el Análisis de Correspondencia Múltiple (ACM) se establecieron seis grupos genéticamente diferentes, con un Gst bajo (0.18), lo cual indica que la variabilidad no se debe a diferencias entre los grupos sino a diferencias dentro de cada grupo. La diversidad genética encontrada fue alta (HT: 0.61692).
Palabra clave: Manihot esculenta, diversidad, distancia genética, heterocigotos.
Using molecular microsatellites analysis, the genetic diversity of 717 cassava genotypes from small formers in the Caribbean region of Colombia was assessed. Six genetically distinct groups was established, with a low Gst (0.18), indicating that the variability was not due to differences between groups but within each group. The genetic diversity was high (Ht: 0.61692).
Key words: Manihot esculenta Crantz, diversity, genetic divergency, heterozygotes.
La yuca, Manihot esculenta Crantz constituye el cuarto producto básico más importante en la alimentación mundial después de arroz, trigo y maíz. Tiene un papel fundamental en la dieta de más de mil millones de personas (Ceballos, 2002), y constituye un importante recurso energético en la alimentación humana.
Esta especie es cultivada, generalmente, por agricultores pequeños. Por su buen comportamiento en condiciones marginales de clima y suelo, es considerada una especie importante que contribuye a la seguridad alimentaria en poblaciones con bajos recursos económicos y en donde otros cultivos no se desarrollan adecuadamente (Iglesias et al., 1994).
En Colombia, la principal zona de producción de yuca es la región Caribe donde se concentra el 42% de la producción nacional (Gottret et al., 2002). Igual que en otros países, el cultivo es manejado por agricultores en pequeñas explotaciones, los cuales utilizan cultivares no identificados conformados por genotipos locales o variedades mejoradas, al desconocer la magnitud de la variabilidad genética.
El conocimiento de la variabilidad genética de los cultivos, incluyendo la yuca, en cualquier región del mundo, facilita la toma de decisiones en los programas de mejoramiento y en la conservación de la especie. La conservación del germoplasma evita la pérdida de especies silvestres y cultivadas, pues prevé o reduce la erosión genética (Jaramillo, 2002).
La variabilidad genética puede ser estimada directamente a partir del genoma, con el uso de marcadores moleculares tales como RFLP, RAPD, minisatélites y microsatélites, entre otros. Cuando la variabilidad es calculada por diferencias alélicas en varios loci a través de electroforesis, los parámetros más usados son el número de alelos y la heterocigosidad (Nei, 1987). Los marcadores moleculares microsatélites SSR – (Simple Sequence Repeat), han sido ampliamente usados para estudios de variabilidad genética de yuca. Se han identificado 36 marcadores microsatélites como los de mayor polimorfismo (Mba et al., 2001) los cuales se han utilizado en investigaciones en países como Sierra Leona (Dixon, 2003), Uganda (Kizito, 2003), Nigeria (Dixon, 2002), Ghana (Okay, 2003), Perú (Alcántara, 2001), Guatemala (Monte, 2003) y Cuba (Beovides, 2004). Además, se han encontrado valores altos de diversidad y evidenciado subestructuras en poblaciones africanas, lo que constituye un aspecto positivo para el mejoramiento del cultivo.
En algunos de los anteriores trabajos se han incluido variedades colombianas provenientes del Banco de Germoplasma del Centro Internacional de Agricultura Tropical-CIAT. Sin embargo, en Colombia no se han hecho estudios de variabilidad genética que incluyan exclusivamente introducciones de yuca colectadas en el país o en alguna región de éste.
El objetivo de este trabajo fue analizar la variabilidad genética de yuca cultivada por pequeños agricultores de la región Caribe de Colombia, mediante marcadores moleculares del tipo microsatélites.
La colección de germoplasma se realizó en fincas pequeñas que tenían un área comprendida entre 0.5 y 12 ha dentro de los departamentos de Córdoba, Sucre, Atlántico y Magdalena, ubicados en la región Caribe colombiana. El muestreo fue de tipo conglomerado polietápico y se considera al grupo de cultivadores existente en cada municipio, como conglomerado. Primero se seleccionó aleatoriamente el municipio dentro de cada departamento y después las fincas dentro de cada municipio.
Se seleccionaron 40 municipios (conglomerados) y luego 10 fincas dentro de cada municipio, para un total de 400 muestras de fincas (Cuadro 1).
Para determinar el tamaño de muestra (no) se consideró el siguiente modelo:
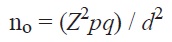
Donde:
p: 50% de fincas con variabilidad genética de yuca sembrada.
q: 50% de fincas sin variabilidad genética de yuca sembrada.
Z: nivel de confianza de 90%: 1.64
d: error esperado por parte del investigador: 0.06
La encuesta estuvo enfocada a detectar el conocimiento, por parte del agricultor, de las variedades sembradas en su finca. Se tomaron muestras de estacas o propágulos que fueron trasladadas al CIAT y sembradas en invernadero. De las plantas obtenidas en esta fase se tomaron muestras de hojas jóvenes y cogollos, los cuales se secaron en horno a 40° durante tres días para la posterior extracción del ADN con uso del método propuesto por Dellaporta et al., 1983.
La calidad del ADN fue observada en geles de agarosa al 1%. Se cuantificó en fluorómetro antes de realizar diluciones a una concentración de 10 ng/µl de ADN. Las amplificaciones fueron hechas en los termocicladores PTC-100 vía PCR usando el programa 30 ciclos a: 94 °C por 30"; 55 °C por 30"; 72°C por 1 min, y una extensión de 5 min a 72 °C. El producto de la amplificación se corrió en geles denaturantes de poliacrilamida 6% acrilamida y se hizo un revelado de tinción con plata. Posteriormente la lectura de los geles y la toma de datos para el análisis estadístico molecular.
Se usaron nueve marcadores microsatélites, seleccionados del grupo de 36 SSRY establecidos en trabajos anteriores de diversidad como los de mayor polimorfismo para análisis de diversidad y diferenciación genética (Mba et al., 2001). El procedimiento para seleccionar los nueve marcadores consistió en un análisis preliminar de 30 muestras de la población de los 36 marcadores microsatélites. Después de obtener las lecturas de los geles de poliacrilamida se descartaron los monomórficos, y de los polimórficos se seleccionaron visualmente los 18 SSRY que presentaron mayores polimorfismos.
Estos 18 SSRY fueron evaluados en el programa sistematizado Cervus para escoger los nueve con el PIC Polymorphic Information Content (Contenido de Información Polimórfica más alto), adicionalmente se consideró la ubicación de estos marcadores en el mapa genético de yuca para tener una buena cobertura del genoma. Los marcadores seleccionados fueron: SSRY 12, SSRY 51, SSRY 63, SSRY 82, SSRY 100, SSRY 135, SSRY 151, SSRY 155, SSRY 179.
Para el estudio molecular también se adicionaron 14 genotipos del Banco de Germoplasma de Yuca del CIAT, que corresponden a variedades locales y mejoradas originales, liberadas en la región y provenientes del Programa de Mejoramiento de Yuca del CIAT. Estas muestras fueron usadas como control para comprobar su existencia en la región. Las variedades fueron: Corpoica Gines CM 4843-1, Corpoica Colombiana CM 3306-19, Corpoica Sucreña CM 3555-6, Corpoica Verónica CM 4919-1, ICA Costeña CG 1141-1, ICA Negrita CM 3306-4, SM 1433-4, ICA P-12 MCOL 1505, Cubana CM 4574-7, Venezolana MCOL 2215, Blanca Mona MCOL 2253, Secundina MCOL 2063, MTAI-8, y MVEN-25.
Las gráficas de ordenación fueron construidas con base en las coordenadas obtenidas para los individuos con un análisis de correspondencia múltiple (ACM), utilizando programas elaborados en SAS en un ambiente UNIX CIAT, SAS: Versión 9.1.3.
Los parámetros de variabilidad genética: porcentaje de loci polimórficos, número medio de alelos por locus polimórfico, heterocigosidad promedio observada Ho, y diversidad genética promedio He fueron estimados con el programa SAS.
Se colectaron 1.045 genotipos de yuca, de los cuales 717 presentaron información molecular completa con los nueve marcadores microsatélites seleccionados y con los cuales se hizo el análisis molecular.
Relaciones genéticas entre genotipos
En el análisis de correspondencia múltiple (ACM) se definieron seis grupos genéticamente diferentes, los cuales explicaron el 81% de variación (Figura 1).
En el Grupo 1, el 85.5% de los genotipos corresponden a la variedad Venezolana; en el Grupo 3, el 85% equivale a la variedad ICA Costeña; en el Grupo 6, el 71.3% es de la variedad Banca Mona y en el Grupo 5, el 43.3% corresponde a la variedad ICA P-12 (Figura 2). Teniendo en cuenta los porcentajes de presencia de estas variedades en cada grupo, fue posible considerarlas como representativas de cada uno de ellos. Los restantes genotipos, que se ubican en cada uno de estos grupos y que se encuentran en menor proporción, tienen relación genética con estas variedades. Los grupo 2 y 4 (Figura 2) tienen el mayor número de genotipos y se presentan en bajos porcentajes, no obstante, debido a su variabilidad es difícil establecer características específicas distintivas o discriminantes que los identifiquen.
En la Figura 3 se observan las variedades más sembradas por los pequeños agricultores en la región Caribe de Colombia, se destaca la variedad Venezolana con 26%, seguida de ICA Costeña 9% y Blanca Mona 7%, y en menor proporción las variedades P-12, MTAI-8, Cubana y Secundina con 5%. El 46% de la yuca sembrada equivale a otros genotipos mencionados por los agricultores.
Variabilidad genética
El promedio de la heterocigosidad observada (HI) fue alta con un valor de 0.56087, lo cual confirma el entrecruzamiento en la yuca y su naturaleza altamente heterocigota. El promedio ponderado de la heterocigosidad esperada (Hs) fue 0.50280, que representa el nivel de heterocigosidad que se encontraría si se dieran cruzamientos al azar. La heterocigosidad total (HT) para todos los genotipos fue alta, con un valor de 0.61692, es decir, la heterocigosidad esperada de un individuo en toda la población (Cuadro 2).
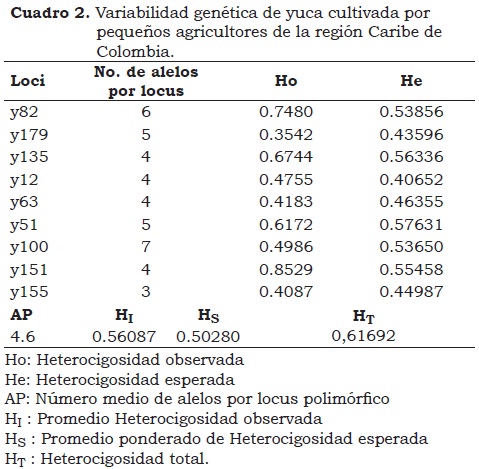
Aunque se establecieron seis grupos genéticamente diferentes en el ACM, el coeficiente de diferenciación fue bajo (Gst) 0.18498, es decir, que la variabilidad no se debe a diferencias genéticas entre grupos, sino dentro de los grupos, lo que se confirma con los altos valores de heterocigosidad esperada (He) dentro de cada grupo (Cuadro 3). Se observa también que el Grupo 1 presenta la más baja He 0.37751 lo cual está relacionado con el alto porcentaje de la presencia de la variedad Venezolana y muy bajo de otras variedades. Igualmente, ocurre con los grupos 3, y 6. Caso contrario, los grupos 2 y 4 presentaron los valores más altos de He y efectivamente con el mayor número de genotipos, convirtiéndose en los de mayor variabilidad genética (Figura 2).

La variabilidad genética de la yuca está relacionada con la introducción, intercambio y con la polinización cruzada, cada individuo es naturalmente un híbrido con altos niveles de heterocigosidad (Ceballos, 2002). Además de la alta alogamia y su reproducción sexual, se debe tener en cuenta también la reproducción asexual de esta especie y su amplia distribución geográfica. Se sabe que la yuca nativa está distribuida en todo el sector de la costa Caribe colombiana, entre los ríos Atrato y Magdalena, donde se han encontrado variedades como Cartagenera, Momposina, Samaria, Pie de paloma, Gallinazo, Batea, Solita, Riogrande, Algodón, Camarón, Pie de perdiz, Chingale y Pascualita (Patiño, 1964).
La reproducción asexual es la forma de propagación más utilizada, pero también puede estar combinada con formas de reproducción sexual, que permiten la recombinación y favorecen la generación de nuevas formas o combinaciones.
La evolución de la estructura genética de una especie y su mantenimiento en el tiempo y en el espacio, están sujetos a la acción de tres fuerzas evolutivas con efectos divergentes. Por un lado, la selección natural y la deriva genética –fuerzas eminentemente perturbadoras– que favorecen la diferenciación genética de una población y por el otro, el flujo genético que permite el intercambio de material genético entre poblaciones (Nason, 2002).
La migración también beneficia los cambios de las frecuencias alélicas. Desde una perspectiva genética, el intercambio de genes entre poblaciones debido a la migración de los individuos es un factor importante de cambio genético (Barbadilla, 2009). En la región Caribe es posible considerar, como caso de migración, la introducción de la variedad Venezolana, traida desde otro país por los agricultores; al igual que las variedades mejoradas y liberadas en la región, o aquellos materiales que en las pruebas regionales de ensayos del programa de mejoramiento, no demostraron ser mejores que otros materiales evaluados, se han difundido espontáneamente entre los agricultores a pesar de no haber sido liberadas como variedades (Ceballos, 2002). Todas estas causas contribuyen a la alta variabilidad encontrada.
Como se observa en la Figura 3, el 46% de la yuca cultivada por pequeños agricultores equivale a diversos genotipos, la variabilidad en finca no es estática sino que con el paso del tiempo hay genotipos que salen y otros que son incorporados al sistema de producción del agricultor. Esto refleja el hecho de que los agricultores tienen una disposición de aprobar nuevos materiales, observarlos y con el tiempo incorporarlos o rechazarlos. Las fuentes de variabilidad que manejan son de dos tipos: variedades suministradas por los vecinos o parientes, y clones nacidos en el campo debido a cruces entre variedades locales involuntarios. Estos procesos pueden no ser advertidos en el corto plazo, pero han constituido la base para la evolución del cultivo (Iglesias et al., 1994).
Los resultados de la variabilidad genética (He, Ho, AP, HI, HS, HT) obtenida en este estudio fueron similares a los encontrados en otros trabajos de variabilidad genética de yuca realizados en áfrica y algunos países en los cuales se usaron microsatélites y se estimaron los mismos parámetro. En general, estos trabajos concluyen en confirmar el comportamiento heterocigoto de la yuca y su alta variabilidad genética.
- Se detectaron seis grupos genéticamente diferentes que explican el 81% de la variación, con un coeficiente de diferenciación bajo (Gst: 0.18), esto indica que la variabilidad se debe a diferencias dentro de los grupos y no entre ellos.
- De los seis grupos diferenciados, los grupos 1, 3, 5 y 6 estuvieron representados en un alto porcentaje por una sola variedad. Los grupos 2 y 4 presentaron el mayor número de genotipos y la mayor variabilidad genética de ellos, por lo que no fue posible establecer una variedad específica que los identificara.
- Las medidas de variación genética demostraron que, en los cultivos de yuca de pequeños agricultores de la región Caribe de Colombia, existe alta heterocigosidad (0.56087) lo cual confirma el entrecruzamiento en la yuca y su naturaleza altamente heterocigota. Su condición alógama favorece la segregación y la generación de nueva variabilidad genética. También se encontró alta heterocigosidad total (HT: 0.61692) y alto nivel de heterocigosidad esperada (HS: 0.50280).
Al Fondo Conmemorativo de Becas Ginés-Mera para Estudios de Postgrado en Diversidad Biológica por la financiación del proyecto. Al Programa de Mejoramiento de Yuca de CIAT.
Alcántara, J.; Guarino, L.; Fregene, M. 2001. Análisis de la diversidad genética en Manihot esculenta yuca' mediante el uso de marcadores microsatélites–SSR. CIAT. Cali, Colombia. p. 9. [ Links ]
Barbadilla A 2009. Genética de Poblaciones. Departamento de Genética y Microbiología. Universidad Autónoma de Barcelona. http://biologia.uab.es/divulgacio/genpob.html#factores. Acceso: 6-04-2009 [ Links ]
Beovides, Y.; Barrera, E.; Gutiérrez, J. P.; Buitrago, C.; Marin J.; y Fregene, M.. 2004. Characterization of genetic diversity: Simple sequence repeat SSR assessment of genetic diversity of local cassava varieties from Cuba. Funding: Cassava Biotechnology Network CBN. CIAT. Cali. Annual Report. Output 12 – 34. [ Links ]
Ceballos H.; Morante, N.; Calle, F.; Lenis, J.; Jaramillo, G.; Perez, J. 2002. Mejoramiento genético de la yuca. En: La yuca en el tercer milenio: Sistemas modernos de producción, procesamiento, utilización y comercialización. Publicación CIAT. Cali, Colombia, p. 295 - 325. [ Links ]
Dellaporta, S. L.; Wood, J.; Hicks Jr. 1983. A plant DNA minipreparation: version II. Plant Mol Biol. Rep. 1:19 - 21. [ Links ]
Dixon, A.; Raji, B.; Marín, J.; Ospina, C.; Buitrago, C.; Fregene, M. 2003. Characterization of genetic diversity: Simple sequence repeat SSR assessment of genetic diversity of local cassava varieties from Sierra Leone. Annual Report: Development and use of biotechnology tools for cassava improvement. CIAT, Cali, Colombia. Output 8 - 43. [ Links ]
Dixon, et al. 2002. Simple sequence repeat SSR marker diversity in cassava Manihot esculenta Crantz landraces form Nigeria. Annual Report: Development and use of biotechnology tools for cassava improvement. CIAT. Cali. Annual Report: Output 8 - 43. [ Links ]
Gottret, M. V.; Escobar, Z.; Pérez S. 2002. El sector yuquero en Colombia: Desarrollo y competitividad. En: La yuca en el tercer milenio: Sistemas modernos de producción, procesamiento, utilización y comercialización. Publicación CIAT. Cali, Colombia, p. 340. [ Links ]
Iglesias, C.; Hernández, L. A. 1994. Introducción de diversidad genética mejorada a nivel de campos de agricultores. En: Interfase entre los programas de mejoramiento, los campos de los agricultores y los mercados de la yuca en Latinoamérica. Centro Internacional de Agricultura Tropical CIAT, Cali, Colombia. p. 151 - 157. [ Links ]
Jaramillo, G. 2002. Recursos genéticos de Manihot en el Centro Internacional de Agricultura Tropical CIAT. En: La yuca en el tercer milenio: Sistemas modernos de producción, procesamiento, utilización y comercialización. Publicación CIAT. Cali, Colombia. p. 271 - 294. [ Links ]
Kizito, E.; Gullberg, U.; Bua, A. Omara, J.; Egwang, M.; Castelblanco, W.; Gutierrez, J.; Buitrago, C.; Fregene, M. 2003. Simple sequence repeat SSR assessment of genetic diversity of local cassava varieties from Uganda. Annual Report: Development and use of biotechnology tools for cassava improvement. CIAT, Cali - Colombia. Annual Report: Output 8 - 36. [ Links ]
Mba, R. E. C.; Stephenson, P.; Edwards, K.; Melzer, S.; Nkumbira ,J.; Gullberg, U.; Apel, K.; Gale, M.; Tohme, J.; Fregene M. 2001. Simple sequence repeat SSR markers survey of the cassava Manihot esculenta Crantz genome: towards an SSR-based molecular genetic map of cassava. Theor. Appl. Gen. 102:21 – 31 [ Links ]
Monte Luis et al. 2003. Simple sequence repeat SSR assessment of genetic diversity of local cassava varieties from Guatemala. Annual Report: Development and use of biotechnology tools for cassava improvement. CIAT, Cali - Colombia. Annual Report: Output 8 - 21. [ Links ]
Nason, J. D. 2002. La estructura genética de las poblaciones de árboles. En: Guariguata, M. R. y Kattan, G. H. (eds.). Ecología y conservación de bosques neotropicales. CR, Editorial Tecnologica. p. 299 - 327. [ Links ]
Nei, M. 1987. Molecular evolutionary genetics. Columbia University Press, Nueva York, p. 512. [ Links ]
Okay, E.; Otoo, J.; Buitrago, C.; Fregene, M.; Dixon. A. 2003. Characterization of genetic diversity: Simple sequence repeat SSR assessment of genetic diversity of local cassava varieties from Ghana and predictability of heterosis. Annual Report: Development and use of biotechnology tools for cassava improvement. CIAT, Cali - Colombia. Annual Report. Output 8 – 28. [ Links ]
Patiño, V. M. 1964. Plantas cultivadas y animales domésticos en América equinoccial. Tomo 2. Plantas alimenticias, 1. Imprenta departamental, Cali, Colombia, 364 p. [ Links ]














