Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.60 no.2 Palmira Apr./June 2011
1Department of plant Pathology, Kansas State University, USA. 2Facultad de Agronomía, Universidad Nacional de Colombia, Bogotá, Colombia. 3Departamento de Biología, Universidad Pedagógica Nacional, Bogotá, Colombia. *Autor para correspondencia: galigarretom@unal.edu.co; †mcgiraldo@ksu.edu; ‡dgcayons@unal.edu.co; ††lmelo@pedagogica.edu.co
La Colección Colombiana de Musáceas (CCM) es la única a nivel mundial que representa un alto valor por ser la que posee introducciones andinas de altura (> 1500 m.s.n.m.). La caracterización de este germoplasma puede generar valor agregado para su utilización en procesos de selección clonal y para el mejoramiento genético de la especie, mediante el uso de materiales diploides con características transmisibles de importancia. Por esta razón, 33 clones de la CCM conservadas in vitro, fueron evaluadas bioquímicamente mediante 10 enzimas, de las cuales cuatro fueron polimórficas: glutamato oxaloacetil transaminasa (GOT), ab-esterasa (ab-EST), peroxidasa (PRX) y diaforasa (DIAP). La enzima GOT fue la más discriminante entre grupos genómicos particulares. PRX, DIAP y ab-EST permitieron evaluar la variabilidad al interior de cada grupo. El estudio facilita el entendimiento de la estructura genética de los genotipos de plátano y banano cultivados en Colombia.
Palabra clave: Caracterización morfológica, Colombia, isoenzimas, marcadores genéticos, Musa, Musaceae.
The Colombian Collection of Musaceae (CCM) represents a great value to the world agriculture for being the only one with high Andean germplasm. The characterization of this germplasm can generate added value to use in processes of clone selection and breeding of the species by using diploid materials with desirable traits to be transferred and thus help alleviate food problems in poor countries. For this reason the CCM 33 clones conserved in vitro, were assessed biochemically using 10 enzymes of which four were polimorfics: oxaloacetil transaminasa glutamate (GOT), ab-esterase (ab-EST), peroxidase (PRX) and diaphorase (DIAP). The GOT enzyme was the most discriminating among individual genomic groups. PRX, ab-EST and DIAP allowed us to evaluate the variability within each group. The study provided a better understanding of the genetic structure of the genotypes of plantain and banana grown in Colombia.
Key words: Colombia, genetic markers, isoenzymes, morphological characterization, Musa, Musaceae.
Los plátanos y bananos (Musa spp) son considerados componentes básicos en la alimentación de más de 400 millones de habitantes en las regiones tropicales y subtropicales (Perea, 2003). Colombia es el mayor productor de plátanos de América Latina con 400.000 ha sembradas y una producción de 3,000,000 t/año.
Botánicamente son hierbas gigantes, monocotiledóneas y perennes pertenecientes a la familia Musaceae que comprende los géneros Ensete Horan y Musa L. El género Ensete es monocárpico, con nueve cromosomas básicos y se caracteriza por la ausencia de retoños y frutos no-comestibles. El género Musa es el más ampliamente distribuido y comprende cuatro secciones (Eumusa, Rhodochlamys, Callimusa y Australimusa). Callimusa y Australimusa tienen un número básico de cromosomas de 10 (2n = 20), mientras Eumusa y Rhodochlamys tienen un número básico de 11 (2n = 22). Las especies de Rhodochlamys y Callimusa son principalmente de importancia ornamental y las especies de Eumusa y Australimusa son las de mayor importancia por producir frutos comestibles (Chandel y Anuradha, 2000; Horry, 2000). La sección Eumusa, que incluye bananos y plátanos, muestra una gran variabilidad entre sus especies expresada en la cantidad de formas triploides y tetraploides, amplia distribución geográfica y mayor adopción como cultivo (Belalcázar, 1991; Chandel y Anuradha, 2000). La existencia de esta poliploidía ha originado alguna disparidad en la clasificación exacta entre las subespecies (Simmonds, 1966).
Las especies de bananos y plátanos comestibles se originaron de los cruces interespecíficos de las silvestres Musa acuminata Colla (Genoma A) y Musa balbisiana Colla (Genoma B), de las cuales esta última presenta menos variabilidad (Chandel y Anuradha, 2000). Musa acuminata sola o en combinación con Musa balbisiana son las especies progenitoras de la mayoría de bananos cultivados, los cuales pueden ser diploides (2n = 22), triploides (2n = 33) o tetraploides (2n = 44). En Musa las plantas poliploides son más vigorosas, resistentes y de mayor productividad y adaptación que las diploides. La poliploidía se puede presentar en varios niveles: triploides de M. acuminata pura AAA, triploides híbridos de las fórmulas AAB y ABB, tetraploides AAAA, ABBB, AAAB y AABB (Bakry et al., 2001). Según Simmonds (1979) los clones triploides son resultado de cruces entre diploides, donde uno de los dos produce gametos no reducidos; los tetraploides se desarrollan de un gameto triploide no reducido y uno diploide reducido o de gametos diploides no reducidos, aspecto que es de gran importancia si se quiere determinar a partir de cuáles diploides silvestres se originaron los nuevos grupos y proponer una adecuada taxonomía. Los triploides se designan según los códigos siguientes: (1) AAA para bananos con muy bajo contenido de almidón y mayor contenido de azúcares, (2) AAB para plátanos de cocción con dominancia acuminata, (3) ABB para plátanos con dominancia balbisiana con alto contenido de almidón y bajo contenido de azúcares. Estos últimos han resultado ser resistentes y tolerantes a sequías y a enfermedades infecciosas como sigatoka negra, causada por el hongo Mycosphaerella fijiensis var. difformis, sigatoka amarilla causada por Mycosphaerella musicola y moko, causado por la bacteria Ralstonia solanacearum (Purseglove,1985; Gómez et al., 1987).
Las semillas son muy raras en los bananos y plátanos cultivados y la esterilidad, combinada con la partenocarpia son características de calidad favorables del fruto que, unidas a la propagación vegetativa, han hecho de estas plantas cultivos de interés comercial de primera importancia entre los frutales (León, 1987). El germoplasma natural de estas plantas directamente disponible es un conjunto de aproximadamente 500 clones (De Langhe, 1987), el cual es distribuido por algunas redes de investigación (Bioversity International) a diferentes regiones del mundo, para ser empleado en fomento e investigación (Bakry et al., 2001). Sin embargo, el movimiento acelerado de este germoplasma y la carencia de un acuerdo común entre cultivadores y campesinos sobre la nomenclatura de las diferentes variedades, ha limitado la identificación correcta y completa de cada uno de ellos y el entendimiento de la distribución de su diversidad genética.
La mayor parte de los esfuerzos de la investigación en Musa se ha concentrado en mejorar la calidad de la fruta, la condición de enanismo en la planta para contrarrestar su volcamiento, la resistencia a enfermedades y plagas (Bakry et al., 2001; Rodríguez y Cayón, 2008) y la propagación clonal (Perea, 2001). Con el fin de optimizar la investigación en Musa se han generado programas de conservación en bancos de germoplasma en condiciones de campo e in vitro, ya que ambas facilitan un acceso continuo a los materiales para estudios de sistemática y fitomejoramiento y para el mantenimiento de una base genética amplia (De Langhe, 1987). La Colección Colombiana de Musáceas (CCM) en condiciones de campo, ubicada anteriormente en el Centro Experimental El Agrado (Montenegro, Quindío) y actualmente en el Centro de Investigación Palmira (Palmira, Valle) cuenta con más de 140 introducciones entre diploides (AA, BB, AB), triploides (AAA, AAB, ABB) y tetraploides (AAAA, AAAB, AABB, ABBB) todas ellas caracterizadas por métodos morfológicos, agronómicos y moleculares. La CCM en condiciones in vitro se encuentra en el Centro de Investigación Tibaitatá (Mosquera, Cundinamarca) y tiene 145 materiales clonales, la mayoría provenientes de la CCM y establecida en campo.
En las especies de reproducción exclusivamente vegetativa como plátano y banano, las colecciones de germoplasma son de vital importancia para la identificación, conservación, intercambio, caracterización y evaluación de los genotipos que las integran y deben ser objeto de estudios continuos para evaluar su rango de diversidad genética. Estas colecciones deben ser mantenidas en condiciones de campo e in vitro ya que son los métodos de conservación necesarios para la propagación vegetativa de las plantas. Los riesgos de la conservación in vitro radican en la presencia usual de variación somaclonal y la necesidad de regeneración frecuente (Karamura, 1999); mientras que en las colecciones de campo estos riesgos están representados por plagas, enfermedades y desastres naturales (Horry, 2000).
Tradicionalmente, la caracterización y clasificación de las musáceas se ha logrado mediante el uso de descriptores morfológicos (Simmonds y Shepherd, 1955; Stover y Simmonds, 1962) lo que implica la comparación del material genético basada en fenotipos visibles (forma, tamaño y colores) del seudotallo, hojas, racimo y frutos. Aunque estas características clásicas son todavía muy útiles, la eficiencia de identificación de las plantas puede reducirse por la edad, la etapa de desarrollo o los efectos ambientales sobre las características medidas (Bhat et al., 1995a). Debido a la naturaleza subjetiva de esta clasificación, los científicos están ahora complementándola con técnicas moleculares para la caracterización de los grupos de Musa que incluyen la diversidad de contenidos y el perfil de antocianinas (Horry y Jay, 1988), polimorfismo de enzimas (Bonner et al., 1974; Bhat et al., 1992a), heterogeneidad de longitud de espacios ('spacer-length') de rARN (Lannaud et al., 1992), polimorfismos de ADN de cloroplastos (Gawel et al., 1992), RFLP (polimorfismo de la longitud de fragmentos de restricción) (Jarret et al., 1992; Bhat et al., 1995b), RAPD (ADN polimórfico amplificado al azar) (Jarret et al., 1993; Bhat y Jarret, 1995), VNTR (número variable de repeticiones en tándem) (Kaemmer et al., 1993), SSR (secuencias simples repetitivas) y AFLP (polimorfismo de la longitud de fragmentos amplificados) (Nadal et al., 2008). El desarrollo de estos nuevos métodos de clasificación genética, particularmente el polimorfismo de enzimas, RFLP y RAPD, ha permitido el conocimiento más detallado y profundo del género Musa (Quiroz, 1991). El empleo de isoenzimas como marcadores genéticos ha resultado útil para la detección de duplicados, estimación en poco tiempo de la variabilidad genética e identificación clonal de un gran número de materiales con una inversión económica menor que la demandada por técnicas moleculares, no obstante, la información genética puede ser en algunos casos de menor veracidad comparada con algunas técnicas moleculares, dado que el proceso de extracción puede tener algún grado de efecto de ambiente.
El objetivo de este estudio fue analizar la variabilidad genética de la Colección Colombiana de Musáceas (CCM) mediante la caracterización bioquímica de 33 clones del banco base in vitro.
Material vegetal
En el proceso de conformación del banco base de germoplasma in vitro de la CCM, formado por 145 materiales clonales, se incorporaron algunos materiales que por parámetros morfológicos y fisiológicos representan la alta variabilidad de la colección. Se micropropagaron 66 clones provenientes de la CCM en campo del Centro Experimental El Agrado (Montenegro, Quindío), para ser incluidos en el banco base in vitro, y de estos se emplearon para la caracterización bioquímica los 33 clones que presentaron mayor respuesta en la conservación in vitro (Cuadro 1).
Antes de la extracción de las muestras de tejidos para análisis bioquímicos se evaluaron previamente las condiciones fisiológicas y físicas del material conservado in vitro y las de almacenamiento (temperatura y luz). Generalmente, cualquier tejido de la planta (hojas, raíces, tallos, semillas, organelos celulares) sirve para estudios enzimáticos, no obstante es importante tener en cuenta que los metabolitos secundarios pueden causar oxidación de los tejidos. Para la extracción se tomaron muestras de tejido de 0.5 g de toda la plántula y se maceraron con buffer de extracción Tris HCl 0,05M pH 8.3 (Ramírez et al., 1987) en proporción 1:2. Los extractos crudos fueron centrifugados a 14 000 gn y 4 °C por 20 min y posteriormente se tomaron alícuotas del sobrenadante en dos tubos Eppendorf que se almacenaron a 0 °C para su uso posterior.
Corrida electroforética
Se utilizó un sistema discontinuo en geles de poliacrilamida a 10% (p/v) y 4% (p/v), con el sistema tampón Tris-Borato pH:9.0 propuesto por Ramírez et al. (1987). Las condiciones utilizadas en la electroforesis fueron las siguientes: 250 V y 25 W, iniciando el proceso con una corriente de 15 mA hasta que las muestras entraron al gel de separación y luego se incrementó 5 mA cada 10 min hasta alcanzar 25 mA finales por gel. La electroforesis se terminó 15 min después que el indicador de corrida (azul de bromofenol) comenzó a salir del gel de separación, entre 6 y 7 h más tarde.
Como marcadores enzimáticos se evaluaron diez enzimas: glutamato oxaloacetil transaminasa (GO T), shikímico dehodrogenasa (SKDH), enzima málica (ME), ab-esterasa (ab-EST), peroxidasa (PRX), diaforasa (DIAP), 6-fosfogluconato dehidrogenasa (6-PGDH), malato dehidrogenasa (MDH), fosfogluconato isomerasa (PGI) y fosfatasa ácida (ACP); los protocolos de tinción enzimática utilizados fueron los propuestos por Ramírez et al. (1987).
Análisis estadístico
Con base en los zimogramas obtenidos con cada una de las enzimas analizadas, se realizó la lectura de los patrones de bandeo, expresando numéricamente como 1 = presencia ó, 0 = ausencia. Con estos datos se organizó una matriz básica de datos (MBD). Luego se analizaron los datos de las enzimas más polimórficas por medio del programa NTSYSpc (Rohlf, 2007), con el uso del coeficiente de similitud de Nei (Dice), con el cual se efectuaron los agrupamientos con el método de ligamiento promedio no-ponderado (UPGM A) (Crisci, 1983).
En el análisis bioquímico se observó que el extracto más eficiente para la separación y definición electroforética se obtuvo a partir de una plántula de tres semanas de propagación in vitro, aproximadamente, lo que disminuyó el porcentaje de oxidación en las muestras. De los diez sistemas enzimáticos utilizados como marcadores, (GO T, ab-EST, DIAP, PRX, ME, SKDH, MDH, 6-PGDH, PGI y ACP); los sistemas PGI y ACP no presentaron actividad enzimática y las enzimas 6-PGDH, ME, SKDH y MDH no revelaron patrones polimórficos consistentes. Por consiguiente, la colección se analizó por parámetros estadísticos usando matrices de datos de las cuatro enzimas más polimórficas (GOT, ab-EST, PRX y DIAP), y en este orden se presentan los resultados obtenidos con cada enzima.
Glutamato oxaloacetato transaminasa (GOT)
Se encontraron tres zonas de actividad que corresponden a los tres loci reportados por Jarret y Litz (1986) (Figura 1a) y al igual que ellos, los loci GO T-1 y GO T-2 presentaron un comportamiento monomórfico, por tanto para el análisis se tomó sólo el locus GOT-3, de estructura dimérica, presentando un patrón compuesto por tres bandas polimórficas, como se representa en la Figura 1b.

ab-Esterasa (ab-EST)
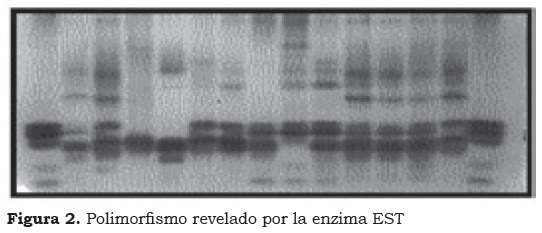
De todas las enzimas evaluadas fue la más polimórfica, con un número total de catorce bandas, como se observa en la Figura 2. Para esta enzima se definieron siete zonas de actividad que corresponden a: (1) zona A, para las bandas 1 y 2; (2) zona B, bandas 3 y 4; (3) zona C, corresponde a las bandas 5 y 6; (4) zona D: para las bandas 7 y 8; (5) zona E, para las bandas 9 y 10; (6) zona F, a las bandas 11 y 12 y (7) zona G, que corresponde a las bandas 13 y 14. La banda 9 es única para el material M. laterita y la banda 10 se evidencia sólo en los híbridos Rose D'ekona y Dominico Común (Figura 2a).
Peroxidasa (PRX)
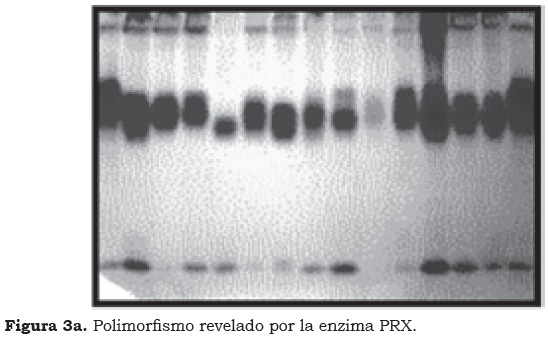
Se definieron tres zonas de actividad A, B y C (Figura 3a), de las cuales se tomó para el análisis estadístico la zona C con dos bandas polimórficas como se observa en la Figura 3b, debido a que las zonas A y B no presentaron patrones consistentes para el análisis.
Diaforasa (DIAP)
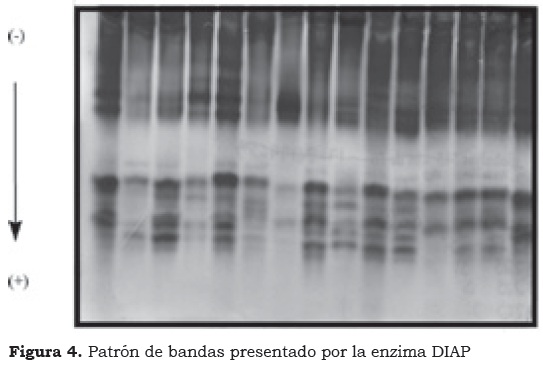
Se definieron cuatro zonas de actividad, A, B, C y D (Figura 4), donde las bandas polimórficas 1 y 2 corresponden a la zona A; 3 y 4 a la zona B; la zona C, inconsistente, comprende las bandas 5 y 6; y la zona D está constituida por las bandas polimórficas 7 y 8. Para el análisis se tomaron las zonas A, B, y D, por ser las que presentaron mayor polimorfismo, resolución y consistencia en los patrones de bandeo (Figura 4a).
En el Cuadro 2 se hace referencia al número de alelos por locus o zona de actividad revelados por las cuatro enzimas polimórficas evaluadas. Del análisis estadístico con todas las enzimas se obtuvieron nueve grupos, en los cuales se separan los grupos genómicos así: Plantain (AAB), Popoulou (AAB), Pelipita (ABB) y la especie M. laterita, y se agrupan otros materiales como Bocadillo Chileno (AA) y FHIA 03 (AABB), IC2-Gros Michel (AAAA), Saba (ABB) y Cachaco Enano (ABB) (Figura 5).
A partir de estos resultados se puede inferir que las enzimas ME y SKDH no son recomendables para estudios de diversidad en Musa por presentar un patrón de bandas monomórfico que no permite evaluar el grado de variabilidad ínter ó intraespecífica, al igual que MDH y 6-PGDH ya que se comportaron de forma inconsistente en el revelado. Las enzimas ab-EST, PRX y DIAP presentaron un polimorfismo aprovechable para la caracterización, posibilitando una mayor discriminación dentro de los grupos. La enzima GOT, aunque reveló un polimorfismo menor que las anteriores, permite evaluar la variabilidad entre los diferentes grupos genómicos de la CCM.
El polimorfismo obtenido con las cuatro enzimas consideradas permite predecir que al interior de la CCM existe una variabilidad significativa y que es posible detectar la variabilidad presente de los diferentes grupos genómicos con las enzimas DIAP, PRX y ab- EST. A partir del patrón de bandeo presentado por la enzima ab-EST, sin un estudio de herencia, se puede deducir que, posiblemente, la estructura de la enzima para las siete zonas (A, B, C, D, E, F y G) es monomérica (Figura 2). Dentro del grupo Plantain (AAB) con las cuatro enzimas utilizadas para el análisis estadístico fue posible discriminar algunos genotipos y detectar tres grupos como posibles duplicados: uno constituido por Bend Mossendjo y Dominico Ancuyano, otro por Plantain 17 y Hartón Birracimo, y el tercero compuesto por Dominico Maqueño y Dominico 300 (Figura 5).
La banda 10 ab-EST (Figura 2a) sugiere la existencia de una característica única en los clones Rose D'ekona y Dominico Común, lo que podría ser interesante si se logra hacer una correlación de esta banda con un carácter morfoagronómico de importancia. Adicionalmente, el polimorfismo presentado por esta enzima permite diferenciar claramente a la especie M. laterita, de la sección Rhodochlamys (Figura 2a) por la banda 9, única de los clones pertenecientes a la sección Eumusa.
Los patrones presentados por la enzima DIAP en las zonas A, B, C y D corresponden, posiblemente, a una estructura monomérica (Figura 4a). De todas las enzimas evaluadas, DIAP es la única que permite evaluar la variabilidad al interior del grupo Plantain, separando los materiales Dominico Común, Dominico Enano, Dominico Guaicoso y Diby como clones únicos dentro del núcleo común de los Plantain; adicionalmente, muestra cuatro materiales de otros grupos genómicos interesantes por presentar patrones únicos como son: Cachaco Enano (Blugooe), Bocadillo Chileno (Sucrier), Popoulou (Popoulou) y Pompo Comino (Popoulou). Lo anterior indica que se trata de materiales con una variabilidad muy útil para programas de mejoramiento genético, por tanto es interesante conocer otras características fisiológicas y/o morfológicas que permitan hacer correlaciones de los mismos con los patrones bioquímicos únicos que los separan del resto de la colección.
El locus GOT-3 y la zona C de la enzima PRX reflejan, posiblemente, una estructura dimérica y monomérica, respectivamente, como se observa en las Figuras 1 y 3. El polimorfismo mostrado por la enzima GOT es insuficiente para un estudio de caracterización ya que sólo permite observar diferencias entre los grupos genómicos y no al interior de cada uno de ellos. Esta enzima presenta una expresión estable que hace posible comparar los resultados, independientemente de las condiciones necesarias para su obtención, como son: buffer de extracción, buffer de corrida y clase de gel, siendo éste un comportamiento similar al conseguido por Jarret y Litz (1986).
El polimorfismo observado con la enzima PRX permite separar los materiales FHIA 02 y Pompo Comino, al presentar un patrón de bandas peculiar que los hace formar grupos distintos y únicos, lo que sugiere la riqueza genética que encierran estos clones. Un estudio del polimorfismo de isoenzimas en setenta cultivares de Musa, pertenecientes a los grupos genómicos AB, AAA, AAB y ABB, mostró que siete sistemas enzimáticos fueron apropiados para la identificación de los cultivares (Bhat et al., 1992 a,b). Aunque la ampliación del polimorfismo observado fue alta, no fue suficiente para distinguir todos los cultivares analizados ya que el 25% de estos compartieron el mismo perfil isoenzimático.
En el análisis elaborado con todas las enzimas, los materiales de banano se distribuyeron formando tres grupos: (1) constituido por FHIA 03 y Bocadillo Chileno, (2) constituido por Cachaco Enano, Saba e IC2, Gros Michel y (3) constituido solo por FHIA 02 (Figura 5). Para el primer grupo es de esperar que el material FHIA 03 se agrupe con Bocadillo Chileno, ya que es un clon híbrido derivado de dos progenitores de genoma AAB y AA, y Bocadillo Chileno es de genoma AA. Esto permite observar la posible dominancia del genoma acuminata, lo cual sería de interés para cruces en los que se busque aumentar la variabilidad de los bananos comerciales, si se tiene en cuenta que un diploide AA, como es el caso de Bocadillo Chileno, representa un genoma más cercano a los parientes silvestres o ancestrales de los bananos cultivados actuales. En el segundo grupo, los materiales Cachaco Enano y Saba, plátanos con genoma ABB, se agrupan con el banano IC2-GrosMichel, de genoma AAAA, un banano calidad exportación. Si se tiene en cuenta que Saba es uno de los pocos plátanos que se pueden consumir maduros como fruta y que los resultados bioquímicos lo agrupan en calidad exportación, se estaría confirmando la dominancia del genoma acuminata para estos dos plátanos, lo cual los hace interesantes para el mejoramiento. El tercer grupo, conformado por FHIA 02, confirma que este material es de gran relevancia para el mejoramiento en musáceas. La caracterización isoenzimática con electroforesis de isoenzimas permite agrupar los plátanos colombianos con un mínimo de variabilidad genética, como descendientes de una o dos ramas comunes, diferenciándolas de las introducciones recientes del Oeste africano, las cuales también revelan entre sí un agrupamiento estrecho. Esto muestra la existencia de plátanos que han evolucionado, adaptándose a diferentes agroecosistemas colombianos (Rosales y Pocasangre, 2002).
Estos resultados indican que existe alta variabilidad al interior del grupo de los bananos ya que, a pesar de tener un bajo número de clones representados en la colección de estudio, la mayoría se separan como materiales únicos al realizar el análisis con todas las enzimas. También se encontró que las isoenzimas tienen buen potencial para la discriminación de los cultivares de Musa, estudios que se pueden complementar con los marcadores moleculares para la diferenciación entre los grupos acuminata y balbisiana y para entender las relaciones estrechas de variabilidad entre los cultivares de plátano (Sánchez et al., 1998). Los resultados de este trabajo también confirman lo expuesto por Chandel y Anuradha (2000) quienes sostienen que la caracterización genética es esencial para el desarrollo de estrategias científicas para la conservación del germoplasma de plátano y banano en colecciones.
- Las enzimas ab-EST, PRX y DIAP son óptimas para evaluar variabilidad en Musa spp y podrían ser marcadores enzimáticos eficientes, mientras GOT es una enzima estable y su expresión no depende de las condiciones de extracción o corrida.
- Los clones FHIA 02 y Pompo Comino representan una fuente importante de variabilidad dentro de la CCM, mientras que entre los clones Plantain 17 y Hartón Birracimo, Dominico 300 y Dominico Maqueño, al igual que entre Bend Mossendjo y Dominico Ancuyano, posiblemente exista una relación de duplicación.
- El número de clones presente por cada grupo genómico en la CCM y los resultados de los análisis con todas las enzimas sugieren la necesidad de incorporar un mayor número de clones tradicionales cultivados y silvestres para confirmar la clasificación de los materiales ya existentes y, adicionalmente, aumentar la variabilidad genética por grupo genómico en la Colección Colombiana de Musáceas.
Bakry, F.; Carreel, F.; Caruana, M. L.; Cóte, F.; Jenny, C.; y Tezenas, H. 2001. Banana. En: Charrier, A.; Jacquot, M. Hamon, S.; y Nicolas, D. (eds.). Tropical plant breeding, CIRAD-SPI, Montpellier, France. p.1-29. [ Links ]
Bhat, K. V.; Bhat, S. R.; y Chandel, K. P. 1992a. Survey of isozime polymorphism for clonal identification in Musa. I. Esterase, acid phosphatase and catalase. J. Hort. Sci. 67:501 - 507. [ Links ]
Bhat, K. V.; Bhat, S. R.; Chandel, K. P. 1992b. Survey of isozime polymorphism for clonal identification in Musa. II. Peroxidase, superoxide dismutase, shikimate dehydrogenase and malate dehydrogenase. J. Hort. Sci. 67:737 - 744. [ Links ]
Bhat, K. V. y Jarret, R. L. 1995. Random amplified polymorphic DNA and genetic diversity in Indian Musa germplasm. Genet. Res. Crop Evol. 42:107 - 118. [ Links ]
Bhat, K. V.; Jarret, R. L.; y Rana, R. S. 1995a. DNA profiling of banana and plantain cultivars using random amplified polymorphic DNA (RAPD) and restriction fragment length polymorphism (RFLP) markers. Electrophoresis 16:1736 - 1745. [ Links ]
Bhat, K. V.; Bhat, S. R.; Chandel, K. P.; Lakhanpaul, S.; y Ali, S. 1995b. DNA fingerprinting in Musa cultivars with oligodeoxyribonucleotide probes specific for simple repeat motifs. Genet. Anal. 12: 45-51. [ Links ]
Belalcázar, S. (ed.). 1991. El Cultivo de plátano (Musa AAB Simmonds) en el trópico. Manual de asistencia técnica No. 50. Instituto Colombiano Agropecuario (ICA), Ed. Feriva. Cali. p. 45-89 [ Links ]
Bonner, J. W.; Warner, R. M.; y Brewmarker, J. L. 1974. A chemostatic study of Musa cultivars. HortSci. 9:325 - 328. [ Links ]
Chandel, K. P. y Anuradha, A. 2000. Genetic resources of banana in India: collection in vitro conservation and characterization. En: Shing, H.P. y Chadha, K.L. (eds.). Banana - Improvement, Production & Utilization. Proceedings of the Conference on Challenges for Banana and Utilization in 21st Century. Association for the Improvement in Production and Utilization of Banana (AUIPUB). National Research Centre on Banana (NRCB), Tricy, India, p. 128-135. [ Links ]
Crisci, J.V. 1983. Introducción a la teoría y práctica de la taxonomía numérica, Washington, D.C., 133 p. [ Links ]
De Langhe, E. 1987. Necesidad de una estrategia Internacional para el mejoramiento genético del banano y del plátano. En: Jaramillo, R. y Mateo, N. (eds.). Memorias de la reunión regional del INIBAP para América Latina y el Caribe. Costa Rica. p. 181-199. [ Links ]
Gawel, N. J.; Jarret, R. L.; y Wittermore, A. 1992. Restriction fragment length polymorphism (RFLP)- based phylogenetic analysis of Musa. Theor. Appl. Genet. 84:286 - 290. [ Links ]
Horry, J. P. y Jay, M., 1988. Distribution of anthocyanin in wild and cultivated banana varieties. Phytochemical 27:2667 - 2672. [ Links ]
Horry, J.P . 2000. Status and characterization of banana genetic resources. En: Shing, H.P. y Chadha, K. L. (eds.). Banana - Improvement, Production & Utilization. Proceedings of the Conference on Challenges for Banana and Utilization in 21st Century. Association for the Improvement in Production and Utilization of Banana (AUIPUB). National Research Centre on Banana (NRCB), Tricy, India. p. 117 - 127. [ Links ]
Jarret, R. L. y Litz, R. E. 1986. Isoenzymes as genetic markers in bananas and plantains. Euphytica 35:539 - 549. [ Links ]
Jarret, R. L.; Gawel, N. J.; y Wittermore, A. 1992. RFLP-based phylogeny of Musa species in Papua New Guinea. Theor. Appl. Genet. 84:579 - 584. [ Links ]
Jarret, R. L.; Vulysteke, D.; Gawel, N. J.; Pimentel, R.; y Dunbar, L. 1993. Detecting genetic diversity in diploid bananas using PCR and primers from a highly repetitive DNA sequence. Euphytica 68:69 - 76. [ Links ]
Kaemmer, D.; Afza, R.;Weisang, K.; Kahl, G.; y Novak, F. J. 1993. Oligonucleotide and amplification fingerprinting of wild species and cultivars of banana (Musa spp.). Biotechnol. 10:1030 - 1035. [ Links ]
Karamura, D. A. 1999. Numerical taxonomic studies of the east African highland bananas (Musa AAA-East Africa) in Uganda. Ph.D. Thesis, The University of Reading. IPGRI, INIBAP, Montpellier, Francia. 192 p. [ Links ]
Lannaud, C.; Tezenas, H.; Jolivot, M.P.; Glaszmann, J.C.; y González de León, D. 1992. Variation of ribosomal gene spacer length among wild and cultivated bananas. Hered. 68:147 - 156. [ Links ]
Gómez, P. L.; Belalcázar, S.; y Martínez, A. 1987. Programa de plátano y banano en Colombia - Marco Orientador-. En: Jaramillo, R. y Mateo, N. (eds.). Memorias de la reunión regional del INIBAP para América Latina y el Caribe, San José, Costa Rica. p. 52-74. [ Links ]
León, J. 1987. Botánica de los cultivos tropicales. Instituto Interamericano de Cooperación para la Agricultura (IICA). San José, Costa Rica. p. 88-92. [ Links ]
Nadal, R.; Manzo, G.; Orozco, J.; Orozco, M.; y Guzmán, S. 2008. Diversidad genética de clones de bananos y plátanos (Musa spp.) usando marcadores RAPD. En: XVIII Reunión de la Asociación para la Cooperación en Investigaciones de Banano en el Caribe y en América Tropical (ACORBAT), Guayaquil, Ecuador. 9 p. Memorias CD Rom [ Links ]
Perea, M. 2001. La biotecnología como soporte en el mejoramiento de las musáceas. En: Biotecnología agrícola, un enfoque hacia el mejoramiento de plantas. Editora Guadalupe, Bogotá, Colombia. p. 138 - 149. [ Links ]
Perea, M. 2003. Biotecnología, bananos y plátanos. Editora Guadalupe, Bogotá. 228 p. [ Links ]
Purseglove, J. W. 1985. Crops Monocotyledons. Longman Group Limited, Singapore. p. 343 - 384. [ Links ]
Quiroz, C. F. 1991. Isoenzimas como marcadores genéticos para identificar híbridos en el cultivo de tejidos. En: Roca, W. y Mroginski L.A. (eds.). Cultivo de tejidos vegetales en la agricultura: Fundamentos y Aplicaciones. Centro Internacional de Agricultura Tropical (CIAT), Cali. p. 858 - 876. [ Links ]
Ramírez, H.; Hussain, A.; Roca, W.; y Bushuk, W. 1987 Isozyme electrophoregrams of sixteen enzymes in five tissues of cassava (Manihot esculenta Crantz) varieties. Euphytica 36:39 - 48. [ Links ]
Rosales, F. y Pocasangre, L. 2002. Oferta tecnológica de banano y plátano para América Latina y el Caribe. Una contribución de Musalac a la investigación y desarrollo de las Musáceas. Red Internacional para el Mejoramiento del Banano y el Plátano, INIBAP. Montpellier, Francia. 29 p. [ Links ]
Rodríguez, P. A. y Cayón, G. 2008. Efecto de Mycosphaerella fijiensis sobre la fisiología de la hoja de banano. Agronomía Colombiana 2 (2):256 - 265. [ Links ]
Rohlf, F. J. 2007. NTSYS. Numerical taxonomy and multivariate analysis system. Versión 2.1. Exeter Publications, Setauket, Nueva York. 31 p. [ Links ]
Sánchez, I.; Gaviria, D.; Gallego, G.; Reyes, L. M.; Giraldo, M.C.; Fajardo, D.; Valencia, J. A.; Lobo, M.; Tohme, J.; y Roca, W.. 1998. Caracterización bioquímica y molecular de la colección colombiana de musáceas. En: Giraldo, M. J.; Belalcázar, S. L.; Cayón, D. G.; y Botero, R. G. (eds.). Seminario Internacional sobre Producción de Plátano. Corpoica, Universidad del Quindío, Comité de Cafeteros del Quindío, Sena, Armenia, Colombia. Memorias. p. 26 - 31. [ Links ]
Simmonds, N. W. y Shepherd, K. 1955. The taxonomy and origins of cultivated bananas. J. Linn. Soc. (Bot). Londres 55:302 - 312. [ Links ]
Simmonds, N. W. 1966. Bananas. Longman, Londres. 468 p. [ Links ]
Simmonds, N.W. 1979. Evolutions of crop plants. Edinburhg School of Agrculture. Longman, Londres. [ Links ]
Stover, R. H. y Simmonds, N. W. 1962. Bananas. 3rd ed. Longman, Nueva York. [ Links ]














