Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.63 no.4 Palmira Oct./Dec. 2014
https://doi.org/10.15446/acag.v63n4.42928
Doi: http://dx.doi.org/10.15446/acag.v63n4.42928
Fitomejoramiento y Recursos Genéticos
Caracterización con RAMs de la colección de durazno (Prunus persica L. Batsch) existente en la Universidad Pedagógica y Tecnológica de Colombia
Characterization with RAMs of the peaches (Prunus persica L. Batsch) collection at the Pedagogical and Technological University of Colombia
Ana Cruz Morillo Coronado1*, Yacenia Morillo Coronado2 y Elberth Hernando Pinzón Sandoval1
1Universidad Pedagógica y Tecnológica de Colombia, Tunja, Boyacá, Colombia; 2Universidad de los Llanos, Villavicencio, Meta, Colombia *Autor para correspondencia: ana.morillo@uptc.edu.co
Rec.:21.06.2014 Acep:09.07.2014
Resumen
Utilizando ocho cebadores Microsatélites Amplificados al Azar (RAMs) fue evaluada la diversidad genética de 31 materiales de Prunus de la colección de caducifolios existente en la Universidad Pedagógica y Tecnológica de Colombia. Se generaron un total de 121 amplicones con pesos moleculares entre 260 y 1000 Kb. Con un coeficiente de similitud de 0.75, se formaron tres grupos, de acuerdo principalmente con las características del fruto, encontrando en el grupo 2 las variedades de importancia económica. El número de loci polimórficos varió entre 5 y 16 para los cebadores GT y CCA, respectivamente. El valor promedio de heterocigosidad fue de 0.22, más bajo que los encontrados en otros estudios de diversidad genética en el género Prunus; por tanto, se deben buscar estrategias para incrementar la variabilidad genética de la colección como la introducción de nuevos materiales o híbridos interespecíficos. La técnica RAMs es útil para evaluar la diversidad genética en especies caducifolias.
Palabras clave: Marcadores genéticos, Prunus, diversidad genética, microsatélites RAMs, caducifolios.
Abstract
A sample of 31 Prunus materials from the deciduous collection of the Pedagogical and Technological University of Colombia was selected to evaluate its genetic diversity using eight primers for Random Amplified Microsatellite (RAMs). A total of 121 amplicons were generated with molecular weights ranging between 260 and 1000 Kb. A similarity coefficient of 0.75, three groups were formed, according to fruit characteristics, all economically important varieties in one group. The average value of heterozygosity was 0.22, much lower than values found in other studies. Therefore, strategies must be found to increase genetic variability in the collection, such as introduction or new or interespecific hybrid exemplars among others. The RAMs technique is a useful tool for assessing genetic diversity in the genus Prunus.
Key words: Genetic markers, Prunus, genetic diversity, Microsatellites RAMs, deciduous fruit trees.
Introducción
El durazno (Prunus persica L. Batsch), también llamado melocotonero, es una de las especies frutales caducifolias más cultivadas en las zonas templadas del mundo. Su fruto presenta buenas características nutritivas, lo que lo hace un alimento saludable con importante uso agroindustrial en la elaboración de conservas, pulpa, néctar, mermelada y deshidratados (Mendoza et al., 2010).
Boyacá es uno de los departamentos de Colombia pioneros en la producción nacional de caducifolios de clima frío, por lo que la Facultad de Agronomía de la Universidad Pedagógica de Colombia (UPTC) y la Universidad Técnica de Berlín desarrollaron en la granja experimental de Tunguavita, Paipa, un proyecto para el establecimiento de una colección de materiales caducifolios importados de diferentes pisos térmicos (Fisher y Torres, 1990). En 2008 Puentes et al. (2008), realizaron un estudio de caducifolias teniendo en cuenta sus ventajas comerciales y hallaron que el durazno y el ciruelo son una alternativa productiva para este departamento.
La identificación molecular usando marcadores de ADN se ha convertido en la principal herramienta para la caracterización y manejo de las colecciones de germoplasma de muchas especies frutales. Los marcadores Microsatélites (SSRs) son la técnica preferida para los estudios de diversidad genética entre las especies cultivadas, debido a su alto polimorfismo, abundancia y herencia codominante. En Prunus esta técnica ha sido utilizada para análisis genéticos (Ercisli et al., 2011) y en la identificación y 'finger-printing' de cultivares (Aranzana et al., 2010), estudios de autoincompatibilidad y estructura poblacional (Vaughan et al., 2007), creación de mapas genéticos de ligamiento (Cao et al., 2012), identificación de QTLs (Campoy et al., 2011), análisis de secuencias del genoma (Scalibrin et al., 2013) y en la selección asistida por marcadores moleculares (MAS) (Pozzi y Vecchietti, 2009).
Los RAMs, entre los marcadores microsatélites, son muy útiles para medir la diversidad genética en plantas y animales e identificar diferencias entre familias, especies y al interior de la especie; además esta metodología es factible en pequeños laboratorios (Muñoz et al., 2008). Trabajos sobre caracterización de diversidad genética vegetal sugieren que esta técnica es útil para identificar duplicados y establecer relaciones entre y dentro de especies (Sanabria et al., 2006; Bonilla et al., 2008; Morillo et al., 2009). En este contexto, este trabajo de investigación tuvo como objetivo principal estudiar la diversidad genética de la colección de duraznos en la Universidad Pedagógica y Tecnológica de Tunja, empleando los marcadores moleculares RAMs, con el propósito de utilizar los resultados en el desarrollo de estrategias de mejoramiento, así como en un futuro contribuir a la identificación de materiales que respondan a las necesidades del mercado.
Materiales y métodos
Se evaluaron un total de 28 accesiones de durazno, dos de albaricoque (Prunus armeniaca L.) y una de almendro (Prunus dulcis (Mill.) D. A. Webb) establecidas en la granja experimental de Tunguavita, Paipa, a 36 km de la ciudad de Tunja con una latitud norte de 5° 47' 04" y longitud oeste de 73° 07' 17", temperatura, promedio de 15 °C (Cuadro 1).
Caracterización molecular
La caracterización molecular se hizo en los Laboratorios de Investigación en Biología Molecular, Bioplasma y Gebimol, de la Universidad Pedagógica de Colombia, Tunja. Para la extracción de ADN se utilizó el protocolo de Dellaporta et al. (1983). Los ADN totales se visualizaron en geles de agarosa a 0.8%, en una cámara Maxicell Primo EC-340 Electroforesis Gel System. Para determinar la concentración de ADN se utilizó el fluorómetro Hoefer Dyna Quant 200 y se diluyó en agua tipo HPLC a un volumen total de 100 µl a 10 ng/µl y se almacenó a -20 °C. Para el análisis RAMs se utilizaron ocho cebadores sintetizados por Technologies Inc. Bioneer (Cuadro 2). Para la reacción de amplificación con RAMs se preparó el cóctel en un tubo estéril de microcentrífuga (1.5 ml) para un volumen final de 25 µl. La mezcla de reacción se preparó con buffer 1X, MgCl2 1.5 mM, dNTPs 0.2 mM, Taq Polimerasa 1U, cebador 2 µM y ADN genómico 10ng.
La amplificación se llevó a cabo en un termociclador PTC 100 Programmable Termal Controller (MJ. Research, Inc), en el laboratorio de molecular. La desnaturalización inicial fue a 95 °C durante 5 min; desnaturalización a 95 °C por 30 seg, hibridación a 50 °C (cebador AG y CA), 55 °C (cebador CCATG-CT) y 58 °C (cebador GT-CGA) durante 45 seg, una extensión de 72 °C por 2 min, 37 ciclos desde la desnaturalización a extensión y por último, una extensión a 72 °C durante 7 min. Los productos amplificados fueron separados por electroforesis en geles de poliacrilamida 37:1 (acrilamida:bisacrilamida) al 7% a 150 v por 1 h en una cámara pequeña de DNA Sequencing System, FB-SEQ-3545 de Fisher Biotechnologies. La tinción se realizó usando sales de plata y siguiendo el protocolo de Crest et al. (2001).
Análisis estadístico de datos
Se generó una matriz binaria de ausencia (cero) y presencia (uno). La similitud genética entre los individuos se calculó utilizando el coeficiente de similitud de Nei y Li (1979). El análisis de agrupamiento se realizó por el método UPGMA y se generó un dendrograma empleando el paquete estadístico NTSYS (Numerical Taxonomy System for personal Computer, versión 2.02 PC). Para evaluar la diversidad genética se estimó la heterocigosidad insesgada y el porcentaje de loci polimórficos con el paquete estadístico TFPGA (Tools For Population Genetic Analysis, versión 1.3, 1997). Se determinó el valor 'F' estadístico insesgado con un intervalo de confianza de 95 %.
Resultados y discusión
El análisis mediante el coeficiente de Nei-Li a un nivel de similitud de 0.75 permitió diferenciar los materiales en tres grupos (Figura 1). En el Grupo I se encuentran algunas variedades introducidas desde Ecuador entre ellas, Conservero de piel y pulpa amarilla y otras provenientes de Venezuela, como Jarrillo de piel rojiza con tonalidades amarillas, forma ligeramente redondeada y de tamaño grande con peso aproximado de 150 g. Este último durazno se estableció con buena adaptación en el departamento del Norte de Santander, especialmente en los municipios de Pamplona, Chitagá, Silos y Cácota (Campos, 2013). Dentro de este grupo se encuentra Camuezo uno de los materiales utilizado con éxito como porta-injerto de todas las variedades de durazno y algunas de ciruelo, es de piel y pulpa amarilla, forma redondeada, tamaño mediano y peso promedio de 125 g, con una producción prolífica pero ligeramente tardía aunque susceptible a daños en la manipulación poscosecha (Campos, 2013).
En el mundo, existen más de 15.000 variedades de durazno (Castro et al., 1998). En Colombia se destacan las variedades Fresa morado, Pezón de Venus, Fresa blanco, Elberta gigante, Robidoux, Frank, Ventura, Diamante, Riograndense, Dorado, Conservero amarillo y Zapallo (Ordóñez, 2007), algunas de las cuales se encuentran dentro del Grupo II, donde también se incluyen las variedades de durazno propiamente dichas (Blanco común, Florida, Riograndense, Dorado y Diamante) y las de durazno-melocotón (Rubidoux, Rey Negro) (Campos, 2013).
La variedad Florida se adapta a las condiciones del departamento del Cauca (Colombia) donde se cultiva a 2.400 m.s.n.m en los municipios de Silvia y Sotará. Sus frutos son de buena calidad, muy jugosos de color rojo y pulpa amarilla, la producción empieza tres años después de la siembra y puede producir durante 20 años (Serna y Oliva, 1999).
Las variedades Dorado y Diamante se caracterizan por su piel amarilla con ligera pigmentación roja, pulpa amarilla-oro y coloración rojiza alrededor del hueso de la semilla. La primera presenta buenas características para la industrialización y por su contenido de azúcar es excelente para el consumo en fresco; mientras que la segunda se destaca por ser plantas muy vigorosas y productivas. La variedad Oro con un color amarillo atractivo, muestra buenas propiedades organolépticas (Baiza, 2004).
El durazno blanco posee una pulpa ligeramente insípida y se utiliza básicamente en la industria de mermeladas, compotas y otras. Su semilla es requerida para la obtención de patrones tanto para durazno como para ciruelos, aunque su producción es muy baja, la planta no defolia y en algunas zonas del país florece en épocas de lluvias (Campos, 2013).
La variedad Rubidoux, dentro de las variedades de durazno-melocotón, es resistente al manejo poscosecha, tiene pulpa amarilla con coloración rojiza y una forma ligeramente redondeada. En estas mismas variedades, Rey Negro presenta piel amarilla con manchas rojas opacas, presencia de un pezón muy pronunciado (Foto 1) que dificulta su manejo poscosecha (Campos, 2013).
En el Grupo III se encuentra el albaricoque (Prunus armeniaca L.) subgénero Prunophora. Su importancia económica reside en sus frutos, con más de 100 especies cultivadas, aunque también existen variedades de determinadas especies cuyo uso principal es como planta ornamental (Rodrigo y Herrero, 2002). Dentro de las variedades más importantes se encuentra Bulida, de origen español, adaptado a varios tipo de suelos y alto contenido de jugo; otra variedad es Canina, con fruto de buena calidad, alta comercialización (Martínez et al., 2004) siendo la de menor similitud con el resto de los materiales evaluados. El almendro es una especie rústica que se adapta a las condiciones extremas de los climas secos, es de clima templado por lo que su floración es temprana y poco exigente en temperaturas bajas, su afinidad con albaricoque es muy alta (Gallego, 2010) como se observa en el dendrograma de este estudio.
En términos generales, los agrupamientos de los materiales de duraznos evaluados con marcadores RAMs corresponden con las características morfológicas del fruto, lo que ha sido encontrado en otros estudios de diversidad genética usando marcadores microsatélites en Prunus (Stanys et al., 2008). En relación con las variedades de durazno, Alvarado (2003) sugiere el estudio de variedades de bajos requerimientos de temperatura fría con potencial para cultivo en condiciones ambientales diferentes, entre ellas Flor da Prince, Tropic Beauty, Tropic Sweet y Rayon.
Los ocho cebadores RAMs utilizados para la caracterización molecular en durazno, generaron un total de 121 alelos que fluctuaron entre 9 para el cebador GT y 26 para el cebador ACA, con pesos moleculares entre 260 y 1.000 kb. El número de loci polimórficos varió entre 5 y 16 para los cebadores GT y CGA, respectivamente (Cuadro 3). El número de alelos se puede considerar adecuado para estimar parámetros genéticos, si se compara con los resultados obtenidos en otras especies de Prunus (Stanys et al., 2012; Fernández et al., 2009). El cebador CCA hizo el mayor aporte a la variación genética observada, Fst de 0.85, lo que significa que puede ser útil para una mayor diferenciación entre los materiales del género Prunus.
Los valores de heterocigosidad variaron entre 0.23 para el cebador ACA y 0.38 para CCA. El valor promedio para todos los materiales evaluados fue de 0.22, similar al encontrado por Bouhadida et al. (2011) (0.23) pero más bajo que los hallados en duraznos por Aranzana et al. (2012) (0.31), Li et al. (2013) (0.60), Turkoglu et al. (2010) (0.72) y en otras especies de Prunus (Rogatis et al. (2013) (0.70), Bourguiba et al. (2010) (0.56), y Stanys et al. (2012) (0.68)). No obstante, fue más alto que los valores observados en la caracterización de diversidad y estructura genética de poblaciones naturales de P. davidiana (0.17) (Cheng et al., 2011) e igual a la heterocigosidad presente en poblaciones de durazno por Martínez et al. (2003).
Los bajos niveles de heterocigosidad se atribuyen al sistema de cruzamiento, ya que la mayoría de cultivares de durazno son autocompatibles lo que favorece los procesos de autofecundación (Sassa et al., 1992). Estos bajos niveles de polimorfismos también se han hallado en materiales de cereza dulce (Boskovic et al., 1998), lo que probablemente refleja una estrecha base genética en el germoplasma de ciruela dulce y de durazno. Imbert y Lefèvre (2003), por otra parte, consideran que un exceso de heterocigosidad no es frecuentemente usual en especies arbóreas y generalmente esta es favorecida por la autoincompatibilidad y por los cruzamientos interespecíficos (Stoeckel et al., 2006). L a estrecha base genética utilizada para el desarrollo de cultivares y la existencia de a ncestros comunes en los pedigree de duraznos y ciruelas, podrían explicar los resultados encontrados (Okie y Hancock, 2008). E n general los bajos niveles de variabilidad son consistentes con los obtenidos usando diferentes marcadores moleculares en especies del género Prunus (Aranzana et al., 2010).
Los resultados son corroborados con el coeficiente de diferenciación genética (Fst) obtenido al evaluar los 31 materiales de Prunus con ocho marcadores RAMs, el cual fue de 0.44 con una desviación estándar de 0.04 (Cuadro 3). Según Wright (1978) valores de 0.25 muestran gran diferenciación genética, lo cual puede estar reflejado en el alto grado de domesticación que han sufrido estos materiales, ya que la mayoría corresponden a variedades comerciales. El índice de fijación (F) y el poder de discriminación de los marcadores fueron más bajos que los reportados en otros estudios en durazno (Bouhadida et al., 2011), las diferencias pueden ser debidas tanto al tipo de materiales como al bajo número de marcadores microsatélites RAMs utilizados. La mayor parte de los duraznos evaluados corresponden a variedades donde la variabilidad genética es menor y los resultados son consistentes con los observados en especies autoincompatibles como cereza (Mariette et al., 2010) donde los cultivares locales son más diversos que las variedades debido, en parte, al proceso evolutivo que sufren las especies propagadas vegetativamente (McKey et al., 2010). Es necesario señalar que el durazno es una de las especies menos polimórficas dentro del género Prunus debido a su incompatibilidad (Font et al., 2014).
Los marcadores RAMs fueron útiles para estudiar la diversidad genética en materiales de durazno, al formar tres grupos a distancias genéticas muy reducidas, incluso con los materiales de albaricoque y almendro (0.74 a 0.80) y por ende con la menor variabilidad genética entre los materiales evaluados. Yoon et al. (2006) y Fernández et al. (2009) encontraron distancias genéticas más reducidas en duraznos y nectarinas (0.34) y almendros (0.36). Browicz y Zohary, (1996) también observaron similitudes genéticas entre especies de Prunus y almendro y/o albaricoque.
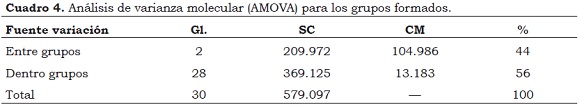
El análisis de varianza para los tres grupos mostró que la variación en los materiales evaluados se encuentra dentro de cada grupo, con un valor de 56% (Cuadro 4). Esta alta variación podría indicar la existencia de niveles de subdivisión y jerarquización mayores que los evaluados en este estudio. El 44% restante se debió al componente de varianza genética entre grupos, ésta fue significativa (P < 0.001) lo que indica la existencia de diferenciación genética molecular entre los tres grupos, la cual puede ser aprovechada en los procesos de conservación y mejoramiento genético de la especie. Con el uso de marcadores SNPs y análisis de secuencias se han realizado estudios más detallados de la variabilidad genética del genoma del durazno, que muestran las complejas relaciones genéticas que pueden existir entre y dentro de las especies del género Prunus (Scalabrin et al., 2013; Vicedomini et al., 2013).
Los análisis SSR de este germoplasma ofrecen oportunidades para una determinación más precisa de las relaciones genéticas y pueden ser una importante herramienta para la transferencia de genes asistida por marcadores, al identificar esas variantes a nivel del genoma. La introgresión de características de interés mediante la hibridación interespecífica y la conservación y el estudio de materiales silvestres, son algunas de las alternativas que pueden ser implementadas en la colección de duraznos estudiada, para ampliar su base genética y hacer que los procesos de selección puedan ser más eficientes y permitan, en un futuro próximo, identificar materiales élite adaptados a las condiciones del departamento y que satisfagan las necesidades del agricultor, del productor y del consumidor.
Conclusiones
- Utilizando la técnica RAMs se encontró una baja diversidad genética en la colección de duraznos en la Universidad Pedagógica y Tecnológica de Colombia debido principalmente a que la mayoría son variedades; por tanto es necesario implementar estrategias como la introducción de nuevos materiales o realizar cruces interespecíficos con el fin de incrementar su variabilidad.
Agradecimientos
Los autores agradecen la colaboración de los laboratorios de investigación Bioplasma y Gebimol, a la granja experimental de Tunguavita y la Dirección de Investigaciones, DIN, de la Universidad Pedagógica y Tecnológica de Tunja
Referencias
Alvarado, H. 2003. El Cultivo del Melocotón. Presentación - Capacitación. Instituto Interamericano de Cooperación Agrícola (IICA) - Frutal ES, Nueva San Salvador. 25 p. [ Links ]
Aranzana, M.; Abbassi, E.; Howad, W.; y Arus, P. 2010. Genetic variation, population structure and linkage disequilibrium in peach commercial varieties. BMC. Genet. 11:69 - 79. [ Links ]
Aranzana, M.; Illa, E.; Howad, W.; y Arús, P. 2012. A first insight into peach (Prunus persica (L.) Batsch) SNP variability. Tree. Genet. Genom. 8:1359 - 1369. [ Links ]
Baíza, V. 2004. Guía técnica del cultivo del melocotón. Programa Nacional de Frutas del Salvador. 46 p. [ Links ]
Bonilla, M.; Espinosa, K.; Posso, A.; Vásquez, H.; y Muñoz, J. 2008. Caracterización molecular de 43 accesiones de uchuva de seis departamentos de Colombia. Act. Agron. 57:109 - 115. [ Links ]
Boskovic, R.; Russell, K.; y Tobutt, K. 1998. Inheritance of stylar ribonucleases in cherry progenies, and reassignment of incompatibility alleles to two incompatibility groups. Euphy. 95:221 - 228. [ Links ]
Bouhadida, M.; Moreno, M.; Gonzalo, M.; Alonso, J.; y Gogorcena, Y. 2011. Genetic variability of introduced and local Spanish peach cultivars determined by SSR markers. Tree Genet. Genom. 7:257 - 270. [ Links ]
Bourguiba, H.; Krichen, L.; Audergon, J.; Khadari, B.; y Farah, N.2010. Impact of mapped SSR markers on the genetic diversity of apricot (Prunus armeniaca L.) in Tunisia. Plant. Mol. Biol. Rep. 28:578 - 587. [ Links ]
Browicz, K. y Zohary, D. 1996. The genus Amygdalus L. (Rosaceae): Species relationships, distribution and evolution under domestication. Genet. Resour. Crop Evol. 43:229 - 247. [ Links ]
Campos, E.; Miranda, D.; Fischer, G.; y Carranza, C.; 2013. Los frutales caducifolios en Colombia. Especies y Variedades de hoja caduca en Colombia. Dvini S.A, Bogotá. 47 - 65p. [ Links ]
Campoy, J.; Ruíz, D.; Egea, J.; Rees, D.; Celton, J.; y Martínez, P. 2011. Inheritance of flowering time in apricot (Prunus armeniaca L.) and analysis of linked Quantitative Trait Loci (QTLs) using Simple Sequence Repeat (SSR) markers. Plant. Mol. Biol. Rep. 29:404 - 410. [ Links ]
Cao, K.; Wang, L.; Zhu, G.; Fang, W.; Chen, C.; y Luo, J. 2012. Genetic diversity, linkage disequilibrium, and association mapping analyses of peach (Prunus persica) landraces in China. Tree. Genet. Genom. 8:975 - 990. [ Links ]
Castro, S.; Delgado, Z.; Ramirez, F.; y Puentes, M. 1998. Manejo post-cosecha y comercialización de durazno (Prunus pérsica (L.) Batsch). Serie de paquete de Capacitación sobre Manejo de Postcosecha de Frutas y Hortalizas. NRI, DFID, SENA, Convenio SENA Reino Unido. OP Gráficas, Bogotá, Colombia. 372 p. [ Links ]
Chaparro, P.; Sánchez, L.; Pinzón, E.; Sleby, J.; y Mariño, L. 2010. Portafolio de servicios área agrícola, granja experimental Tunguavita. Universidad Pedagógica y Tecnológica de Colombia. 9 p. [ Links ]
Cheng, Z.; Gasic, K.; Wang, Z.; y Chen, X. 2011. Genetic diversity and genetic structure in natural populations of Prunus davidiana germoplasm by SSR markers. J. Agric. Sci. 3:113-125. [ Links ]
Crest, S.; Tulman-Neto, A.; y Figueira, A. 2001. Detection off single sequence repeat polymorphisms in denaturing polyacrilamide sequencing gels by silver staining. Plant. Mol. Biol. Repor. 19:299 - 306. [ Links ]
Dellaporta, S.; Wood, J.; y Hicks, J. 1983. A plant DNA minipreparation: Versión II. Plant Mol. Biol. Rep. 14:19 - 21. [ Links ]
De Vicente, M.; Truco, M.; Egea, J.; Burgos, L.; y Arús, P. 1998. RFLP variability in apricot (Prunus armeniaca L.). Plant. Breed. 117:153 - 158. [ Links ]
Ercisli, S.; Agar, G.; Yildirim, N.; Duralija, B.; Vokurka, A.; y Karlidag, H. 2011. Genetic diversity in wild sweet cherries (Prunus avium) in Turkey revealed by SSR markers. Genet. Mol. Res. 10:1211 - 1219. [ Links ]
Fernández, A.; Martí, J.; Alonso, M.; Espiau, M.; Rubio, M.; y Socias, R. 2009. J. Amer. Soc. Hort. Sci. 134:535 - 542. [ Links ]
Fisher, G. y Torres, F. 1990. Simposio Internacional de Fruticultura en los Altiplanos Tropicales. Tunja, 1990. 23 p. [ Links ]
Font, C.; Gradziel, T.; Gogorcena Y; y Moreno, M. 2014. Phenotypic diversity among local spanish and foreign peach and nectarine (Prunus persica L.) Batsch) accessions. Euphy. 197:261 - 277. [ Links ]
Gallego, A. 2010. Manual sobre el cultivo del almendro en producción ecológica. Asociación para el Desarrollo Sostenible del Poniente Granadino. CAAE. España. 16 p. [ Links ]
Imbert, E. y Lefèvre, F. 2003. Dispersal and gene flow of Populus nigra (Salicaceae) along a dynamic river system. J. Ecol. 91:447 - 456. [ Links ]
Li, X.; Meng, X.; Jia, H.; Yu, M.; Ma, R. et al. 2013. Peach genetic resources: diversity, population structure and linkage disequilibrium. BMC. Genet. 14:84 - 100. [ Links ]
Mariette, S.; Tavaud, M.; Arunyawat, U.; Capdeville, G.; Millan, M.; y Salin, F. 2010. Population structure and genetic bottleneck in sweet cherry estimated with SSRs, and the gametophytic selfincompatibility locus. BMC. Genet. 11:77 - 84. [ Links ]
Martínez, J.; Badene, M.; y Llácer G. 2004. Estado actual del programa de mejoramiento genético de albaricoquero (Prunus armeniaca L.) en la comunidad Valenciana. Frut. Prof. 144:7 - 11. [ Links ]
Martínez, G.; Sozzi, G.; Sánchez, R.; y Rubio, M. 2003. New approaches to Prunus tree crop breeding. Food. Agricult. Environ. 1:52 - 63. [ Links ]
McKey, D.; Elias, M.; Pujol, B.; y Duputié, A. 2010. Ecological approaches to crop domestication. In: GeptsP, BettingerR, Brush SB, FamulaT, McGuirePE, QualsetCO, eds. Biodiversity in agriculture: domestication, evolution and sustainability. Cambridge, UK: Cambridge University Press. 456 p. [ Links ]
Mendoza, A.; Hernández, R.; Ramírez, H.; y Sandoval, A. 2010. Tratado de Botánica Económica Moderna. Universidad Autónoma Agraria Antonio Narro. Buenavista, Saltillo, Coah. 332 p. [ Links ]
Morillo, A.; Morillo, Y.; Chagüeza, Y.; Caicedo, A.; Jaramillo, J.; Muñoz, O.; Arcos, A.; Vásquez, A.; y Muñoz, J. 2009. Caracterización de la diversidad genética en naranja y comparación del polimorfismo de microsatélites amplificados al azar (RAMs) usando electroforesis de poliacrilamida y agarosa. Act. Agron. 58(2): 234 - 244. [ Links ]
Muñoz, J.; Morillo, A.; y Morillo, Y. 2008. Microsatélites Amplificados al Azar (RAMs) en estudios de diversidad genética vegetal. Act. Agron. 57(2):219 - 226. [ Links ]
Nei, M. y Li, W.1979. Mathematical model for studying genetic variation in terms of restricción endonucleasa. Proc. Nat. Acad. Sci. 79:5267 - 5273. [ Links ]
Okie, W. y J.F. Hancock. 2008. Plums. In: Hancock J.F. (ed.). Temperate fruit crop breeding: germplasm to genomics. Kluwer Academic Publisher. Dordrecht, Holanda. p. 337 - 358p. [ Links ]
Ordóñez, O. 2007. Monografía del durazno, Secretaria de desarrollo Rural. Estado de Puebla. Disponible en: http://www.sdr.gob.mx/Contenido/CadenasProductivas.pdf. 26-06-2013. [ Links ]
Pozzi, C.; y Vecchietti, A. 2009. Peach structural genomics. In: Folta K.M. y Gardiner S.E. (eds.). Genetics and genomics of Rosaceae. Springer, Nueva York, p. 235 - 257. [ Links ]
Puentes, G.; Rodríguez, L.; y Bermúdez, L. 2008. Análisis de grupo de las empresas productoras de frutales caducifolios del departamento de Boyacá. Agron. Col. 26(2):146 - 154. [ Links ]
Rodrigo, J. y Herrero M. 2002. Effects of pre-blossom temperatures on flower development and fruit set in apricot. Scien. Hort. 92:125 - 135. [ Links ]
Rogatis, A.; Ferrazzini D.; Ducci, F.; Guerri, S.; y Belleti, P. 2013. Genetic variation in Italian wild cherry (Prunus avium L.) as characterized by nSSR markers. Forestry 86:391 - 400. [ Links ]
Sanabria, H.; García M.; Díaz, H.; y Muñoz, J. 2006. Caracterización molecular con marcadores RAM de árboles nativos de Psidium guajava (guayaba) en el Valle del Cauca. Act. Agron. 55:23 - 30. [ Links ]
Sassa, H.; Hirano, H.; y Ikehashi H. 1992. Self-incompatibility- related RNases in styles of Japanese pear (Pyrus serotina Rehd.). Plant. Cell. Physiol. 33:811 - 814. [ Links ]
Scalabrin, S.; Policriti, A.; Nadalin, F.; Pinosio, S. et al. 2013. A catalog of molecular diversity of Prunus germoplasm gathered from aligning NGS reads to the peach reference sequence: Bioinformatic approaches and challenges. Act. Hort. 976:169 - 176. [ Links ]
Serna, D. y Oliva, L. 1999. El cultivo del durazno. Cartilla divulgativa elaborada por CorpoTunía, Corporación para el Desarrollo de Tunía. 25 p. [ Links ]
Stanys, V.; Frercks, B.; Siksnianiene, B.; Stepulaitiene, I.; Gelvonauskiene, D.; Staniene, G.; y Bobinas, C. 2012. Identification of sweet cherry (Prunus avium L.) cultivars using AFLP and SSR markers. Zem. Agric. 99:437 - 444. [ Links ]
Stanys, V.; Stanytè, R.; Stanienè, G.; y Vinskienè, J. 2008. S-allele identification by PCR analysis in Lithuanian sweet cherries. Biol. 54:22 - 26. [ Links ]
Stoeckel, S.; Grange, J.; Fernández, J. Bilger, I.; Frascaria, N.; y Mariette S. 2006. Heterozygote excess in a self-incompatible and partially clonal forest tree species Prunus avium L. Mol. Ecol. 15:2109 - 2118. [ Links ]
Turkoglu, Z.; Bilgener, S.; Ercisli, S.; y Bakir, M.2010. Simple sequence repeat-based assessment of genetic relationships among Prunus rootstocks. Genet. Mol. Res. 9:2156 - 2165. [ Links ]
Vaughan, S.; Cottrell, J.; Moodley, D.; Connolly, T.; y Russel, K. 2007. Clonal structure and recruitment in British wild cherry (Prunus avium L.). Forest Ecol. Manag. 242:419 - 430. [ Links ]
Vicedomini, R.; Vezzi, F.; Scalabrin, S.; Arvestad, L.; y Policriti, A. 2013. GAM-NGS: genomic assemblies merger for next generation sequencing. BMC. Bioinforma. 14:1471 - 1481. [ Links ]
Wright, S, 1978. Evolution and the genetics of populations, variability within and among natural populations. vol 4. University of Chicago Press. Chicago. [ Links ]
Yoon, J.; Dongcheng, L.; Wonseob, S.; Weisheng, L.; Aimin, Z.; y Shaohua, L. 2006. Genetic diversity and ecogeographical phylogenetic relationships among Peach and Nectarine cultivars based on Simple Sequence Repeat (SSR) Markers. J. Amer. Soc. Hort. Sci. 131:513 - 521. [ Links ]