Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Acta Agronómica
versão impressa ISSN 0120-2812
Acta Agron. vol.64 no.1 Palmira jan./jun. 2015
https://doi.org/10.15446/acag.v64n1.40724
Fitomejoramiento y Recursos Fitogéneticos doi:http://dx.doi.org/10.15446/acag.v64n1.40724 e-ISSN 2323-0118
Estudio molecular preliminar de accesiones de maíz (Zea maysL.) criollo e indígena Colombiano, utilizando una región de ADN cloroplástico
A molecular preliminary study of Colombian maize (Zea mays L.) accessions by using a cpDNA region
Ediel Armando Revelo Portilla, Carlos Iván Cardozo Conde y Creucí María Caetano
Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia, A.A. 237 Palmira, Valle del Cauca, Colombia. Autor para correspondencia: rediel78@yahoo.es
Rec.: 07.11.2013 Acep.: 02.04.2014
Resumen
Se exploró en forma preliminar la diversidad genética existente en las 23 razas de maíz criollo e indígena descritas para Colombia por Roberts y colaboradores (1957), para el efecto se evaluaron 28 cebadores nucleares y cloroplásticos. Catorce de ellos amplificaron en la reacción en cadena de la polimerasa (PCR) y fueron enviados para secuenciar a Macrogen Inc. (Corea). Mediante programas bioinformáticos (BioEdit 7.1.0, ClustalW versión 1.81, EditPlus Text Editor versión 3.20 y Gblock 0.91b 8) se encontró que ocho de estos cebadores presentaron un nivel alto de polimorfismo. La región genómica cloroplástica AtpB-1-RbcL-1 mostró el mayor polimorfismo y por tanto se utilizó para evaluar 23 materiales representativos de las 23 razas conservadas en el Banco de Germoplasma del CIMMYT (Centro Internacional de Mejoramiento de Maíz y Trigo) en México. Con el análisis de secuencias se revalidaron y confrontaron los grupos raciales obtenidos en el presente estudio -tres razas primitivas, siete razas probablemente introducidas, y 13 razas híbridas colombianas- con aquellos establecidos por Roberts y colaboradores, en los cuales se encontraron dos razas primitivas, nueve probablemente introducidas y 12 híbridas colombianas y con los grupos establecidos por Cardona (2010) utilizando la metodología Ward-MLM para los mismos caracteres descritos por Roberts et al. (1957), donde se encontraron cinco razas primitivas, siete probablemente introducidas y nueve híbridas colombianas. Con base en lo anterior, en el presente trabajo se hizo un aporte metodológico para revalidar datos históricos y redefinir grupos raciales.
Palabras clave:ADNcp, diversidad genética, Zea mays L., razas criollas, Colombia.
Abstract
In order to preliminarily explore the genetic diversity in the 23 Colombian races of maize described by Roberts and co-workers (1957), this study evaluate 28 nuclear and chloroplast primers. Of these, 14 amplified in the PCR, which were sent to sequencing to Macrogen Inc. (Korea). Bioinformatics programs revealed that six of these primers had a high level of polymorphism. It was considered that the genomic chloroplast region atpB-rbcL-1-1 showed the highest polymorphism, therefore was used to evaluate 23 materials, representative of the 23 races, stored in the CIMMYT germplasm bank, Mexico. By analyzing sequences we could defend and confront racial groups obtained in the present study ('primitive races' three, 'probably introduced' seven, 'Colombian hybrid', 13) with those established by Roberts and co-workers at 50, shown two 'primitive', nine 'probably introduced' and twelve 'Colombian hybrid' and the groups set by Cardona (2010), applying the strategy Ward-MLM for the same characters described by Roberts et al. (1957), which shows five 'primitive', seven 'probably introduced' and nine 'Colombian hybrid' races. Thus, it makes a methodological contribution to validate historical data and redefine racial groups.
Key words: cpDNA, genetic diversity, Zea mays L. maíz criollo, razas criollas, Colombia.
Introducción
La alta diversidad de los maíces en Suramérica guarda relación con su geografía e historia. El desarrollo de las culturas de los diferentes pueblos americanos, sus migraciones, el descubrimiento de América y el subsiguiente movimiento de europeos, fueron factores decisivos en la creación de la diversidad del germoplasma de maíz (Goodman y Bird, 1977).
Los continuos intercambios de genes entre las poblaciones, la posterior selección tanto natural como por el hombre, han mantenido activo el proceso de diversificación mediante el cual se han seleccionado y modificado características genotípicas que dieron formación a nuevas poblaciones adaptadas a diversos climas y tipos de suelos. Sánchez et al. (2000) proponen que la interacción ambiente x genotipo y el aislamiento geográfico también favorecieron la distribución geográfica de nuevas variantes de maíz, principalmente en las regiones altas (Roberts et al., 1957).
Anderson y Cutler (1942) propusieron una clasificación natural de la diversidad racial del maíz e introdujeron el concepto de raza como el conjunto de individuos con un número suficiente de caracteres en común que permite su reconocimiento como grupo.
En Colombia se han identificadas 23 razas de maíz, agrupadas en tres categorías en relación con su origen probable, reconocidas por Roberts et al. (1957). Se definen dos razas primitivas, nueve razas probablemente introducidas y 12 razas híbridas originadas en Colombia. Estos autores identificaron, además, cuatro factores de evolución que contribuyeron a la formación de dichas razas: aislamiento geográfico, hibridación interracial, hibridación con maíces contaminados con teocintle procedentes de México, e hibridación de maíz con su pariente silvestre Tripsacum
.Actualmente, factores como el cambio de uso del suelo en función del mercado, éxodos de población debidos a fenómenos sociales y naturales, la ausencia de programas públicos de conservación in situ, y algunos eventos relacionados con los procesos evolutivos propios de especies panmíticas como deriva genética, mutaciones o hibridación con materiales mejorados, posiblemente favorecieron una redistribución geográfica o la pérdida en su diversidad genética de las razas identificadas por Roberts et al. (1957).
Los análisis moleculares con secuencias de ADN en maíz criollo e indígena colombiano son escasas. Para el análisis de la diversidad genética, Peñaranda et al. (2005) realizaron una caracterización molecular con marcadores de tipo SSR de 22 accesiones criollas colombianas de maíz, para determinar los contenidos de aceite en grano.
El objetivo principal en este trabajo fue evaluar una metodología para estudiar la diversidad genética en maíz criollo colombiano, con una técnica molecular a partir de una región genómica de alto polimorfismo, y revalidar los grupos raciales propuestos por Roberts et al. (1957) y Cardona (2010).
Materiales y métodos
Se utilizaron accesiones representativas de las 23 razas caracterizadas por Roberts et al. (1957) en el territorio colombiano (Cuadro 1, Figura 1). Las semillas de estas accesiones fueron tomadas de las colecciones almacenadas en el Banco de Germoplasma del Centro Internacional de Mejoramiento de Maíz y Trigo (CIMMYT) en México.
La producción de plántulas se realizó en el Laboratorio de Citogenética y el estudio molecular en el Laboratorio de Biología Molecular de la Universidad Nacional de Colombia sede Palmira. Para el efecto se utilizaron 20 semillas por accesión, las cuales se colocaron a germinar en papel periódico y agua. Cinco días después de la germinación se extrajo el ADN de hojas jóvenes poco desarrolladas. La extracción se realizó mediante los métodos propuestos por Dellaporta et al. (1983) modificado por el Laboratorio de Biología Molecular de la Universidad Nacional sede Palmira, Doyle y Doyle (1987) modificado por CIMMYT (1996), Fulton et al. (1995), y el kit dNTPs Promega de extracción de ADN para plantas, no obstante el método de Doyle y Doyle (1987) modificado por CIMMYT (2006) fue el que permitió extraer ADN de buena calidad, con una concentración, promedio de 50ng/ul en todas las introducciones evaluadas.
Para evaluar la cantidad y la calidad del ADN se prepararon geles de agarosa al 0.8% corridos en tampón TBE 0.5X (Tris-borato 0045M; EDTA 0.001M) y teñidos con bromuro de etidio a una concentración final de 0.5 ng/ml. Las concentraciones se determinaron por comparación de concentraciones de ADN del bacteriófago Lambda a 30, 60 y 90 ng/ ul. Se efectuó una base de datos computarizada donde se registró la mayor cantidad de información disponible.
Para la estandarización de las condiciones de cada uno de los marcadores, se preparó una mezcla de reactivos en un tubo estéril de microcentrífuga (1.5 ml) para un volumen final de 25 µl. Se utilizaron 2 µl de ADN genómico a 50 ng/ul para la PCR con concentraciones finales de tampón taq 1X, dNTPs 0.2 mM, cebador F Y R 0,1uM, MgCl2 de 3 mM, y 2.5 unidades de Taq polimerasa (Cuadro 2). Para la amplificación de los cebadores utilizados en el estudio, se manejó la técnica de reacción en cadena de la polimerasa (PCR) (Cuadro 3).
Para la visualización de los productos PCR bajo luz ultravioleta se utilizó gel de agarosa a una concentración de 1.2% teñido con bromuro de etidio, en una cámara de electroforesis para gel horizontal CBS Scientific SGE-020-02 y un equipo digital de captura de imagen marca Uvitec. Se preparó el soporte del gel con cinta adhesiva, sellando los lados abiertos con refuerzos en los márgenes inferiores y las esquinas del soporte del gel. Se adicionó 1.2 g de agarosa por 150 ml de volumen de TBE 0.5X requerido. Se llevó al microondas hasta que la solución quedó disuelta completamente y se agregó (1µl) de bromuro de etidio por cada 100 ml de solución de agarosa preparada. A continuación se enfrió hasta que polimerizará en la cabina extractora de gases. Se adicionó la mezcla DNA + buffer de corrida dentro de los pozos del gel sumergidos en la cámara de electroforesis. Se corrió la electroforesis por 40 min a 80 voltios. Finalmente, se observaron las bandas en un foto-documentador con luz ultravioleta UV y se tomaron fotos a las bandas de ácidos nucleicos excitadas por el bromuro de etidio. La secuenciación fue realizada en Macrogen Inc. (Corea) conjuntamente con la purificación del ADN.
Para el análisis bioinformático, las secuencias fueron editadas y ensambladas con el programa BioEdit 7.1.0, un software para Windows 95/98. Cada región de ADN evaluada fue alineada usando el programa ClustalW versión 1.81, con los parámetros estándar de Larkin et al. (2007). Los alineamientos modificados manualmente fueron realizados con EditPlus Text Editor versión 3.20 para Windows 7/vista/2000/2003/XP. Para la identificación de bloques no informativos y regiones divergentes del alineamiento, se utilizó el programa Gblock 0.91b 8 Castresana (2000). Cada una de las secuencias fueron revisadas en la base de datos NCBI con el algoritmo BLASTn para estimar el 'score' (puntuación) y confirmar la especie en estudio.
Para la evaluación de los marcadores se usaron cinco criterios: (1) se identificaron los marcadores con un grado de facilidad en su amplificación y se seleccionaron 5 individuos por marcador, para evaluar la calidad de las secuencias; (2) se calculó para cada locus el número de haplotipos, número de sitios polimórficos y la diversidad haplotípica de Nei (Nei, 1973), con el soporte del programa DNAsp (Rozas y Rozas, 1999); (3) se evaluaron los modelos de sustitución nucleotídica a partir de los individuos que representan las razas criollas de maíz colombianas, con el objetivo de describir la consanguinidad existente; (4) con el mejor marcador en la capacidad de discriminar a nivel de grupo racial se construyó un árbol de distancia genética, con el apoyo del programa MEGA versión 5 (Tamura et al., 2011); y (5) el marcador seleccionado como altamente informativo, se evaluó con todas las secuencias para observar su eficiencia en la discriminación de especies y grupo racial. El valor de Bootstrap de apoyo (BS) para cada clado individual se calculó mediante la ejecución de 500 replicaciones en el arranque de los datos. Todas las posiciones que contenían gaps o datos faltantes fueron eliminadas de la base de datos.
Resultados y discusión
Entre los métodos ensayados para la extracción de ADN en maíz, el de Doyle y Doyle (1987) modificado por CIMMYT (2006) permitió extraer ADN de buena calidad, con una concentración promedio de 50 ng/ul para todos los materiales evaluados. Esto coincide con lo reportado por Wang et al. (1993), Weir et al. (1996), Henry (1997), Jewit et al. (1998) entre otros, que han utilizado CTAB para aislamiento de ADN de hojas maduras para estudios de caracterización de especies por RAPD.
La tasa de éxito en la amplificación por PCR de los cebadores mostró que seis de ellos presentaron problemas en su amplificación. Estos incluyen: ITS4-ITS5A, Adhc-P1-Adhc-P2, GPDX7F-GPDX9R, TabC-TabF, rpL32-R-ndhF y nad1-BF2-nad1-BF3. Amplificaron los cebadores rpS16x2F2-trnK(UUU)x, psbA3'f-trnHf, trnL(UAG)-rpL32-F, rbcLa-rbcLb, trnQ(UUG)-rpS16x1, trnG2G-F-trnG-R, TabE-TabF y atpB-1-rbcL-1 (Figura 2).
Prueba preliminar de variación de secuencias
La estadística descriptiva para los ocho cebadores evaluados se presenta en el Cuadro 4. El marcador AtpB-1-RbcL-1 presentó el mayor número de sitios polimórficos (182), siendo el más informativo entre los evaluados. Posteriormente, con este par de cebadores se hicieron las evaluaciones en las 23 accesiones utilizadas en este estudio.
Modelo de sustitución y frecuencia nucleotídica
Para la evaluación se eliminaron todas las posiciones que contienen 'gaps' (vacíos) y datos faltantes. En total se evaluaron 24 modelos de sustitución nucleotídica a partir de los representantes de las razas colombianas de maíz. De acuerdo con los criterios de sustitución nucleotídica, el modelo con el puntaje más bajo de BIC (Criterio de Información Bayesiana) se considera el más adecuado para describir la consanguinidad existente entre los individuos y crear el árbol filogenético para la investigación. En este estudio el modelo T92 (Tamura 3-parameter) obtuvo un BIC de 4338.8, siendo el valor más bajo entre los 24 modelos evaluados.
La frecuencia nucleotídica estima el contenido de adenina (A), citosina (C) guanina (G) y tiamina (T). Para la región AtpB-1-RbcL-1 se observó un contenido alto de (A) y (T) con una frecuencia del 67.56%, respectivamente. Sin embargo, para C y G se consideró una frecuencia del 32.44% siendo menor que la relación (A-T). Para la evaluación se estimó un total de 897 posiciones en el conjunto de datos finales (Cuadro 5).
Análisis descriptivo
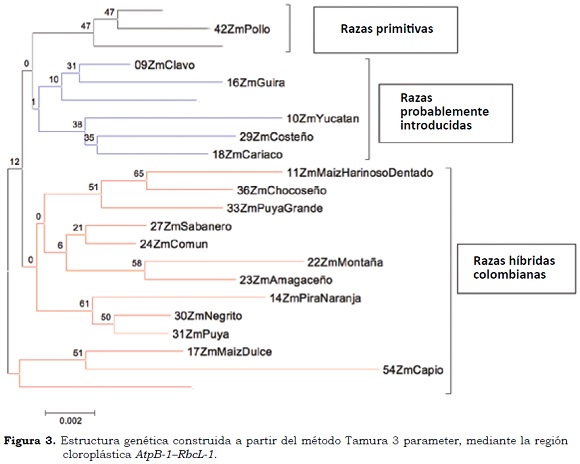
De acuerdo con la evaluación de los marcadores, AtpB-1-RbcL-1 ofreció mayor favorabilidad al momento de realizar un estudio filogenético a nivel de especie para Z. mays, esto con base en la facilidad de amplificación y mayor número de sitios informativos respecto a los demás marcadores evaluados. Se construyó un árbol filogenético para observar el nivel de consanguinidad. Según el dendrograma, basado en el método de Tamura 3-parameter, se observó un patrón claro de separación a nivel de grupo racial, considerando los tres grupos raciales propuestos por Roberts et al. (1957): 'Primitivas', 'Probablemente Introducidas' e 'Híbridas Colombianas' (Figura 3); no obstante, se encontró diferencia en la ubicación de algunas introducciones de maíces a lo propuesto inicialmente por Roberts et al. (1957).
El primer agrupamiento identificó un nivel de consanguinidad entre Imbricado, Pollo y Pira, clasificándolas como primitivas. Un segundo agrupamiento asoció a Clavo, Güirúa, Cabuya, Yucatán, Costeño y Cariaco como razas probablemente introducidas. Por último, se conformó un grupo entre maíz Harinoso Dentado, Puya Grande, Chococeño, Común, Sabanero, Montaña, Amagaceño, Pira Naranja, Negrito, Puya, Maíz Dulce, Capio y Cacao como razas híbridas colombianas. El valor de Bootstrap para la conformación del árbol filogenético fue de 500 repeticiones (Figura 3).
Cardona (2010) utilizando la estrategia Ward-MLM (Franco y Crossa 2002), clasificó los grupos raciales de acuerdo con métodos estadísticos cuantitativos tomando como referencia el trabajo de Roberts et al. (1957). El presente estudio contrastó las tres clasificaciones, en donde se observan similitudes y diferencias. Roberts et al. (1957) clasificaron las razas Pollo y Pira como primitivas, mientras que Cardona (2010) amplió este grupo con tres razas más: Pira Naranja, Clavo e Imbricado; sin embargo, en el presente estudio se identificaron Pollo, Pira e Imbricado como razas primitivas (Cuadro 5).
En el segundo grupo racial, de acuerdo con Roberts et al. (1957), siete razas son probablemente introducidas: Güirúa, Andaquí, Pira Naranja, Cariaco, Clavo, Imbricado y Sabanero. Cardona (2010), por otra parte, conforma este grupo con igual número de razas (7), pero sólo dos (Güirúa y Andaquí) están en el mismo grupo propuesto por Roberts et al. (1957). Las otras cinco están conformadas por Amagaceño, Común, Yucatán, Cacao y Puya Grande. En este trabajo, comparado con el trabajo de Roberts et al. (1957), se tienen en común las razas Güirúa, Clavo y Cariaco. El grupo lo completan las razas Yucatán, Costeño y Cabuya (Cuadro 5).
El tercer y último grupo racial, de acuerdo con Roberts et al. (1957), está conformado por catorce razas (Montaña, Cabuya, Capio, Costeño, Negrito, Puya, Chococeño, Amagaceño, Común, Yucatán, Puya grande, Cacao, Harinoso Dentado, Maíz Dulce) y de acuerdo con este trabajo se tiene una similaridad de once razas con respecto a la referencia. A su vez, el estudio de Cardona (2010) sólo presentó similaridad con cinco razas (Montaña, Capio, Negrito, Puya y Chococeño) (Cuadro 6).
Conclusiones
- El análisis entre grupos raciales mostró similitudes y diferencias de acuerdo con los estudios realizados por Roberts et al. (1957) mediante caracterización morfológica y Cardona (2010) utilizando la estrategia Ward-MLM (Franco y Crossa 2002). Sin embargo, se conservan los tres grupos raciales colombianos identificados inicialmente por Roberts et al. (1957), aunque con variaciones entre las razas que conforman los grupos.
- Según la evaluación realizada en el estudio a nivel de marcadores moleculares, AtpB-1-RbcL-1 de origen cloroplástico, presentó eficiencia en la identificación a nivel de grupos raciales, demostrando ser un potente marcador para futuros trabajos en diversidad genética para maíz.
Agradecimientos
A los doctores Suketoshi Taba (CIMMYT, México) y Luis Alberto Narro (CIMMYT Suramérica), Jaime Eduardo Muñoz (UN-Palmira).
Referencias
Anderson E. y Cutler H. C. 1942. Races of Zea mays L.: I. Their recognition and classification. Ann. Missouri Bot. Gard. 29:69 - 89. [ Links ]
Cardona, J. O. 2010. Análisis de diversidad genética de las razas colombianas de maíz a partir de datos Roberts et al. (1957) usando la estrategia Ward-MLM. CienciAgro 2(1). [ Links ]
Castresana J. 2000. Selection of conserved blocks from multiple alignments for their use in phylogenetic analysis. Mol. Biol. Evol. CIMMYT (Centro Internacional de Mejoramiento de Maíz y Trigo). 2006. Protocolos de laboratorio: Laboratorio de Genética Molecular Aplicada del CIMMYT. Tercera edición. México, D.F.: CIMMYT. [ Links ]
Dellaporta S. L.; Wood J. J. y Hicks, J. B. 1983. A plant DNA minipreparation: Versión II. Plant Mol. Biol. Rep. 1(14):19 - 21. [ Links ]
Doyle, J. J. y Doyle J. L. 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem. Bull. 19:11 - 15. [ Links ]
Franco, J. y Crossa, J. 2002. The modified location model for classifying genetics resources: I. Association between categorical and continuous variables. Crop Sci. 42:1719 - 1726. [ Links ]
Fulton, T. M.; Chunwongse, J.; y Tanksley, S .D. 1995. Micropep protocol for extraction of DNA from tomato and other herbaceous. Plant Mol. Biol. Rep. 13 (3):207 - 209. [ Links ]
Goodman M. M. y Bird R. M. 1977. Races of maize IV: tentative groupings of 219 latin american races. Econ. Bot. 31:204 - 221. [ Links ]
Henry, R. J. 1997. Useful routine protocols in plant molecular biology. En: Practical applications of plant molecular biology. Chapman and Hall, Londres. [ Links ]
Jewit, S.; Puddephat, I.; Coveney, J.; Davey, M. R.; Alderson, P. G.; Lowe K. C.; y Power, J. B. 1998. Extraction of Quercus DNA from leaves is suitable for RAPD-PCR studies. The biotechnology towards the millennium. CAB Abstracts (1998). p. 351 - 357. [ Links ]
Larkin, M. A. et al. 2007. Clustal W and Clustal X version 2.0 In: Bioinformatics. Bd. 23, S. 2947 - 2948 [ Links ]
Nei, M. 1973. Analysis of gene diversity in subdivided populations. Proceedings of the Natural Academy of Sciences. EE.UU. 70:3321 - 3323. [ Links ]
Peñaranda, M. y Navas, A. 2005. Caracterización molecular y evaluación bioquímica de cultivares colombianos y germoplasma elite de maíz según contenido de aceite Programa Nacional de Recursos Genéticos y Biotecnología Vegetal. Corporación Colombiana de Investigación Agropecuaria (Corpoica). C. I. Tibaitatá, Bogotá [ Links ].
Rozas, J. y Rozas, R. 1999. DnaSP, Version 3: An integrated program for molecular population genetics and molecular evolution analysis. Bioinformatics 15:174 - 175. [ Links ]
Roberts, L. M.; Grant, U. J.; Ramírez, R. E.; Hatheway, W. H.; Smith, D. L.; y Mangelsdorf, P. C. 1957. Razas de maíz en Colombia. Departamento de Investigación Agrícola (DIA). Colombia. Ministerio de Agricultura. Editorial Máxima. Bogotá. Bol. Tec. No. 2. [ Links ]
Sánchez, G.; Stuber, C. W.; y Goodman, M. M. 2000. Isozymatic diversity of the races of maize of Americas. Maydica 45:185 - 203 [ Links ]
Tamura, K.; Peterson, D.; Peterson, N.; Stecher, G.; Nei, M.; y Kumar, S. 2011. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol. Biol. Evol. 28:2731 - 2739. [ Links ]
Wang, H.; Meiging, Q. A.; y Cutler, J. 1993. A simple method of preparing plant simples for PCR. Nucleic Acids. Res. 21:4153 - 4154. [ Links ]
Weir, B. R.; Pierre, P.; y Chibbar, N. 1996. Isolation of DNA for RAPD. Analysis from leaves of the saskatoon (Amelanchier alnifolia Nutt) and other horticultural crops. Can. J. Plant Sci. 76:819 - 824. [ Links ]