Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.64 no.4 Palmira Oct./Dec. 2015
https://doi.org/10.15446/acag.v64n4.47772
http://dx.doi.org/10.15446/acag.v64n4.47772
Evaluación de las herramientas de secuenciación masiva (NGS) para identificar genes asociados con tolerancia al estrés hídrico en caña de azúcar
Assessment of the new Next Generation Sequencing (NGS) tools to identify genes associated with tolerance to water deficit in sugarcane
John Jaime Riascos-Arcos*, Héctor Fabio Espitia Navarro y Jershon López Gerena
Centro de Investigación de la Caña de Azúcar de Colombia, Cenicaña. Florida, Valle del Cauca. Colombia. *Autor para correspondencia: jjriascos@cenicana.org
Rec.:10.12.2014 Acep.: 09.06.2015
Resumen
En la investigación se evaluó la utilidad de las nuevas tecnologías de secuenciación masiva NGS en la identificación de genes expresados en variedades de caña, bajo condiciones de estrés por déficit hídrico o por anegamiento. No obstante que en la actualidad la secuenciación masiva NGS es la metodología preferida en estudios de transcriptómica comparativa, en el caso de la caña de azúcar (Saccharum spp.) es necesario verificar su utilidad, si se tiene en cuenta la complejidad de su genoma y que herramientas útiles, como un genoma de referencia, no se encuentran disponibles. Para el estudio se seleccionaron dos variedades de caña para cada tipo de estrés (una tolerante y una susceptible) tanto en el caso del déficit hídrico como en el caso del anegamiento. Cada una de estas variedades se mantuvo bajo condiciones de estrés (niveles medio o severo) por déficit de agua o por anegamiento para inducir la expresión de los ARNm de interés. Para cada una se crearon tres bibliotecas de ADNc a partir de tejido foliar (para un total de 12 bibliotecas), las cuales se secuenciaron usando la metodología Illumina-RNA-Seq. Los resultados de expresión diferencial obtenidos a partir de estos análisis mostraron ortólogos de genes previamente identificados como contribuyentes a la tolerancia causada por el déficit hídrico o el anegamiento. También fue posible diferenciar entre los niveles de expresión de transcritos altamente similares. Los resultados aquí presentados permiten concluir la utilidad de las metodologías NGS en estudios de transcriptómica comparativa de caña de azúcar.
Palabras clave: Caña de azúcar, déficit hídrico, anegamiento, ADNc, secuenciación masiva NGS, transcriptoma.
Abstract
The aim of this research was to evaluate the performance of the new Next Generation Sequencing (NGS) technologies in comparative transcriptomics experiments, aiming to identify genes associated with tolerance mechanisms to abiotic stress such water deficit or flooding in sugarcane. Despite being widely used in most of current comparative transcriptomics studies, it is important to test the utility of NGS technologies in species such as sugarcane considering its genome complexity and the fact that there is no reference genome, which could be of use in this type of studies. For this purpose, in this investigation, varieties tolerant and susceptible to drought or flooding were selected and independently subjected to stress (medium or severe levels) due to drought or flooding, in order to induce the production of mRNAs of interest. For each of these, leaves were collected and cDNA libraries produced (a total of 12). Each library was sequenced using NGS methodologies (Illumina-RNA-Seq) and data were analyzed using specialized bioinformatics software. Among the genes that were observed as differentially expressed it was possible to identify orthologs of those previously associated with tolerance for the traits of interest. Also, it was possible to detect differences in expression levels of highly similar transcripts. Our results provide evidence that support the use of NGS technologies in transcriptomics studies in genetically complex species such as sugarcane.
Key words: Sugarcane, drought, flooding, cDNA, next generation sequencing, transcriptome.
Introducción
En el valle del río Cauca existen 230,303 ha cultivadas con caña de azúcar (Saccharum spp.) de las cuales 121,830 ha (52.9%) corresponden a zonas secas o con déficit hídrico (DH); mientras que 26,370 ha (11.45%) corresponden a zonas con niveles freáticos más altos (zonas anegadas). El área restante corresponde a zonas semisecas (68,630 ha) que representan 29.8% y zonas de piedemonte (13,818 ha) que representan 6%. Se estima que las pérdidas en la producción de caña por causa del déficit hídrico pueden llegar hasta 42%, mientras que aquellas generadas por suelos excesivamente húmedos alcanzan 54% (Cruz et al., 2000, 2009).
Hasta el momento, el estrés por DH o por anegamiento se ha manejado con éxito mediante el uso de sistemas apropiados de riegos y drenajes, los cuales se han implementado en más del 80% del área cultivada en la región y han permitido reducir la cantidad de agua utilizada en el caso de las zonas que necesitan riego y mantener altos niveles de producción en toda el área cultivada. No obstante, los costos de estos sistemas son altos y representan, aproximadamente, 45% de los costos totales de producción (Campos et al., 2009).
En la actualidad es necesario disponer de genotipos de caña de azúcar con capacidad de mantener buenos niveles de producción bajo condiciones de estrés abiótico, principalmente déficit hídrico o anegamiento. Para enfrentar estas condiciones, el área de Mejoramiento del Centro de Investigación de la Caña de Azúcar de Colombia (Cenicaña) trabaja en la evaluación de su banco de germoplasma. En forma paralela se busca implementar herramientas biotecnológicas a fin de acelerar el proceso de mejoramiento genético de las variedades de caña.
En los últimos años los avances en las metodologías de secuenciación masiva NGS (del inglés Next Generation Sequencing), específicamente la secuenciación RNA-Seq, han permitido realizar numerosos estudios de transcriptómica comparativa en busca de identificar genes responsables o que contribuyan al desarrollo de una característica específica. Este tipo de estudios parte de secuencias cortas (‘short reads’) de ADNc (ácido Desoxiribonucleico complementario), generadas a partir de ARN (ácido Ribonucleico) celular del organismo de interés y, usualmente, compara la expresión génica de un mismo individuo o de un grupo de individuos bajo diferentes tratamientos (Trapnell et al., 2012). Mediante metodologías informáticas, las cuales requieren un poder computacional significativo, es posible cuantificar el número de secuencias cortas que corresponden con los genes o transcritos de una condición o individuo en particular y a través de comparaciones, identificar los genes con expresión diferencial significativa. En este tipo de análisis con frecuencia se usa un genoma o transcriptoma conocido como punto de referencia, sin embargo también es posible realizarlo sin tener información conocida de la especie que se estudia (‘non-model species’) gracias a las técnicas de ensamblaje de novo aplicadas a datos NGS.
No obstante que el concepto de transcriptómica comparativa apoyado en la secuenciación RNA-Seq ha sido utilizado en especies no modelo, como por ejemplo el fríjol común (Phaseolus vulgaris L.) (Wu et al., 2014), la variedad de uva cabernet sauvignon (Vitis vinifera) (Li et al., 2014) y la especie de trigo diploide escaña cultivada (Triticum monococcum) (Fox et al., 2014), el caso de la caña de azúcar es particular ya que ésta tiene un genoma poliploide (octa-decaploide), aneuploide (2n = 80-130), de gran tamaño, >10 GB (Revisado en D'Hont, 2005), lo cual podría dificultar el análisis de la información genómica producida. Debido a esto, el objetivo en esta investigación fue probar la utilidad de las nuevas herramientas de secuenciación para identificar genes asociados con tolerancia al estrés por DH y/o el anegamiento en variedades de caña de azúcar, con el fin de confirmar su utilidad en estrategias de mejoramiento varietal apoyado en biotecnología. Considerando que en otras especies de cultivos se han identificado genes que contribuyen a la tolerancia del estrés causado por el DH y el anegamiento, se decidió utilizar esta información para evaluar la utilidad de resultados obtenidos en los análisis del presente estudio.
Materiales y métodos
Material vegetal e inducción de estrés
Se utilizaron las variedades SP 71-6949 y MZC 74-275, previamente caracterizadas (Viveros, 2011) como tolerantes y susceptibles, respectivamente, al estrés por déficit hídrico (DH). Para el caso del estrés debido a anegamiento se utilizaron las variedades CC 01-1940 y CC 93-4418 catalogadas por el Programa de Variedades de Cenicaña como tolerante y susceptible para esta característica, respectivamente.
Inducción de estrés por déficit hídrico
La inducción de estrés por DH se hizo en invernadero, en plantas de 5 meses de edad, las cuales se sembraron en tanques de 0.8 m. de profundidad y 2 m. de diámetro. Las variedades se sembraron usando un modelo completamente al azar con tres repeticiones por cada tratamiento. Las muestras de tejido foliar, aproximadamente 5 g, fueron recolectadas en plantas cultivadas en condiciones normales de irrigación (control) o en condiciones de estrés, específicamente cuando el nivel de humedad en el suelo había agotado aproximadamente 75% del agua aprovechable (estrés medio) y cuando había alcanzado un nivel de humedad cercano al punto de marchitez permanente (estrés severo). Para el monitoreo de los niveles de humedad en el suelo se usó la sonda Diviner-2000 (Sentek, Australia). Antes de recolectar el tejido foliar para confirmar la inducción de estrés en cada una de las plantas, se hicieron mediciones de temperatura de la hojas, conductancia estomática, fluorescencia del fotosistema II y contenido de clorofila, usando los instrumentos Termómetro High Temperature IR Thermometer (Extech Instruments, USA), porometro SC-1 (Decagon Devices, USA), FluorPenFP 100 modelo Z990 (Qubit Systems, Canadá) y Clorofilómetro SPAD 502 Konica Minolta (Sensing Americas, USA), repectivamente.
Inducción de estrés por anegamiento
Para esta inducción se utilizaron plantas de 4 meses de edad sembradas en lisímetros (1.2 m de largo x 1.5 m. de ancho x 2 m. de profundidad) que fueron anegadas por un período de 2 y 14 días o mantenidas bajo condiciones normales de humedad. El experimento se sembró usando un modelo de bloques completos al azar con tres repeticiones por cada tratamiento. Las muestras de tejido foliar fueron recolectadas a partir de plantas cultivadas en condiciones normales de irrigación (control) o en condiciones de estrés, específicamente después de 2 días (estrés medio) y de 14 días bajo anegamiento (estrés severo). Para monitorear los cambios fenotípicos indicativos de estrés, se evaluó la presencia de raíces superficiales al momento de la cosecha.
Extracción de ARN
El ARN total de cada una de las variedades fue extraído a partir de 100 mg de tejido foliar, usando el reactivo Trizol (Invitrogen, USA) y siguiendo las indicaciones del fabricante. El ARN, una vez extraído, fue disuelto en agua ultrapura, libre de ARNasas, para determinar su concentración en un Nanodrop 2000 Spectophotometer (Thermo Scientific, USA). Adicionalmente, 5 µg de ARN fueron tratados con ADNasa I (Ambion, cat #: AM 2222) disueltos en agua ultrapura, los cuales fueron utilizados para la construcción de las bibliotecas de ADNc.
Construcción de bibliotecas de ADNc y secuenciación masiva Illumina
Muestras de ARN total, tratado con DNAsa I, fueron enviadas a la Universidad de Illinois en Urbana-Champaign (http://www.igb.illinois.edu/biotech/htdna) para el proceso de construcción y secuenciación de las librerías de ADNc. Un total de 12 librerías, correspondientes a dos variedades bajo tres tratamientos (control, estrés medio y estrés severo) para el caso de DH y dos variedades bajo tres tratamientos (control, estrés medio y estrés severo) para el caso de EH, fueron construidas. Cada librería fue secuenciada por cada extremo (paired-end sequenced), usando el kit de secuenciación TruSeq SBS (versión 3) y el paquete Casava1.8 (pipeline 1.9). El proceso de secuenciación se llevó a cabo en una plataforma de secuenciación HiSeq1000.
Análisis bioinformático y estadístico
Una vez obtenidas las secuencias, varios paquetes informáticos fueron usados para determinar los genes que presentaron expresión diferencial entre los tratamientos de cada experimento (EH o DH) Entre ellos, el paquete CLC Genomics workbench, version 4.6.1 (CLC bio, Aarhus, Denmark) y la base de datos de EST de caña de azúcar, http://sucest-fun.org, (Vettore et al., 2003) se usaron para el proceso de alineamiento y estimación de valores RPKM (del inglés Reads Per Kilobase of transcript per Million reads mapped). Los valores de RPKM estimados para cada una de las secuencias fueron utilizados en el programa DEGseq (Wang et al., 2010) para identificar los genes que presentaron una expresión diferencial (P < 0.001) entre los tratamientos (Control vs. E. Medio y Control vs E. Severo) dentro de cada experimento (DH y anegamiento). Finalmente, la anotación de las secuencias de interés se realizó con el programa Blast2GO (Conesa et al., 2005). Los análisis estadísticos se hicieron con el paquete SAS, versión 9.3.
Resultados y discusión
Factores abióticos como el déficit de agua o el anegamiento promueven cambios bioquímicos y fisiológicos en las plantas, por ejemplo, cambios en la apertura o cierre de las estomas y captación de CO2, cambios en la tasa de división celular y fotosíntesis, entre otros. Estos cambios ocurren como una adaptación a las nuevas condiciones ambientales y son el producto de la activación o inactivación de un determinado número de genes. Debido a su función durante periodos de estrés, estos genes podrían ser considerados como los responsables de la tolerancia a estos tipos de estrés (Shinozaki y Yamaguchi-Shinozaki, 2007). La identificación de genes que contribuyen a la tolerancia por estrés hídrico representa un claro beneficio en la agricultura, ya que brindaría la posibilidad de generar nuevas variedades con un consumo menor de agua en áreas donde este recurso es escaso, reducir las pérdidas en producción debidas al estrés abiótico y disminuir los costos de producción del cultivo.
En el presente trabajo se evaluó la utilidad de las metodologías de secuenciación NGS en la identificación de genes asociados con la tolerancia al estrés hídrico, como el DH y el anegamiento, en la caña de azúcar. Esto, considerando que el gran tamaño del genoma de la caña (aproximadamente 10 GB) y su nivel de ploidía podría dificultar, entre otras características, la identificación confiable de genes expresados diferencialmente (GED). Es importante mencionar que en el proceso de análisis de datos NGS-RNA-Seq, el primer paso requiere un alineamiento de las secuencias producidas a un transcriptoma de referencia o, en caso de que éste no exista, se debe producir uno a partir del ensamblaje de novo de las secuencias cortas. Es a partir de esta primera reconstrucción de los genes contenidos en las secuencias NGS, que es posible cuantificar el número de fragmentos que hacen parte de un gen y de esta manera determinar si los genes reconstruidos han variado su expresión entre los tratamientos en estudio (Trapnell et al., 2012). En este estudio también se evaluó la hipótesis que es posible diferenciar entre genes muy similares, expresados bajo la misma condición de interés, en un genoma o transcriptoma de gran tamaño como el de la caña de azúcar.
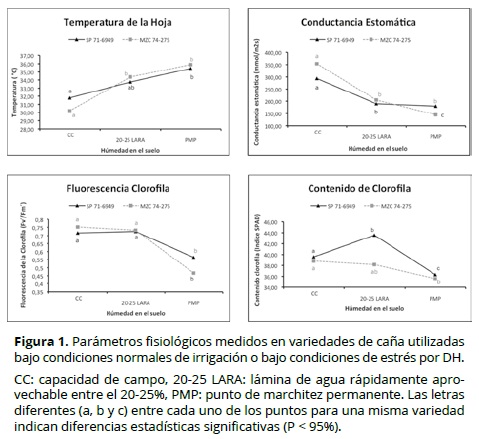
Con el objeto de promover los cambios en la expresión de los genes de interés, variedades de caña de azúcar previamente categorizadas como tolerantes o susceptibles al estrés por DH o al anegamiento fueron mantenidas, de manera independiente, bajo condiciones control o de estrés. Lo anterior, con el fin de comparar la expresión génica de ambos tratamientos y de identificar aquellos genes que se expresaban diferencialmente. La inducción de la condición de estrés en cada uno de los experimentos se confirmó por monitoreo de variables fisiológicas como la temperatura de la hoja, la conductancia estomática, la fluorescencia del fotosistema II y el contenido de clorofila de las hojas, en el caso del DH; y el desarrollo de raíces superficiales en el caso del anegamiento. En la Figura 1 se observa que en el caso del DH, la temperatura de la hoja aumentó en la medida que incrementaba el estrés, mientras que en el caso de la conductancia estomática, la fluorescencia del fotosistema II y el contenido de clorofila de las hojas, estas tendieron a disminuir. Estos resultados eran esperados y han sido documentados como cambios típicos de plantas bajo estrés por DH y son una consecuencia del cierre estomático y la reducción en el intercambio gaseoso (Barbosa et al., 2013; de Almeida Silva et al., 2011; Liu et al., 2011). En el caso de anegamiento, la inducción del estrés fue observada en el desarrollo de las raíces, teniendo en cuenta que el estrés por anegamiento no necesariamente genera alteraciones en la temperatura de las hojas, intercambio gaseoso o fotosíntesis (Glaz et al., 2004). En contraste, al disminuir la concentración de oxígeno en el suelo, como consecuencia del exceso de agua, se observan cambios como el desarrollo de raíces superficiales con mayor aerénquima, los cuales facilitan la captura de oxígeno (Eavis, 1972) (Figura 2).
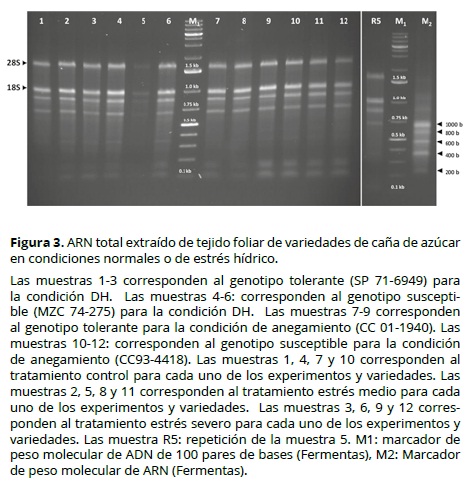
El ARN foliar se utilizó como punto de partida para evaluar la utilidad de las herramientas de secuenciación masiva para estudios de expresión génica en caña de azúcar. Esto debido a la simplicidad de extraer ARN foliar y a la disponibilidad de datos provenientes de otros estudios (Tougou 2012; Vettore et al., 2003) que evaluaron la expresión génica en hojas y que permiten establecer comparaciones con los resultados del presente trabajo. No obstante, es necesario destacar que la expresión génica es influenciada por el ambiente y se considera específica de un tejido es decir, los genes que se expresan en un momento dado en las hojas pueden llegar a ser diferentes de aquellos que se expresan en ese momento en la raíz lo cual sugiere que tanto en el caso del DH como en el anegamiento, la evaluación de la expresión génica en las raíces podría aportar información adicional a la aquí presentada. La calidad del ARN extraído de las hojas y utilizado en la construcción y secuenciación de las bibliotecas RNA-Seq fue evaluada mediante los rangos de absorbancia 260/280 y la evaluación visual de las bandas ribosomales 28S y 18S obtenidos para todas las muestras. En la Figura 3 se observa que cada una de las muestras presentaba bandas ribosomales sólidas. En conjunto con los rangos de observancia obtenidos, los cuales se mantuvieron entre 1.8 - 2.0 (datos no mostrados), se puede concluir que el ARN usado para la construcción de las bibliotecas fue de alta calidad.
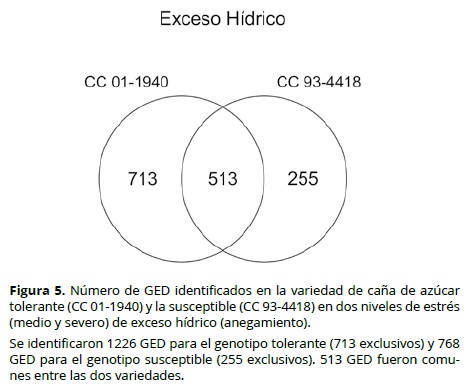
En este estudio se secuenció un mínimo de 9,461,897 secuencias por cada una de las 12 bibliotecas construidas (Tabla 1). Adicionalmente, se usó el transcriptoma público de caña de azúcar, denominado SUCEST, como punto de referencia. Aunque SUCEST no representa de forma fiel el transcriptoma de la caña de azúcar, ya que en la actualidad no existe un genoma secuenciado, sí es una excelente herramienta que reúne 43,143 transcritos (que en este caso se podrían interpretar como genes) y sus posibles variantes, que no están muy distantes de lo que se esperaría observar en un genoma como el de la caña de azúcar. Del total de posibles transcritos almacenados en SUCEST se lograron reconstruir para cada biblioteca entre 30,219 - 33,419, los cuales fueron utilizados para identificar aquellos que se expresaron de manera diferencial. En otros términos, se identificaron tanto aquellos genes que ser sobre-expresaron o se reprimieron en una variedad cuando esta se encontraba bajo condiciones de estrés. Los resultados de la cuantificación de la expresión mostraron un total de 3193 GED en el caso del estrés por DH y 1481 en el caso del estrés por anegamiento. En las Figuras 4 y 5 aparece un resumen de esta información y se establecen diferencias entre el número de GED presentes, tanto en variedades tolerantes y susceptibles o de manera exclusiva en alguno de estos dos grupos.
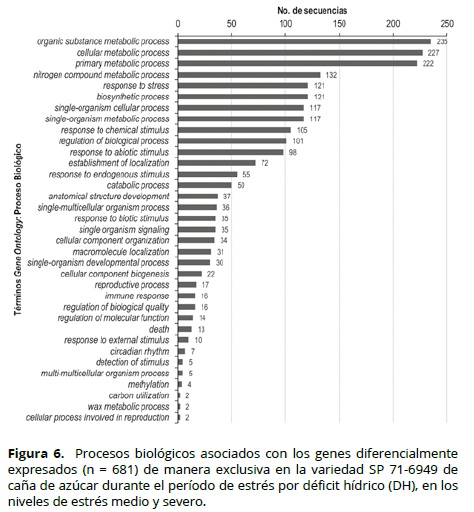
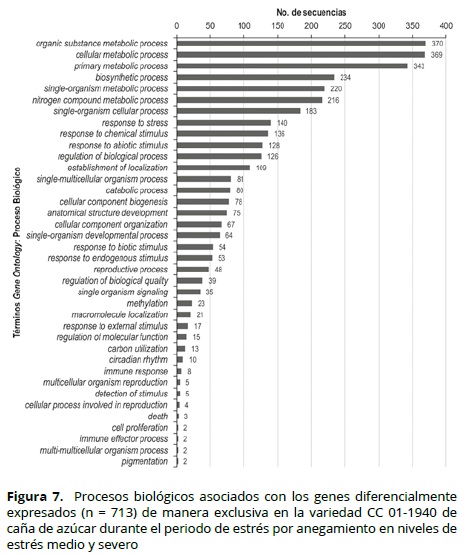
La función más posible en los diferentes procesos biológicos celulares de los genes fue evaluada a través de comparaciones con los genes almacenados en la base de datos de proteínas no redundantes (nr: Non-redundant GenBank CDS translations + PDB + SwissProt + PIR + PRF) del NCBI (www.ncbi.nlm.nih.gov/), usando el programa Blat2GO. En las Figuras 6 y 7 se observa esta clase de información para los GED en los genotipos tolerantes (SP 71-6949 y CC 01-1940) usados en este estudio. Dentro de las rutas metabólicas activadas durante el proceso de estrés se encuentran genes asociados con respuesta a estrés y estímulos abióticos, entre otros, los cuales confirman lo observado en otros estudios similares (Aparecida-Rodrigues et al, 2009; Vettore et al, 2003).
Finalmente, para evaluar la posibilidad de identificar GED con alta similitud se monitoreó la expresión de los genes DREB (Dehydration Responsive Element Binding) y ERF (Ethylene Responsive Factor) en el caso de estrés por DH, y la familia ADH (Alcohol Dehydrogenase) en el caso del estrés por anegamiento. Dentro de los GED identificados se encontraron siete transcritos categorizados como DREB, 29 como ERF y 3 como ADH. Cada una de estas familias de genes han sido extensivamente caracterizadas como contribuyentes a la tolerancia a DH y anegamiento en varias especies de monocotiledóneas y dicotiledóneas (Tougou et al., 2012; Quan et al., 2010; Shinozaki y Yamaguchi-Shinozaki 2007; Li et al, 2005). Adicionalmente, en el caso de la familia DREB, la similitud entre los siete GED varió entre 43.9% y 90.5%. En el caso de los ERF el rango de similitud entre los 29 GED fue de 31.2% a 96.4%. Para anegamiento, los tres GED identificados mostraron una similitud entre ellos de 39% a 57%. En el Tabla 2 se presenta un listado parcial de los GED de DH pertenecientes a las familias DREB y ERF, mientras que en el Tabla 3 se relaciona el listado de los tres GED pertenecientes a la familia ADH en los experimentos de anegamiento. Estos resultados son importantes porque confirman la posibilidad de diferenciar entre GED altamente similares los cuales han sido previamente asociados con las características de interés en otros cultivos.
Conclusiones
- La consecución de variedades de caña de azúcar con capacidad para un uso más eficiente del recurso hídrico, tendría una importante repercusión en la industria azucarera colombiana, ya que permitiría reducir los costos y las pérdidas en producción en zonas donde ocurre estrés por déficit o sobre-oferta del recurso hídrico. En este sentido, Cenicaña trabaja en la identificación de genes de interés de tal manera que se puedan implementar estrategias de mejoramiento genético basado en transformación genética o selección varietal usando marcadores moleculares.
- Los resultados preliminares presentados en este documento son el primer paso en la identificación de estos genes y confirman la utilidad de las metodologías NGS, en una especie genéticamente compleja como la caña de azúcar.
- Aunque los resultados observados hasta el momento son promisorios, es de resaltar que estos requieren de repeticiones biológicas que confirmen la expresión de los genes aquí identificados como expresados diferencialmente y que podrían servir como candidatos en estrategias de mejoramiento molecular.
Referencias
Aparecida-Rodrigues, F.; de Laia, M.L.; y Zingaretti, S.M. 2009. Analysis of gene expression profiles under water stress in tolerant and sensitive sugarcane plants. Plant Sci. 176:286 - 302. [ Links ]
Barbosa, D.; Ciríaco E.; Custódio, R.; Menossi, M.; y Silveira, M. 2013. Physiological limitations in two sugarcane varieties under water suppression and after recovering. Theoretical and Experimental Plant Phys. 25(3):213 - 222. [ Links ]
Campos, R. A.; Cruz, D. M.; y Torres, J. S. 2009. Riego con caudal reducido. Cenicaña. Cali, Colombia. Documento de Trabajo No. 694. 14 p. [ Links ]
Conesa, A.; Götz, S.; García-Gomez, J. M.; Terol, J.; Talón, M.; y Robles, M. 2005. Blast2GO: a universal tool for annotation, visualization and analysis in functional genomics research, Bioinformatics 21:3674 - 3676. [ Links ]
Cruz, J.R.; Silva, M.L.; y Torres, J. 2000. Nuevas opciones de drenaje. p 13. En: Memorias del V Congreso de la Asociación Colombiana de Técnicos de la Caña de Azúcar (Tecnicaña). Cali. Colombia. 4-6 de octubre de 2000. Tenicaña. Cali, Colombia. p. 13. [ Links ]
Cruz, J. R.; Torres, J. S.; Besosa, R.; y Rojas, R. 2009. Función de respuesta de la caña al agua. En: Memorias del VIII Congreso de la Asociación Colombiana de Técnicos de la Caña de Azúcar. Cali.; Colombia. 16-18 de septiembre de 2009. Tenicaña. Cali, Colombia. p. 256 - 262. [ Links ]
D'Hont, A. 2005. Unraveling the genome structure of polyploids using FISH and GISH; examples of sugarcane and bananas. Cytog. Genome Res.109:27 - 33. [ Links ]
De Almeida Silva, M.; Jifon, J. L.; Sharma, V. Da Silva J. A.; Caputo, M. M.; Damaj, M. B.; Guimarães E. R.; y Ferro, M. I. 2013. Use of physiological parameters in screening drought tolerance in sugarcane genotypes. Sugar Tech. 13(3):191 - 197. [ Links ]
Eavis, B. W. 1972. Effects of flooding on sugarcane growth 2. Benefits during subsequent drought. Proc. Int. Soc. Sugarcane Techn. 14:715 - 721. [ Links ]
Fox, S. E.; Geniza, M.; Hanumappa, M.; Naithani, S. et al. 2014. De novo transcriptome assembly and analyses of gene expression during photomorphogenesis in diploid wheat Triticum monococcum. PloS One 9(5): e96855. [ Links ]
Glaz, B.; Morris, D. R.; y Daroub, S. H. 2004. Sugarcane photosynthesis, transpiration, and stomatal conductance due to flooding and water table. Crop Sci. 44/:1633 - 1641. [ Links ]
Quan, R.; Hu, S.; Zhang, Z.; Zhang, H.; Zhang, Z.; y Huang, R. 2010. Overexpression of an ERF transcription factor TRSF1 improves rice drought tolerance. Plant Biot. J. 8:476 - 488. [ Links ]
Li, Q.; He, F.; Zhu, B. Q.; Liu, B.; Sun, R. Z.; Duan, C.Q.; Reeves, M. J.; y Wang, J. 2014. Comparison of distinct transcriptional expression patterns of flavonoid biosynthesis in Cabernet Sauvignon grapes from east and west China. Plant Phys. Bioch. 84:45 - 56. [ Links ]
Li, X. P.; Tian, A. G.; Luo, G. Z.; Gong, Z. Z.; Zhang, J. S.; y Chen, S. Y. 2005. Soybean DRE-binding transcription factors that are responsive to abiotic stresses. Theor. Appl. Gen. 110:1355 - 1362. [ Links ]
Liu, Y.; Subhash, C.; Yang, J.; Song, C.; Zhao, J.; y Li, J. 2011. Maize leaf temperature responses to drought: Thermal imaging and quantitative trait loci (QTL) mapping. Envir. Exp. Bot. 71(2):158 - 165. [ Links ]
SAS Institute Inc. 1991. SAS System for Linear Models (3rd Edition). Cary, NC. SAS Institute Inc. p 329. [ Links ]
Shinozaki, K.; Yamaguchi-Shinozaki, K. 2007. Gene networks involved in drought stress response and tolerance. J. Exp. Bot. 58(2):221 - 227. [ Links ]
Tougou, M.; Hashiguchi, A.; Yukawa, K.; Nanjo, Y. et al. 2012. Responses to flooding stress in soybean seedlings with the alcohol dehydrogenase transgene. Plant Biot. 29:301 - 305. [ Links ]
Trapnell, C.; Roberts, A.; Goff, L.; Pertea, G. et al. 2012. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nature Protocols 7(3):562 - 578. [ Links ]
Vettore, A.L.; da Silva, F.R.; Kemper, E.L.; Souza, G.M. et al. 2003. Analysis and functional annotation of an expressed sequence tag collection for the tropical crop sugarcane. Genome Res. 13:2725 - 2735. [ Links ]
Viveros, C A. 2011. Identificación de características asociadas con la mayor eficiencia en el uso de agua para la producción de caña de azúcar. Tesis Doctoral. Universidad Nacional de Colombia, Sede Palmira. Palmira, Valle, Colombia. p. 120. [ Links ]
Wang, L.; Feng, Z.; Wang, X.; Wang, X.; y Zhang, X. 2010. DEGseq: an R package for identifying differentially expressed genes from RNA-seq data. Bioinformatics 26 136 - 138. [ Links ]
Wu, J.; Wang, L.; Li, L.; y Wang, S. 2014. De novo assembly of the common bean transcriptome using short reads for the discovery of drought-responsive genes. PloS One. 9(10):e109262. [ Links ]