Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.65 no.1 Palmira Jan./Mar. 2016
https://doi.org/10.15446/acag.v65n1.47496
http://dx.doi.org/10.15446/acag.v65n1.47496
Análisis filogenético del murciélago hematófago Desmodus rotundus en el Valle del Cauca Colombia
Phylogenetic analyses of the hematophagous bat Desmodus rotundus in Valle del Cauca, Colombia
Fernando Favian Castro Castro 1*, Jaime Eduardo Muñoz Flores 1, y Wilson Uieda 2
1 Facultad de Ciencias Agropecuarias, Universidad Nacional de Colombia sede Palmira, Colombia. 2 Departamento de Zoología, Instituto de Biociencias. Universidade Estadual Paulista, Botucatu, Sao Paulo, Brasil. *Autor para correspondencia: fernandomorcegos@gmail.com
Rec.: 25.11.2014 Acep.: 09.06.2015
Resumen
En los municipios La Victoria, Águila, Obando, Cartago, Zarzal, San Pedro, Cerrito y Palmira del departamento Valle del Cauca; Mercaderes en el Cauca; y Puerto Nariño en el departamento del Amazonas, se realizó el análisis de filogenia del murciélago hematófago Desmodus rotundus. Las muestras de tejido epitelial fueron tomadas en el patagio del quiróptero, amplificadas por PCR utilizando cebadores 16SL y 16SH, se amplificó ADN mitocondrial, y se secuenciaron 50 individuos de murciélagos hematófagos. Después de la secuenciación se encontraron ocho haplotipos y se encontró que el más frecuente se presenta en 43 individuos. Se utilizaron dos métodos, máxima verisimilitud y parsimonia. Utilizando el Genbank se revisaron secuencias de marcadores mitocondriales s16 rRNA de Brasil, Venezuela y Costa Rica. Se encontró un acercamiento de estos individuos de Costa Rica y Venezuela con murciélagos hematófagos de los departamentos del Valle del Cauca y Cauca, ubicados en el sur occidente de Colombia. Se revisan aspectos genéticos como relaciones filogeográficas entre las poblaciones y se encontró en el estudio que existe una conexión entre los diferentes haplotipos de los clados existentes reflejado en los árboles filogénicos y red de haplotipos, que se formó en varias generaciones producto posiblemente de migraciones de los murciélagos hematófagos a lo largo de las cordilleras y ríos que atraviesan Colombia.
Palabras clave: ADN, Clados, Genbank, Haplotipos, Patagio, PCR, 16SL, 16SH
Abstract
Phylogenetic analyses of the hematophagous bat Desmodus rotundus were performed in villages belonging to Valle del Cauca, Cauca and Amazonas regions. Samples were mainly collected at hematophagous bat shelters located in Victoria, Águila, Obando, Cartago, Zarzal, San Pedro, Cerrito and Palmira villages belonging to Valle del Cauca region, two samples at Mercaredes which belong to Cauca, two samples at Puerto Nariño which is part of Amazonas region. Epithelial tissue samples were collected from bat's patagium, and submitted to PCR using primers 16 SL and 16SH amplifying mitochondrial DNA of 50 vampires, these products were sequencing resulting in eight haplotypes being the most common presented in 43 individuals. Two methods to reach maximum likelihood and parsimony were used. GENBANK sequences of mitochondrial markers s16 rRNA from Brazil, Venezuela and Costa Rica were used for comparison. Higher similarity between vampire bats from Valle del Cauca and Cauca region, located in the southwest Colombia and vampire bats from Costa Rica and Venezuela was found. In the present study the genetic profiles correlated with the phylogeographic aspects of different populations was performed and a connection between different haplotypes of existing clades was found according to the phylogenetic tree and haplotype net, possible being generated during hematophagous bats migration over the mountain–chain and rivers crossing Colombia.
Keywords: DNA, clades, Genbank, Haplotypes, patagium, PCR, 16SL, 16SH
Introducción
Los murciélagos son los únicos mamíferos con capacidad de vuelo debido a la transformación de sus brazos en alas (Villa, 1976), pertenecen al orden Chiroptera que contiene casi 1000 especies, siendo los más numerosos después de los roedores (Grennhall, 1993). Están distribuidos en 18 familias, 168 géneros dos subórdenes megachiroptera y microchiroptera. El suborden megachiroptera se encuentra conformado por quirópteros de gran tamaño, con envergaduras superiores a 1 m que viven en África, Asia y Oceanía (Bredt et al., 1998). El suborden Microchiroptera es de amplia distribución geográfica, se encuentra en todos los continentes y su tamaño es pequeño; incluye la familia Phyllostomatidae, la subfamilia: Desmodontinae y las especies Desmodus rotundus, Diphylla ecaudata y Diaemus youngi (Bredt et al., 1998).
El murciélago hematófago D. rotundus, sub–familia Desmodontinae (Chiroptera, Phyllostomidae) es una de las tres especies propias de América. La distribución geográfica se extiende desde el norte en México a Sudamérica a lo largo de la Costa Pacífica en Chile y a la costa atlántica de Uruguay, hasta el extremo de Brasil (Villa, 1976). Se alimenta de sangre de animales de corral, domésticos, silvestres y en ocasiones extremas del hombre (Grennhall, 1993).
D. rotundus es la especie más estudiada (Uieda, 1986). Su hábitat se encuentra en cavernas, árboles con tallos huecos, techos de casas abandonadas y aljibes (Paz et al, 1990). Se desplaza a lo largo de grandes ríos (ICA, 2002), tiene hábitos migratorios y su vuelo puede alcanzar hasta 100 km. En Colombia esta especie es la más abundante entre 0 y 1800 m.s.n.m. (ICA, 2002).
De acuerdo con Brass (1995) el murciélago hematófago es uno de los principales transmisores de enfermedades como la rabia en bovinos que es una amenaza para el hombre y como tal una zoonosis de alta importancia en América Latina. Según Flores (1978) en Latinoamérica esta enfermedad afecta anualmente un millón de cabezas de bovinos. Entre 1998 y 2003 en Colombia se han incrementado los focos de rabia, pasando de 44 a 194 en este periodo, lo que significa un incremento de 400%, aproximadamente (ICA, 2014). Uno de los de departamentos más afectados es el Valle del Cauca, especialmente en zonas ganaderas cercanas a los grandes ríos (Castro, 2014).
La filogeografía estudia la distribución geográfica de linajes genealógicos de una especie (Torres et al., 2010). En este estudio esta técnica se aplicó en el caso del murciélago hematófago D. rotundus en el departamento del Valle del Cauca, para lo cual se utilizaron marcadores moleculares ADN mitocondrial (Avise, 2000) que permiten establecer el flujo genético entre poblaciones (Slatkin ,1987) y estimar la expansión de territorio en relación con la localización de supuestos refugios en zonas de expansión (Rogers et al., 1992).
Dentro de los marcadores moleculares más utilizados se encuentran las aloenzimas, PCR, RFLP, RAPD, microsatélites, minisatélites, secuenciamiento del DNA y SNP. La escogencia del marcador depende de factores como la magnitud del daño ocasionado, el tipo de material biológico y recursos financieros disponibles (Costa ,2011). Wilkinson (1985) utilizó aloenzimas para cuantificar y explicar la estructura molecular del D. rotundus en Costa Rica y encontró que los machos subordinados son más dispersantes de genes y contribuyen más a la variabilidad genética de la población que los dominantes. Piaggio et al. (2008) desarrollaron marcadores nucleares microsatélites para doce loci, lo que permite un nuevo abordaje principalmente a nivel intraespecífico del murciélago hematófago D. rotundus, especie donde se observa relación entre individuos.
Los primeros estudios utilizando marcadores moleculares nucleares y mitocondriales se realizaron en Brasil y permitieron entender la variabilidad genética y la distribución geográfica de una especie (Martins et al., 2007). Pinto (2009) en Ecuador, utilizando marcadores moleculares nucleares y mitocondriales, estudio el efecto de la cordillera de Los Andes como barrera geográfica y cuantificó su impacto en la diversificación y el aislamiento de la flora y fauna neotropical y las grandes divergencias entre las poblaciones de murciélagos hematófagos del este y el oeste. Dischfield (2000) en el bosque de la zona atlántica de Brasil encontró haplotipos en D. rotundus altamente divergentes para citocromo b en localidades relativamente próximas, lo que hace suponer la existencia de heterogeneidad en la distribución geográfica de la especie. Martins et al. (2007) hallaron un patrón filogeográfico, genético y morfológico para el D. rotundus con formación de cuatro poblaciones geográficas circunscritas, a saber: (1) Mata Atlántica do Norte, (2) Mata Atlántica do Sur, (3) Pantanal y (4) Cerrado, lo que indica que los ejemplares de D. rotundus son morfológicamente semejantes, pero genéticamente diferentes. En el estudio se reconocen dos líneas atribuidas a D. rotundus como subespecies diferentes, una al este (Mata atlántica) y otra al oeste (Cerrado y la amazonia) de Brasil. Según Goncalves (2011) los especímenes de la Amazonia y de América Central forman una población homogénea. Castela et al. (2001) observaron diferenciación poblacional entre colonias de hibernación localizadas en los polos opuestos de los Alpes de Europa, cuando analizaron la región control de ADN mitocondrial en murciélagos de las especies Myotis myotis, sin embargo, cuando usaron marcadores nucleares microsatelites no encontraron diferenciación entre ellas.
Se realizó un estudio en India (Chinnasamy et al., 2013) de la variación en la estructura genética del murciélago de nariz roja Hipoosidero speoris especie no migratoria, utilizando la secuencia de 16s ARNr y microsatélites. Los datos obtenidos ofrecen diferentes patrones de estructura genética dentro de las poblaciones ubicadas cerca de un rango de 250 km, teniendo en cuenta la filopatría marcada de las hembras y el aislamiento de las poblaciones.
En Colombia no existen estudios morfológicos a nivel molecular sobre migración y difusión entre refugios de murciélagos hematófagos en una zona determinada, con el fin de conocer sus desplazamientos y conectividad a nivel de filogenia de los refugios y orientar los programas sobre conservación y control de la rabia en bovinos.
Materiales y métodos
Obtención de muestras
Las capturas periódicas de murciélagos hematófagos (D. rotundus) se realizaron con la colaboración del Instituto Colombiano Agropecuario (ICA) en diez refugios localizados en dos fincas georreferenciadas, utilizando para tal fin mallas de niebla colocadas cerca a dichos refugios. La identificación de los ejemplares capturados se hizo con base en la experiencia en campo de personal capacitado del ICA y con el uso de claves de identificación (Aguirre et al., 2001).
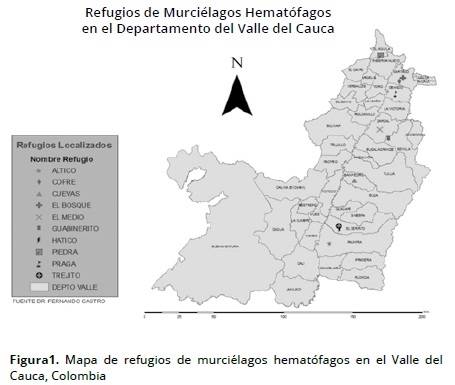
La toma de muestras se realiza tanto en machos como hembras adultos; para ello se utilizó el patagio del ala izquierda previamente desinfectado de donde se extrajeron 4 mm del dactilopatagio. Posteriormente el tejido recolectado fue colocado en un tubo desinfectado con alcohol absoluto a 96%, refrigerado en nevera plástica y acondicionado para traslado al Laboratorio de Biología Molecular y Genética en de la Universidad Nacional de Colombia sede Palmira, lo que tarda normalmente entre 12 y 24 h. Una vez finalizado el estudio los animales en perfectas condiciones eran liberados al ambiente. Las características de los predios seleccionados donde se realizaron las capturas aparecen en la Tabla 1 y Figura 1.
Obtención de secuencias
Para la extracción de ADN se utilizó un ‘kit’ comercial Wizard® Genomic DNA Purification Kit (Promega). Este kit está diseñado para el aislamiento de ADN de células blancas de la sangre (leucocitos), cultivos celulares, tejido animal, plantas y bacterias. El procedimiento de extracción de ADN se basa en: (1) proceso de purificación de ADN. Consiste en la lisis celular, donde se rompe la membrana celular liberando el material genético, a su vez, ocurre una lisis del núcleo usando una solución especial (Nuclei Anlysis Solution); (2) digestión, opcional, de RNAs; (3) las proteínas celulares son removidas por precipitación en sal, aunque conservan el ADN; y (4) el ADN genómico es desalinizado y concentrado por medio de la precipitación en isopropanol, estado que en el cual es estable por un tiempo aproximado de 1 año (Wizard®, 2010).
La cuantificación de ADN de las muestras de tejido se hizo utilizando un equipo Nano Drop 2000 Thermo scientfi espectrophotometro. La calidad del ADN obtenido en las muestras se comparó con concentraciones conocidas de ADN del bacteriófago Lamda, en gel de agarosa a 0.8% y corridas en tampón TBE 0.5X (Tris–borato 0045M; EDTA 0.001M) a un voltaje constante de 80 voltios durante 1 h (Muñoz, 2011).
La amplificación del gen 16s de mamíferos se hizo en varios análisis filogenéticos por su función y distribución universal, facilidad de aislar y caracterizar, así como por el contenido tanto regiones altamente variables como conservadas. El par de primers fueron: forward 16SL (5'–GCCTCGCCTGTTACCAAAAAC–3') y revers 16SH (5'–CCGGTCTGAACTCAGATCACGT–3'), el cual amplifica una región de 340 pb, aproximadamente (Palumbi et. al, 1996). El programa tiene como parámetros: 30 ciclos de 30 segundos a 94 °C para desnaturación; 1 min a 50 °C para hibridización; 2 min a 72 °C para extensión. Un ciclo inicial de desnaturación de 5 min a 94 °C, en el final de reacción una extensión de 7 min a 72 °C que se llevó a cabo en un termocilcador Biocycler–Biosystems®
Para la amplificación se utilizaron DNA 1 ng/µl DNTP, 4 µl solución tampón 2.5 µl MgCl 0,5 µl, Primers 0,25 µl,Taq polimerasa 0.20 U/ µl agua 16.55 para un volumen total de 25 µl. Los productos de la amplificación de los genes en estudio fueron analizados por electroforesis en gel de agarosa a 1%, detectados por coloración con bromuro de etídio y visualizados sobre luz UV.
Las secuencias fueron realizadas a través del método didesoxiterminal (Sanger et al., 1997) en secuenciador automático de DNA ABI prism 3500 de Life Technologies en Genbimol laboratorio de biología molecular de la Universidad Estadual de Marañao en Brasil. Los programas utilizados para analizar las secuencias fueron: Seq Scaner software v.1.0 Applied Biosystems; Secuencia Scanner Software que permite graficar y editar datos de impresión y exportar secuencias generadas usando instrumentos analizadores que generan informes gráficos, permiten alinear y analizar los picos de datos en bruto y aquellos fuera de escala.
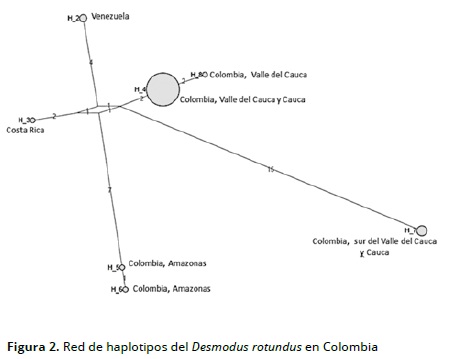
La mega versión 5 permite hacer alineaciones de las secuencias manuales y automatizadas con editor de archivo de rastreo y arboles filogenéticos utilizando el algoritmo de Neighbor Joining (NJ), lo que se hizo mediante la construcción de un árbol con un bootstrap de 1000 autoréplicas y un grupo externo Diphylla ecaudata. Con Network versión 4.6.1.0 se hizo el análisis de filogenia intraespecífica utilizando redes de haplotipos con el algoritmo Median joining en el Departamento de Genética la Universidad del Valle en Cali Colombia. (figura 2)
La comparación de las muestras se hizo con el Banco de Datos del Genbank tomando individuos de otros países latinoamericanos de la especie Dyphilla ecaudata (Outgrup). Se usaron la secuencia con acceso [Diphylla ecaudata_AF411533, Venezuela [D. rotundus1_HG003310, D. rotundus 2_NC_022423] y Costa Rica [D. rotundus 3_AF263228], resultando en total 54 muestras con el objeto de analizar distancias filogenéticas entre todos los individuos en estudio, tomando el Genbank como punto de referencia.
Resultados
Análisis filogenético
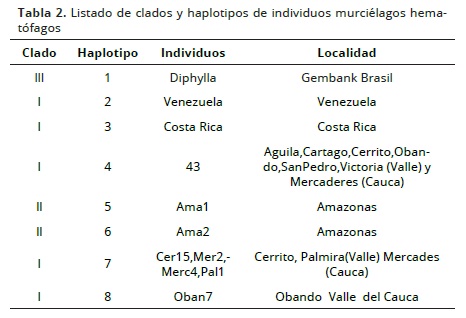
En 54 murciélagos hematófagos D. rotundus se encontraron 8 haplotipos que formaron 3 clados:
- Clado 1 comprende cinco haplotipos 2, 3, 4, 7 y 8 individuos del Valle del Cauca, Cauca, Costa Rica y Venezuela. Con individuos en los municipios del Águila, Cartago; Cerrito, Obando y La Victoria en el Valle del Cauca; Mercaderes en el Cauca; en Venezuela y Costa Rica.
- Clado 2 con individuos en el departamento del Amazonas, haplotipo 5 y 6 (98) del municipio de Puerto Nariño.
- Clado 3, con haplotipo 1 de la especie D. ecaudata (Outgrup).
Los cuales generaron los datos siguientes:
Existen clados que tienen conectividad. En el Clado 1 se evidencia que los animales de Valle y Cauca, y Venezuela y Costa Rica tienen proximidad; mientras que en el Clado 2 los individuos del Amazonas tienen conectividad entre sí. El haplotipo 4 fue el principal y comprendió los municipios del departamento del Valle del Cauca (El Águila ,Cartago, Cerrito, Obando, San Pedro, La Victoria y Zarzal) y Mercaderes en el Cauca (Figura 3 y Tabla 2).
Se realizó un análisis de red de haplotipos con el programa Networdk 4.51.0 con el fin de identificar el haplotipo principal, que es el número 4 con 43 individuos y los derivados haplotipos 5 y 6 del departamento del Amazonas. Los haplotipos 2 de Venezuela y 3 del Costa Rica están diferenciados y el haplotipo 7, con individuos del sur del departamento del Valle y los municipios de Palmira y Cerrito y el municipio de Mercaderes en el Cauca, es el haplotipo mas derivado porque contiene mayor número de pasos mutacionales. El haplotipo 8 es del municipio de Obando en el departamento del Valle del Cauca y derivado del haplotipo 4 (Figura 4).
Discusión
Los estudios filogeográficos sobre murciélagos hematófagos en Latinoamérica son limitados (Goncalves, 2010), aún más, en Colombia no existen. Con este estudio se presenta la primera visión para determinar varios aspectos genéticos de la especie D. rotundus en el país para iniciar aproximaciones al estudio de migraciones en poblaciones de murciélagos colombianas.
Se encontraron relaciones entre los individuos de las localidades en los departamentos del Valle del Cauca y Cauca y de estos con individuos en Venezuela y Costa Rica, producto de las migraciones de esta especie durante varias décadas, lo que confirma porqué el vampiro es clasificado como una especie migratoria de tercer tipo característico de la familia phyllosmatidae (IGAC, 2009). Se observa que el haplotipo 4 es posiblemente el haplotipo ancestral debido a que se encuentra más conservado y tiene la mayoría de individuos, alcanzando 43 del total de 54 en la muestra, los demás son haplotipos satélites. Se observó conexión entre los clados debido a factores geográficos, hidrográficos, ecológicos y ambientales.
Los geográficos son importantes debido a la topografía del terreno y las existencias de barreras geográficas, lo que afecta o beneficia la movilización de los murciélagos. En el caso de este estudio la topografía plana es favorable debido a la presencia del valle interandino formado por las cordilleras occidental y central, que se expande por los departamento de Cauca y Valle del Cauca (IGAC, 2014).
La hidrografía es importante en la migración de quirópteros ya que por ausencia de obstáculos utilizan los ríos para el desplazamiento durante el vuelo (ICA, 2002). En este caso la existencia del Macizo colombiano, donde se bifurcan las cordilleras Central y Occidental y nacen los ríos Cauca y Magdalena. El Cauca atraviesa ambos departamentos favoreciendo las migraciones de los murciélagos (ICA, 2002).
Es importante tener en cuenta que esta especie hace grandes desplazamientos, especialmente los machos que tienen un patrón de dispersión más evidente, y donde machos juveniles son expulsados por los residentes cuando tienen entre 12 y 18 meses de edad por lo que deben buscar un refugio nuevo (Wilkinson, 1985).
Se observó una relación entre localidades estudiadas debido a la existencia de migraciones de animales, como es el caso de la proximidad de individuos del Valle del Cauca y Cauca con individuos de Centro América (Costa Rica) e individuos de Venezuela.
La variabilidad genética de D. rotundus en el Valle del Cauca y Cauca se debe, posiblemente, a la migración activa que realizan machos y hembras en busca de alimento. Se observa la existencia de una migración forzada favorecida por la actividad antrópica que destruye ecosistema obligando al murciélago a ocupar refugios artificiales cercanos a centros urbanos (Castro, 2014).
Referencias
Díaz, M. M.; L. F. Aguirre, L. F.; y Barquez, R. M. 2011. Clave de identificación de los murciélagos del cono sur de Sudamérica. Centro de Estudios en Biología Teórica y Aplicada. Cochabamba, Bolivia. 94 p. [ Links ]
Avise, J. C. 2000. Phylogeography: The History and Formation of Species. Harvard Univ. Press, Cambridge, MA. 447 p. [ Links ]
Brass, D. A. 1995). Rabies in bats: natural history and public health implications. Epidemiol Infect, 114(3): 522. Recuperado de http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2271301/page 336 [ Links ]
Bredt, A. F.; Araújo, J.; Caetano, J. R.; Rodrigues, G. R.; Yoshikawa, M.; y Silva,M. M. 1998. Morcegos em áreas urbanas e rurais: manual de manejo e controle. Brasília, Fundação Nacional de Saúde. 117 p. [ Links ]
Castella,V.; Ruedi. M.; y Excoffier. L. 2001. Contrasted patterns of mitochondrial and nuclear structure among nursery colonies of the bat Myotis miotys. J. Evol. Biol. 14–708–720. [ Links ]
Castro,C. F. 2014. Análisis molecular y aspectos ecológicos del murciélago hematofago Desmodes rotundus en el departamento del Valle del Cauca. Universidad Nacional de Colombia, Palmira, Ph.D. Tesis. 136 p. [ Links ]
Costa, P. P. 2011. Ecologia molecular de Desmodus rotundus (chiroptera: phyllostomidae) no parque estadual de campinhos, Paraná, Brasil (Tesis de Maestría). Universidade Federal do Paraná, Brasil. Recuperado de http://www.iap.pr.gov.br/arquivos/File/Pesquisa%20em%20UCs/resultados%20de%20pesquisa/14_07_POLLYANA_COSTA_%20Ecologia_Molecular_de_Desmodus_rotundus_(Chiroptera,Phyllostomidae)_no_Parque_Estadual_de_Campinhos_Parana_Brasil.pdf [ Links ]
Clare, E. L. 2011. Cryptic species? Patterns of maternal and paternal gene flow in eight neotropical bats. PloS ONE, vol. 6(7): e21460. Recuperado de http://www.ncbi.nlm.nih.gov/pmc/articles/PMC3144194/. [ Links ]
MADR (Ministerio de Ambiente, Vivienda y Desarrollo Territorial. 2009. Plan Nacional de las especies migratorias. Diagnóstico e identificación de acciones para la conservación y el manejo sostenible de las especies migratorias de la biodiversidad en Colombia. 1ª. ed. Colombia. Comunicaciones y Equipo de Conservación –WWF Colombia. Recuperado de https://www.minambiente.gov.co/images/BosquesBiodiversidadyServiciosEcosistemicos/pdf/Planes–para–la–conservacion–y–uso–de–la–biodiversidad/211010_plan_especies_migratorias.pdf [ Links ]
IGAC (Instituto Geográfico Agustin Codazi). IGAC. Recuperado de http://www.igac.gov.co/igac . consulta 14 noviembre 2014 [ Links ]
Costa Rica. Instituto Geográfico Nacional. Recuperado de http://unstats.un.org/unsd/geoinfo/RCC/docs/rcca10/E%20Conf_103_30_Costa%20Rica_Ponencia_UNRCCA_Max%20A%20Lobo–Hernandez. pdf. 14 noviembre 2014 [ Links ]
Chinnasamy, K.; Pitchamuthu, M.; Doss, P.S.; Marimuthu, G. & Rajan, K. E. (2013). Genetic diversity and population structure of leaf–nosed bat Hipposideros speoris (Chiroptera: Hipposideridae) in Indian subcontinent. African Journal of Biotechnology, vol. 10(8): 1320–1328. Recuperado de http://www.ajol.info/index.php/ajb/article/view/92937. [ Links ]
Ditchfield, A. D. 2000. The comparative phylogeography of neotropical Mammals:patterns of intraspecific mitochondrial DNA variation among bats contrasted to nonvolant small mammals. Mol. Ecol. 1307 – 1318. [ Links ]
Flores–Flores–Crespo, R. (ed.). 1978. La rabia en las diferentes especies, sus transmisores y su control. (1ª. ed.). México. Inifap, Sagar. No. Pág.34 [ Links ]
Gonçalves de Andrade, F. A. 2009. Variabilidade morfométrica e molecular em desmodus rotundus (chiroptera, phyllostomidae) de diferentes áreas de risco para raiva rural no Estado Do Pará, Brasil (Tesis de doctorado). Universidade Federal Do Pará. Recuperado de http://www.ufpa.br/lama/pub/doutorado/Tese%20Fernanda.pdf [ Links ]
Greenhall, A. M. 1993. Bats and Rabies Edition fundation Marcel Meriux,No. pág 3–57. [ Links ]
ICA (Instituto Colombiano Agropecuario). 2002. Guía metodológica para la prevención y el control de la rabia de origen silvestre, Bogotá. 63 p. [ Links ]
Larsen, R. J.; Knapp, M. C.; Genoways, H. H.; Khan, F. A. A.; Larsen, P. A. et al. 2012. Genetic diversity of neotropical myotis (Chiroptera: Vespertilionidae) with an emphasis on South American species. Public Library of Science. 7(10):9. Recuperado de http://www.plosone.org/article/info%3Adoi%2F10.1371%2Fjournal.pone.0046578 [ Links ]
Martins, F. M.; Ditchfield, A. D.; Meyer, D.; y Morgante, J. S. Mitochondrial DNA phylogeography reveals marked population structure in the common vampire bat, Desmodus rotundus (Phyllostomidae). J. Zool. Syst. Evol. Res. 45(4):372 – 378. [ Links ]
Ministerio del Poder Popular para Ecosocialismo, Hábitat y Vivienda, IGVSB. Venezuela. Recuperado de http://www.igvsb.gob.ve/ . consulta 14 noviembre 2014 [ Links ]
MINSA (Ministerio de Salud). 2010. Norma técnica de prevención y control de la rabia en el Peru. vol. 1 [ Links ]
Slatkin, M. 1987. Gene flow and the geographic structure of natural populations. Science 236:787 – 792. [ Links ]
Muñoz, F. J. 2011. Diversidad genética, estructura poblacional y selección de clones superiores de Guadua angustifolia Kunth en la eco–región cafetera de Colombia. (Tesis de Doctorado). Universidad Nacional de Colombia, Palmira. Colombia. Recuperado de http://www.bdigital.unal.edu.co/5321/1/16246522.2011.pdf [ Links ]
Rogers, A. R. 1992. Harpending, h. Population growth makes waves in the distribution of pairwise genetic differences. Mol. Biol. Evol. 9:552 – 569. [ Links ]
Palumbi, S,R.(1996). Nucleic acids II: The polymerase chain reaction. In: Hillis DM, Moritz C, Mable BK (eds) Molecular systematics. Sinauer Associates, Inc , pp. 205–247 [ Links ]
Paz, O.; Benzal, M.; y Fernández, R. 1990. Criterios de valoración de refugios para murciélagos: aplicación al inventario nacional. No. 4, Ecologia 4, ICONA. Madrid. p. 191 – 206. [ Links ]
Piaggio, A. J.; Johnston, J. J., y Perkins, S. L. 2008. Development of polymorphic microsatellite loci for the common vampire bat, Desmodus rotundus (Chiroptera: Phylostomidae). Mol. Ecol. Res. 8:440 – 442. [ Links ]
Pinto, M. 2009. Genetic diversity of the common vampire bat Desmodus rotundus in Ecuador: testing cross–andean gene flow (Tesis de Maestría). Texas Tech University. Texas. Recuperado https://repositories.tdl.org/ttuir/bitstream/handle/2346/11055/Pinto_Miguel_Thesis.pdf?sequence=1 [ Links ]
Tamura, K.; Peterson, D.; Peterson, N.; Stecher, G.; Nei, M.; et al. 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Mol. Biol. Evol. 28(10):2731 – 2739. [ Links ]
Torres Flores, J. W. y Guevara Chumacero, L. M. 2010. Perspectivas sobre el origen y la filogenia de los murciélagos. ContactoS (77):5 – 9. Recuperado de http://www.izt.uam.mx/newpage/contactos/anterior/n77ne/murcielago.pdf [ Links ]
Sanger, F.; Nicklen, S.; y Coulson, A. R. 1977. DNA Sequencing with chain terminating inhibitors. Proc. Nat. Acad. Sci. 74(12):5463 – 5467. [ Links ]
Uieda, W. 1987. Morcegos hematófagos e a raiva dos herbívoros no Brasil.Seminario de Ciencias da FIUDE, Ubera, p. 13 – 29. [ Links ]
Uieda, W. 1986). Morcegos hematófagos e a raiva dos herbívoros no Brasil. En: Seminario de Ciencias da FIUDE, Uberaba. [ Links ]
VILLA, B. 1976. Biología de los murciélagos hematófagos. Laboratorio de mastozoología, instituto de Biología UNAM, México DF: p. 94– 99. [ Links ]
Weaver, K. N., Alfano, S. E., Kronquist, A. R.; y Reeder, D. M. 2009. Healing rates of wing punch wounds in free–ranging little brown myotis (Myotis lucifugus). Acta Chiropterologica 11(1):220 – 223. [ Links ]
Wilkinson, G. S. 1985. Altruismo y la cooperación en los murciélagos. En: Avances recientes en el estudio de los murciélagos (Fenton, MB, Racey, PA y Rayner, JMV, eds.). Cambridge University Press, Editorial Cambridge. Cambridge:1985, p. 299 – 323. [ Links ]
Wizard® 2010. Genomic DNA Purification Kit, technical manual. [ Links ]