Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Acta Agronómica
versão impressa ISSN 0120-2812
Acta Agron. vol.65 no.3 Palmira jul./set. 2016
https://doi.org/10.15446/acag.v65n3.52212
http://dx.doi.org/10.15446/acag.v65n3.52212
Aplicación de visión por computadora en la evaluación del desarrollo in vitro de protocormos de Oncidium sphacelatum Lindl., (Orchidaceae)
Application of computer vision in the assessment of the in vitro development of Oncidium sphacelatum Lindl., (Orchidaceae) protocorms
Vincenzo Bertolini 1*, Javier Francisco Valle Mora1 y Ángel Natanael Rojas Velásquez 2
1El Colegio de la Frontera Sur (ECOSUR), Tapachula. Laboratorio de Ecología y Cultivo Sustentable de las Orquídeas del Soconusco., México. 2 Facultad de Agronomía de la Universidad Autónoma de San Luis Potosí, San Luis Potosí, México. *Autor para correspondencia: vin.bertolini@gmail.com
Rec.: 30.07.2015 Acep.: 09.09.2015
Resumen
Se presenta una aplicación de visión por computadora diseñada para monitorear el desarrollo in vitro de protocormos de la orquídea Oncidium sphacelatum Lindl. A partir de fotografías digitales, el crecimiento y el desarrollo de los protocormos se evaluó mediante parámetros como su tamaño (área en pixeles), el cambio de etapa de desarrollo dentro del proceso de germinación (medido a través del factor de redondez) y el estado de salud (por tonalidad de color). Usando la plataforma Matlab R2012a, la programación desarrollada para analizar estos parámetros se compone de una etapa de segmentación para discernir los pixeles de interés (protocormos) del fondo, una de entrenamiento–aprendizaje para establecer criterios de segregación de los objetos de interés, y una última de clasificación del color. Las características obtenidas por el sistema proporcionan información útil para la evaluación del proceso de germinación de orquídeas en condiciones in vitro, con un ahorro significativo en el tiempo y esfuerzo dedicados por el operador humano.
Palabras clave: Clasificación de patrones, cambios morfológicos, clasificador de color LDA, conjunto de semilla, conservación de especies amenazadas.
Abstract
We report an application of computer vision designed to monitor the in vitro development of Oncidium sphacelatum Lindl., orchid protocorms. Growth and development of protocorms was evaluated in terms of size (measured through the area in pixels), changing in its shape within the germination process (measured by the roundness factor) and state of health (color tone), using digital photos. Programming the software Matlab R2012a, we developed a type of analysis, consists of a first stage as segmentation to discern the pixels of interest (protocorms), a second stage of training and learning to establish criteria for segregation of interest object and a final stage as classification. Programming is performed by R2012a Matlab platform. The characteristics obtained by our system provide useful information for evaluating the germination process of in vitro propagation by orchid grains, with significant savings in terms of hours/work of the human operator.
Keywords: Patterns classification, morphological changes, LDA color classifier, seed set, conservation of endangered species.
Introducción
Las semillas de las orquídeas se encuentran entre las más pequeñas del reino vegetal, básicamente consisten en un embrión y la testa, careciendo de una reserva de nutrientes (Arditti y Abdul–Ghani, 2000). Por ello, para su germinación requieren de una relación simbiótica obligada con hongos micorrízicos tipo Rhizoctonia (Hadley, 1997; Suárez et al., 2006; Valadares et al., 2013). En orquídeas el proceso de germinación empieza cuando la semilla se hidrata y toma un color verde, lo que origina un tejido indiferenciado denominado protocormo, del cual se origina la plántula después de pasar por cinco etapas de desarrollo (Seaton y Ramsay, 2005): (1) formación del protocormo, (2) aparición de los rizoides, (3) aparición del meristemo apical, (4) desarrollo de la primera hoja y (5) aparición de la primera raíz. A partir de la etapa 5 se considera ya formada la plántula (Zettler y McInnis, 1994).
La germinación in vitro de semillas de orquídeas es vista como una herramienta que ayuda en la conservación de especies amenazadas. De la misma manera, conocer el proceso de germinación es básico para mejorar los protocolos de propagación in vitro para estas especies. Sin embargo, monitorear el proceso de germinación in vitro de semillas de orquídeas, conlleva un cierto grado de dificultad debido al tamaño diminuto tanto de la semilla como de los protocormos, implicando además costos en término de horas laborales y el esfuerzo dedicado por quienes lo llevan a cabo. Además, factores como la etapa de desarrollo y el color de los protocormos son subjetivos y dependen del punto de vista de cada operador.
Actualmente existen herramientas de la informática capaces de capturar y procesar imágenes que analizan con exactitud la calidad y clasificación de semillas. Sin embargo, existe un creciente interés en desarrollar aún más estos métodos computarizados para reemplazar los métodos de inspección tradicionales (ISTA, 1999 y AOSA, 2000). A pesar de este amplio interés, el factor de discrecionalidad humana sigue siendo preponderante (Dell' Aquila, 2009) y por otro lado, debido a las diferencias en tamaño, forma y color de las semillas de especies diferentes, se necesita el desarrollo de un protocolo de análisis computacional para cada una de ellas.
Una alternativa capaz de hacer un mejor análisis visual de la calidad y clasificación de las semillas durante la germinación la representan los enfoques que emplean la visión computacional. Este enfoque busca patrones visuales que se puedan relacionar con los cambios biológicos de una manera precisa y objetiva. Por ejemplo, Urena et al. (2001); han utilizado técnicas de lógica fuzzy para evaluar la germinación; Ducournau et al. (2005), han empleado la visión artificial, asociándola a un sistema de temporización, para monitorear los procesos de germinación en semillas de Helianthus annuus L. (Girasol); comparado con los resultados obtenidos por un operador humano, evidenciando el gran potencial de esta herramienta para la evaluación de los procesos de germinación. También, con estas herramientas se han logrado caracterizar las etapas de germinación de la orquídea Chloraea crispa Lindl., para fines de estudios botánicos y descripción del proceso (Verdugo et al., 2007). Más recientemente, Dell' Aquila (2009), ha empleado el área y factor de redondez de la semilla para determinar etapas en la germinación de Lens culinaris Medik. (Lenteja). Además, para identificar con exactitud el comienzo de la germinación, Dell' Aquila (2009); analizó los cambios temporales en las componentes RGB del color, estableciendo patrones en el cambio de color determinantes para considerar en germinación la semillas. El protocolo resultante es muy efectivo en el caso de la lenteja analizada por este autor. Sin embargo, podemos decir que su uso está limitado a una región predeterminada de la imagen y la discrecionalidad del operador humano resulta determinante en la interpretación de los resultados finales.
Con el fin de incrementar la objetividad en el análisis visual de la germinación de semillas de orquídeas y disminuir los tiempos de operación así como el desgaste físico del operador, aquí se propone un protocolo de programación desarrollado para la medición del área, factor de redondez y la clasificación del color durante el desarrollo de protocormos de Oncidium spachelatum Lindl. (Orchidaceae) cultivados in vitro.
Materiales y métodos
Material Vegetal
Las semillas de Oncidium sphacelatum Lindl., se obtuvieron de una cápsula dehiscente tomada de una planta cultivada en el Jardín Botanico "El Soconusco", ECOSUR unidad Tapachula. Las semillas fueron sembradas según los protocolos de Seaton y Ramsay (2005). Después de dos meses de siembra, se empezaron a tomar fotografías digitales para desarrollar el análisis. El material vegetal se mantuvo en condiciones ambientales controladas a 16 horas de iluminación por día y a una temperatura de 24°C ±2, durante toda la duración del experimento.
Adquisición imágenes computarizadas
Las imágenes de los protocormos en desarrollo fueron capturadas usando un estereoscopio VE–S5C Digital Stereo ZOOM Microscope VELAB™, equipado con una cámara digital de 1.3 MegaPixeles. Para probar el desempeño del protocolo de análisis de imágenes, se tomó una fotografía cada tercer día, un total de 7 imágenes digitales fueron ingresadas al sistema, por un total de 21 días de ensayo.
Protocolo de análisis de las imágenes
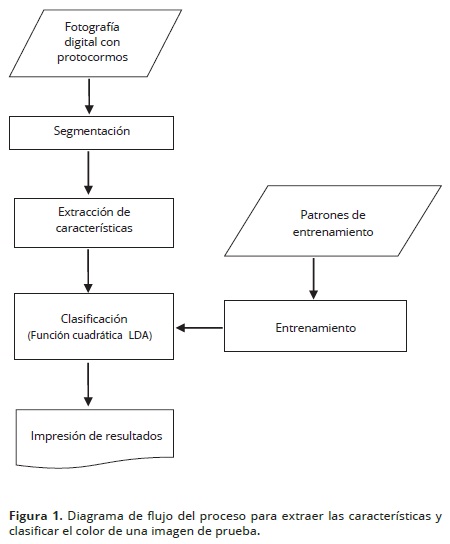
Se diseñó una secuencia de operaciones racionales (Figura 1) para llevar a cabo las operaciones de reconocimiento de los protocormos. Las partes de este protocolo se describen a continuación:
Segmentación
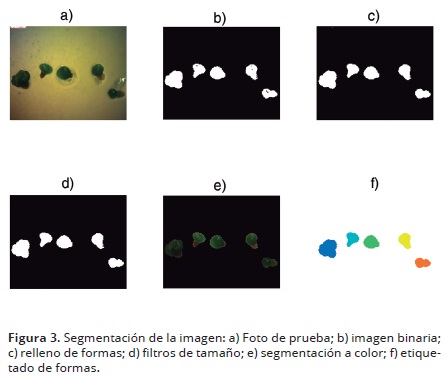
En este bloque, la fotografía digital se dividió en subregiones, cada una correspondió a un protocormo presente en la toma fotográfica; cada subregión se etiquetó con un índice diferente. De esta forma se logró diferenciar los protocormos entre si y del fondo representado por el medio de cultivo. También, se revisaron los valores rojo (R), verde (G) y azul (B) de los protocormos versus el fondo de la imagen, para diferenciar el color típico de los primeros y del segundo. Como resultado, se obtuvo una imagen binaria a la cual se aplicaron operaciones de relleno de formas, eliminación de objetos con contacto sobre los bordes de la imagen, eliminación de áreas de tamaño pequeño (ruido), y finalmente, se etiquetaron las componentes de objetos conectadas.
Extracción de características
Las características o patrones que se extrajeron de cada objeto etiquetado (protocormo) fueron: el área (A), factor de redondez (FR) y las componentes a – b del espacio de color Lab.
Para A se usaron operaciones de conteo de pixeles sobre cada objeto etiquetado. Básicamente se usaron dos ciclos For anidados para recorrer toda la imagen junto con un acumulador para almacenar el número de pixeles de cada objeto. Esa operación determina indirectamente el área o tamaño de cada protocormo.
FR, se originó dividiendo el cuadrado del perímetro del protocormo por el área previamente obtenida multiplicada por 4π, obteniendo así el número de pixeles presentes en el contorno del objeto, según la ecuación empleada por Dell' Aquila (2006):
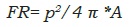
dónde: FR=Factor de redondez, p= perímetro, A= área del objeto.
Relativamente al color, la manera más común de representarlo es en el sistema RGB, sin embargo existen otras representaciones que pueden facilitar aún más el análisis al representar el color solo con 2 variables, así como lo hace el espacio de color Lab. La primera componente de este sistema contiene solo información de luminosidad, dejando toda la información del color en las otras 2 componentes a–b (Mathworks, 2014). Para extraer las componentes de cromaticidad a–b sobre los objetos etiquetados, se realizó el cambio del espacio de color RGB al espacio de color Lab en todos los pixeles, desechando la primera componente por tratarse de información acerca de su luminosidad.
Entrenamiento
El entrenamiento es la fase de aprendizaje que el programa debe de pasar antes de reconocer cada tono a través de características representativas seleccionadas previamente (grupo de entrenamiento). Así, el programa, a través de un criterio de clasificación aprendido, puede reconocer patrones y asociarlos a una determinada clase.
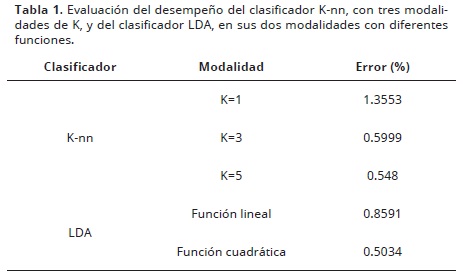
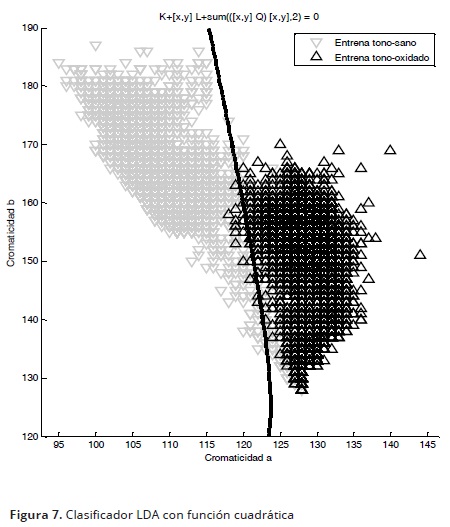
En el caso específico de los protocormos de orquídeas, los componentes de color a–b de cada pixel se agruparon en dos matrices de características, una etiquetada como "tonalidad–sana" y otra etiquetada como "tonalidad–oxidada", con el objetivo de reconocer protocormos viables y saludables. Se evaluó el desempeño de cinco clasificadores al entrenarlos y probarlos con las dos matrices. El primer clasificador evaluado, conocido como K–nn (K–nearest neighbors), realiza la asignación de clase basado en la clase del vecino (K=1) o vecinos (K>1) más cercanos del grupo de entrenamiento. Se realizó la medición de la distancia que existe entre cada uno de los patrones del conjunto de entrenamiento hacia la muestra a clasificar, etiquetándola con la misma clase del que tuvo la menor distancia (caso K=1 un solo vecino). Para valores de K mayores a uno, se asignó la clase que tenía la mayoría de los K vecinos cercanos. Se evaluaron en esta aplicación uno, tres y cinco vecinos (K=1, K=3 y K=5), tomando como muestras a clasificar el mismo grupo que se usó para el entrenamiento. También, se evaluó el clasificador LDA (Linear Discriminant Analysis), utilizando una función lineal y una cuadrática sobre los datos de entrenamiento. Con el fin de determinar el clasificador más eficiente se calculó el porcentaje de pixeles que fueron clasificados exitosamente y aquellos que no lo fueron tanto en las modalidades de K1, K3 y K5 para el clasificador Knn, como para el análisis lineal discriminante, utilizando una función lineal y una cuadrática. Para el clasificador LDA con función cuadrática, se estableció como frontera la siguiente ecuación para separar los 2 colores en las imágenes de prueba:
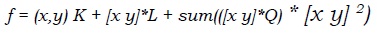
donde: K= –403.7486; L= [–0.8114;8.3282]; Q=[0.0238,–0.0246; –0.0246,–0.0089]
Calculado el desempeño de los clasificadores durante el entrenamiento, se decidió emplear el clasificador que presentó el mejor desempeño.
Clasificación
Para evaluar el estado de salud o vigor de los protocormos (objetos etiquetados), de cada pixel se tomaron los patrones de cromaticidad a–b extraídos en la sección de entrenamiento, se graficaron en el plano de color a–b, clasificando el pixel como "tonalidad–sana" si este caía del lado derecho de la función obtenida durante el entrenamiento, en caso contrario se identificó como "tonalidad–oxidada". De esta manera se obtuvo una estimación del estado de salud de cada protocormo.
Resultados y discusión
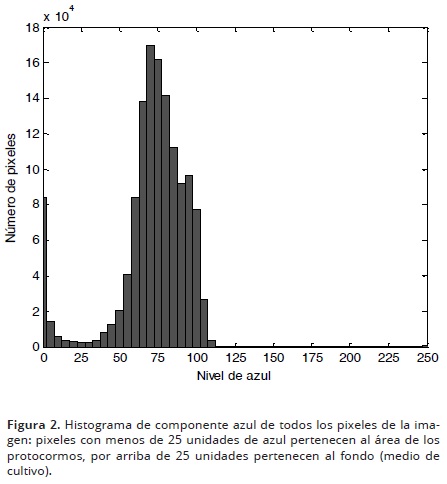
En la segmentación, las componentes roja y verde no presentaron diferencias marcadas entre protocormos y fondo (medio de cultivo). Al contrario, se observó que la gran mayoría de los pixeles correspondientes a los protocormos mostraban valores inferiores a 25 unidades de la componente azul, mientras que el fondo tenía valores superiores de la misma. Esta diferencia permitió establecer este umbral como primer criterio de segmentación, diferenciando entre medio de cultivo y protocormos, y así continuar con las otras operaciones de segmentación (Figuras 2 y 3).
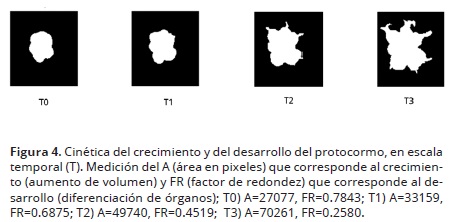
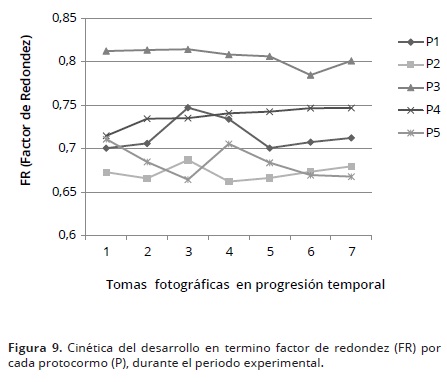
La Figura 4, ejemplifica como varían los parámetros A (área en pixeles) y FR (Factor de Redondez) de la imagen digital durante la germinación. El A, forzosamente indica el espacio bidimensional ocupado por el protocormo, y por ende puede asociarse al aumento o disminución de volumen (crecimiento). El FR puede asociarse al desarrollo de protocormo: en una forma perfectamente esférica el factor de redondez equivale a 1 y disminuye a medida que su forma geométrica se hace más irregular. Eso se debe a que durante las etapas de desarrollo del protocormo se pierde la forma esférica/ovoide al aparecer de hojas y raíces, dependiendo de la etapa de germinación en que se encuentre cada protocormo. Por lo tanto, el FR se puede asociar fácilmente al desarrollo de los protocormos durante la germinación y usar su valor para identificar las etapas de desarrollo descritas por Zettler y McInnis (1994).
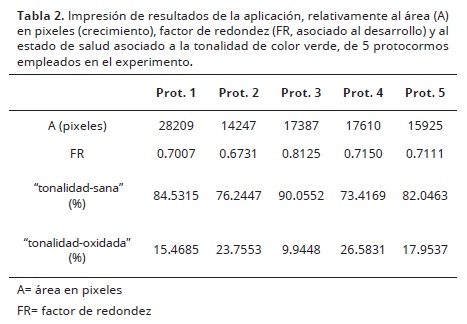
También, con el entrenamiento en función del color se pudo distinguir entre protocormos que presentaban un color brillante y fácilmente asociable a un buen estado de salud contra aquellos en que su tonalidad de verde fuese más oscura o al contrario muy pálida, y fácilmente asociable a protocormos con un mal estado de salud (Figura 5). Además de distinguir entre un protocormo con promedio de color aferente a la tonalidad–oxidado, se pudo distinguir al interior de cada protocormo el porcentaje de pixeles que tienen un tono de color verde opaco, café, poco saludable (Figura 6).
El análisis de color para determinar el estado de salud de los protocormos, la comparación del desempeño de los calificadores evidenció que el clasificador LDA con función cuadrática es el más adecuado produciendo el menor error estadístico (Tabla 1). El clasificador LDA discrimina las dos clases de colores: tonalidad–sana y tonalidad–oxidada, en función de las componente a y b, al analizar una imagen del protocormo (Figura 7). Con este procedimiento se siguió el desarrollo de protocormos de O. sphacelatum durante 21 días, minimizando el esfuerzo en términos de horas de trabajo y desgaste físico. Esta metodología puede ser útil para identificar diferencias entre tratamiento o entre individuos, en términos de crecimiento, desarrollo y estado de oxidación o vitrificación de los protocormos.
En la Tabla 2, se muestran los valores de A y FR para la imagen procesada de la Figura 6, así como los porcentajes para cada clase de tonalidad obtenida al aplicar el clasificador LDA con función cuadrática como criterio de segregación.
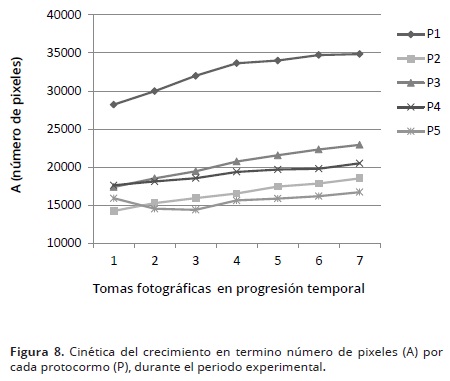
Dando seguimiento temporal al crecimiento de cada protocormos y analizando la variación temporal de A y FR durante el desarrollo de los protocormos, permite evidenciar los cambios en la tasa de crecimiento de los mismos (Figuras 8 y 9).
En la aplicación de esta metodología se debe hacer una buena elección de los protocormos para evaluar su desarrollo. Eso se debe a las variaciones entre individuos en los tiempos de germinación de sus semillas y desarrollo de los protocormos resultantes. Por ejemplo, en las Figuras 8, el protocormo 1 presenta un valor de A (28,761 pixeles) superior con respecto a los demás (entre 15,000 y 16,000) desde la toma inicial, pues su tiempo de germinación fue más rápido; en la Figura 9, el protocormo 3, empieza con un valor del FR (0.81) superior que los demás, por cuestiones que no desarrollo rápidamente primordios foliares. No obstante, esta situación no representa una limitante: un conjunto de semilla tiene una cierta variabilidad genética a pesar que pertenezcan a la misma especie, lo cual produce comportamientos distintos de las semilla en relaciones a las mismas condiciones ambientales, influenciando así el crecimiento y el desarrollo de cada protocormo. Por lo tanto estadísticamente hay que considerar un adecuado número de repeticiones para extrapolar información congruente con la realidad. Además, FR es un parámetro cuyo empleo reduce el juicio subjetivo de un observador que trata de determinar la etapa de desarrollo de cada semilla. Por otro lado, el registro del crecimiento de los protocormos a través de A permite hacer una evaluación de la tasa de crecimiento de éstos sin recurrir a pruebas destructivas. Esto resulta ser una gran ventaja considerando que el número de protocormos suele ser reducido y se encuentran creciendo en condiciones asépticas; de otra manera, la toma de datos métricos implicaría extraer el protocormo del medio de cultivo.
Conclusiones
- El empleo del clasificador de color LDA (Linear Discriminant Analysis) de cada protocormo resultó ser un parámetro que permite determinar el estado de salud del mismo.
- La aplicación de la metodología aquí propuesta permitirá obtener datos objetivos, de forma rápida y fácil sobre la germinación en orquídeas, para mejorar el desempeño y disminuir los costos asociados con la mano de obra relativos al tiempo de análisis manual.
Agradecimientos
Se agradece la Biol. Mariana Yadira López Chávez por la toma de la imagines digitales y al Lic. en Informática Luis Francisco Barbosa por la asesoría técnica.
Referencias
AOSA (Association of Official Seed Analysis). (1983). Seed Vigor Testing Handbook. Contribution No. 32 to the Handbook on Seed Testing. Association of Official Seed Analysis, Springfield, IL, USA. 93 p. [ Links ]
AOSA (Association of Official Seed Analysis). (2000). Rules for testing seeds. Association of Official Seed Analysis, Springfield, IL, USA. 106 p. [ Links ]
Arditti. J. & Abdul–Ghani. A. K. (2000). Transley Review No. 110. Numerical and physical properties of orchid seeds and their biological implications. New Phytol, 145(1), 367 – 421. doi: 10.1046/j.1469–8137.2000.00587.x [ Links ]
Dell' Aquila. A. (2006a). Computerized seed imaging: a new tool to evaluate germination quality. Commun Biometry Crop Sci, 1(1), 20–31. [ Links ]
Dell' Aquila. A. (2009b). Digital imaging information technology applied to seed germination testing. A review. Ital J Agron, 29(1), 213–221. doi: 10.1051/agro:2008039. [ Links ]
Ducournau. S. A. Feutry. P. Plainchault. P. Revollon. B. Vigouroux. & M.H. Wagner. (2005). Using computer vision to monitor germination time course of sunflower (Heliantus annuus L.) seeds. Seed Sci & Technol, 33, 329–340. doi: 10.15258/sst.2005.33.2.06.
Hadley. G. (1997). Orchid mycorrhiza. En: Arditti, J. y Pridgeon, A. M. (Eds.). Orchid Biology: Reviews and Perspectives vol. 2. Cornell University Press. Ithaca, Nueva York. 83 – 118 p. [ Links ]
ISTA (International Seed Testing Association). (1999). International Rules for Seed Testing Rules. Seed Sci. & Technol, 27 (Supp), 27–31. [ Links ]
Knudson. L. (1922a). No symbiotic germination of orchid seeds. Bot Gaz, 78(1), 1 – 25. [ Links ]
Knudson. L. (1946b). A new nutrient solution for orchid seed germination. American Orchid Society Bulletin, 15, 214 – 217. [ Links ]
Knudson. L. (1951c). Nutrient solutions for orchids. Bot Gaz, 112(4)528 – 532. [ Links ]
Seaton. P. T. & Ramsay. M. (2005). Growing orchid seed. Royal Botanic Gardens Kew, London. 128 p. [ Links ]
Sharma. R. N. Sharma. B. & De. K. K. (2004). Micropropagation of Dendrobium fimbriatum Hook, an endangered orchid of Sikkim Himalaya. J Hill Res, 17(2), 65–67. [ Links ]
Suárez. J.P. Weib. M. Abele. A. Garnica. S. Oberwinkler. F. & Kottke. I. (2006). Diverse tulasnelloid fungi form mycorrhizas with epiphytic orchids in an Andean cloud forest. Mycol Res, 110, 1257–1270. doi: 10.1016/j.mycres.2006.08.004. [ Links ]
Urena. R. F. Rodriguez. & M. Berenguel. (2001). A machine vision system for seeds germination quality evaluation using fuzzy logic. Computer and Electronics in Agriculture, 32(1), 1–20. doi: 10.1016/S0168–1699(01)00150–8. [ Links ]
Valadares. R.B.S. Perotto. S. Santos. E.C. & Lambais. M.R. (2013). Proteome changes in Oncidium sphacelatum (Orchidaceae), at different trophic stages of symbiotic germination. Mycorrhiza, 24(5), 349– 360. doi: 10.1007/s00572–013–0547–2. [ Links ]
Verdugo. G. Marchant. J. Cisternas. M. Calderón. X. Peñaloza. P. (2007). Caracterización morfométrica de la germinación de Chloraea crispa Lindl. (Orchidaceae) usando análisis de imágenes. Gayana Bot, 64 (2), 232–238. doi :10.4067/S0717–66432007000200008. [ Links ]
Zettler. L. W. & McInnis. T. M. (1994). Light enhancement of symbiotic seed germination and development of an endangered terrestrial orchid (Platanthera integrilabia). Plant Sci, 102(2) ,133 – 138. doi: 10.1016/0168–9452(94)90030–2. [ Links ]