Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.65 no.3 Palmira July/Sep. 2016
https://doi.org/10.15446/acag.v65n3.45661
http://dx.doi.org/10.15446/acag.v65n3.45661
Estructura genética y caracterización molecular del cerdo criollo (Sus scrofa domestica) de Ecuador, utilizando marcadores microsatélites
Molecular characterization and genetic structure of the local criollo pig breeds (Sus scrofa domestica) from Ecuador, using microsatellite markers
Julio César Vargas Burgos 1, Francisco Jesús Velázquez Rodriguez 1* y Edilberto Chacón Marcheco 2
1Universidad Estatal Amazónica, Pastaza, Ecuador. 2 Universidad Técnica de Cotopaxi – La Maná, Ecuador. *Autor para correspondencia: franciscovelazquezrodriquez@gmail.com
Rec.: 23.09.2014 Acep.: 14.11.2014
Resumen
Los marcadores moleculares han mostrado su gran utilidad en la caracterización de los animales domésticos, por ello, el objetivo de este trabajo fue caracterizar genéticamente al cerdo criollo de Ecuador mediante el uso de microsatélites. Se recolectaron muestras de pelo de 15 animales. Se utilizó un panel de 25 microsatélites, la amplificación de los mismos se realizó mediante la reacción en cadena de la polimerasa y los fragmentos amplificados, separados por electroforesis en geles de poliacrilamida. Se calcularon el número medio de alelos por locus (MNA), las frecuencias alélicas, las heterocigosidades esperada (He) y observada (Ho), el contenido de información polimórfica (PIC), así como el equilibrio Hardy–Weinberg (EHW) y el FIS por marcador. El número medio de alelos encontrado por locus ha sido de 6.2 y el porcentaje de individuos heterocigotos se comportó por encima de del 60%. De la totalidad de los loci estudiados el 68 % mostró un PIC elevado. Estos resultados muestran que el cerdo Criollo ecuatoriano posee una elevada diversidad genética, información esencial para optimizar las estrategias nacionales de conservación y mejora de este genotipo en el Ecuador.
Palabras clave: Diversidad genética, frecuencias alélicas, heterocigosidad esperada, heterocigosidad observada, contenido de información polimórfica.
Abstract
The molecular markers have shown their great utility in the characterization of the domestic animals, hence, the objective of this work was to characterize, genetically, the Creole pig of Ecuador by means of microsatellites. Samples of hair of 15 animals were gathered. A panel of 25 microsatellites, the amplification of the same was carried out by means of the reaction in chain of the polymerase and the amplified fragments, separated by electrophoresis in gels of polyacrylamide. In addition, were calculated the half number of alleles by locus (MNA), the allelic frequencies, the expected (He) and the observed (Ho) heterozygosity , the content of polymorphic information (PIC), as well as the balance Hardy–Weinberg (EHW) and the FIS for marker. The half number of alleles found by locus has been of 6.2 and the percentage of individual heterozygote behaved above of 60%. Of the entirety of the studied loci the 68% showed a high PIC. These results show that the Ecuadorian Creole pig possesses a high genetic diversity, essential information to optimize the national strategies of conservation and it improvement of this genotype in Ecuador.
Keywords: Genetic diversity, allelic frequencies, expected heterozygosity, observed heterozygosity, content of polymorphic information.
Introducción
Los cerdos criollos han desarrollado un papel socioeconómico muy importante, principalmente en el medio rural. El conocimiento científico con respecto a estos animales es bajo; sin embargo, se deben hacer esfuerzos que permitan conservar este valioso recurso genético. Las características de rusticidad y probable resistencia a enfermedades, su diversidad en la alimentación y su poca exigencia en el manejo lo hacen una alternativa en los sistemas de producción sustentable. El manejo efectivo de los recursos genéticos animales requiere conocimiento comprensivo de las características de raza, incluso los datos en el tamaño de la población y estructura, distribución geográfica, el ambiente de la producción, y la diversidad genética dentro y entre razas (FAO, 2012).
Entre todos los tipos de marcadores moleculares, los microsatélite son los más usados para estudios de la diversidad genética y estructura de la población de ganado (Jordana et al., 2003; Yang et al., 2003; Martínez et al., 2006; Peter et al., 2007).
Por tal razón la utilización de marcadores microsatélites constituye la herramienta fundamental de este trabajo, con el objetivo de conocer la diversidad genética de las poblaciones del cerdo criollo de Ecuador, debido a que es una especie poco conocida, analizada y valorada, además de desplazada en el territorio nacional ecuatoriano.
Materiales y métodos
Muestreo y extracción de ADN
La investigación se realizó en las provincias Los Ríos, Cotopaxi y Pichincha, Ecuador. Se recolectaron muestras de pelo de 15 animales de cerdo Criollo de Ecuador y se sometieron a los análisis respectivos.
Al igual que en otros estudios de diversidad genética de razas criollas latinoamericanas (Oslinger et al., 2006), se trabajó con un tamaño de muestra que pudiera considerarse pequeño debido a dificultades en la obtención de las muestras por el reducido número de animales, reflejo del estado de peligro de extinción en que se encuentra la raza y la localización de las poblaciones en zonas de difícil acceso en la costa y en la sierra Ecuatoriana.
Las muestras de pelo fueron recogidas en sobres de papel identificados con los datos de cada animal y mantenidas a temperatura ambiente hasta su envío al laboratorio.
Los trabajos de laboratorio se realizaron en el Laboratorio de Genética Molecular Aplicada de la empresa Animal Breeding Consulting S.L. (ABC) de la Universidad de Córdoba, España.
El ADN se extrajo de muestras de pelo conforme a los lineamientos del siguiente método:
Material Empleado (TE: Tris–HCl 10 mM, EDTA 1 mM pH=8; Tampón K: 0,372 g de KCl, 0,051g de MgCl2, 1 ml de Tris–HCl 1 M (pH=8,5), 0,5 ml de Tween 20 y 98 ml de H2O); Se añadieron 100 ug/ml de Proteinasa K justo en el momento en que se va a utilizar; Muestra de pelo con raíz.
Método (Se lavaron de 3 a 5 pelos con raíz de cada animal con agua bidestilada y después con etanol 100%; hasta secarse; se cortaron las raíces de los pelos con unas tijeras esterilizadas con alcohol e introducirlos en microtubos; posteriormente se añadieron 100 µl de tampón K; se incubó a 56 °C durante 45 minutos; se elevó la temperatura a 95 °C y se incubó nuevamente durante 10 minutos y finalmente se conservó el ADN extraído a –20 °C hasta su uso).
Microsatélites caracterizados
Se analizaron mediante PCR 25 microsatélites (Tabla 1), 19 de ellos recomendados por el comité de expertos de la FAO/ISAG (Food and Agriculture Organization of the United Nations/International Society of Animal Genetics) para estudios de diversidad genética en la especie porcina (FAO, 2012) y otros con probada utilidad en estudios de diversidad genética al estudiar poblaciones de cerdos de Cuba, España y la India (Martínez et al., 2006 y Kumar et al., 2013). Los fragmentos obtenidos mediante la PCR fueron sometido a una electroforesis en gel de poliacrilamida en un secuenciador automático ABI Prism 377XL (Applied Biosystems, Foster City, CA, USA). El análisis de los fragmentos y el genotipado se realizó mediante los programas informáticos Genescan Analysis® 3.1.2 y Genotyper® 2.5, respectivamente.
Análisis de la diversidad genética intraracial
El cálculo del número medio de alelos por locus (MNA), las frecuencias alélicas, las heterocigosis esperada (He) y observada (Ho) y el contenido de información polimórfica (PIC) con el software microsatellite toolkit ®.
Los valores de FIS (coeficiente de consanguinidad) con un intervalo de confianza del 95% se calcularon con el programa informático GENETIX v. 4.05 (Belkhir et al., 2004).
Para determinar el equilibrio Hardy–Weinberg (EHW) por marcador se desarolló un test exacto de Fisher, usando el método en cadena de Monte Carlo–Markov (Guo y Thompson, 1992), los cálculos se realizaron mediante el software Genepop versión 3.4®.
Resultados y discusión
Las frecuencias alélicas, expresadas en porcentajes de los 25 microsatélites en la población de cerdos criollos de Ecuador se muestra en la Tabla 2, resultando polimórficos todos los marcadores estudiados, con valores por locus que oscilan entre un mínimo de 2 alelos (SW951) y 14 (S0005). Se observa en general una moderada diversidad alélica.
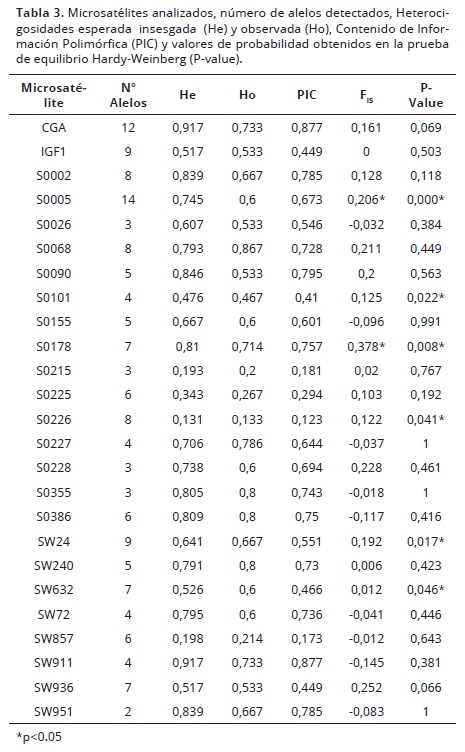
La Tabla 3, recoge los valores de heterocigosidad esperada y Heterocigosidad observada Contenido de Información Polimórfica y los valores de probabilidad obtenidos en la prueba de equilibrio Hardy–Weinberg (P–value), por marcador analizado. Los valores de heterocigosidad esperada (He) por marcador analizado, varían entre un mínimo de 0.131 para el marcador S0226 y un máximo de 0,917 para (CGA y SW911). Por su parte la heterocigosidad observada (Ho) se comportó con valores entre 0,133 para el marcador S0226 y 0,867 para el S0068. Los marcadores S0178, CGA, SW911, S0227, S0355, S0386, SW240 y S0068 muestran mayor polimorfismo, con valores de heterocigosidad observada superiores al 70%.
Por los valores Contenido de Información Polimórfica obtenidos, todos los microsatélites son medianamente o muy informativos exceptuando cuatro microsatélites (S0215, S0225, S0226 y S0857) que son poco informativos en esta población.
Uno de los primeros pasos en el estudio de la estructura de una población es la detección de desviaciones de las proporciones de Hardy–Weinberg, reveladoras de la ocurrencia de procesos de selección, migración, apareamientos no aleatorios, etc. Los valores de probabilidad obtenidos en la prueba de equilibrio Hardy–Weinberg para todas las combinaciones locus/población, revelan que para 6 marcadores (S0005, S0101, S0178, S0226, SW24 y SW632) el P–value es inferior a 0.05, indicando que estos microsatélites se encuentran desequilibrados en esta población.
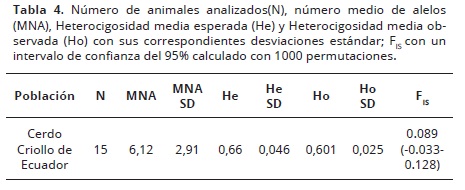
En la Tabla 4, se recogen los valores del número medio de alelos y la heterocigosidad media esperada (He) y heterocigosidad media por recuento directo (Ho) en esta población. El número medio de alelos fue de (6.2) y el porcentaje de individuos heterocigotos osciló entre 60.1% para la heterocigosidad media observada y 65.8% para la heterocigosidad media esperada.
El valor de FIS (0,089) no es significativo, lo que es indicativo de que la población no muestra una desviación significativa del HWE.
Este estudio definió la estructura genética del cerdo criollo ecuatoriano analizando 25 marcadores microsatélite. El conocimiento de la variabilidad genética servirá para la implementación de programas de conservación y posterior mejora de la raza.
La posibilidad de utilizar un importante número de microsatélites recomendados por la FAO para este tipo de estudios (FAO, 2012), permite plantear que los resultados encontrados en las poblaciones aquí estudiadas son fiables y consistentes para el cerdo Criollo de Ecuador.
El número medio de alelos por población, y la heterocigosidad observada y esperada, son los parámetros más usados en la evaluación de la diversidad intrarracial (FAO, 2012).
Toro et al. (2008), propuso una alternativa basada en la idea de que entre más mayor sea el número de alelos, mayor será la diversidad potencial de una raza.
Los cerdos criollos ecuatorianos exhibieron niveles altos de diversidad genética, con un número medio de alelos (6,12), superior a los reportados por Kim et al. (2005) en cerdos nativos de Korea (3,44), igualmente supera a los valores encontrados por Rodrigáñez et al. (2008) en las razas ibéricas Guadyerbas (4,5) y Torbiscal (3,7), Pardo et al. (2014) en el cerdo doméstico de Momil, Córdoba, Colombia (5.2). Muy próximos a los referidos por Meléndez et al. (2014) en una población de cerdos en Cereté, Colombia (6,7).
La heterocigosidad media observada (0,601) en cerdos criollos ecuatorianos supera a los descritos por Oslinger et al. (2006), en cerdos criollos colombianos (0.201); Rodrigáñez et al. (2008) en la razas porcinas autóctonas españolas Guadyerbas (0,439) y Torbiscal (0,579); Swart et al. (2010), en cerdos de Namibia (0,531) y por Pardo et al. (2014) y Meléndez et al. (2014) en poblaciones de cerdos en Momil y Cereté, Colombia (0,522 y 0,547), respectivamente.
A su vez están dentro del rango de diversidad porcina informada en anteriores estudios por: Yang et al. (2003), en razas de China; Martínez et al. (2006), en el cerdo Criollo Cubano; en el cerdo pelón mexicano; Wang et al. (2011), en razas de China y Kumar et al. (2013), en razas de la India.
Basados en los análisis de cerdos criollos mexicano y brasileños Sollero et al. (2009), sugieren que valores altos de heterocigosidad pudieran ser explicados por la baja presión de selección a que han estado sometidas estas poblaciones locales y la falta de programas de cruzamientos, lo que también ocurre con el cerdo criollo ecuatoriano.
Los valores de las heterocigosidades y del PIC para los 25 marcadores tienen comportamientos bastante similares, coincidiendo con Vaiman et al. (1994), quienes plantearon la existencia de una relación directa entre el PIC y la Heterocigosidad, en el sentido de que cuando aumenta uno también lo hace el otro y que al encontrarse cercanos, indican a priori que se realizó un adecuado muestreo y confirman la calidad de los marcadores seleccionados para el estudio de la diversidad genética de esta raza.
De los 25 microsatélites analizados, 17 presentaron un PIC superior a 0.5%, lo que permite optimizar esta batería para futuros estudios dentro de los programas de conservación de las poblaciones de cerdos criollos de Ecuador. El hecho de que la población evaluada no mostrara una desviación significativa del HWE basados en el valor que toma el FIS, es indicativo de una adecuada proporción de individuos heterocigotos y de un bajo nivel de endogamia.
Contrario al resultado obtenido en este trabajo, autores como Kim et al. (2005), al estudiar la estructura genética de razas porcinas de Korea y China encontraron varios casos donde se relaciona la desviación del equilibrio HW con los valores positivos tomados por el FIS, indicativo de un déficit de heterocigosis en esas poblaciones.
Conclusiones
- La caracterización genética del cerdo Criollo ecuatoriano reveló que la raza posee una elevada diversidad genética, medida a partir del promedio de alelos y los valores de heterocigosidad encontrados. Estos resultados constituyen una importante contribución para la implementación de programas nacionales de conservación y mejora de este genotipo en el Ecuador.
Agradecimientos
Los autores agradecen la colaboración de los funcionarios del Ministerio de Agricultura y criadores del cerdo Criollo de (Quevedo, La Maná, Zumbahua y Mejías) de la República del Ecuador.
Referencias
Belkhir. K. Borsa. P. Chikhi. L. Raufaste. N. & Bonhomme. F. (2004). GENETIX 4.05, logiciel sous Windows TM pour la genetique des populations. Laboratoire Genome, Populations, Interactions, CNRS UMR 5000, Universite de Montpellier II, Montpellier (France). pp 1996–2004. [ Links ]
FAO. (2012). Status and Trends of Animal Genetic Resources–2012. In: Information Document, Rome. http://www.fao.org/docrep/meeting/027/mg046e.pdf. 17.10.2014. [ Links ]
González. S. A.A. Clemente– Lemus. F.C. Mejía. M. K. Rodríguez. C. J.G. Orozco. B. M.G. Barreras. S.A. (2011). Diversidad genética en cerdos criollos mexicanos con genes candidatos asociados a características productivas. Pesq Agropec Bras, 46(1), 44–50. doi: 10.1590/S0100–204X2011000100006. [ Links ]
Guo. S. W. & Thompson. E. A. (1992). Performing the exact test of Hardy–Weinberg proportions for multiple alleles. Biometrics, 48(2), 361–372. doi: 10.2307/2532296. [ Links ]
Jordana. J. Alexandrino. P. Beja–Pereira. A. Bessa. I. Canon. J. Carretero. Y. Dunner. S. Laloe. D. Moazami–Goudarz. K. Sanchez. A. & Ferrand. N. (2003). Genetic structure of eighteen local south European beef cattle breeds by comparative F–statistics analysis. J Anim Breed Genet, 120(2), 73–87. doi: 10.1046/j.1439–0388.2003.00384.x. [ Links ]
Kim. T. H. Kim. K. S. Choi. B. H. Yoon. D. H. Jang. G. W. Lee. K. T. Chung. H. Y. Lee. H. Y. Park. H. S. & Lee. J. W. (2005). Genetic structure of pig breeds from Korea and China using microsatellite loci analysis. J Anim Sci, 83(10), 2255–2263. [ Links ]
Kumar. D. A. Jeyakumar. S. Kundu. A. Kundu. M.S. Sunder. Jai. & Ramachandran. M. (2013). Genetic characterization of Andaman Desi pig, an indigenous pig germplasm of Andaman and Nicobar group of islands, India by microsatellite markers. Vet World, 6(10), 750–753. doi: 10.14202/vetworld.2013.750–753. [ Links ]
Martínez. A. M. Acosta. J. Vega–Pla. J. L. & Delgado. J. V. (2006). Analysis of the genetic structure of the canary goat populations using microsatellites. Livest Sci, 102(1–2), 140–145. doi: 10.1016/j.livsci.2005.12.002. [ Links ]
Meléndez. G. I. Pardo. P. E. & Cavadia. M. T. (2014). Genetic characterization of the domestic pig (Sus scrofa domestica) in Cerete–Colombia, using microsatellite markers. Rev. MVZ. Córdoba, 19(2), 4150–4157. [ Links ]
Oslinger. A. Muñoz. J. E. Álvarez. L. A. Ariza. F. Moreno. F. & Posso. A. (2006). Caracterización de cerdos criollos colombianos mediante la técnica molecular RAMs. Acta Agron, 55(4) ,45–50. [ Links ]
Pardo. E. Cavadía. T. & Meléndez. I. (2014). Microsatellite Characterization of the Momil Cordoba (Colombia) Domestic Pig. Arch. Zootec, 63(241), 215–218. doi: 10.4321/S0004–05922014000100024. [ Links ]
Peter. C. Burford. M. Pérez. T. Dalamitra. S. & Erhardt. G. (2007). Genetic diversity and subdivision of 57 European and Middle–Eastern sheep breeds. J Anim Breed Genet, 38(1), 37 – 44. doi: 10.1111/j.1365–2052.2007.01561.x. [ Links ]
Rodrigáñez. J. Barragán. C. Alves. E. Gortázar. C. Toro. M. A. & Silió. L. (2008). Genetic diversity and allelic richness in Spanish wild and domestic pig population estimated from microsatellite markers. Span J Agric Res, 6(Sp. Iss.),107–115. [ Links ]
Sollero. B. P. Paiva. S. R. Faria. D. A. Guimarães. S. E. F. Castro. S. T. R. Egito. A. A. Albuquerque. M. S. M. Piovezan. U. Bertani. G. R. & Mariante. da S. A. (2009). Genetic diversity of Brazilian pig breeds evidenced by microsatellite markers. Livest Sci, 123(1), 8–15. doi: 10.1016/j.livsci.2008.09.025. [ Links ]
Swart. H. Kotze. A. Olivier. P. A. S. & Grobler. J. P. (2010). Microsatellite–based characterization of Southern African domestic pigs (Sus scrofa domestica).S Afr J Anim Sci, 40(2), 121–132. doi:10.4314/sajas.v40i2.57280. [ Links ]
Toro. M. A. Fernández. J. & Caballero. A. (2009). Molecular characterization of breeds and its use in conservation. Livest Sci, 120(3), 174–195. doi: 10.1016/j.livsci.2008.07.003. [ Links ]
Vaiman. D. Mercier. D. Moazami–Goudarzi. K. Eggen. A. Ciampolini. R. Lepingle. A. Velmala. R. Kaukinen. J. Varvio. S. L. & Martin. P. (1994). A set of 99 cattle microsatellites: characterization, synteny mapping, and polymorphism. Mamm. Genome, 5(5), 288–297. doi: 10.1007/BF00389543. [ Links ]
Wang. J. Y. Guo. J. F. Zhang. Q. Hu. H. M. Lin. H. C. Cheng. W. Yin. Z. & Wu. Y. (2011). Genetic Diversity of Chinese Indigenous Pig Breeds in Shandong Province Using Microsatellite Markers. Asian–Aust J Anim Sci, 24(1), 28–36. doi: 10.5713/ajas.2011.10091. [ Links ]
Yang. S. Wang. Z. Liu. B. Zhang. G. Zaho. S. Yu. M. Fan. B. Li. M. Xiong. T. & Li. K. (2003). Genetic variation and relationships of eighteen Chinese indigenous pig breeds. Genet Sel Evol, 35(6), 657–671. doi: 10.1051/gse:2003045. [ Links ]