Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Acta Agronómica
Print version ISSN 0120-2812
Acta Agron. vol.65 no.4 Palmira Oct./Dec. 2016
https://doi.org/10.15446/acag.v65n4.51714
http://dx.doi.org/10.15446/acag.v65n4.51714
Caracterización molecular, análisis morfológico y colonización micorrízica en la rizósfera del aguacate (Persea americana Mill) en Caldas, Colombia
Molecular characterization, morphological analysis and mycorrhizal colonization in the avocado (Persea americana Mill) rhizosphere in Caldas, Colombia
Fredy Arvey Rivera Páez*, Viviana González Salazar, Jorge Giovany González Acosta y Paula Andrea Ossa López
Grupo de Investigación GEBIOME. Departamento de Ciencias Biológicas, Universidad de Caldas. Manizales, Caldas –Colombia. *Autor para correspondencia: fredy.rivera@ucaldas.edu.co
Rec.:05.07.2015 Acep.: 09.09.2015
Resumen
Se evaluaron familias de hongos formadores de micorriza arbuscular (HMA) asociados a dos variedades de aguacate Persea americana Mill (hass y lorena), en 12 muestras de suelo rizosférico y raíces en Caldas, Colombia. Las esporas se obtuvieron por tamizado húmedo y centrifugación en gradiente de sacarosa, y montadas en los reactivos Polivinilactoglicerol y Melzer para determinar su morfología, y las raíces tratadas con hidróxido de potasio, ácido acético, azul de tripano y lactoglicerol, para establecer su colonización. El ADN fue extraído por DNeasy Plant Mini Kit (Qiagen™) y amplificado por PCR utilizando iniciadores sobre los genes 18S y 28S del ADNr nuclear, visualizado y purificado en geles de agarosa, y secuenciado por Macrogen Advancing Through Genomics–Corea del Sur. Los resultados muestran presencia de 12 morfotipos pertenecientes a las familias Acaulosporaceae, Gigasporaceae, Diversisporaceae y Glomeraceae, y una colonización micorrízica superior al 90% en ambas variedades de aguacate. Se logró aislar ADN de nueve morfotipos y amplificar siete. El alineamiento de las secuencias de ADN se realizó en el programa ClustalW incluido en el programa Mega 6, junto con secuencias depositadas en el GenBank, utilizando el parámetro de distancia Kimura 2 (K2P), permitieron construir un árbol de similitud por el método Neighbor–Joining (NJ) agrupando las muestras determinadas por morfología en las familias correspondientes. Estos resultados permitieron estandarizar protocolos para familias de este grupo, donde su taxonomía es dificultosa y controversial, registrando familias citadas en la literatura como de elevada importancia para la protección e incorporación de nutrientes en la planta.
Palabras clave: Alineamiento de secuencias de ADN, genes ribosomales, morfotipos HMA, simbiosis, taxonomía.
Abstract
Families of arbuscular mycorrhizal fungi (AMF) associated to two varieties of avocado Persea americana Mill (hass and lorena), in 12 samples of rhizosphere soil and roots in Caldas, Colombia were evaluated. Spores were obtained by wet sieving and sucrose gradient centrifugation, and mounted on polyvinyl alcohol using glycerol and Melzer's reagents, to determine their morphology, and roots treated con potassium hydroxide, acetic acid, Trypan Blue and lactoglycerol, to establish colonization. The DNA was extracted by DNeasy Plant Mini Kit (Qiagen™) and amplified by PCR using primers for genes 18S and 28S nuclear rDNA, visualized and purified on agarose gels and sequenced by Macrogen Advancing throughout Genomics–South Korea. The results show presence of 12 morphotypes belonging to the families Acaulosporaceae, Gigasporaceae, Diversisporaceae and Glomeraceae, and mycorrhizal colonization higher 90% in both varieties of avocado. It was possible to isolate DNA of nine morphotypes and amplify seven. Alignment of DNA sequences carried out in the ClustaW software included in the program Mega 6, together with sequences deposited in GenBank, using the distance parameter Kimura 2 (K2P), they allowed to build a tree of similarity for the method Neighbor–Joining (NJ) clustering the samples determined by morphology in the corresponding taxonomical families. These results allowed to standardize protocols for families in this group, where his taxonomy is difficult and controversial, registering families cited in the literature as highly important for the protection and incorporation of nutrients into the plant.
Keywords: AMF morphotypes, DNA sequence alignment, ribosomal genes, symbiosis, taxonomy.
Introducción
Colombia ocupa el quinto lugar a nivel mundial en producción de aguacate. Las estrategias para aumentar la producción de este cultivo contemplan programas de mejoramiento genético, así como la implementación de biofertilizantes que mejoran la disponibilidad de nutrientes, disminuyen los costos de producción, protegen el medio ambiente y mantienen la conservación del suelo desde el punto de vista de fertilidad y biodiversidad.
Dentro de los organismos empleados como biofertilizantes se encuentran los hongos formadores de micorriza arbuscular (HMA). Un grupo de hongos simbiontes pertenecientes al Phylum Glomeromycota, los cuales realizan asociación con la mayoría de especies vegetales presentes, derivando su nombre de la formación de unas estructuras ramificadas denominadas arbúsculos, ubicados en el interior de la células corticales de las raíces de las plantas, donde se lleva a cabo el intercambio de nutrientes (Schüßler et al. 2001). Es la simbiosis fúngica más extendida en las plantas, con cerca de 240000 especies capaces de establecer este tipo de asociación con más de 6000 especies de hongos (Barea, 1998). La absorción y translocación de agua y nutrientes poco móviles como fosfato, amonio y micronutrientes, es una de las más sobresalientes adaptaciones de la raíz para desenvolverse adecuadamente en el ambiente edáfico, aumentando el área superficial efectiva de absorción.
Los HMA intervienen en el crecimiento y establecimiento exitoso de especies cultivables, ayudan en el control biológico de hongos patógenos y nematodos, inmovilizan metales pesados, proveen resistencia a diferentes condiciones de estrés y en conjunción con otros microorganismos edáficos contribuyen a la formación de agregados estables; necesarios para mantener la estructura y calidad del suelo, siendo útiles en la restauración de sitios degradados con un efecto positivo en la creación de comunidades de plantas (Peterson et al. 2004).
Tradicionalmente la clasificación taxonómica de este grupo de organismos se ha realizado con base a la morfología de sus esporas y en ausencia de ellas, la estructura intraradical permite la identificación a nivel de familia. Sin embargo, la determinación de especies por morfología es complicada y es así, que recientemente se han implementado técnicas moleculares a partir del análisis de secuencias de ADN, las cuales han apoyado la sistemática de estos organismos, que pasaron de estar inicialmente incluidos en un solo género hasta llegar a nivel de orden. La última clasificación comprende alrededor de 248 especies distribuidas en 18 géneros, 11 familias y 4 órdenes (Schüßler & Walker, 2010).
En Colombia, el estudio de estos organismos ha sido escaso, las investigaciones han estado centradas a unas cuantas especies productivas dentro de las hortalizas, frutales, cultivos tradicionales (maíz, sorgo, soya y caña) y algunas especies forestales, por lo que los inventarios de HMA son pocos y el conocimiento de la diversidad biológica es aún muy limitado. En este contexto el objetivo de la presente investigación es contribuir al conocimiento de las familias de HMA asociados a dos variedades de Persea americana en el departamento de Caldas, Colombia mediante el uso de análisis morfológicos y herramientas moleculares, que puedan ser utilizados en futuras investigaciones de carácter productivo y de simbiosis específicas.
Materiales y Métodos
Zona y material de estudio
La zona de estudio comprendió un cultivo perteneciente a la variedad Hass con una edad de tres años y medio (Finca "el Chagualo", Vereda San Rafael, 1930 m.s.n.m. 75° 30' 33.45'' N, 5° 12' 3.77'' W, Aranzazu – Caldas, Colombia) y un cultivo perteneciente a la variedad Lorena con una edad de seis años (Finca "Aquí no es", Vereda San Pedro, 1610 m.s.n.m. 75° 47' 50.37'' N, 5° 14' 12.15'' W, Anserma – Caldas, Colombia). Las zonas de estudio se encuentran ubicadas dentro de la zona de vida bmh–PM (Bosque muy húmedo–premontano). El patrón nativo del portainjerto para la variedad Hass, corresponde a una variedad de aguacate de origen Californiano y Guatemalteco, traído en el año de 1988 por la Federación Nacional de Cafeteros de Colombia, mientras que para la variedad Lorena, el portainjerto corresponde a un Patrón Criollo.
Se evaluaron 12 muestras de suelo rizosférico y raíces de aguacate colectadas en ambos municipios, seis pertenecientes a cada zona de estudio en época de sequía. El muestreo de suelo se llevó a cabo seleccionando simultáneamente raíces finas para su posterior tinción y evaluación de la simbiosis.
Análisis morfológico y colonización micorrízica
Las muestras se trasladaron al laboratorio de Genética de la Universidad de Caldas y cada una fue procesada para aislar las esporas nativas presentes mediante el tamizado húmedo y centrifugación en gradiente de sacarosa. Una parte de la muestra se envió para el análisis fisicoquímico al laboratorio de química y fertilidad de suelos en la Universidad de Caldas, Colombia.
Los morfotipos fueron separados con la ayuda de microscopio de luz, a partir del reconocimiento de parámetros morfológicos usados en la taxonomía de este grupo como el color, forma, tamaño de las esporas, características de sus paredes (grosor, color, presencia de ornamentaciones) y conexión hifal. A cada morfotipo se le realizó montajes en los reactivos P.V.G.L (Polivinilactoglicerol) y Melzer, para observar la estructura y composición de la espora y la reacción positiva o negativa. En la evaluación de simbiosis, se realizó un conteo de esporas de la rizósfera (número de esporas.20g de suelo-1), utilizando el mismo método de aislamiento descrito anteriormente y ubicándolas en una caja de Petri para proceder a contarlas con la ayuda del microscopio de luz; realizando tres repeticiones por cada árbol de aguacate y el debido proceso de tinción de raíces. Para evaluar la colonización de los HMA al interior de las raíces. Terminado éste último proceso, se cortaron fragmentos de raíces teñidas de 1 cm de longitud y se montaron paralelamente en placas, realizando tres repeticiones por cada árbol de aguacate. El porcentaje de colonización total, por vesículas y por arbúsculos se determinó estimando el número de fragmentos colonizados sobre el número de fragmentos totales (=10 fragmentos totales por placa) y finalmente sacar un promedio. Los morfotipos y la colonización micorrízica fue fotografiada utilizando Microscopio Nikon – Modelo Eclipse 80i.
Análisis Molecular
La extracción del ADN se realizó con el DNeasy® Plant Mini Kit (Qiagen™) conforme al protocolo standard sugerido por el fabricante, a partir de un número variable de esporas, tiempo de incubación en buffer de lisis y el uso de nitrógeno líquido. La cantidad y calidad del ADN fue determinada con el espectrofotómetro – Nanovue Plus.
El ADN extraído de cada muestra fue sometido a la reacción en cadena de la polimerasa (PCR Duplex y anidados), amplificando ADNr nuclear correspondientes a regiones SSU–ITS–LSU, utilizando los iniciadores propuestos por Krüger et al. (2009) SSUmA1/LSUmA3 (F) 5'–TGGGTAATCTTTTGAAACTTYA–3' y (R) 5'–TGCTCTTACTCAAATCTATCAAA–3'; SSUmA2/LSUmA1 (F) 5'–TGGGTAATCTTRTGAAACTTCA–3' y (R) 5'–GCTCACACTCAAATCTATCAAA–3' que amplifican un fragmento de 1800 pb que sirven de molde para amplificar posteriormente un fragmento final de 1500 pb (el cual es considerado de ADN "Barcoding" en Glomeromycota), utilizando los iniciadores SSUmC3/LSUmB1 (F) 5'– TATTGCTCTTNAACGAGGAATC–3' y (R) 5'–DAACACTCGCATATATGTTAGA–3'; SSUmC1/LSUmB5 (F) 5'–TCGCTCTTCAACGAGGAAT–3' y (R) 5'–AACACTCGCATATATGCTAGA–3'.
La reacción de amplificación por PCR se realizó en microtubos de 200 µl, que contenían 4 µl de buffer 5X, 1.6 µl de la mezcla (10 mM) de dNTP's, 0.3 µl de cada primer (25 µM), 1 unidad de GoTaq Flexi DNA polimerasa (Promega), 1.2 – 2.0 µl de MgCl2 (25mM), 2 µl de ADN (aproximadamente 100 ng de ADN), y aforando a 20 µl con agua ultrapura (en la PCR anidada se tomaban 2 µl de la primera reacción de amplificación en remplazo de los 2 µl de ADN).
La amplificación de los genes mitocondriales se realizó en un termociclador (Techne – TC Plus), utilizando el perfil térmico: desnaturalización inicial a 94 °C por 3 min, 35 ciclos de desnaturalización a 94 °C por 45 s, anillamiento en un rango de (48 °C, 53 °C, 55 °C y 58 °C) por 1 min10 s y extensión a 72 °C por 1 min30s, seguido de una extensión final por 10 min a 72 °C.
Todos los productos de PCR fueron sometidos a electroforesis horizontal en gel de agarosa 2% con tampón de corrido TBE 1X pH 8.0 a 110v/50mA, coloreado con bromuro de etidio y visualizado en fotodocumentador Gel Doc–It2 310 (UVP). Los productos de la PCR fueron purificados utilizando QIAquick PCR purification y QIAquick Gel Extraction (Qiagen™), según las instrucciones del fabricante y enviados para obtener las secuencias de ADN a Macrogen Advancing Through Genomics – Corea del Sur (las secuencias obtenidas serán depositadas en el GenBank).
Análisis de datos
Se realizó un análisis de dependencia para evaluar la correlación entre el porcentaje de colonización de raíces con respecto a la concentración de fósforo en el suelo. El análisis de la calidad de las secuencias de ADN fue realizado en el software Geneious v8.14 (Drummond et al. 2009). Con las secuencias de buena calidad se realizó un BLAST con la base de datos de micorrizas MaarjAM (http://maarjam.botany.ut.ee/?action=sBlast&method=create), lo que confirmó la identidad de los aislados. Los alineamientos de las secuencias se realizaron en el programa ClustalW (Thompson et al. 1997) incluido en el sotware Mega 6 (Tamura et al. 2013). La identificación y confirmación de familias y géneros se realizó mediante la estimativa de similaridad entre las secuencias obtenidas en el estudio y las secuencias depositadas en el GenBank por Krüger et al. (2009, 2012). La divergencia de las secuencia de ADN fue estimada utilizando el parámetro de distancia Kimura 2(K2P), un árbol fue construido por el método Neighbor–Joining (NJ). Los análisis de soporte por bootstrap fueron realizadas en 1000 réplicas, el parámetro K2P fue seleccionado como modelo de distancia genética en el programa Mega 6 (Tamura et al. 2013).
Resultados y discusión
Análisis morfológico y colonización micorrízica
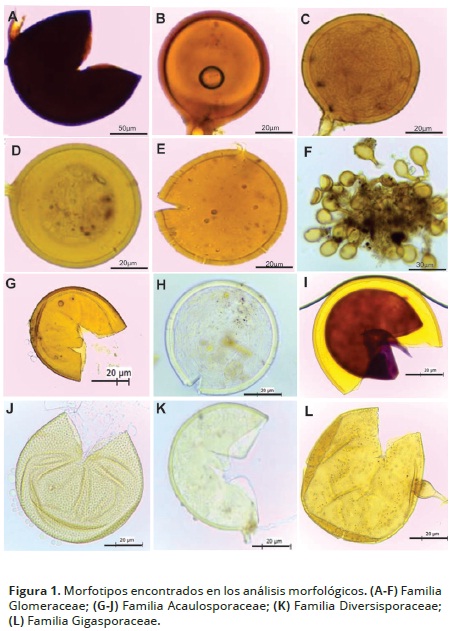
Un total de 12 morfotipos de HMA se registraron en las zonas de estudio, pertenecientes a las familias Glomeraceae, Acaulosporaceae, Gigasporaceae y Diversisporaceae (Figura 1). La familia Glomeraceae estuvo representada por seis morfotipos en los dos cultivos de aguacate, lo que es acorde con la literatura que la registra como la familia con mayor distribución geográfica y especies cosmopolitas. En cultivos de aguacate ha sido reportada en Israel, California, México y Colombia (Hass & Menge, 1990; Montañez et al. 2010). Especies de esta familia han sido utilizadas como inóculo en plántulas micropropagadas y semillas sometidas a estrés, mejorando la formación del sistema radical, incrementando significativamente el desarrollo y nutrición de las plantas y su resistencia al estrés generado por el trasplante (Azcón–Aguilar et al. 1992; Banuelos et al. 2013).
La Familia Acaulosporaceae actualmente con un único género (Acaulospora), estuvo representado por la presencia de cuatro morfotipos en ambos cultivos. Este número de morfotipos está acorde a lo registrado en la literatura, donde sus especies después de Glomeraceae son las que presentan mayor registro en todo el mundo, con una distribución muy amplia y se ha reportado como la más abundante en inventarios de cultivos de aguacate en México, y segunda familia más abundante en los llanos Orientales de Colombia (Montañez et al. 2010).
Un morfotipo de la familia Gigasporaceae fue encontrado en ambos cultivos, especies muy estudiadas en plantas forestales, y solo reportada en cultivos de aguacate en México pero con un número muy bajo de especies. En Colombia, ha sido encontrado en los Departamentos de Amazonas, Caquetá y Guaviare asociada a leguminosas, pastos y arreglos agroforestales respectivamente (Peña–Venegas et al. 2006).
La familia Diversisporaceae, presentó un único morfotipo asociado exclusivamente a la variedad lorena. La familia se distribuye a lo largo de Europa, reportada principalmente en Suiza (Oehl et al. 2009) en diferentes agroecosistemas. En América es asociada a especies forestales (Rodríguez–Morelos et al. 2014), pero no existen registros para cultivos de aguacate y tampoco para Colombia.
La distribución de los morfotipos entre los arboles de aguacate en ambos sitios fue muy similar, el cultivo de la variedad Lorena presentó el mayor número de morfotipos (12). La variedad Hass (9), en la cual no se encontró la presencia de la familia Diversisporaceae, así como un morfotipo de la familia Acaulosporaceae y otro de la familia Glomeraceae. Esto puede deberse a que la variedad lorena tiene casi el doble de la edad de Hass y coincide con los reportes de Rodríguez–Morelos et al. (2014), en especies forestales; en donde se indica que la edad influye en la diversidad de HMA, ya que la riqueza de géneros y especies de HMA fue dos veces mayor en árboles maduros que en plántulas y árboles jóvenes, o también estar relacionado con el origen del portainjerto Californiano y Guatemalteco para la variedad has y Criollo para la variedad Lorena. Es de resaltar que las variantes registradas en la literatura como altamente relevantes en la diversidad de HMA, no presentaron diferencias significativas para las dos variedades de aguacate, donde la textura del suelo en ambos cultivos fue Franco Arenosa (FA), pH entre 4.6–5.2 para la variedad Hass y 4.6–5.5 para la variedad Lorena, y materia orgánica entre 15.2 – 19.36.
La simbiosis entre HMA y P. americana, mostraron un número de esporas en los tres conteos para cada uno de los árboles en un rango entre 73 – 124 en la variedad Lorena y 121 – 265 en la variedad Hass por cada 20g de suelo, valores muy altos comparados con estudios en donde a partir de 25 g de suelo, se encuentra entre 0 y 40 esporas de HMA en cultivos de maíz y soya. Sin embargo, en condiciones controladas se pueden obtener poblaciones más abundantes (Douds et al. 1995). Los valores encontrados indican un buen establecimiento de las poblaciones de HMA, esto debido en gran parte a la acidez del suelo que influye tanto en la solubilidad del fósforo como en la abundancia de estos organismos (Porter et al.1987).
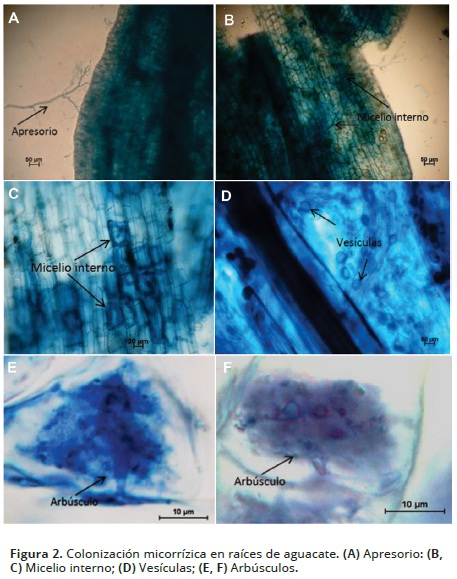
La presencia de esporas en la rizósfera indica la riqueza de especies que se pueden encontrar asociadas al aguacate, sin embargo para saber si realmente la planta está siendo colonizada por los HMA y la simbiosis se encuentra activa, es necesario evaluar las estructuras al interior de la raíz, lo cual fue comprobado al evidenciar estructuras como la formación del apresorio, la presencia de micelio interno intercelular, vesículas y arbúsculos al interior de las células corticales (Figura 2).
Todas las raíces de aguacate evaluadas presentaron colonización por estructuras de HMA, con porcentajes de colonización total por encima del 90% para ambas variedades, lo que demuestra la capacidad tan elevada que tiene esta especie para realizar este tipo de asociaciones, dada por la ocurrencia de arbúsculos y vesículas (Tabla 1). Así mismo por la presencia de hifas al interior de las células corticales de las raíces (Figuras 2B–2C).
El porcentaje de colonización fue alto, lo que se comprueba en el elevado número de vesículas y arbúsculos formados dentro de la raíz, y teniendo en cuenta que se trata de estructuras de reserva, se puede sugerir la alta dependencia del aguacate con los HMA que lo colonizan (Dorantes et al. 2012). La alta presencia de arbúsculos en ambas variedades a pesar de tener una vida media tan corta (4 a 15 días), indica lo activa que se encontraba la simbiosis al momento del muestreo. Teniendo en cuenta que la literatura registra que el principal nutriente implicado en la asociación micorrízica es el fósforo (Azcón–Aguilar et al. 1992; Barea, 1998; Oehl et al. 2009; Montañez et al. 2010), el análisis de dependencia realizado mostró que no se presenta correlación entre el porcentaje de colonización de raíces con respecto a la concentración de fósforo.
Análisis Molecular
El ADN extraído por el DNeasy® Plant Mini Kit (Qiagen™), siguiendo las instrucciones del fabricante con la cepa Glomus manihotis IVIC 476, (actualmente Rhizopagus manihotis), variando la cantidad de esporas (5, 10, 20, 40) para determinar el número mínimo con la concentración adecuada de ADN para llevar a cabo la amplificación por PCR, registraron los mejores resultados a partir de extracciones con 20 esporas.
Con el número mínimo de esporas definido, con el fin de verificar la eficiencia en el rompimiento de la pared celular y así optimizar el tiempo y calidad del proceso, los resultados de los ensayos en la maceración con Buffer AP1 y ARNasa suministrados por el kit y en nitrógeno líquido, mostraron que después de una incubación por 1 hora las esporas quedaron intactas, después de 12 horas algunas se rompen pero la mayoría quedaron enteras y después de 24 horas se logra ver la mayoría de las esporas rotas. En contraste las esporas maceradas en nitrógeno líquido se rompen de inmediato, sin embargo la más alta concentración de ADN se obtuvo a partir de un tiempo de incubación equivalente a 40 minutos. Todos los morfotipos se trabajaron con base en estos resultados, consiguiendo extraer el ADN de nueve morfotipos, con una concentración de ADN que oscilo en un rango entre 18.6 ng/µl y 73.1 ng/µl.
El tipo de amplificación que mejor funcionó fue la PCR Duplex y anidada, modificada de Römpler et al. (2006), utilizando cuatro parejas de iniciadores, aumentando el volumen de los dNTPs, MgCl2 y Taq polimerasa al doble, y una temperatura de anillamiento de 55 ° C. Con estos resultados, se procedió a realizar las amplificaciones por PCR de todos los morfotipos.
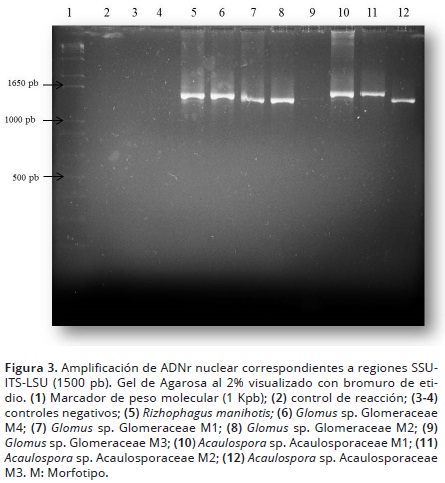
De los 12 morfotipos registrados, 9 de estos presentaron el número y condición de las esporas que permitieron realizar la extracción de su ADN. La amplificación por PCR Duplex con los iniciadores SSUmA1/LSUmA3 y SSUmA2/LSUmA1 que amplifican un fragmento de 1800 pb, y posteriormente realizando un PCR anidado y Duplex con los iniciadores SSUmC3/LSUmB1 y SSUmC1/LSUmB5 que amplifican un producto final de 1500 pb (el cual es considerado de ADN "Barcoding" en Glomeromycota), consiguió amplificar 7 morfotipos, cuatro de ellos pertenecientes a la familia Glomeraceae y los otros tres a la familia Acaulosporaceae (Figura 3). Al comparar las secuencias obtenidas de estos hongos con las que se encuentran depositadas en la base de datos MaarjAM se confirmó la identidad de la familia Glomeraceae y se determinó que todos los aislados pertenecen al género Glomus, así mismo se confirmó la identidad para los tres aislados de la familia Acaulosporaceae, y su taxón quedó determinado como Acaulospora sp.
Los alineamientos de las secuencias en el programa ClustalW y la divergencia estimada utilizando el parámetro de distancia Kimura 2 (K2P), permitieron construir un árbol por el método Neighbor–Joining (NJ) con las secuencias depositadas en el GenBank por Krüger et al. (2009, 2012), y las secuencias que presentaron una calidad, identidad y cobertura superior al 90% (dos aislados pertenecientes a la familia Glomeromycota, dos aislados de la familia Acaulosporaceae), así como dos secuencias del aislado de referencia Rizhophagus manihotis, que confirmaron la identificación morfológica de los morfotipos en las familias, géneros y especie correspondientes (Figura 4).
Las secuencias de los morfotipos dentro de la familia Glomeraceae, presentaron una divergencia elevada, que merecen atención porque aún no corresponden a una familia bien determinada, esto se evidencia con las recientes investigaciones donde se han retirado géneros o especies de esta familia, así como Schüßler y Walker (2010) crearon la nueva familia Claroideoglomeraceae con especies previamente reportadas como Glomeraceae.
La clasificación molecular de este grupo de organismos es aún compleja, lo que se dificulta llegar a la determinación de especie, aún más teniendo en cuenta que de las 248 especies descritas actualmente, 132 se encuentran en posición incierta a la espera de datos moleculares relevantes que permitan ubicarlas correctamente.
Conclusiones
- Se evidenció la simbiosis de HMA en dos variedades de P. americana, con altos porcentajes de colonización al interior de las raíces, así como también una cantidad significativa de esporas por gramo de suelo. Las herramientas morfológicas y moleculares permitieron caracterizar morfotipos de HMA asociados a la rizósfera de las dos variedades de P. americana, así como estandarizar protocolos para familias de este grupo, donde su taxonomía es complicada y controversial.
Agradecimientos
COLCIENCIAS financiación del proyecto "Establecimiento del cultivo monoxénico y caracterización molecular de aislados de Glomus asociados a cultivos y la rizósfera de comino crespo Aniba perutilis". Instituto Amazónico de Investigaciones Científicas SINCHI.
Referencias
Azcón–Aguilar. C. Barceló. A. Vidal. M. & de La Viña. G. (1992). Further studies on the influence of mycorrhizae on growth and development of micropopagated avocado plants. Agronomie, 12(10), 837–840. [ Links ]
Banuelos. J. Trejo. D. Lara. L. Gavito. M. & Carreón. Y. (2013). Effects of seven different mycorrhizal inoculum in Persea americana in sterile and non–sterile soil. Trop Subtrop Agroecosyt, 16(3), 423–429. [ Links ]
Barea. J.M. (1998). Biología de la Rizósfera. Invest Cienc, 256, 74–81. [ Links ]
Dorantes. N. Martínez. M. & Carreón. Y. (2012). Utilización del ADN ribosomal 18S para la identificación de hongos micorrízicos arbusculares que colonizan plantas de aguacate (Persea americana Mill.). Biológicas, 14 (2), 42 – 47. [ Links ]
Douds. J.R. Galvez. L. Janke. R. & Wagoner. P. (1995). Effect of tillage and farming system upon populations and distribution of vesicular–arbuscular mycorrhizal fungi. Agr Ecosyst Environ, 52(2–3), 111–118. DOI: 10.1016/0167–8809(94)00550–X. [ Links ]
Drummond. A.J. Ashton. B. Cheung. M. Heled. J. Kearse. M. Moir. R. Stones. H.S. Thierer. T. & Wilson. A. (2009). Geneious v.8.14. http://www.geneious.com. 05.05.2015. [ Links ]
Hass. H. & Menge. A. (1990). VA–mycorrhizal fungi and soil characteristics in avocado (Persea americana Mill) orchard soils. Plant Soil, 127(2), 207–212. doi: 10.1007/BF00014427. [ Links ]
Krüger. M. Stockinger. H. Krüger. C. & Schüßler. A. (2009). DNA–based species level detection of Glomeromycota: one PCR primer set for all arbuscular mycorrhizal fungi. New Phytol, 183(1), 212–223. doi: 10.1111/j.1469–8137.2009.02835.x. [ Links ]
Krüger. M. krüger. C. Walker. C. Stockinger. H. & Schüssler. A. (2012). Phylogenetic reference data for systematics and phylotaxonomy of arbuscular mycorrhizal fungi from phylum to species level. New Phytol, 193(4) ,970–984. doi: 10.1111/j.1469–8137.2011.03962.x. [ Links ]
Montañez. I. Vargas. C. Cabezas. M. Cuervo. J. (2010). Colonización micorrícica en plantas de aguacate (Persea americana L.). rev udcaactual divulg cient, 13 (2), 51–60. [ Links ]
Oehl. F. Sieverding. E. Ineichen. K. Mäder. P. Wiemken. A. & Boller. T. (2009). Distinct sporulation dynamics of arbuscular mycorrhizal fungal communities from different agroecosystems in long–term microcosms. Agric Ecosys Environ, 134(3–4), 257–268. doi:10.1016/j.agee.2009.07.008. [ Links ]
Peña–Venegas. C.P. Cardona. G.I. Mazorra. A. Arguellez. J.H. Arcos. A.L. (2006). Micorrizas arbusculares de la Amazonía Colombiana. Catálogo ilustrado. Instituto Amazónico de Investigaciones Científicas SINCHI. 90 p. http://www.sinchi.org.co/images/pdf/dfpublicaciones/catalogo_micorrizas%20para%20web.pdf. 05.05.2015. [ Links ]
Peterson. R. Massicotte. H. & Melville. L. (2004). Mycorrhizas: anatomy and cell biology. Ontario Canada: National Research Council of Canada, Ottawa. 176 p. [ Links ]
Porter. W.M. Robson. A.D. Abbott. L.K. (1987). Field survey of the distribution of vesicular – arbuscular mycorrhizal fungi in relation to soil pH. J Appl Ecol, 24(2), 659–662. doi: 10.2307/2403900. [ Links ]
Rodríguez–Morelos. V.H. Soto–Estrada. A. Pérez–Moreno. J. Franco–Ramírez. A. & Díaz–Rivera. P. (2014). Arbuscular mycorrhizal fungi associated with the rhizosphere of seedlings and mature trees of Swietenia macrophylla (Magnoliophyta: Meliaceae) in Los Tuxtlas, Veracruz, México. Rev Chil Hist Nat, 87, 9–10. doi: 10.1186/s40693–014–0009–z. [ Links ]
Römpler. H. Dear. P. Krause. J. Meyer. M. Rohland. N. Schöneberg. T. Spriggs. H. Stiller. M. Hofreiter. M. (2006). Multiplex amplification of ancient DNA. Nat Protoc, 1, 720–728. doi:10.1038/nprot.2006.84. [ Links ]
Schüßler. A. Schwarzott. D. & Walker. C. (2001). A new fungal phylum, the Glomeromycota: phylogeny and evolution. Mycological Research, 105(12), 1413–1421. doi: 10.1017/S0953756201005196. [ Links ]
Schüßler. A. & Walker. C. (2010). The Glomeromycota: a species list with new families and genera. Edinburgh & Kew, UK: The Royal Botanic Garden; Munich, Germany: Botanische Staatssammlung Munich; Oregon, USA: Oregon State University. 59p. http://schuessler.userweb.mwn.de/amphylo/Schuessler&Walker2010_Glomeromycota.pdf. 05.05.2015. [ Links ]
Tamura. K. Stecher. G. Peterson. D. Filipski. A. & Kumar. S. (2013). MEGA 6: Molecular Evolutionary Genetics Analysis version 6.0. Mol Biol Evo, 30(12), 2725–2729. doi: 10.1093/molbev/mst197. [ Links ]
Thompson. J.D. Gibson. T.J. Plewniak. F. Jeanmougin. F. & Higgins. D.G. (1997). The CLUSTAL X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Res, 25(24) ,4876–4882. [ Links ]