Introducción
Los virus del género Begomovirus (familia Geminiviridae) tienen el genoma de ADN circular de cadena sencilla y son transmitidos por el complejo críptico de especies de la mosca blanca Bemisia tabaci (Gennadius, 1889) (Hemiptera: Aleyrodidae) a plantas dicotiledóneas (Stanley et al., 2005; Brown et al., 2015).
Las arvenses asociadas a los cultivos se consideran el sitio ideal para que los begomovirus puedan sobrevivir, conservar su estructura genética, participar en procesos de intercambio genético y la aparición de nuevas especies virales (Padidam, Sawyer y Fauquet, 1999; Ramos et al., 2003; Jovel et al., 2004; García-Andrés, Monci, Navas-Castillo y Moriones, 2006). Diversas arvenses son conocidas como hospederas de begomovirus en la regiones tropicales y subtropicales a nivel mundial y han sido estudiadas en América Central y el Caribe (Brown, Lastra y Bird, 1991; Roye, Mclaughlin, Nakhla, y Maxwell, 1997; Umaharan, Padidam, Phelps, Beachy, y Fauquet, 1998; Solórzano-Morales et al., 2017).
En 1983, en la región Azuero de Panamá, se observaron las primeras plantas infestadas con B. tabaci que presentaban síntomas de infección por begomovirus. Sin embargo, solo fue en 1991 cuando se registraron elevadas pérdidas económicas en la producción de tomate de tipo industrial, atribuidas al incremento en la población de las especies del complejo de la mosca blanca en asociación con el begomovirus Potato yellow mosaic Panama virus (PYMPV) (Engel, Fernández, Jeske, y Frischmuth, 1998; Anderson y Morales, 2005). La provincia de Los Santos pertenece a la Región de Azuero y según el informe de producción agropecuaria del Instituto Nacional de Estadística y Censo de Panamá (2017) durante la época seca produce el 87% del tomate industrial del país.
En el distrito Los Santos, Fernández, Osorio y Frischmuth, (1998)) identificaron las arvenses en los cultivos de tomate con infección de begomovirus que pertenecen a las familias Malvaceae (Herissantia crispa, Sida acuta y Sida rhombifolia), Euphorbiaceae (Euphorbia heterophylla), Tiliaceae (Corchorus orinocensis), Fabaceae (R. mínima) y Asteraceae (Eleutheranthera ruderalis). Durante la época lluviosa, las parcelas de cultivo son dejadas en barbecho o son usadas para pastoreo. La alternancia entre temporada de cultivo y de barbecho puede estimular a la mosca blanca a utilizar hospederos alternos, siendo estos, generalmente, las arvenses asociadas al cultivo (Bezerra, De Oliveira y Vasconcelos, 2004). Este trabajo se desarrolló con el objetivo de identificar los reservorios de begomovirus en las parcelas dedicadas al cultivo de tomate, en las épocas de barbecho y de cultivo en el distrito de Los Santos, provincia de Los Santos (Panamá). La identificación de nuevos hospederos alternativos de begomovirus en las áreas de cultivo de tomate permitirá mejorar las estrategias de manejo de las arvenses, incluyendo la época de barbecho en la planificación.
Materiales y métodos
Area de muestreo
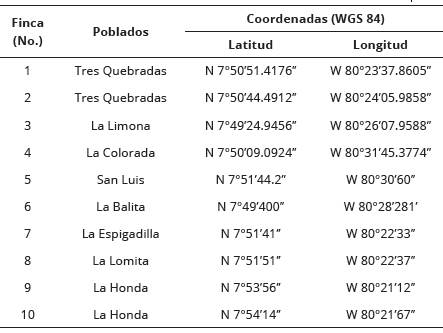
Fueron seleccionadas 10 fincas dedicadas al cultivo de tomate industrial entre los productores asociados a la Cooperativa El Progreso R. L. del Corregimiento de Agua Buena del Distrito de Los Santos. La ubicación de las parcelas se determinó mediante un sistema de posicionamiento global (GPS) (Etrex, Summit HC, Garmin®, Shijr, Taiwan), en los poblados de Tres Quebradas, La Limona, La Colorada, San Luis, La Balita, La Espigadilla, La Lomita y La Honda Los Santos que pertenecen al distrito de Los Santos, provincia de Los Santos, República de Panamá (Tabla 1).
Recolección de plantas arvenses
Las recolecciones fueron realizadas en parcelas de tomate de tipo industrial var. IDIAP T-8, en el periodo comprendido entre octubre de 2016 y abril de 2017. Las recolecciones en campos en barbecho fueron realizadas entre mayo y septiembre de 2016.
El recorrido de las parcelas para la toma de muestras se hizo tratando de abarcar una superficie representativa. Durante este recorrido, fueron recolectadas 110 muestras de ápices de arvenses dicotiledóneas sintomáticas y asintomáticas, 78 en época de cultivo y 32 en época de barbecho (Tabla 2). Se consideró como plantas sintomáticas aquellas con la presencia de síntomas característicos de virosis, tales como: mosaico foliar amarillo, moteado foliar amarillo, entrenudos cortos, hojas enrolladas o rizadas (Engel et al., 1998).
Procesamiento de muestras
Las muestras fueron colocadas en tubos de poliuretano de 50 ml con gel de silicio (Scharlau®, Barcelona, España) como secante para garantizar su conservación. La identificación taxonómica preliminar se realizó en campo, mediante observación de las plantas con flores. Luego fueron codificadas e identificadas en el Laboratorio del Instituto de Investigación Agropecuaria de Panamá (IDIAP). Su identificación fue verificada en el Herbario de la Universidad Autónoma de Chiriquí (UNACHI).
Extracción de ácidos nucleicos totales
Se obtuvo el ADN total a partir de 0.05 g de tejido vegetal como lo indica el protocolo de Dellaporta (Dellaporta, Wood y Hicks, 1983). El producto obtenido fue disuelto en 100 μL de agua bidestilada estéril y conservado a -20 °C hasta su posterior uso en PCR.
(pcr)
La solución para la PCR consistió en 6.25 μL de PCR Master Mix® (Promega®, Madison WI, U.S.A), 0.20 μL de cada cebador degenerado AVCORE 494 y ACCORE 1048 que amplifican universalmente la región conservada de aproximadamente 550 pb, ubicada en la parte central de la secuencia de ADN que codifica para la proteína de la cápside de los begomovirus (Wyatt y Brown, 1996). Se completó con 2.25 pL de agua destilada estéril para un volumen de reacción final de 12.5 μL que incluía 2.5 μL de ADN total de cada muestra. La amplificación se realizó en un termociclador modelo 2720 Applied Biosystems Thermal Cycler versión 2.09 (ThermoFisher Scientific, CA, U.S.A). El programa de PCR consistió en 2 min a 94 °C, 30 ciclos que comprendían tres etapas cada uno: 94 °C durante 1 min, 58 °C durante 1 min y 72 °C por 1 min. Los resultados se visualizaron en gel de agarosa (Type I, Sigma-Aldrich) al 1.5% en TAE 1.0% y tinción de bromuro de etidio. Como patrón se usó el marcador de peso molecular 1 kb DNA Ladder (Promega®, Madison WI, U.S.A) (Figura 1). Como control positivo se usaron plantas de tomate del cultivo con síntomas de infección por begomovirus. Como control negativo se usaron plantas de tomate cultivadas en invernadero.
Análisis estadísticos
La comparación de la presencia de begomovirus en las arvenses fue realizada entre temporada de cultivo y temporada de barbecho, mediante una Prueba de t de Student para demostrar que la prevalencia de malezas infectadas por begomovirus varía durante la época de barbecho (μ1) con respecto a la época de cultivo (μ2), es decir: Ho2: μ1 - μ2 = 0; en donde: μ1 es el valor promedio de la prevalencia de plantas infectadas por begomovirus durante el barbecho y μ2 es el valor promedio de la prevalencia durante la temporada de cultivo (Tabla 3). Para verificar que las diferencias entre las medias no fueron debidas al azar, se usó un nivel de significancia de 5%. El proceso de selección de las muestras de tejido vegetal en campo se realizó de manera aleatorizada y los valores de las prevalencias de las plantas infectadas por begomovirus fueron transformadas con para compensar la falta de normalidad de los datos. En esta investigación la variable de estudio fue la prevalencia de plantas infectadas por begomovirus.
Resultados y discusión
Se analizaron un total de 110 muestras mediante PCR, la observación de una banda entre 550 y 600 pares de bases indicó la presencia de begomovirus en las muestras como señalan Wyatt y Brown (1996)) (Figura 1). En la época de cultivo de tomate de 2016 (Tabla 1) fueron evaluadas en total 35 muestras de arvenses para detectar la presencia o ausencia de begomovirus. Las muestras evaluadas durante esta época pertenecían a 12 familias, representadas por 18 especies. Los resultados obtenidos mostraron la infección de 12 arvenses de las especies R. mínima, Calopogonium mucunoides, Mimosa sp. y Physalis. angulata. Es decir, un total del 34% de plantas estaban infectadas (Tabla 2).
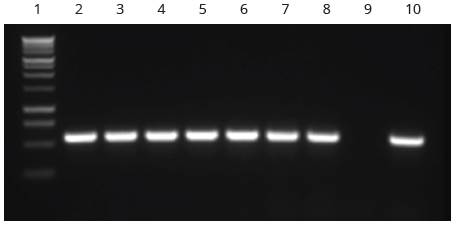
Figura 1 Resultados de la amplificación por PCR de un fragmento del componente A de begomovirus de aproximadamente 550 pb con los cebadores AVCORE y ACCORE. Líneas: 1. Marcador de peso molecular 1 kb Ladder (Promega), 2. P. angulata, 3. C. mucunoides, 4. R. minima, 5. M. alceifolia, 6. A. spinosus, 7. M. americanum, 8. C. viscosa, 9. Control negativo, 10. Control positivo. Electroforesis en gel de agarosa 1.0% con tinción de Bromuro de etidio.
En la época de barbecho del 2016, (Tabla 1) fueron evaluadas 32 muestras. Los resultados obtenidos indicaron que 13 muestras pertenecientes a siete especies fueron positivas a infección por begomovirus. Estas fueron: A. dubius, Cleome viscosa., C. mucunoides, R. minima, Malvastrum americanum, P. angulata y Hybanthus. attenuatus, para un total del 41% de plantas infectadas durante esta temporada (Tabla 2).
En la época de cultivo de 2017, se tomaron muestras en cuatro fincas (Tabla 1) para un total de 43 muestras evaluadas. Los resultados obtenidos fueron: 12 muestras positivas de las especies A. spinosus, C. viscosa, R. minima, Malachra alceifolia y P. angulata, para un total de 28% (Tabla 2).
Los resultados muestran la presencia de arvenses infectadas por begomovirus en fincas dedicadas al cultivo de tomate industrial en el distrito de Los Santos, tanto en épocas de cultivo como de barbecho. Las especies de arvenses que resultaron con mayor número de plantas infectadas por begomovirus fueron: P. angulata. (100%), Mimosa spp. (100%), A. dubius (100%), R. mínima (70%), C. viscosa (60%), A. spinosus (50%), H. attenuatus (50%), C. mucunoides (44.4%), M. alceifolia(28%), M. americanum(25%). En general, el 33.64% de las arvenses resultaron infectadas por begomovirus. Las plantas positivas pertenecen a las familias Amaranthaceae, Cleomaceae, Fabaceae, Malvaceae, Solanaceae y Violaceae.
Las arvenses A. dubius, A. spinosus, C. mucunoides, C. viscosa, H. attenuatus, M. alceifolia, M. americanum, Mimosa sp. y P. angulata fueron registradas por primera vez como hospederas de begomovirus asociadas al cultivo de tomate industrial en el distrito de Los Santos y en la República de Panamá.
Los síntomas de infección con begomovirus, como mosaico dorado, fueron observadas en C. mucunoides, R. mínima y Sida spp. tanto en la época de cultivo como de barbecho. Las arvenses M. alceifolia, M. americanum y Euphorbia spp. no fueron recolectadas durante la época de cultivo, ya que por tratarse de arbustos de mayor tamaño, son eliminadas por los productores de las áreas de cultivo. No obstante durante la época de barbecho fue posible recolectar plantas con síntomas de mosaico amarillo y clorosis en la hoja. La presencia de arvenses con clorosis de la hoja y negativas a begomovirus, pudo ser debido a que estaban infectadas con otros patógenos o presentaban carencias nutricionales. Las plantas con alto contenido de mucílago como Sida spp. y Euphorbia spp., no dieron resultados positivos, a pesar de que se les extrajo ADN total siguiendo protocolos específicos (Echevarria-Machado et al., 2005; Ghosh, Paul, Ghosh, Roy, 2009).
El 100% de las arvenses identificadas como P. angulata que fueron recolectadas, tanto en época de cultivo como de barbecho, fueron asintomáticas y presentaron infección por begomovirus. Este dato es importante porque esta especie, igual que S. lycopersicum, pertenecen a la familia Solanaceae, lo que aumenta el riesgo de ser un hospedero alternativo de los begomovirus que infectan al tomate o una fuente de variabilidad genética para estos (Briddon, Patil, Bagewadi, Nawaz-ul-rehman, y Fauquet, 2010). La presencia de begomovirus que infectan arvenses y plantas de interés económico en Centroamérica ha sido documentada entre Fabaceae en el caso de frijol y Calopogonium golden mosaic virus en Nicaragua y Costa Rica (Diaz, Maxwell, Karkashian y Ramírez, 2002; Karkashian, Ramos-Reynoso, Maxwell y Ramírez, 2011; Fiallo-Olivé, Navas-Castillo, Moriones y Martínez- Zubiaur, 2012). Los resultados presentados indican que el 26% de las arvenses infectadas por begomovirus son asintomáticas (Tabla 2), lo que permite suponer que la cantidad de inóculo presente en el agroecosistema al momento de establecer el cultivo puede ser subestimada por el productor. Una práctica frecuente es la remoción de las arvenses infectadas por virus y que se encuentran presentes en los alrededores de cultivos de interés económico. Sin embargo, esto no siempre reduce la prevalencia de virus en un cultivo inclusive, en algunos casos puede incrementar la infección, ya que induce al vector a trasladarse desde las arvenses hasta el cultivo (Bezerra et al., 2004).
Presencia de begomovirus en las arvenses en temporadas de cultivo y de barbecho
La gran capacidad innata de sobrevivencia de las arvenses les permite persistir en el ecosistema a través de las épocas del año. No obstante, esta situación las expone a infecciones por begomovirus provenientes de las moscas blancas virulíferas de la zona. Este hecho, permite la propagación y recombinación de los begomovirus en hospederos alternativos, aún en épocas con ausencia de los hospederos de interés económico, como señalan Gilbertson, Rojas, Russell y Maxwell (1991)). La comparación de la presencia de begomovirus en las arvenses entre temporada de cultivo y temporada de barbecho, indicó que la prevalencia de malezas infectadas no varió (P > 0.05) a través del año (Tabla 3). Los resultados obtenidos indican que, tanto en época de cultivo como de barbecho, se pueden encontrar arvenses infectadas en las parcelas de cultivo de tomate, lo que muestra el potencial que tienen como reservorio y hospedero alternativo de begomovirus, que infecten las plantas de este cultivo de una temporada a la siguiente.
Tabla 2 Presencia de begomovirus en arvenses asociadas al cultivo de tomate, en el distrito de Los Santos, Panamá.
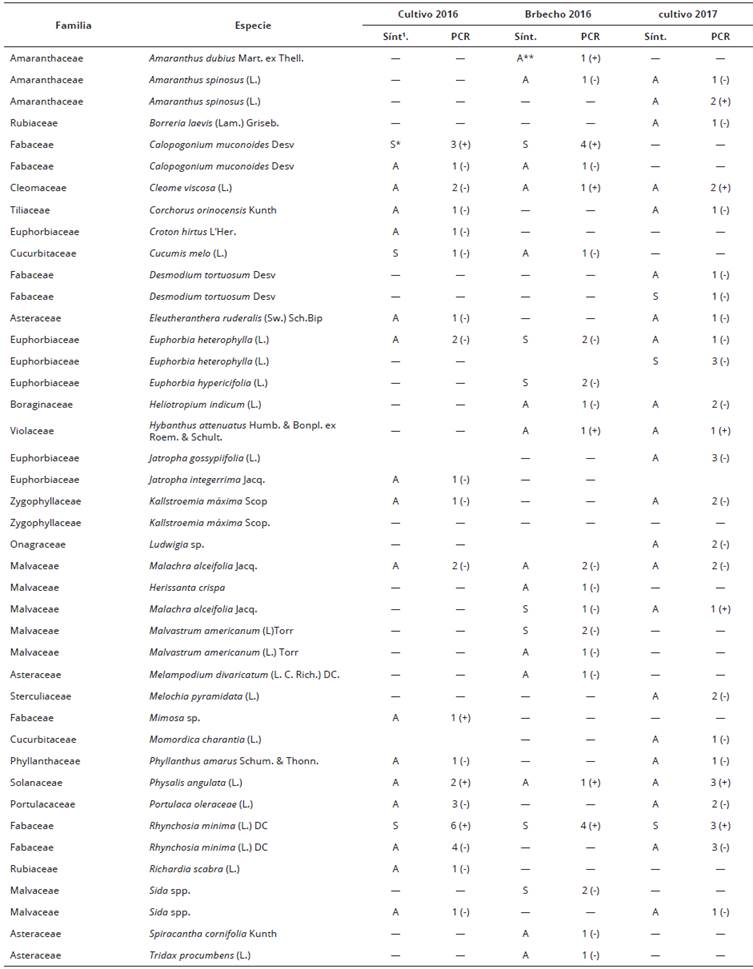
*S: Sintomática: Presencia de mosaico dorado, clorosis de la hoja, enrollamiento de la hoja, clorosis de las venas. **A: Asintomática: Ausencia de los síntomas descritos para la infección con begomovirus. 1.Sínt. = Síntomas.
Tabla 3 Prevalencia de begomovirus en las malezas durante las temporadas de cultivo y de barbecho (2016-17). distrito de Los Santos, Panamá.
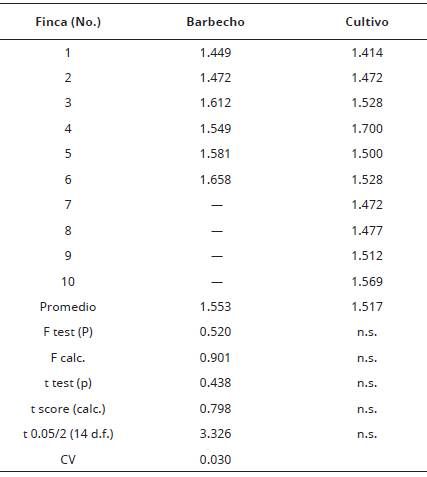
*P<0.05, **P<0.01 y n.s. P>0.05 Datos transformados con la función Vx+2.
+ Fuente: Biometrika tables for statisticians (Pearson y Hartley, 1966).
Conclusiones
Los resultados indican que las especies de arvenses asociadas al cultivo de tomate en el Distrito de Los Santos, son hospederas de begomovirus durante las épocas de cultivo y de barbecho. Las especies de plantas infectadas con begomovirus fueron: P. angulata, A. dubius, A. spinosus, R. minima C. viscosa, H. attenuatus, C. mucunoides, M. alceifolia, M. americanum y Mimosa sp.
Las arvenses A. dubius, A. spinosus, C. mucunoides, C. viscosa, H. attenuatus, M. alceifolia, M. americanum, Mimosa sp. y P. angulata, fueron registradas por primera vez en este estudio como hospederas de begomovirus en el distrito de Los Santos, República de Panamá.
Las especies sintomáticas R. minima y C. mucunoides y la asintomática P. angulata presentaron resultados positivos a infección por begomovirus tanto en la temporada de cultivo como de barbecho. No se encontraron diferencias (P > 0.05) en los valores de prevalencia de infección por begomovirus entre las arvenses recolectadas durante la temporada de cultivo y la de barbecho.