Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista de la Facultad de Medicina Veterinaria y de Zootecnia
Print version ISSN 0120-2952
Rev. Med. Vet. Zoot. vol.57 no.1 Bogotá Jan./Apr. 2010
HIBRIDACIÓN IN SITU PARA LA DETECCIÓN DE Streptococcus agalactiae
EN TEJIDOS DE TILAPIA (Oreochromis sp.)
IN SITU HYBRIDIZATION TECHNIQUE FOR Streptococcus agalactiae
DETECTION IN TILAPIA TISSUES (Oreochromis sp.)
E. A. Pulido1, C. A. Iregui2
Laboratorio de Patología
Facultad de Medicina veterinaria y de Zootecnia
Universidad nacional de Colombia
1eapulidob@unal.edu.co, 2cairegui@unal.edu.co
Recibido: 8 de marzo de 2010; aprobado: 21 de abril de 2010
RESUMENLa estreptococosis es uno de los problemas sanitarios más serios en la acuicultura mundial. En Colombia la enfermedad afecta de manera importante las explotaciones de tilapia. Se estandarizó la técnica de hibridación in situ (HIS) en tejidos de tilapia previamente identificados como positivos a la presencia de S. agalactiae por la técnica de inmunoperoxidasa indirecta (IPI) y microbiología. Se obtuvo señal positiva en el interior de los granulomas con una especificidad del 100%. Se logró amplificar significativamente la señal mediante el uso de la tiramida. La HIS en tejidos de tilapia puede ser usada para el diagnóstico y estudios de patogenésis y epidemiológicos con este microorganismo. Se requieren futuras investigaciones para optimizar la marcación de las bacterias libres en los tejidos y evaluar la sensibilidad de la técnica.
Palabras clave: hibridación in situ, Streptococcus agalactiae, tilapia, biología molecular.
ABSTRACTStreptococcosis is one of the most important health problems in world aquaculture. In Colombia, the disease affects significantly tilapia farms. The standardization of the HIS technique was achieved in tilapia tissues previously identified as positive for the presence of S. agalactiae by other techniques as indirect immunoperoxidase technique (IPI) and microbiology. Positive signal was obtained within the granulomas with specificity of 100%. The usefulness of the tiramide about increasing the signal intensity was confirmed. The HIS in tilapia tissues can be used in the diagnosis, pathogenesis and epidemiological studies of the disease. Further research is required to optimize the hybridization of bacteria located free in the tissues and to assess the technique sensitivity.
Key words: in situ hybridization, Streptococcus agalactiae, tilapia, molecular biology.
INTRODUCCIÓN
El S. agalactiae es reconocido como el principal agente etiológico responsable de septicemia y meningoencefalitis en peces (1, 2). En Colombia se detectó la bacteria en híbridos de tilapia roja (Orechromis spp.) inicialmente en el departamento del Tolima, posteriormente se ha identificado en otros departamentos y además se ha aislado de otras especies de tilapia como la nilotica (O. niloticus) y trucha arco iris (Oncorhynchus mykiss). Hasta el momento, de los streptococcus reportados internacionalmente, sólo se ha logrado identificar en el país el S. agalactiae, por medio de la reacción en cadena de la polimerasa (PCR) (3, 4, 5, 6). Su diagnóstico se basa en los signos clínicos, las lesiones histopatológicas y el aislamiento microbiológico; sin embargo, su identificación específica requiere de técnicas moleculares (1, 7, 8, 9, 10). Las lesiones histopatológicas van desde una reacción necrótico-inflamatoria hasta granulomatosa.
En el laboratorio de patología de la Facultad de Medicina veterinaria y de Zootecnia de la Universidad nacional de Colombia, además de las técnicas citadas, se dispone de las técnicas de PCR e inmunoperoxidasa indirecta (IPI) para la identificación de esta bacteria; sin embargo, para realizar estudios de patogénesis y epidemiológicos más sensibles, se hace necesario implementar técnicas moleculares más específicas que se puedan aplicar directamente en los tejidos de los peces y que permitan el seguimiento del microorganismo en el interior del hospedero. Tal es el caso de la hibridización in situ (HIS), la cual permite la identificación específica de microorganismos en los tejidos mediante sondas genómicas con las cuales se puedan observar simultáneamente los cambios histopatológicos y la ubicación topográfica precisa de los patógenos (11, 12, 13). En la presente investigación se intentó estandarizar la HIS para la detección del S. agalactiae en tejidos de tilapia roja (Oreochromis spp.) afectada con la enfermedad, y se intentó evaluar su uso potencial en estudios epidemiológicos y de patogénesis. Hasta el momento no se tienen reportes del uso de esta técnica para la detección de S. agalactiae en tejidos de peces.
MATERIALES Y MÉTODOS
Bacterias
La bacteria que se usó para los ensayos iniciales correspondió a un aislamiento obtenido de tilapias rojas (Oreochromis sp.) con estreptococosis provenientes de la represa de Betania, el cual fue identificado como S. agalactiae mediante la técnica de PCR según lo descrito por iregui (4); fue mantenido hasta su uso a –70oC en caldo Todd-Hewitt con glicerol al 20% (v/v).
Preparación de la sonda
Se adquirió un kit comercial con sondas de oligonucleótidos para la detección del 16s rRNA del S. agalactiae (S. agalactiae 16s rRNA Oligonucleotide probe pack -Genedetect ®), el cual incluye la sonda denominada saga (5´GTA AAC ACC AAA CCT CAG CG-3´) reportada por Trebesius et ál. (reportada por Kempf) (14) para la detección de esta bacteria. Se siguieron las recomendaciones del proveedor para su preparación. La dilución de trabajo más apropiada de la sonda y del conjugado estreptavidina-peroxidasa se evaluó llevando a cabo algunas variaciones a la técnica descrita por anderson (15) para la hibridación en membrana (Dot blot hybridization) y a la inmuno dot indirecta reportada por Bohórquez y Botero (16) y díaz (17) en la técnica de inmunoperoxidasa indirecta. La sonda se ensayó en cuatro diluciones (1:10, 1:100, 1:1000 y 1:10000), fijadas al pa-pel de nitrocelulosa por calentamiento a 80oC por 10 minutos y posterior exposición a luz ultravioleta por 10 minutos, y su detección con dos diluciones del conjugado streptavidina-peroxidasa (1:50 y 1:100).
Estandarización inicial de la técnica
La estandarización inicial de la técnica se llevó a cabo en tejidos positivos a S. agalactiae por las técnicas de inmunoperoxidasa indirecta (IPI) y microbiología, los cuales fueron desincluidos de bloques de parafina, cortados de acuerdo con los protocolos usuales de histopatología y colocados sobre láminas tratadas con poly-L-lysina.
Se siguió de manera general el protocolo descrito por Genedetect Limited (18) para la hibridación in situ (HIS) usando sondas de oligonucleótidos marcadas con biotina y su detección con el conjugado streptavidina-peroxidasa. De acuerdo con los resultados iniciales, se hicieron todos los ajustes necesarios al protocolo original, incluyendo algunos pasos adicionales, especialmente en la permeabilización de los tejidos. Se definió el siguiente protocolo para aplicar en los siguientes ensayos, brevemente (cuando no se mencione lo contrario los pasos son a temperatura ambiente): los cortes de los tejidos fueron puestos sobre láminas previamente tratadas con poly-L-lysina, desparafinadas con tres lavados con xylol, rehidratadas con concentraciones descendentes de etanol; se bloquearon las RNasas presentes mediante lavados con DEPC-H2O (diethilpirocarbonato), se hizo una posfijación con paraformaldehído (4% en 0,1M PB), se inactivaron las peroxidasas endógenas con H2O2 en PBS (0,3%), se incubaron con anhídrido acético en buffer TEA 0,1 M (0,5% v/v). Para la permeabilización de los tejidos se empleó proteinasa K 4 µl (100 µg/ml en buffer THCL 50mM, 10mM CaCl2, pH 8,0 x 60 min a 37oC), lisozima (23 mg/ml en buffer TE 1X, pH 8,0, x 90 min a 37oC) y buffer TrisSDS (100mM Tris HCL pH 9,0, y 4% de SDS x 30 min a 37oC). Para bloquear las posibles biotinas endógenas se hizo una incubación con estreptavidina. Se incubaron a 39oC por 2 horas en la solución de prehibridación compuesta de (20 ml total): 20 XSSC (4 ml), dextrán sulfato (4 g), formamida desionizada (10 ml), PolyA (10 mg/ml, 0,5 ml), "herring sperm" (10 mg/ml, 0,5 ml), tRNA (10 mg/ml, 0,5 ml), DTT (de una sol. 1M, 2 ml) y solución denhardt´s (50X, 0,2 ml). Se agregó la sonda a la solución de prehibridación (800 ng/ml) y se incubó a 39oC x 18-24 horas. Los lavados poshibridación consistieron en un lava-do rápido con 1XSSC (10 mM DTT), dos lavados con 1XSSC (10 mM DTT) x 15 min c/u. a 42oC, dos lavados con 0,5XSSC (10mM DTT) x 15 min c/u. a 42oC y un lavado con 0,5XSSC (10mM DTT) x 10 min. Se incubó en la solución de bloqueo (10 mg/ml en PBS), con la solución del conjugado estreptavidina-peroxidasa (1:200) en reactivo de bloqueo (1% en PBS) y en la solución de detección (daB 0,6 mg/ml, THCL 50 mM pH 7,6, H2O2 0,2%). Se hizo un lavado con hematoxilina, lavados con agua corriente hasta que salió la coloración, pases por alcohol ácido al 0,4%, agua destilada, agua amoniacal y agua destilada; se deshidrató con concentraciones ascendentes de etanol, dos lavados con xylol y se hizo su montaje final con entellan.
Evaluación de la especificidadPara la determinación de la especificidad se utilizó la técnica de PCR como prueba de referencia, con la cual se identificó el S. agalactiae proveniente de la represa de Betania y las cepas de referencia ATCC empleadas en este ensayo. De acuerdo con la definición de especificidad, en el presente caso se calculó como el cociente entre el número de muestras sin S. agalactiae detectadas como negativas por HIS (verdaderos negativos) y el número de muestras que realmente no tenían S. agalactiae.
Especificidad = Verdaderos negativos x 100
Realmente negativos
Para su determinación se emplearon pelets de bacteria a los cuales, previo a su inclusión en parafina, se le aplicaron los pasos de permeabilización definidos en la estandarización de la técnica en tejidos, concretamente incubación de los pelets con soluciones de proteinasa K, lisozima y buffer Tris-SDS; después de cada paso los pelets fueron recuperados por centrifugación a 12.000 rPM x 10 min a 4oC. Después de esto, los pellets fueron procesados por la técnica usual de histopatología, puestos sobre láminas tratadas con poly-L-lysina y tratados con los pasos restantes descritos en el protocolo previamente definido. El anterior procedimiento se le aplicó al S. agalactiae obtenido de la represa de Betania y a los siguientes cocos gram (+):
-S. agalactiae ATCC 13813
-S. pneumoniae ATCC 6303
-S. dysgalactiae ATCC 12394
-S. uberis ATCC 700407
-Enterococcus faecalis ATCC 6056
Evaluación de la sensibilidad y del uso de la amplificación con tiramida
En el presente trabajo se empleó el término de sensibilidad como la determinación del límite de detección por HIS del S. agalactiae en UFC/ml. Para esto se probó la técnica en porciones de tejidos inoculados artificialmente con diluciones consecutivas en base 10 de S. agalactiae, procesados igualmente por la técnica usual de histopatología, puestos sobre láminas tratadas con poly-L-lysina y tratados con el mismo protocolo previamente definido.
Para evaluar el uso de la tiramida como método de amplificación de la señal, se adquirió un kit comercial (Tyramide signal amplification kit -TSA Kit ·NRO. 21®, Molecular Probes); brevemente, este procedimiento se basa en la deposición mediada por peroxidasa de moléculas de tiramida conjugadas con biotina, las cuales se unen en sitios cercanos donde se ha localizado la actividad de la peroxidasa. De este modo, si las sondas marcadas con biotina son detectadas con peroxidasa, una cantidad extra de moléculas de biotina (acopladas a la tiramida) pueden ser introducidas en el sitio de la hibridización in situ, amplificando de esta forma la señal. Esta técnica se probó en los tejidos anteriores y en cortes positivos a S. agalactiae, de acuerdo con las recomendaciones del proveedor.
En todos los ensayos se incluyeron los siguientes controles:
-Controles negativos:
- Tejidos negativos a la presencia de la bacteria.
- Tejidos sin la aplicación de la sonda.
- Tejidos positivos a la presencia de la bacteria.
- Tejidos con la aplicación de una sonda genérica (PolyDT) marcada con biotina.
Análisis estadístico
Se planeó hacer estadística descriptiva, especialmente para comparar los resultados obtenidos en las pruebas de sensibilidad del presente trabajo con lo obtenido en otros estudios con técnicas como IPI y microbiología. Se buscaba establecer si existían diferencias significativas entre los valores de sensibilidad y entre el número de muestras con resultados positivos y negativos.
RESULTADOS
Mediante la técnica de dot Blot, con algunas modificaciones, se definió como dilución óptima de trabajo de la sonda 1:100, y del conjugado estreptavidinaperoxidasa 1:200.
Mediante el uso del protocolo anteriormente mencionado se obtuvo una señal positiva en el interior de los granulomas, tanto en aquellos bien definidos como en algunos en formación (figuras 1 y 2).
Para la evaluación de la especificidad se obtuvo hibridación positiva con S.agalactiae proveniente de la represa de Betania, y con el S. agalactiae ATCC 13813 (figuras 3 y 4). Con los pellets de los otros cuatro microorganismos gram (+) como S. pneumoniae ATCC 6303, S. dysgalactiae ATCC 12394, S. uberis ATCC 700407 y Enterococcus faecalis ATCC 6056, no se obtuvo señal alguna (figura 5).
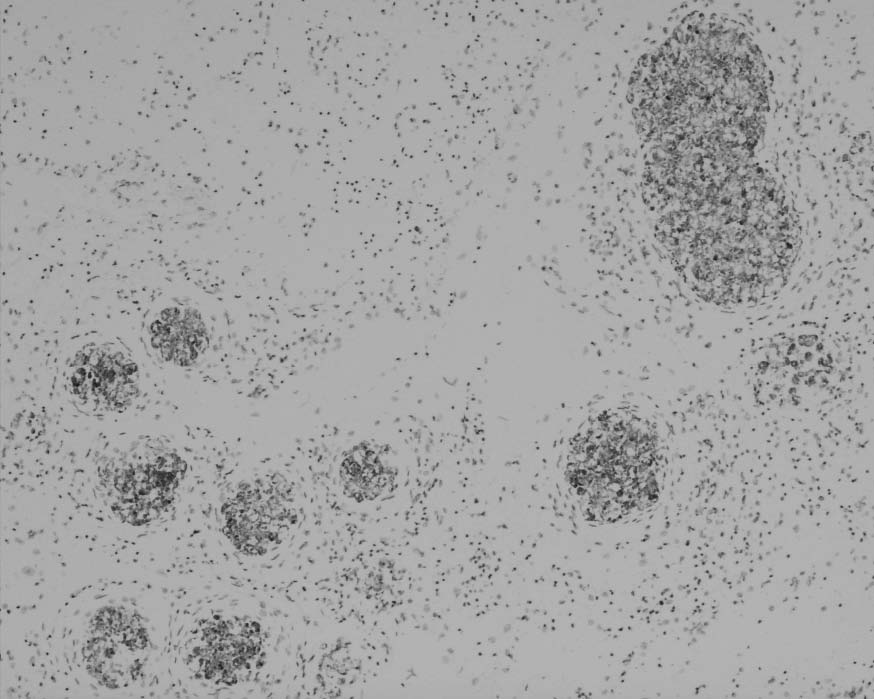
FIGURA 1. HIS de S. agalactiae. Focos positivos de HIS
en el interior de los granulomas (20X).
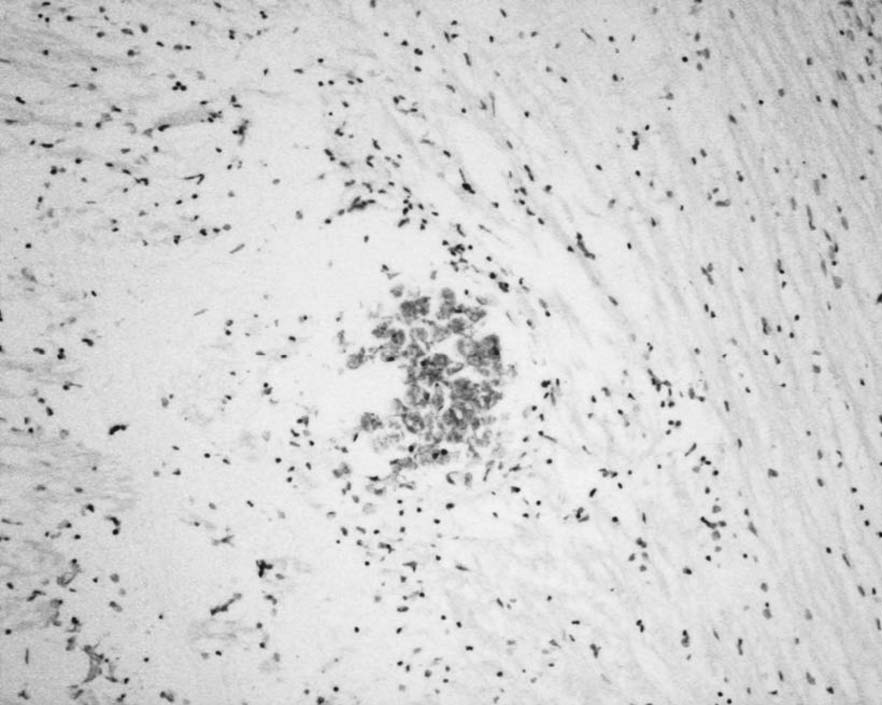
FIGURA 2. HIS de S. agalactiae. Focos positivos de HIS
en el interior de un granuloma en formación (40X).
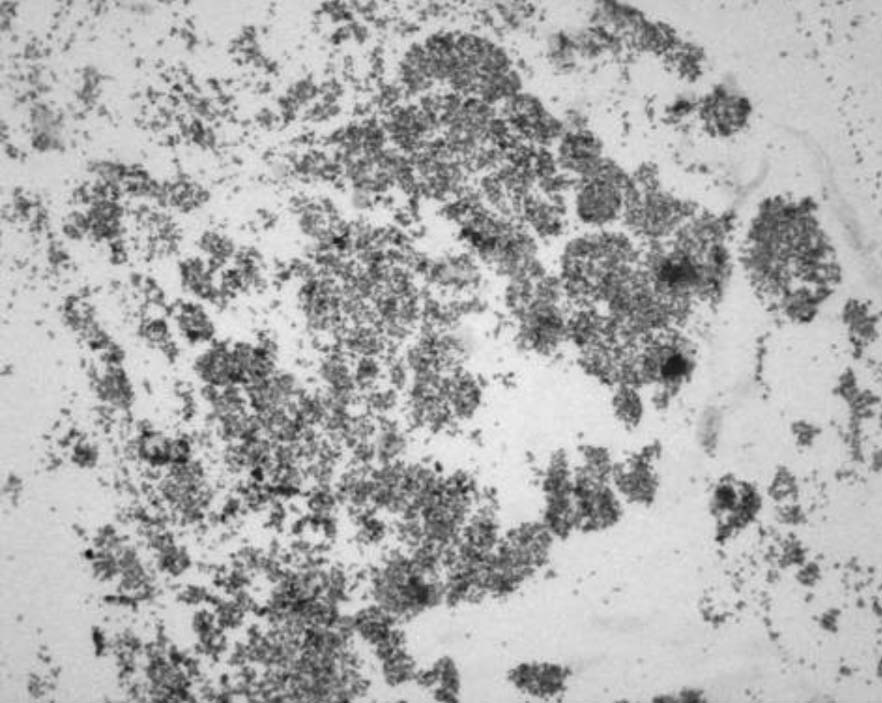
FIGURA 3. HIS positiva con pellets de S. agalactiae
proveniente de la represa de Betania (40X).
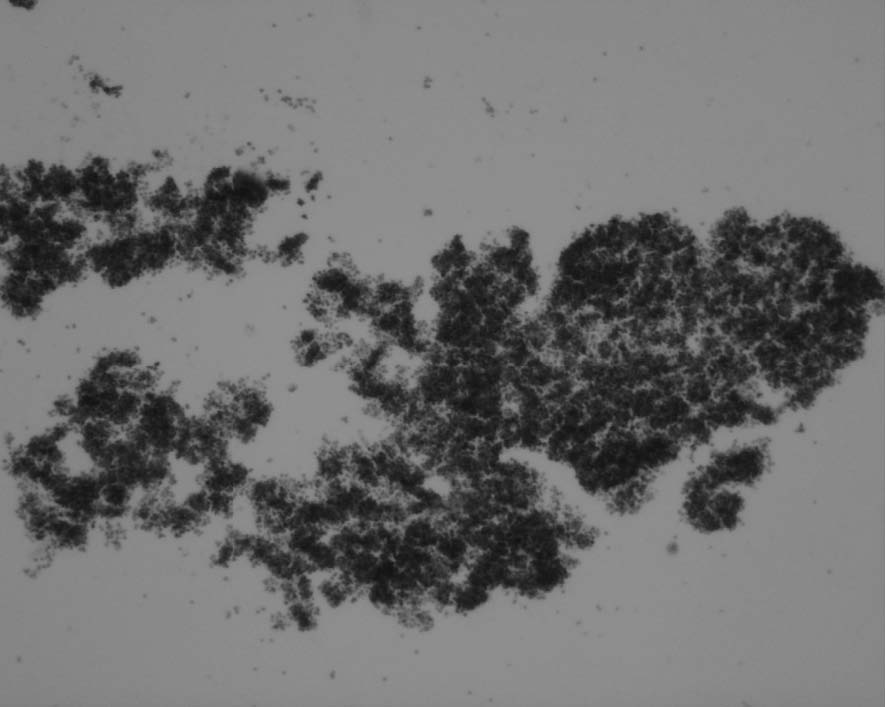
FIGURA 4. HIS positiva con pellets de S. agalactiae ATCC 13813 (40X).

FIGURA 5. Ausencia de HIS con pellets de
S. dysgalactiae ATCC 12394 (40X).
De acuerdo con lo anterior, el valor de la especificidad se calculó de la siguiente forma:
- Muestras evaluadas: 6
- Realmente negativos (negativos por PCR): 4
- Negativos por HIS y negativos por PCR (verdaderos negativos): 4
Especificidad = 4/4 x100 =100%
Con la inclusión de los pasos recomendados por el proveedor para el uso de la tiramida, dentro del protocolo previamente establecido para la HIS del S. agalactiae, se observó una significativa mayor intensidad de la señal (figuras 6 y 7).
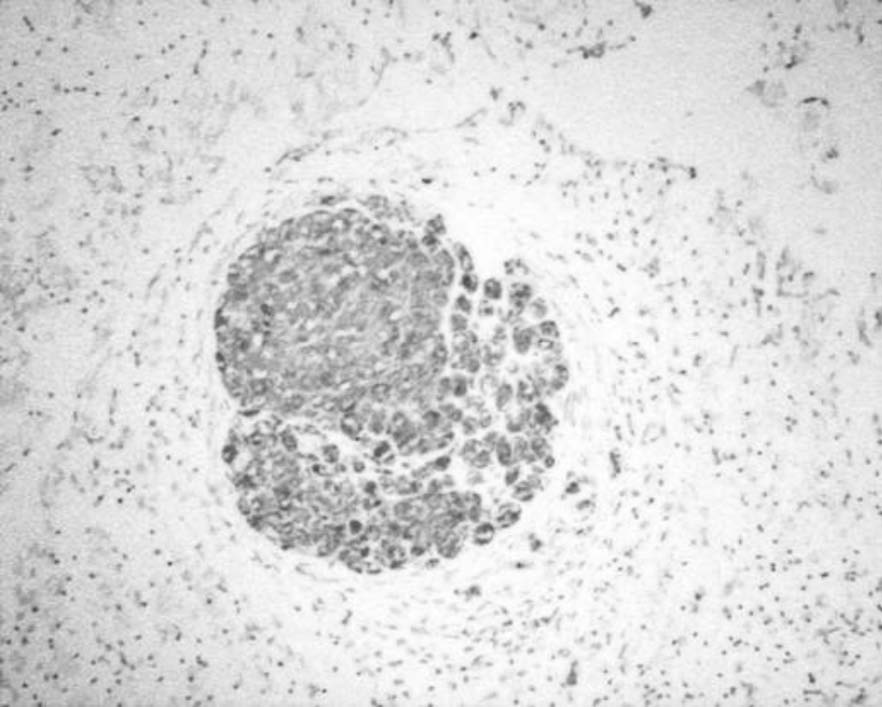
FIGURA 6. HIS de S. agalactiae. Marcación positiva en el interior
de un granuloma sin el uso de tiramida (20X).
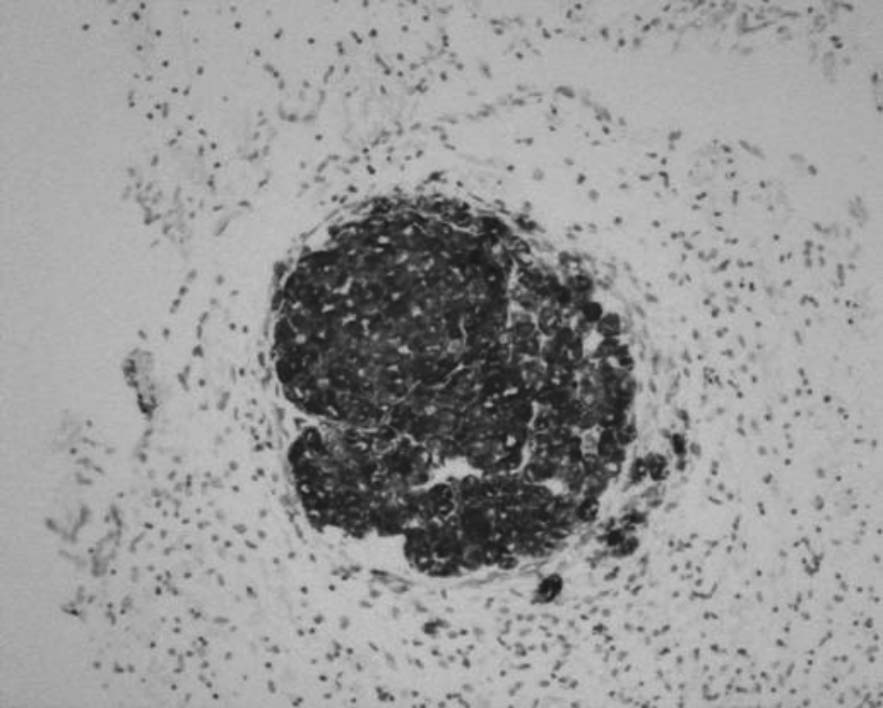
FIGURA 7. HIS de S. agalactiae. Marcación positiva en el interior de un granuloma
con la amplificación de la señal con tiramida (20X).
A pesar de todos los ensayos que se llevaron a cabo probando especialmente diferentes opciones para optimizar la permeabilización, no fue posible obtener una señal positiva de los S. agalactiae que se encontraban libres en los tejidos de los peces enfermos —a pesar de su presencia abundante—, así como tampoco en los tejidos inoculados artificialmente con diluciones consecutivas en base 10 de S. agalactiae. Por esta razón no fue posible evaluar la sensibilidad de la técnica y hacer una mayor evaluación de la eficacia del uso de la amplificación con tiramida. Debido a lo anterior no se pudo llevar a cabo el análisis estadístico que se tenía previsto.
DISCUSIÓN
Por medio de la técnica de HIS , con sondas de oligonucleótidos marcadas con biotina, fue posible la detección de S. agalactiae en tejidos de tilapia afectadas con la enfermedad, y en las que hubiese formación de granulomas, y los cuales habían sido incluidos en parafina. Según la información consultada, este sería el primer reporte del desarrollo y uso de esta técnica para la detección de S. agalactiae en tejidos de peces. asimismo, no se conocen reportes del desarrollo y empleo de esta técnica para la detección de este patógeno en otras especies animales en tejidos embebidos en parafina. Se tienen reportes del uso de la técnica de hibridación in situ fluorescente (FISH) para la detección de S. agalactiae en frotis de cultivo de sangre (14), frotis vaginales (19), frotis de cultivos de bacterias obtenidos de líquido cefalorraquídeo (20) y de abscesos peritonsilares en humanos (21).
En tejidos embebidos en parafina existen reportes de la aplicación de la técnica en otras especies de streptococcus como S. suis (22), y en otros géneros de bacterias como Haemobartonella felis (23), ehrlichias (24), Haemophilus parasuis (13), Francisella spp. (25) y H. pilori (26), entre otros.
En esta investigación se obtuvo una señal claramente positiva en el interior de los granulomas de los tejidos infectados por S. agalactiae y positivos por otras técnicas como inmunoperoxidasa indirecta (IPI) y microbiología; con todo, y a pesar de muchos ensayos, no fue posible la hibridación de las bacterias cuando se encontraban libres en los tejidos. La explicación más probable de la situación anterior, y sobre la cual se centraron la mayor parte de los esfuerzos por superar este inconveniente, tendría que ver con una característica propia de este tipo de cocos, cual es la presencia de una cápsula prominente sobre su superficie, posibilidad mencionada por algunos autores para este tipo de bacterias (27).
La cápsula abundante, compuesta principalmente de polisacáridos de alto peso molecular con unidades repetitivas de glucosa, galactosa, n-acetilgalactosamina y ácido siálico, es reconocida como uno de los principales factores de virulencia del S. agalactiae (28, 29), del S. iniae, L. garviae, S. suis y S. pneumoniae (30, 31, 32, 33, 34), entre otros. Mediante microscopia electrónica de transmisión se demostró la presencia de una prominente cápsula del S. agalactiae dentro de macrófagos en tejidos de tilapia infectados de manera natural con este microorganismo (3).
Mientras que la cápsula del S. agalactiae previo a ser fagocitado muy seguramente ha sufrido pocos cambios y, en consecuencia, impediría el acceso de la sonda al interior de la bacteria y su hibridación con la secuencia homóloga, en el interior de los granulomas la situación sería diferente, puesto que en esta ubicación, debido al procesamiento y la destrucción que sufre el microorganismo por el sistema defensivo del hospedero, muy probablemente su pared sería más permeable haciendo más accesible el RNA del patógeno a la sonda y, por ende, propicio para la hibridación con la misma.
Adicionalmente, hay que tener en cuenta que la expresión de la cápsula puede variar significativamente de acuerdo con estímulos ambientales y con la conveniencia de la bacteria. Existen reportes que sugieren cómo estas bacterias modulan la expresión de su cápsula dependiendo su localización en el hospedero, así cuando no están dentro de los tejidos no necesitarían tener una cápsula prominente y, de este modo, exponer sus proteínas de superficie necesarias para la adhesión y el ingreso; una vez dentro de los tejidos, por el contrario, sí necesitaría una buena cantidad de cápsula que le permitiera evadir la respuesta inmune (35, 36, 37, 38, 39, 40). De acuerdo con lo anterior, es posible que la diferencia en el éxito de la detección entre los autores que han reportado la HIS de S. agalactiae y lo obtenido en la presente investigación se deba a diferencias en la cantidad y la calidad de la cápsula presente al momento de su procesamiento; como se mencionó, estos autores han empleado la técnica con preparados en fresco como frotis de cultivo de sangre, de hisopados vaginales, de cultivos de bacterias obtenidos de líquido cefalorraquídeo y de abscesos peritonsilares en humanos, pero no en tejidos embebidos en parafina (14, 19, 20, 21).
Inicialmente sería dable pensar que la solución al inconveniente sería emplear tratamientos de permeabilización más drásticos que con las bacterias en solución; sin embargo, una de las principales consideraciones que debe observarse con la HIS es el balance que debe existir entre una óptima permeabilización y la conservación de la integridad de los tejidos.
Por ello no es indicado, por ejemplo, incrementar excesivamente las concentraciones de las enzimas o demás reactivos ya que el resultado puede ser igualmente negativo o alterarse la morfología de los tejidos de manera significativa. Infortunadamente, los autores que reportan la hibridación en tejidos embebidos en parafina con otros microorganismos gram (+) no son suficientemente explícitos en lo atinente a los tratamientos de permeabilización (13, 22, 23, 24, 25, 26).
En la presente investigación se ensayaron múltiples combinaciones en las concentraciones, la temperatura y los tiempos de incubación de los diversos reactivos recomendados para la permeabilización de los tejidos como la proteinasa K, lisozima, SDS, Triton X-100 y ácido metílico. Con respecto a la proteinasa K, por ejemplo, se hicieron ensayos a concentraciones de: 10, 100, 300, 500, 1000 µg/ml con diferentes tiempos y temperaturas. Finalmente, de acuerdo con el mejor resultado de marcación, se definió como el mejor tratamiento: 100 µg/ml x 60 min a 37oC. Esta concentración es mayor que la reportada por la mayoría de autores, que por lo general es de 1 a 25 µg/ml (24, 41, 42, 43, 44, 45).
Algunos autores reportan el uso de otras alternativas y combinaciones de tratamientos para optimizar la permeabilización, tales como el horno microondas por 15 minutos en buffer citrato (10mM, pH 6,0), la combinación de hidrólisis ácida y el tratamiento con enzimas, usando mutanolysina / lysozima o lipasa / proteinasa K (42, 46, 47), la mayoría de los cuales fueron probados en la presente investigación sin resultados positivos.
La especificidad de la sonda utilizada en el presente estudio pudo comprobarse de manera clara, la cual, con la metodología y los géneros y especies de bacterias incluidas, fue del 100%; este valor está de acuerdo con lo reportado por diversos autores para la HIS en éste y otros microorganismos similares (14, 21, 26).
Como se mencionó en los resultados, el hecho de no obtener hibridación positiva de las bacterias libres en los tejidos imposibilitó la determinación de la sensibilidad de la HIS y su comparación con los resultados de otras técnicas. artz et ál. (19), para la detección de S. agalactiae, encontraron en frotis vaginales una sensibilidad del 98,3% con la técnica de FISH, significativamente más alta que la encontrada mediante el cultivo bacteriano (64,4%). En el caso de otros patógenos, la HIS ha mostrado igualmente una mayor sensibilidad que el cultivo para diferenciar cepas patógenas y no patógenas de H. pilori en biopsias gástricas (45).
A pesar de la imposibilidad de hacer la evaluación efectiva del uso de la tiramida como método para la amplificación de la señal, debido a la ausencia de marcación de las bacterias libres en los tejidos, su efecto positivo pudo visualizarse a través de la mayor señal obtenida en el interior de los granulomas. Con este método se logra aumentar la sensibilidad en un rango de 2 a 100 veces (48, 49, 50, 51).
Son necesarios futuros estudios para optimizar la marcación de las bacterias libres en los tejidos y de este modo poder evaluar la sensibilidad de la técnica.
CONCLUSIONES
- Se obtuvo marcación con una especificidad del 100% del S. agalactiae en tejidos de tilapia (Oreochromis spp.) por medio de la técnica de hibridación in situ.
- La amplificación de la señal por el método de Card (catalyzed reporter deposition) mediante el uso de tiramida dio muy buenos resultados en granulomas, se prevén resultados similares con las bacterias individuales.
- La técnica de HIS para la detección del S. agalactiae en tejidos de peces puede ser muy sólida para el diagnóstico, la investigación epidemiológica y experimental.
- Son necesarios futuros estudios para lograr la marcación por medio de la técnica de hibridación in situ de las bacterias libres presentes en los tejidos infectados.
AGRADECIMIENTOS
La presente investigación fue financiada por la dirección nacional de investigaciones de la Universidad Nacional de Colombia. Los autores también agradecen la colaboración de los doctores Fernando Ariza, Noel Nerjan y Judith Figueroa por su soporte científico, y del señor Gilberto Córdoba por su ayuda en el procesamiento de los tejidos.
REFERENCIAS
1. Eldar A, Ghittino C, Bercovier H. Molecular taxonomy clarifies the status of Gram positive cocci fish pathogens. In: Abstracts book. European Association of Fish Pathologists. Seventh International Conference, Diseases of Fish and Shellfish. Palma de Mallorca; 1995. p. 25. [ Links ]
2. Evans JJ, Klesius PH, Shoemaker CA. Streptococcus in warm-water fish. Aquaculture Health International; 2006: 10-14. [ Links ]
3. Pulido EA, Iregui CA, Figueroa J. Reporte de streptococcosis en tilapias cultivadas en Colombia. Memorias II Congreso Suramericano de acuicultura. Puerto la Cruz. Venezuela. Tomo II; 1999. p. 229-239. [ Links ]
4. Iregui C, Hernández E, Jiménez A, Pulido A, Rey A, Comas J, Peña L y Rodríguez M. Primer mapa epidemiológico de las lesiones y enfermedades de los peces en Colombia. Universidad Nacional de Colombia, Facultad de Medicina Veterinaria y de Zootecnia, Grupo de Fisiopatología Veterinaria. Ministerio de Agricultura y Desarrollo Rural. Bogotá D. C.; 2004. [ Links ]
5. Jimenez AP, Rey AL, Penagos LG, Ariza MF, Figueroa J, Iregui CA. Estado actual de la estreptococosis en tilapias cultivadas en Colombia. Rev. Med. Vet. Zoot. 2007; 54: 120-123. [ Links ]
6. Hernández E, Figueroa J, Iregui C. Streptococcosis on a red tilapia, Oreochromis sp., farm: a case study. J Fish Dis 2009; 32: 247-252. [ Links ]
7. Toranzo AE, Devesa A, Heinen P, Riasa A, Nuñez S, Barja JL. Streptococcosis in cultured turbot caused by an Enterococcus-like bacterium. Bull Eur Assoc Fish Pathol 1994; 1: 19-23. [ Links ]
8. Doménech A, Fernández-Garayzábal J, Pascual C, García J, Cutuli M, Moreno M, Collins M, Domínguez L. Streptococcosis in cultured turbot Scophthalmus maximus (L), associated with Streptococcus parauberis. J Fish Dis 1996; 19: 33-38. [ Links ]
9. Eldar A, Ghittino C, Asanta L, Bozzetta E, Goria M, Prearo M, Bercovier H. Enterococcus seriolicida is a junior synonym of Lactococcus garvieae, a causative agent of septicemia and meningoencephalitis in fish. Curr Microbiol 1996; 32: 85-88. [ Links ]
10. Berridge BR, Bercovier H, Frelier PF. Streptococcus agalactiae and Streptococcus difficile 16s-23S intergenic rDNA: genetic homogeneity and species-specific PCR. Vet Microbiol 2001; 78: 165-173. [ Links ]
11. Boehringer Mannheim GmbH, Biochemica. Nonradioactive in situ hybridization application manual. Mannheim 31, Germany; 1992. [ Links ]
12. Cunningham CO. Molecular diagnosis of fish and shellfish diseases: present status and potential use in disease control. Aquaculture 2002; 206: 19-55. [ Links ]
13. Jung K, Chae C. In-situ hybridization for the detection of Haemophilus parasuis in naturally infected pigs. J Comp Path 2004; 130: 294-298. [ Links ]
14. Kempf VA, Trebesius K, Autenrieth IB. Fluorescent in situ hybridization allows rapid identification of microorganisms on blood cultures. J Clin Microbiol 2000; 38: 830-838. [ Links ]
15. Anderson ML. Nucleic acid hybridization. New York: Springer-Verlag; 1999. [ Links ]
16. Bohórquez IA, Botero L. Elaboración de sueros policlonales hiperinmunes contra Pasteurella multocida y Bordetella bronchiseptica y producción de un segundo antisuero conjugado con peroxidasa para su utilización en una técnica de inmunoperoxidasa indirecta. Tesis para optar por el título de Médico veterinario. Universidad nacional de Colombia, Facultad de Medicina Veterinaria y de Zootecnia, Bogotá D. C.; 1993. [ Links ]
17. Díaz CA. Desarrollo y evaluación de procedimientos serológicos e inmunohistoquímicos en el diagnóstico de la leptospirosis bovina. Tesis de maestría. Universidad Nacional de Colombia, Facultad de Medicina Veterinaria y de Zootecnia, Bogotá D. C.; 1995. [ Links ]
18. GeneDetect Limited. In-situ hybridization using GeneDetectTM oligonucleotide probes Greenstar BiOTIN-labeled probe, paraffin tissue sections, detection by AP (alkaline phosphatase) or HRP (Horseradish peroxidase) with or without tyramide signal amplification (TSA). (Last update: 1 june 2001, ver 1.2). Disponible en URL: http://www.genedetect.com/insitu.htm. [ Links ]
19. Artz LA, Kempf VA, Autenrieth IB. Rapid screening for streptococcus agalactiae in vaginal specimens of pregnant women by fluorescent in situ hybridization. J Clin Microbiol 2003; 41: 2170-2173. [ Links ]
20. Poppert S, Essig A, Stoehr B, Steingruber A, Wirths B, Juretschko S, Reischl U, Wellinghausen N. Rapid diagnostic of bacterial meningitis by real-time PCR and fluorescent in situ hybridization. J Clin Microbiol 2005; 43: 3390-3397. [ Links ]
21. Poppert S, Nickel D, Berger A, Yildiz T, Kaestner N, Mauerer S, Spellerberg B. Rapid identification of beta-hemolytic streptococci by fluorescent in situ hybridization (FISH). Int J Med Microbiol 2009; 299: 421-426. [ Links ]
22. Boye M, Feenstra AA, Tergtmeier C, Andresen LO, Rasmussen SR, Hansen VB. Deterction of streptococcus suis by in situ hybridization, indirect immunofluorescent, and peroxidaseantiperoxidase assays in formalin-fixed, paraffin-embedded tissues sections from pigs. J Vet Diag Invest 2000; 12: 224-232. [ Links ]
23. Berent LM, Messick JB,Cooper SK, Cusick PK. Specific in situ hybridization of Haemobartonella felis with a dna probe and tyramide signal amplification. Vet Pathol 2000; 37: 47-53. [ Links ]
24. Okada H, Usuda H, Tajima T, Kawahara M, Yoshino T, Rikihisa Y. Distribution of ehrlichiae in tissues as determined by in-situ hybridization. J Comp Path 2003; 128: 182-187. [ Links ]
25. Hsieh CY, Tung MC, Tu C, Chang CD, Tsai SS. Enzootics of visceral granulomas associated with Francisella-like organism infection in tilapia (Oreochromis spp.). Aquaculture. 2006; 254: 129-138. [ Links ]
26. Samarbaf-Zadeh AR, Tajbakhsh S, Moosavian SM, Sadeghi-Zadeh M, Azmi M, Hashemi J, Masjedi-Zadeh A. Application of fluorescent in situ hybridization (FISH) for the detection of Helicobacter pilori. Med Sci Monit 2006; 12: CR426-430. [ Links ]
27. Moter A, Göbel U. Fluorescence in situ hybridization (FISH) for direct visualization of microorganisms. J Microbiol Methods 2000; 41: 85-112. [ Links ]
28. Molinari G, Chhatwal GS. Streptococcal invasion. Curr Opin Microbiol 1999; 2: 56-6. [ Links ]
29. Spellerberg B. Pathogenesis of neonatal streptococcus agalactiae infections. Microbes Infect 2000; 2: 1733-1742. [ Links ]
30. Koedel U, Scheld WM, Pfister HW. Pathogenesis and pathophysiology of pneumococcal meningitis. Lancet Infect Dis 2002; 2: 721-36. [ Links ]
31. Barnes AC, Ellis AE. Role of capsule in serotypic differences and complement fixation by Lactococcus garvieae. Fish Shellfish Immunol 2004; 16: 207-214. [ Links ]
32. Chabot-Roy G, Willson P, Segura M, La-couture S, Gottschalk M. Phagocytosis and killing of streptococcus suis by porcine neutrophils. Microbial Pathogenesis 2006; 41: 21-32. [ Links ]
33. Vendrell D, Balcázar JL, Ruiz-Zarzuela I, de Blas I, Gironés O, Múzquiz JL. Lactococcus garvieae in fish: a review. Comp Immunol Microbiol Infect Dis 2006; 29: 177-198. [ Links ]
34. Agnew W, Barnes AC. Streptococcus iniae: An aquatic pathogen of global veterinary significance and a challenging candidate for reliable vaccination. Vet Microbiol 2007; 122: 1-15. [ Links ]
35. Wessels MR, Bronze MS. Critical role of the group A streptococcal capsule in pharyngeal colonization and infection in mice. Proc Natl Acad Sci 1994; 91: 12238-12242. [ Links ]
36. Schrager HM, Rheinwald JG, Wessels MR. Hyaluronic acid capsule and the role of streptococcal entry into keratinocytes in invasive skin infection. J Clin Invest 1996; 98: 1954-1958. [ Links ]
37. Adamou JE, Wizemann TM, Barren P, Langermann S. Adherence of Streptococcus pneumoniae to human bronchial epithelial cells (Beas-2B). Infect Immun 1998; 66: 820-822. [ Links ]
38. Schrager HM, Alberti S, Cywes C, Dougherty GJ, Wessels MR. Hyaluronic acid capsule modulates M protein-mediated adherence and acts as a ligand for attachment of group a streptococcus to CD44 on human keratinocytes. J Clin Invest 1998; 101: 1708-1716. [ Links ]
39. Winram SB, Jonas M, Chi E, Rubens CE. Characterization of group B streptococcal invasion of human chorion and amnion epithelial cells in vitro. Infect Immun 1998; 66: 4932-4941. [ Links ]
40. Pöhlmann-Dietze P, Ulrich M, Kiser KB, Döring G, Lee JC, Fournier JM, Botzenhart K and Wolz CH. Adherence of Staphylococcus aureus to endothelial cells: Influence of capsular polysaccharide, global regulator agr, and bacterial growth phase. Infect Immun 2000; 68: 4865-4871. [ Links ]
41. Ray RA, Smith M, Sim R, Nystrom M, Pounder RE and Wakefield AJ. The intracellular polymerase chain reaction for small CMV genomic sequences within heavily infected cellular sections. J Path 1995; 177: 171-180. [ Links ]
42. Oliver KR, Heavens RP, Sirinathsinghji DJ. Quantitative comparison of pretreatment regimens used to sensitive in situ hybridization using oligonucleotide probes on paraffin-embedded brain tissue. J Histochem Cytochem 1997; 45: 1707-1713. [ Links ]
43. Kobayashi K, Takeuchi H, Sasaki M, Hasegawa M, Hirai K. Detection of Epstein-Barr virus infection in the epithelial cells and lymphocytes of non-neoplasic tonsils by in situ hybridization and in situ PCR. Arch Virol 1998; 143: 803-813. [ Links ]
44. Morris DJ, Adams A, Richards RH. In situ hybridization of DNA probes to PKX, the causative organism of proliferative kidney diseases (PKD). J Fish Dis 1999; 22: 161-163. [ Links ]
45. Camorlinga-Ponce M, Romo C, González-Valencia G, Muñoz O, Torres L. Topographical localisation of cagA positive and cagA negative Helicobacter pylori strains in the gastric mucosa; an in situ hybridization study. J Clin Pathol 2004; 57: 822-828. [ Links ]
46. Carr EM, Eales K, Soddell J, Seviour RJ. Improved permeabilization protocols for fluorescent in situ hybridization (FISH) of mycolicacid-containing bacteria found in foams. J Microbiol Methods 2005; 61: 47-54. [ Links ]
47. Kitayama Y, Igarashi H, Kozu T, Nagura K, Ohashi Y, Sugimura H. Repeated fluorescent in situ hybridization by a microwave-enhanced protocol. Pathol Int 2006; 56: 490-493. [ Links ]
48. Komminoth P, Werner M. Target and signal amplification: approaches to increase the sensitivity of in situ hybridization. Histochem Cell Biol 1997; 108: 325-333. [ Links ]
49. Von Wasielewski R, Mengel M, Gignac S, Wilkens L, Werner M, Georgii A. Tyramine amplification technique in routine immunohistochemistry. J Histochem Cytochem 1997; 45: 1455-1459. [ Links ]
50. Speel EJ. Detection and amplification systems for sensitive, multiple-target DNA and RNA in situ hybridization: looking inside cells with a spectrum of colors. Histochem Cell Biol 1999; 112:89-113. [ Links ]
51. Wierdorn KH, Kühl H, Galle J, Caselitz J, Vollmer E. Comparison of in-situ hybridization, direct and indirect in-situ PCR as well as tyramide signal amplification for the detection of HPV. Histochem Cell Biol 1999; 111: 89-95. [ Links ]














