Introducción
El Virus de la Diarrea Viral Bovina (VDVB), perteneciente al género Pes- tivirus, familia Flaviviridae (Wengler et al. 2001), es una infección que afecta a los hatos bovinos con diferentes manifestaciones clínicas que pueden ir desde formas asintomáticas (Fulton et al. 2005), hasta cuadros agudos de carácter entérico, reproductivo o respiratorio (Fulton et al. 2005; Ridpath et al. 2006). Los efectos generados por la enfermedad incluyen bajos índices de crecimiento en terneros (Xue et al. 2010), deficiencias reproductivas y susceptibilidad a la presentación de otras patologías.
Los Pestivirus han sido divididos en 5 especies de acuerdo con sus diferencias genómicas: el Virus de la Peste Porcina Clásica (cepa de referencia Alfort), Enfermedad de la Frontera (BD31), Diarrea Viral Bovina genotipo 1 (NADL), Diarrea Viral Bovina genotipo 2 (890) y un posible virus de Diarrea Viral Bovina genotipo 3 o “HoBi virus” que ha sido recientemente propuesto (Liu et al. 2009; Bauermann et al. 2013). Para clasificar el VDVB en alguno de los tres genotipos se emplea el análisis de la secuencia de la región no traducible 5’UTR del RNA viral, que corresponde a la porción más conservada del genoma (Ridpath 1994; Tajima et al. 2001; Couvreur et al. 2002). Los genotipos 1 y 2 han sido a su vez clasificados en subgenotipos que han mostrado una homología entre el 80-85% (Fulton et al. 2005), los más representativos dentro de cada uno de los genotipos son el VDVB-1a, VDVB-1b, VDVB-2a y VDVB-2b (Ridpath et al. 1994; Flores et al. 2002). Adicionalmente, dentro de cada genotipo se pueden encontrar dos biotipos: no citopático (NCP) y citopático (CP) de acuerdo con la capacidad que estos tengan de inducir lisis en cultivos celulares (Peterhans y Schweizer 2010). El biotipo NCP es el más abundante en la naturaleza, a partir de este se generan las cepas CPs y también es el responsable de una de las manifestaciones más importantes de la enfermedad: la de animales persistentemente infectados (PI) (Brogden 2002).
Hasta la fecha, el estudio realizado en Colombia ha demostrado que el VDVB genotipo 1 (VDVB-1) es el de mayor distribución en el país y el que causa mayores problemas reproductivos (Vargas 2010). Estas cepas son conocidas como las clásicas y son empleadas en la elaboración de vacunas, en pruebas diagnósticas y en investigación (Fulton et al. 2005). Por su parte, los genotipos 2 y 3 tienen menos incidencia a nivel mundial y aún no se ha determinado su presencia y efectos en el territorio nacional.
Además de las diferencias genómicas, los genotipos difieren en su comportamiento patogénico: el VDVB-1 se aisló de animales que presentaron cuadros de diarrea aguda y enfermedad de las mucosas; mientras que el VDVB-2 se aisló de animales que murieron infectados por la forma severa aguda de DVB, en terneros PI y en suero fetal bovino (SFB) contaminado (Ridpath et al. 1994). Análisis filogenéticos de este genotipo han logrado la identificación de dos especies: VDVB-2a y VDVB-2b (Flores et al. 2002; Mishra et al. 2008).
La forma más severa de la infección por VDVB es la asociada con los biotipos NCP, quienes causan una enfermedad clínica aguda, que cursa con trombocitopenia y un sangrado profuso en los bovinos, conocida como síndrome hemorrágico (Ridpath et al. 1994). Este sindrome se reportó inicialmente en Estados Unidos y Canadá (Pellerin et al. 1994); se caracteriza porque los animales presentan fiebre alta, leucopenia, trombocitopenia, diarrea, muerte en terneros adultos (Vargas 2010) y en algunos casos, enfermedad respiratoria severa, epistaxis, petequias, equimosis en mucosas y anemia. Adicionalmente, puede provocar deficiencias en las tasas de fertilización, vacas repetidoras, abortos, malformaciones congénitas y el establecimiento de animales PI que eliminan el VDVB durante largos periodos de tiempo (Bauermann et al. 2013). Por su parte, la infección aguda con el biotipo CP del VDVB-2 resulta en una enfermedad subclínica o con sin- tomatología reproductiva, respiratoria y digestiva (Vargas 2010). Como se mencionó anteriormente, las diferentes cepas del virus varían considerablemente en su virulencia, en general, la mayoría de casos de la forma hemorrágica han sido atribuidos al VDVB-2 (Makoschey et al. 2001).
Metodología
Fincas
Se muestrearon 15 fincas distribuidas en cuatro de las regiones ganaderas más representativas de Colombia ubicadas así: Mosquera, Cundinamarca (2), Medellín, Antioquia (3), Tierra de los Santos, Santander (1) y Cali, Valle del Cauca (9). Las fincas elegidas para el estudio se seleccionaron al azar de acuerdo con la disponibilidad de los propietarios para participar en la investigación.
Animales
El muestreo se realizó durante el periodo comprendido entre febrero a octubre de 2014. Los animales incluidos en el mues- treo correspondieron a hembras con una gestación aproximada de 240-255 días de razas Holstein, Jersey, Pardo Suizo, Simmental, 7 colores y Ayrshire. En la Tabla 1 se detalla la cantidad de animales muestreados por grupo y por región.
Muestras
Se tomaron dos muestras de sangre de hembras bovinas preñadas: la primera, un mes antes del parto, denominada muestra preparto (PP) y la segunda, 20 días después del parto, denominada muestra posparto (POP). Adicionalmente, se tomaron dos muestras de sangre de cada uno de los terneros nacidos: la primera, luego de nacer antes de que el animal consumiera calostro, denominada muestra precalostral (PC) y la segunda, 25 días después del nacimiento (25 DD). Finalmente, se tomaron 2 biopsias de cartílago de oreja de cada uno de los terneros pertenecientes al estudio, denominada muesca de oreja (MO) (Tabla 2).
TABLA 2 Distribución de muestras de suero y cartílago auricular obtenidas
| Hembras | Terneros | |||||
|---|---|---|---|---|---|---|
| Región | PP | POP | PC | 25 DD | MO | |
| Cundinamarca | 13 | 13 | 0 | 0 | 3 | |
| Santander | 101 | 101 | 121 | 90 | 87 | |
| Antioquia | 74 | 74 | 67 | 13 | 35 | |
| Valle del Cauca | 191 | 191 | 86 | 42 | 56 | |
| Total | 379 | 379 | 274 | 145 | 181 | |
PP: Preparto, POP: Posparto, PC: Precalostral, 25DD: 25 días después de nacidos, MO: Muesca de oreja.
Las muestras de sangre de las hembras (7-10 ml) se obtuvieron a partir de la vena coccígea y en los terneros (5-7 ml), a partir de la vena yugular; en los dos casos, las muestras se colectaron en tubos Vacutainer® sin anticoagulante. Una vez colectadas las muestras, se refrigeraron a 4°C y se procesaron dentro de las siguientes 18 horas en el Laboratorio de Virología Animal de la Universidad Nacional de Colombia, Sede Bogotá. Los tubos se centrifugaron entre 1300 a 2000 g durante 10 min y el suero se recolectó y almacenó a -70°C hasta su procesamiento. En la Tabla 2 se describe la distribución de las muestras de suero en hembras (PP y POP) y terneros (PC, 22 DD y MO).
Para obtener las MO de terneros, las orejas se lavaron con agua retirando restos de heces, tinta de tatuaje, leche, entre otros contaminantes que podrían afectar la biopsia (WVDL 2012); posteriormente, se tomó 1 cm del extremo de la oreja (Cornish et al. 2005) empleando la pinza ProSampler® (ProSampler’s single-hole ear punching tool, Scandia, USA) y se depositó en un tubo eppendorf que contenía 200 µl de agua tratada con dietilpirocarbonato (DEPC por sus siglas en inglés). Las muestras se refrigeraron a 4°C y se trasladaron al laboratorio, donde se almacenaron a -70°C para su posterior análisis.
PCR para la detección de los genotipos virales
Las MO se sometieron a disrupción por maceración y se sonicaron a 16 kHz durante 30 seg; posteriormente, se centrifugaron a 8000 rpm durante 5 min y se recuperó el sobrenadante a partir del cual se hizo extracción del ARN empleando el kit QIAamp® Viral RNA (Qiagen CA®). Como control positivo, se hizo extracción a partir de cultivos de células MDBK infectadas independientemente con las cepas VDVB-1 (NADL) y VDVB-2 (VS-260). Como control negativo, se realizó extracción de ARN de agua DEPC. El ARN obtenido se almacenó a -70°C hasta su procesamiento. La síntesis del ADNc a partir del RNA se hizo mediante RT-PCR; para esto, se mezcló el ARN (5 pl) con Random Primers (InvitrogenTM) durante un ciclo de 65°C por 3 min. Posteriormente, se realizó la RT siguiendo el protocolo de Zhang et al. (2012) mediante el empleo de la trans- criptasa reversa M-MVL (Invitrogen®), durante un ciclo de síntesis de ADNc de 60 min a 56°C, seguido de una fase de pre-denaturación a 94°C durante 5 min. La RT-PCR se realizó en el termociclador Biorad® DNA Engine. El ADNc se almacenó a -20°C para posteriores análisis. A través de PCR se amplificó inicialmente un segmento de 288pb de la región 5,UTR del genoma de VDVB común para los Pestivirus empleando los primers: F324: 5’ATGCC- CWTAGTAGGACTAGCA 3’ y R326: 5’TCAACTCCATGTGCCATGTAC 3’ (Vilcek et al. 1994). Como control se utilizó la cepa de referencia NADL. Se tomaron 5µl de ADNc junto con 20 µl de mezcla maestra para PCR (1,5 µl de Cloruro de Magnesio; 12,3µl de Agua; 1,5µl de dNTPs; 2,5 µl de Buffer 10x; 0,2 µl de Taq polimerasa (InvitrogenTM); 1 µl primer forward y 1 µl de primer reverse). Las condiciones de la PCR fueron: denaturación a 94°C durante 30 seg, anillamiento a 53°C por 30 seg y extensión a 72°C durante 30 seg, en 35 ciclos.
Posteriormente, el ADNc de aquellas muestras que resultaron positivas a la amplificación de Pestivirus se sometieron a otra PCR para determinar si correspondían al VDVB-2; para ello, se emplearon primers específicos (Letellier 1999) que amplifican un segmento de 221pb sobre la región 5’UTR (5’UTR F: 5’ ACTAGCGGTAGCAGTGAG 3’ y 5’UTR R: 5’ CTAGCGGAATAGCA- GGTC 3’). Se adicionaron 5µl de ADNc a cada tubo con 20 µl de mezcla para PCR (1,5µl de Cloruro de Magnesio; 12,3µl de Agua; 1,5µl de dNTPs; 2,5µl de Buffer 10x PCR; 0,2µl de Taq polimerasa (Platinum Taq DNA Polymerase, InvitrogenTM; 1 µl de primer forward y 1µl de primer reverse). Las condiciones de PCR fueron: denaturación a 94°C durante 1 min, anillamiento a 51°C por 1 min y extensión a 72°C durante 1 min, en 35 ciclos.
Digestión enzimática para la diferenciación de genotipos 1 y 2
Con el fin de confirmar aquellas muestras que resultaron positivas por PCR para el genotipo 2, se hizo digestión con la enzima BstNI. Para esto, se realizó una nueva PCR empleando otros primers sobre la región 5’UTR (F-OPES13Aj 5’GCTAGCGAT- GCCCTTAGTAGGA3’, R-OPESl4A:5’ ATCAACTCCATGTGCCATTTACAGC 3’) reportados por Stàhl et al. (2005) generando un amplificado de 296 pb. La PCR se realizó en un termociclador Biorad® DNA Engine bajo las siguientes condiciones: 3 min a 94°C, seguido por 35 ciclos de 30 seg a 94°C, 30 seg a 50°C, 30 seg a 72°C y una extensión final de 5 min a 72°C. Posteriormente, los amplicones fueron digeridos tomando 5 µl del producto de la PCR, 3,5 µl de agua DEPC, 1 µl de buffer de la enzima (10X NEB Buffer) y 0,5 µl de BstNI (New England BioLabs Inc.®). Se incubó a 60°C durante 1 hora. Las digestiones se sometieron a electrofo- resis en gel de agarosa (Sigma®) al 1,5%, a un voltaje constante de 90V durante una hora. Los geles se visualizaron mediante revelado por inmersión con el agente intercalante GelRed (Thermo Scientific ®) y se evaluaron en fotodocumentador (Gel Doc 1000, Biorad®, CA). Como controles positivos para la digestión enzimática se emplearon las cepas de referencia para el VDVB-1: NADL y para el VDVB-2: VS-260. Como control negativo se hizo extracción de ARN de agua DEPC. Para realizar la extracción, se empleó el kit QIAamp® Viral RNA (Qiagen CA®) según el procedimiento Qiagen One Step RT-PCR (2002).
Resultados
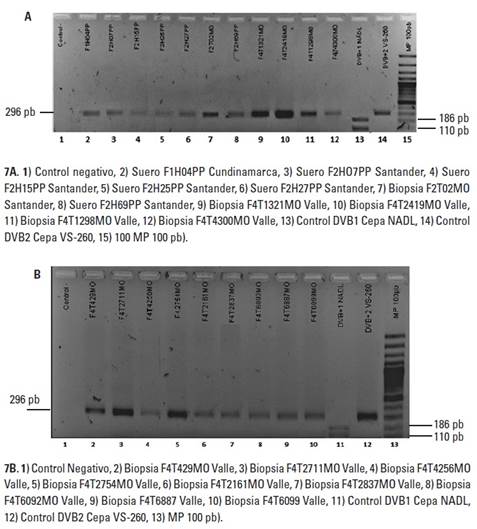
Mediante RT-PCR se amplificaron las diferentes muestras (PP, POP, PC, 25DD y MO) para evidenciar la presencia de un amplicón de 288 pb, correspondiente a Pestivirus (Figura 1A). Posteriormente, se amplificó otro segmento de la región 5’UTR con primers específicos para el VDVB-2, obteniendo como resultados positivos aquellos que amplificaron un segmento de 221 pb (Figura 1B).
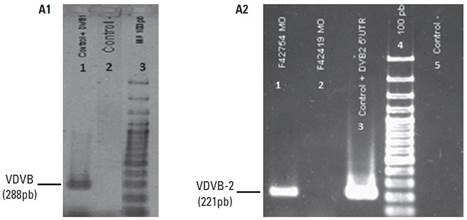
FIGURA 1 Amplificación por RT-PCR para detección del VDVB-2. 1A. (1) Control positivo VDVB Cepa NADL; (2) Control negativo; (3) Marcador de peso molecular (pb). 1B. (1) Muestra positiva de muesca de oreja al VDVB-2; (2) Muestra negativa de muesca de oreja al VDVB-2; (3) Control positivo VDVB-2 Cepa VS-260; (4) Marcador de peso molecular (pb); (5) Control negativo.
En las fincas de la región de Cundinamarca, de 13 muestras de suero PP de hembras, una (7,69%) resultó positiva a Pestivirus y correspondió al VDVB-2. Entre las 4 muescas de oreja de los terneros no se detectaron positivos a Pestivirus.
Por su parte, en la finca de la región de Santander, se encontró que de 101 muestras de suero PP de las hembras, 8 (7,92%) amplificaron para el VDVB, de las cuales, 5 (4,95%) correspondieron al VDVB-2 y 3 (2,97%) al VDVB-1. Por otro lado, de las 121 muestras de suero PC obtenidas de los terneros, 2 (1,65%) fueron positivas al VDVB-1; a su vez, de los 90 sueros colectados 25 días después del nacimiento en los terneros (25 DD), se encontró que 2 (2,22%) resultaron positivos al VDVB-1, correspondiendo a los mismos que fueron positivos en la muestra PC. Finalmente, de las 87 muescas de oreja obtenidas, se encontraron 3 biopsias (3,45%) positivas al VDVB-1 y 1 muestra (1,15%) positiva al VDVB-2 (Figura 2).
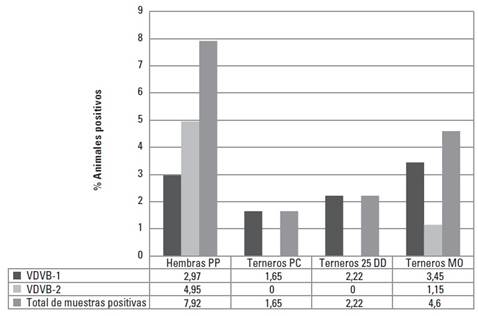
FIGURA 2 Porcentaje de bovinos positivos por RT-PCR al VDVB y VDVB-2 en fincas de la región de Santander. PP: Preparto, PC: Precalostral, 25DD: 25 días después de nacidos, MO: Muesca de oreja.
En cuanto a las fincas de la región del Valle del Cauca se encontró que de 191 sueros PP de hembras, 5 (2,6%) fueron positivos al VDVB-1. En los terneros, de 86 muestras de suero PC, 3 (3,2%) fueron positivas al VDVB-1. En el caso de los sueros 25 DD ninguna de las 42 muestras resultó positiva al VDVB. Por su parte, las muescas de oreja obtenidas de los terneros, demostraron que de 56 biopsias de cartílago auricular, 13 (23,2%) amplificaron para el VDVB y todas correspondían al VDVB-2 (Figura 3).
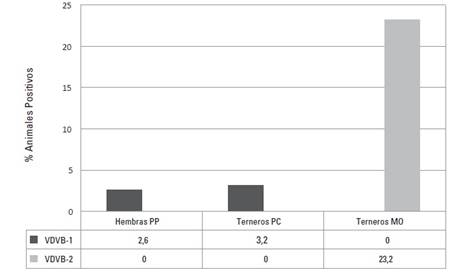
FIGURA 3 Porcentaje de bovinos positivos por RT-PCR para el VDVB-1 y VDVB-2 en fincas del Valle del Cauca. PP: Preparto, PC: Precalostral, MO: Muesca de oreja.
La distribución de los genotipos virales en sueros PP de hembras por zona evaluada, mostró la presencia de ambos genotipos en la finca del departamento de Santander, mientras que en los predios de las regiones Antioquia y Valle del Cauca se encontró únicamente el VDVB-1, en contraste con las fincas evaluadas de la región de Cundinamarca en donde se evidenció solamente el VDVB-2 (Figura 4). Para los terneros, al comparar por zonas de muestreo, se determinó que el VDVB-2 solo se encontró en muestras de MO y estuvo presente únicamente en los departamentos de Santander y del Valle del Cauca con 0,5% y 7,1%, respectivamente (Figura 5).
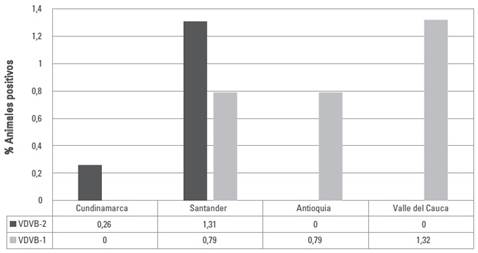
FIGURA 4 Distribución de genotipos virales 1 y 2 del VDVB en sueros preparto (PP) de hembras bovinas en las cuatro regiones evaluadas.
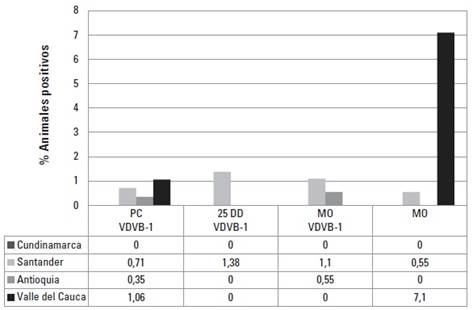
FIGURA 5 Distribución de los genotipos virales 1 y 2 del VDVB en sueros PC, 25 DD y MO en terneros en fincas de las cuatro regiones evaluadas. PC: Precalostral, 25DD: 25 días después de nacidos, MO: Muesca de oreja.
Finalmente, todas las muestras que resultaron positivas por RT-PCR al VDVB-2 se confirmaron mediante digestión de la enzima BstNI, lo que evidenció que la enzima cliva únicamente el genoma del VDVB-1, dividiendo el fragmento en 186 y 110 pb (Figura 6). Según el patrón observado se confirmaron como VDVB-2 13 muestras MO de terneros del Valle del Cauca, 5 muestras de suero sanguíneo PP de hembras de Santander, 1 MO de un ternero de Santander y 1 muestra de suero sanguíneo PP de una hembra procedente de Cundinamarca (Figura 7).
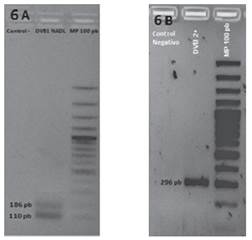
FIGURA 6 Digestión enzimàtica en controles positivos para VDVB-1 y VDVB-2. Digestión realizada empleando la enzima BstNI: 6A. Clivaje del producto del control de la cepa de referencia NADL para el VDVB-1 correspondiente a 186 pb y 110 pb. 6B. Producto del control obtenido de la cepa de referencia VS-260 correspondiente a 296 pb
Discusión
En el presente estudio se determinó la presencia del VDVB en todas las regiones evaluadas, mientras que el VDVB-2 se encontró por primera vez en el país en fincas ubicadas en las regiones de Cundinamarca, Santander y Valle del Cauca. El VDVB-2 se describió inicialmente en Estados Unidos en un caso fatal de la Enfermedad de las mucosas a finales de los años 80’s (Corapi et al. 1989; Carman et al. 1998), en Canadá (USDA 1995), en Europa hace varios años (Wolfmeyer et al. 1997; Van Rijin et al. 1997; Vilcek et al. 2001; Falcone et al. 2001) y en Japón (Nagai et al. 1998). En Suramérica, el VDVB-2 se detectó por primera vez en Brasil, en donde los análisis filogenéticos sugieren que éstas cepas difieren genéticamente del VDVB-2 aislado en Norteamérica (Flores et al. 2002) y en Argentina (Odeón et al. 2003). Recientemente, se detectó en África, específicamente Suráfrica, donde se demostró una similitud en la secuencia con la cepa de referencia 890 proveniente de Norteamérica, (Ularamu et al. 2013).
Los resultados del presente trabajo además de demostrar la presencia del genotipo 2 del VDVB en Colombia, aportan una metodología diagnóstica para la detección del mismo a través de PCR sobre la región 5’UTR del genoma viral (Couvreur et al. 2002; Yilmaza et al. 2012). Esta región constituye la zona del genoma viral más empleada para su caracterización (Wolfmeyer et al. 1997; Flores et al. 2002) ya que es altamente conservada (Cortez et al. 2006) y permite una óptima amplificación (Ridpath 1994; Ridpath y Bolin 1995). Es importante mencionar que en 3 de las 4 regiones muestreadas en este estudio se evidenció la presencia del VDVB-2, lo que constituye un aporte importante para valorar la enfermedad dentro del país. En países en donde se han realizado estudios similares, se han evaluado regiones geográficas específicas como en el caso de Túnez (Thabti et al. 2005), Brasil (Weber et al. 2014), Inglaterra (Wakeley et al. 2004) y Alemania (Tajima et al. 2001), por lo que es pertinente resaltar, que en concordancia, en la presente investigación se evaluó la presentación de los genotipos de forma simultánea en varias regiones geográficas de Colombia.
Aunque la cantidad de muestras de suero obtenidas de hembras en las fincas de la región de Cundinamarca fue pequeña (13) se logró detectar la presencia del VDVB-2, lo que conduce a la hipótesis de que existe una alta probabilidad de que este genotipo se encuentre en otros bovinos de la zona. En la región de Santander se encontró el mayor porcentaje de hembras positivas al VDVB-2 (4,48%), superior comparativamente con los predios de las otras regiones estudiadas, lo que podría indicar una alta circulación del genotipo en la zona. El origen y la fecha de introducción del VDVB-2 en Colombia son inciertos y no se puede establecer mediante el presente estudio, para ello se hace necesaria una investigación retrospectiva. A falta de estos datos varias hipótesis pueden ser propuestas: es probable que la introducción del VDVB-2 sea reciente y se haya producido a través de la importación de ganado o semen (Givens et al. 2003) de países en donde se ha demostrado la presencia de este genotipo, como en Brasil y Argentina. Se podría sospechar también que su introducción pudo deberse al uso de vacunas contaminadas con Pestivirus, como ha sido reportado en la literatura (Falcone et al. 2001). Adicionalmente, puede deberse a la implementación de diferentes técnicas biotecnologías reproductivas en los hatos sin que se realice seguimiento sanitario periódico. Es posible que semen contaminado de toros infectados o toros PI puedan transportar el virus como también es posible que el semen de toros que presenten infecciones testiculares persistentes puedan infectar a las hembras (Martínez y Riviera 2008). Finalmente, gametos y otras células derivadas de algún animal enfermo representan una fuente importante de contaminación cuando estos son empleados en la producción y transferencia de embriones (Martínez y Riviera 2008).
En cuanto a la presencia de animales PI en el estudio, se podría sugerir que aquellos terneros que resultaron positivos al virus por RT-PCR en la muestra de suero PC y en la muestra obtenida a los 25 DD (Finca de la región de Santander) podrían ser eventualmente animales PI, debido a que bovinos con este tipo de infección nacen con viremia persistente, siendo ambas muestras positivas en el tiempo a la detección de genoma viral y negativas a anticuerpos (OIE 2008). Adicionalmente, en el presente estudio se evaluaron muestras de suero y cartílago auricular mediante la técnica de RT-PCR (Amer y Almajhdi 2011), como ya se ha reportado en otros estudios (Hertig et al. 1991). Las muestras de cartílago de oreja se han convertido en el tejido de elección para detección de genoma viral ya que son fáciles de colectar, el equipo necesario es mínimo, no existe alteración de la muestra a causa de anticuerpos pasivos y se pueden emplear en diferentes técnicas diagnósticas como IHC, qRT-PCR, RT- PCR y AgELISA (Ridpath et al. 2006).
En el presente estudio se obtuvo un porcentaje de detección del 9,39% lo que coincide con lo reportado en otras investigaciones como en la realizada por Fulton et al. (2006), donde se evaluaron MO de 21.743 terneros mediante diferentes técnicas y se encontró una prevalencia de animales PI del 0.4%. La tasa de detección para VDVB encontrada en el presente estudio se puede atribuir al hecho de que únicamente se tomaron biopsias de los terneros y no de las hembras incluidas en el muestreo. Sin embargo, lo relevante es que se logró realizar la detección del VDVB-2, particularmente en terneros del Valle del Cauca, lo que permite dar resultados acerca de la dinámica de los genotipos virales en el país. Igualmente, se encontraron únicamente 8 sueros positivos al virus en los terneros evaluados, frente a 20 biopsias de cartílago auricular positivas y llamativamente 13 de esas muestras correspondían al VDVB-2. Este resultado lleva a varias reflexiones: por un lado que la probabilidad de encontrar virus en suero no es muy alta, esto se explica porque la muestra debe coincidir con la presencia de un animal en estado de viremia o de persistencia viral; por otra parte, la MO de acuerdo con nuestros resultados constituye una muestra muy eficiente en detección de genoma viral en animales aparentemente sanos y particularmente para determinar la presencia de genotipos. En efecto, según Ridpath et al. (2006) las biopsias de oreja cuentan con la ventaja de ser relativamente más estables que la sangre debido a que pueden mantenerse almacenadas a temperatura ambiente (25°C) hasta por 7 días, lo que permitiría realizar muestreos en lugares que no cuenten con sistemas de refrigeración y además podrían procesarse posteriormente sin perder la precisión para el diagnóstico del VDVB.
Las muestras positivas al VDVB-2 por RT-PCR se confirmaron mediante digestión enzimática con BstNI como ha sido reportado en varios estudios (Vilcek 1994) para diferenciar entre Pestivirus como VDVB, CSFV y BDV y entre genotipos del VDVB. En el presente estudio, se confirmaron las muestras positivas al genotipo 2 del VDVB, lo que coincide con la literatura reportada por Pizarro-Lucero etal. (2006), en donde se aislaron 33 Pestivirus de animales de diferentes granjas de Chile amplificando la región 5’UTR del genoma viral con una posterior digestión, empleando las enzimas de restricción Bgl I, XhoI y PstI; 16 de los aislamientos se clasificaron como VDVB-1 y 17 como VDVB-2.
En Colombia cualquier investigación realizada en casos individuales y pobla- cionales del VDVB debe mantenerse encaminada a controlar el virus dentro de los hatos, identificando los animales infectados que podrían constituir una amenaza epidemiológica, precisando la detección y genotipificación del VDVB. Es importante resaltar que aquellas muestras que resultaron positivas a Pestivirus podrían corresponder eventualmente al VDVB-3, por lo que se sugiere un estudio que determine la posible presencia de los HoBi-like virus en el país.
Es importante realizar estudios que correlacionen variables tales como el número de parto, alimentación, manejo, condiciones climáticas yregión del país con la presentación del VDVB-2. Así mismo, hacer un seguimiento de la sintomatología clínica de las hembras y terneros dentro de las explotaciones como método para detectar los animales PI. Igualmente, se recomienda realizar un estudio en el cual se implementen técnicas inmunohisto- químicas para el diagnóstico de bovinos PI en las muescas de cartílago de oreja almacenadas debido al reconocido epi- teliotropismo del VDVB. (Thür et al. 1996; Njaa et al. 2000).
Éste estudio ratifica la presencia del VDVB en las zonas de mayor producción ganadera del país, adicionalmente, representa la primera evidencia documentada del genotipo 2 del VDVB en bovinos de Colombia lo que constituye un nuevo elemento para tener en cuenta en los programas de control de la enfermedad en el país, llevando al mejor conocimiento de las cepas que se encuentran dentro y entre hatos e igualmente, contribuye a verificar si los planes de vacunación empleados contra el VDVB en Colombia cubren inmunológicamente a los animales expuestos contra los virus circulantes.
Conclusiones
El presente estudio representa la primera evidencia documentada del genotipo 2 del VDVB en Colombia; el origen y la fecha de introducción de este genotipo en el país son inciertos y no se puede establecer mediante el presente estudio. En 3 de 4 de las regiones geográficas estudiadas se evidenció la presencia del VDVB-2 y fue posible detectar los genotipos 1 y 2 de forma simultánea. En el país son pocos los estudios enfocados a la detección del VDVB mediante técnicas moleculares, el presente es el primero que combina análisis de detección molecular junto con la diferenciación de genotipos en muestras de suero y cartílago de oreja. Aunque la cantidad de muestras de suero de hembras obtenidas de la región de Cundinamarca fue pequeña (13 muestras), éstas permitieron determinar la presencia del VDVB-2, lo que lleva a pensar que existe una considerable probabilidad de que el genotipo 2 esté en otros bovinos de la zona.
Los resultados positivos al VDVB-2 se confirmaron mediante digestión enzimática dado que las endonucleasas de restricción son ampliamente empleadas para diferenciar los genogrupos pertenecientes al género de los Pestivirusl. En el presente estudio se empleó la enzima BstNI con la que se confirmaron las muestras positivas al genotipo 2 del VDVB, lo que coincide con la literatura reportada por Pizarro-Lucero et al. (2006). A partir de ésta investigación, se confirmó que es posible realizar la genotipificación del virus mediante digestión enzimática, al clasificar 16 aislamientos como VDVB-1 y17 aislamientos como VDVB-2. La secuen- ciación realizada mostró una alta similitud con la cepa de referencia VS-123.4 que correspondía a un aislamiento del VDVB-2 de Brasil, lo que podría ser atribuido a la cercanía geográfica con ese país o por la introducción de ganado y/o semen contaminado.
La muestra de elección para detectar el genotipo 2, tanto por su manejo como por la probabilidad de encontrar el virus, es la muesca de oreja que puede ser evaluada mediante RT-PCR o por AgELISA, con niveles de confiabilidad del 95% al ser comparada con otras muestras como suero sanguíneo, saliva e hisopados nasales.
Recomendaciones
Aquellas muestras que resultaron positivas al VDVB-1 podrían corresponder eventualmente al VDVB-3, por lo que se sugiere un estudio que determine la posible presencia de los HoBilike virus en el país. Adicionalmente, se deben realizar estudios posteriores que correlacionen variables tales como el número de parto, alimentación, manejo, condiciones climáticas y región del país con la presentación del VDVB-2. De igual forma, se recomienda realizar un seguimiento de la sintomatología clínica de las hembras y terneros dentro de las explotaciones, como método para detectar los animales PI e implementar técnicas IHC para el diagnóstico de bovinos PI.