INTRODUCCIÓN
Chlamydia psittaci (Cp) es una bacteria intracelular obligada que posee la capacidad de infectar más de 460 especies de aves (Kaleta y Taday 2003). Desde el punto de vista del hospedador posee diferencias filogenéticas, donde existe especificidad a especies aviares y mamíferas (Sachse 2015). Sin embargo, Cp ha diversificado su capacidad de infectar a múltiples especies, y es considerada un factor de riesgo mortal para especies en cautiverio (Frutos et al. 2015). La transmisión de Cp ocurre mediante la inhalación de aerosoles provenientes de secreciones nasales, orina, heces secas, tejidos y plumas de animales portadores de la bacteria (Sachse et al. 2015). Como consecuencia, en los centros de cautiverio existe un riesgo de transmisión permanente para diversas especies animales que de esta manera pueden llegar a desarrollar clamidiosis (Rodríguez-Leo et al. 2017a).
La clamidiosis es una infección del tracto respiratorio superior relacionada con conjuntivitis serosa y sinusitis, puede ser limitada o progresar rápidamente a neumonía, septicemia, hepatitis, esplenitis y en ocasiones muerte súbita del ave. En mamíferos generalmente es sub-clínica, sin embargo, puede generar letargo, anorexia, crecimiento retardado de crías, infertilidad, diarrea, cuadros graves de neumonía, queratoconjuntivitis y poliartritis (Sachse et al. 2015). Recientemente Cp ha adquirido mayor relevancia a nivel mundial en esta clase de animales; Bhardwaj et al. (2015) asociaron a Cp como causante de neumonía (72,7%) y abortos (78,9%) en ovejas y cabras de vida libre en el Himalaya. En Alemania, Sprague et al. (2009) reportaron la presencia de Cp en cuatro caninos de una casa de crianza, las cuales presentaban cuadros recurrentes de queratoconjuntivitis, dificultad respiratoria y mortalidad del 50% en crías, asumiendo que la persistencia del cuadro se debe a la circunstancial introducción de tres psitácidos positivos para Cp.
El contacto previo con aves infectadas principalmente del orden Psittaciforme es un factor predisponente para la adquisición de Cp, por ello son necesarios métodos diagnósticos para la identificación de este agente. La implementación de técnicas moleculares para el diagnóstico de Cp de fauna en cautiverio, permite generar información para la creación de mecanismos epidemiológicos que eviten la diseminación de este microorganismo, sobre todo en Venezuela donde no existe un diagnóstico habitual de este agente en centros de cautiverio. Rodríguez-Leo et al. (2017b) demostraron elevada endemicidad en aves de diversos órdenes y señalan el riesgo de transmisión de Cp a mamíferos adyacentes a las jaulas de las aves positivas para este patógeno. Por ello, el presente trabajo tuvo como objeto la búsqueda de Cp en individuos de hurón sable (Mustella putorios furo) en cautiverio que habitaban junto a una perdiz crestada (Colinus cristatus) con características clínicas de clamidiosis aviar.
MATERIALES Y MÈTODOS
Recolección de muestras
A partir de cinco individuos de hurón sable (Mustelasputorios furo) y una perdiz crestada (Colinus cristatus) en cautiverio, se recolectaron muestras de hisopados rectal y cloacal respectivamente. Para ello, los hurones fueron capturados con guantes de lona para la recolección de muestras de hisopados rectales, se lavó la región anal con una gasa y SSF 0,9% estéril, a continuación se introdujo un hisopo estéril en el recto de cada ejemplar; durante este proceso se realizaron movimientos rotatorios para la recolección de mayor cantidad de células, el mismo procedimiento se realizó para la perdiz. La manipulación de los animales se ejecutó de acuerdo a la Guía para el Cuidado y Uso de los Animales de Laboratorio del NCR (2011). Para ambas especies el procedimiento se realizó por cuadriplicado en el mismo espacio de tiempo, seguidamente, cada hisopo estéril se almacenó en un tubo con 1 mLde SSF 0,9% esteril y se guardó en una cava a temperatura promedio de -4°C para posteriormente ser transportadas al laboratorio N° 8 del Area de Biología Molecular, Instituto de Investigaciones Biomédicas Dr. Francisco Triana Alonso, Universidad de Carabobo, Venezuela.
Procedimiento experimental
La extraccion del ADN bacteriano a partir de las células presentes en las muestras de hisopado rectal y cloacal se realizó a través del protocolo de fenol-cloroformo precipitación con etanol (Rodríguez-Leo et al. 2017a). Se empleó la técnica de PCR anidada optimizada por Rodríguez-Leo et al. (2017a) utilizando en la primera reacción de PCR un par de cebadores específicos sentido Cp1F (ACG GAA TAA TGA CTT CGG) antisentido Cp1R, (TAC CTG GTA CGC TCA ATT. T) dirigidos a los miembros de la familia Chlamydiaceae, específicamente a la secuencia de una porción del gen 16s ADNr, donde se esperaba un amplicon de 436 pb. Posteriormente, en la segunda reacción de amplificación se utilizó un segundo par de cebadores dirigidos a las regiones variables de la especie Cp CpintF sentido (ATA ATG ACT TCG GTT GTT ATT) y CpintR antisentido (TGT TTT AGA TGC CTA AAC AT), que se une a las copias de secuencias de los productos formados en la primera reacción de PCR, a partir de los cebadores dirigidos a los miembros de la familia antes mencionada; en esta segunda reacción se esperaba obtener un amplicon de 126 pb. Se utilizaron dos controles positivos a ADN de Cp (CP018 y CP046) donados por el Laboratorio de Biología Molecular de la Universidad del Zulia, Venezuela.
Las mezclas se realizaron con: Tampón de reacción (Promega®), Tris HCl 20 mM (pH 8,4), KCl 50 mM; 2,5 mM MgCl2 (Promega®), desoxinucleótidostrifosfato (dNTPs, del inglés 2'-desoxynucleoside 5-triphosphate) (10 mM), oligonucleótidos (10pmol/µL), Taq polimerasa (1,25 U/ µL). A las mezclas de reacción se agregaron 01 µL (50ng) de las muestras de ADN extraídas, la mezcla de reacción se sometió a incubación con el siguiente programa de amplificación: desnaturalización inicial a 95°C por 5 minutos, seguido de 35 ciclos de desnaturalización a 94°C por 1 minuto, hibridación a 55°C por 2 minutos, elongación a 72°C por 1 minuto y una extensión final a 72°C por 8 minutos en un termociclador PTC-100™ (MJ Research, INC®). La segunda reacción de PCR se realizó de igual manera usando como ADN molde 02 µL (50ng) de los productos de amplificación de la primera reacción, para un volumen final de reacción de 50 µL. Las condiciones de tiempo y temperatura fueron idénticas a las usadas en la primera reacción de amplificación.
Los productos finales se visualizaron en geles de agarosa al 2%, (Figura 1) y se compararon con el marcador de tamaño molecular 100 pb ADN Promega®. El resultado de la migración se observó en un equipo de fotodocumentación Gel Doc 1000 BIO-RAD® mediante el programa MultiAnalyst versión 1.1 (1996-97).
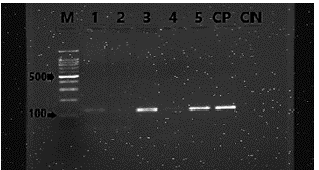
FIGURA 1 Electroforesis Gel de agarosa al 2% en TAE (Tris-acetato 40 Mm, EDTA 1 Mm pH 8) teñido con bromuro de etidio (0,5 ug/ml) de los Productos de amplificación de la subunidad 16s ADNr de Chlamydia psittaci de los reservorios animales positivos en cautiverio. M: Maracador de tamaño molecular 100 pares de bases Promega®), carril 1 Colinus cristaceo, carril 2 - 5 Mustella putorios furo, CP Control Positivo, CN Control negativo.
RESULTADOS Y DISCUSIÓN
Chlamydia psittaci ha sido descrita primordialmente en aves, sin embargo, posee la capacidad de infectar a diversas especies de mamíferos, siendo descrita con mayor frecuencia en porcinos, equinos, caninos, caprinos y ovinos (Reinhold et al. 2012; Rodolakis et al. 2015). En un principio los genotipos de Cp eran considerados especie-específicos; no obstante, en la última década se ha evidenciado que los genotipos han ampliado el rango de hospedadores. Frutos (2015) aisló el genotipo WC en Passeriformes demostrando la variabilidad que Cp presenta y su capacidad de infectar a diferentes especies indistintamente del genotipo. En la presente investigación se identificó ADN compatible para Cp en un ejemplar de Colinus cristatus, dicha ave presentaba signos clínicos característicos de clamidiosis aviar entre ellos: plumas erizadas, depresión, respiración dificultosa, secreción nasal, ocular y diarrea, la cual se presume fue el reservorio y fuente aparente de infección para el resto de las animales en la habitación donde residía, principalmente para los hurones debido a que su jaula se encontraba junto a la de ellos.
Chlamydia psittaci se detectó en 4 de 5 individuos de Mustella putorios furo. Este hallazgo demostró la posibilidad de un nuevo reservorio para esta bacteria, lo que representa el primer reporte de Cp en Mustella putorios furo en Venezuela.
En relación a las condiciones de cautiverio de los hurones, estos eran mantenidos en dos jaulas de 80 x 50 x 70 cm, dentro de una habitación compartida con múltiples especies de aves y mamíferos. En la parte superior se encontraban dos jaulas con Psittacula krameri cuyas excretas se diseminaban por toda la habitación. Estos Psittaciformes no se muestrearon debido a que se encontraban en fase de ovipostura. Este orden de aves ha sido identificado como uno de los que mayor incidencia de Cp presenta (Raso et al. 2015). Por ello, representan un riesgo de transmisión para el resto de las especies de aves y mamíferos albergadas en la misma habitación.
A su vez, en la parte inferior de la habitación se encontraban múltiples jaulas con especies de conejos (Oryctolagus cuniculus), acures (Cavia porcellus y Cavia Sheltie) y erizos morunos (Atelerix algirus vagans). En relación a la ventilación, esta era deficiente ya que la habitación solo poseía la puerta principal y una cúpula en la parte superior, lo que concentraba la cantidad de productos de excreción de los animales y la circulación de Cp.
Adicionalmente, uno de los elementos que posibilitó la transmisión de Cp en los hurones es que la perdiz presentaba signos clínicos de clamidiosis aviar, así que a través de aerosoles provenientes de sus secreciones nasales, orina, heces secas y plumas pudieron transmitir Cp a los primeros. Sin embargo, debido al desconocimiento sobre el tiempo de permanencia de los animales en el centro de cautiverio, la ausencia de un periodo de cuarentena, control del ingreso de especies animales y la ausencia del diagnóstico molecular de Cp, el mecanismo de transmisión es desconocido. En este sentido, es posible que la perdiz pudiera haber sido infectada por el contacto de los productos de excreción de los hurones representando una cepa de Cp de mayor virulencia en aves.
En este sentido, pudieron ser múltiples factores quienes favorecieron la transmisión de Cp a los hurones como la infraestructura sin ventilación, el espacio reducido, las deficiencias sanitarias, la diversidad de especies, así como la presencia de un ave positiva para Cp con signos de clamidiosis aviar. No obstante, la evidencia de ADN complementario para Cp no descarta la posibilidad de coinfección por otros miembros de la familia Chlamydia-ceae. Así pues, es relevante destacar que especies como C. pneumoniae, C. pecorum, y C. abortus poseen la capacidad de infectar aves y mamíferos (Frutos et al. 2015). En Argentina se identificó ADN compatible para Chlamydia sp. en hurones, donde no hubo amplificación para secuencias de C. pneumoniae, C. psittaci y C. abortus (Frutos 2015).
C. psittaci y C. abortus guardan una estrecha relación filogenética, han sido descritos genotipos de Cp más cercanos a C. abortus siendo clasificados como cepas intermedias C psittaci / C. abortus las cuales poseen elevada capacidad de infección a especies en cautiverio y vida libre (Szymanska-Czerwinska et al. 2017). Ma-dani et al. (2013) identificó en Psittacula krameri una única secuencia de ADN con 98% y 92% de identidad con C. abortus y el genotipo F de Cp, respectivamente. En el presente estudio los cinco ejemplares de Mustella putorios furo se encontraban en contacto permanente con las excretas de Psittacula krameri, siendo este un mecanismo de transmisión para cualquier especie de la familia Chlamydiaceae que posea como reservorio Psittaciformes. Por ello, es ineludible la aplicación de técnicas moleculares para la detección de especies Chlamydiales. Opota et al. (2015) diseñaron una PCR altamente específica y sensible para la identificación de las especies de Cp y C. abortus debido al alto impacto que posee no solo para las aves y mamíferos, sino además para la salud pública.
La diversidad genética de Cp y su capacidad de infectar nuevos reservorios apunta hacia una aparente perspectiva evolutiva de esta especie, generando un nuevo abanico de huéspedes susceptibles a estas bacterias con genotipos altamente heterogéneos y con elevada capacidad de dispersión.
En este sentido se debe realizar el análisis filogenético de los aislados de especies clamidiales no solo para identificación de una posible coinfección, sino además, para la identificación del genotipo de Cp que se encuentre circulando. Madani et al. (2013) afirman el advenimiento de nuevos genotipos de Cp donde la caracterización molecular representará una herramienta invaluable para el entendimiento y distribución de esta especie.
En el presente estudio no se logró realizar a la caracterización molecular debido a limitaciones de recursos financieros. No obstante, esta debe ser un instrumento que debe aplicarse debido a la diversidad de agentes clamidiales en fauna en cautiverio y vida libre, donde Venezuela, por ser uno de los países con una enaltecida biodi-versidad debe velar por su conservación y preservación, primordialmente para la fauna en riesgo de extinción.
CONCLUSIONES
Se demostró por primera vez en Venezuela la presencia de ADN de Cp en Mustella putorios furo, donde el contacto con Coli-nus cristatus infectada, el hacinamiento, la inadecuada ventilación y las condiciones sanitarias deficientes favorecieron la infección por Cp. La identificación de Cp en fauna en cautiverio es un procedimiento que debe ser ejecutado de forma habitual, aun cuando el animal no posea signos de clamidiosis, incluyendo especies aviares y mamíferos debido a la diversidad de reservorios que posee Cp.
Recomendaciones
Es necesaria la caracterización molecular de los aislados de Cp para relacionar el genotipo presente en Mustella putorios furo con el de Colinus cristatus y ampliar la identificación de Cp en mamíferos en cautiverio, principalmente los que se encuentren en contacto con aves o con la cercanía de estas a sus jaulas. Por último, es preciso evaluar la presencia de Cp en fauna silvestre de Venezuela para el reconocimiento de los reservorios y su relación con las condiciones geográficas de este país.














