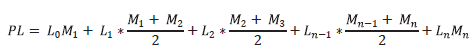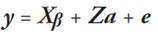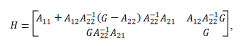INTRODUCCIÓN
La estimación de parámetros genéticos es necesaria para estimar valores de cría, construir índices de selección, predecir la ganancia genética y consecuentemente implementar un programa de mejoramiento genético (Montaldo et al. 2010; Lembeye etal. 2016). La precisión de los parámetros genéticos e índices de selección afecta directamente la respuesta a la selección de las características de interés a mejorar (Falconer y Mackay 1996). Una alternativa para aumentar la precisión de los parámetros y valores genéticos ha sido la disponibilidad y la incorporación de información genómica, lo cual ha sido especialmente más útil para características de baja heredabilidad como las reproductivas (Guarini et al. 2018).
El uso de información genómica también ha generado beneficios en la selección genética para características asociadas a la producción de leche. De esta forma, Gao et al. (2018) obtuvieron mayores confiabilidades para la producción de leche usando modelos que adicionaron información genómica versus modelos convencionales (registros fenotípicos y genealógicos). Incluso, los parámetros genéticos estimados con un número bajo de animales genotipados también mostraron moderados incrementos en las confiabilidades para características asociadas a la producción de leche (Boison et al. 2017).
El desarrollo genético de la raza Simmental (Bos taurus) en Colombia se ha dado por la importación de material genético proveniente principalmente de los Estados Unidos, Canadá, Austria y Alemania. No obstante, no se han realizado evaluaciones genéticas del desempeño fenotípico de la población colombiana en condiciones locales. Por esta razón, y dada la necesidad del conocimiento de los parámetros genéticos para el diseño e implementación de un programa de mejoramiento genético, esta investigación tuvo como objetivo estimar la heredabilidad y las correlaciones genéticas incluyendo o no el parentesco genómico para PL60, PL150, PL210 y PL305 en la primera lactancia de vacas Simmental de Colombia.
MATERIALES Y MÉTODOS
En este estudio no fue necesaria la aprobación por parte de un comité de ética debido a que todos los registros utilizados en los análisis provenían de una base de datos ya existente y no involucraron experimentos o procedimientos con los animales.
La base fenotípica estuvo conformada por 2883 controles lecheros, provenientes de 620 vacas Simmental de primera lactancia. Los partos ocurrieron entre 2009 y 2016 y solamente se incluyeron hembras con al menos 4 controles. Las vacas estuvieron sometidas a condiciones de pastoreo intensivo y con acceso a suplementación balanceada. La suplementación mineral estuvo disponible a voluntad. Las ganaderías evaluadas estuvieron entre los 1500 y 2600 msnm, con topografías onduladas y una temperatura que oscila entre los 13 y 25 °C.
Las PL60, PL150, PL210 y PL305 fueron calculadas a partir de animales que tuvieron una duración en lactancia mayor a los tiempos evaluados. En ese escenario el periodo de lactancia fue cortado y calculado hasta los 60, 150, 210 o 305 días en lactancia. Las producciones fueron calculadas mediante el método de intervalo entre registros recomendado por el Comité Internacional de Registro Animal (ICAR 2014), según la siguiente expresión:
Donde,
PL es la producción de leche acumulada a 60, 150, 210 o 305 días.
L0 es el intervalo en días entre el inicio de la lactancia y el primer control.
M 1 , M 2 , M n son las producciones de leche de 24 horas en el día del control.
L 1, L 2 L n-1 , son los intervalos en días entre fechas de los controles.
El archivo final del pedigrí incluyó 27986 animales, de los cuales 3282 y 9378 eran padres y madres, respectivamente. El número de animales genotipados fue 718 usando el chip GeenSeek Genomic Profiler-LD chip (GGP Bovine LD v4) con 30106 SNPs. Los animales con una tasa de genotipado <90% se removieron de la base genómica. Los SNP removidos de la base no cumplieron con los siguientes criterios: 1) una tasa de genotipado >90%, 2) una frecuencia del alelo de menor frecuencia (MAF) que fuese >0,05% y 3) una significancia estadística para el test equilibrio Hardy-Weinberg >0,001. El número final de los SNP para los análisis genómicos fue 23802. La información genómica fue depurada con los algoritmos incluidos en el software PLINK* (Purcell etal. 2007).
Los componentes de varianza fueron estimados usando el procedimiento de estimación por máxima verosimilitud restringida con el software AIREML* con modelos univariado y bivariado (Misztal et al. 2018). Los modelos incluyeron como efectos fijos: el año de nacimiento, el mes de parto, el año de parto y el hato. Los efectos aleatorios incluidos en todos los modelos fueron el animal y el residual. El modelo para las cuatro características en notación matricial fue el siguiente:
Donde y es el vector de registros productivos; β es el vector d efectos fijos; es el vector de efectos genéticos aditivos directos; e es el vector de efectos residuales; XyZ son las matrices de incidencia relacionando β y α con ү. El modelo asumió que E[y] = xβ; Var(d) = A ⊗ G a ; Var(e) = I ⊗ R, donde A es la matriz de parentesco aditiva entre todos los animales en el archivo de pedigrí, ⊗ es el producto directo de Kronecker, G a es la matriz de (co)varianza de efectos genéticos aditivos directos, Ies la matriz de identidad y i? es la matriz de (co)varianza de efectos residuales.
El modelo mixto con la inclusión de información genómica incluyó los mismos efectos fijos y aleatorios, pero la matriz de parentesco fue reemplazada por una matriz H, así:
donde, . A 11 es una matriz de parentesco aditiva de los animales no geno ripados, y A 22 es una matriz de parentesco por pedigrí de animales genotipados, A 12 al igual queyl21 son matrices que contienen las relaciones de parentesco de los animales genotipados y no genotipados, G es la matriz de parentesco genómica. Por lo tanto, la matriz H combina las relaciones de parentesco con base en el pedigrí y en las diferencias encontradas entre el parentesco genómico y por pedigrí (Aguilar et al. 2010).
RESULTADOS Y DISCUSIÓN
La media para PL60, PL150, PL210 y PL305 fue 1029 ± 293, 2478 ± 688, 3352 ± 905 y 3931 ± 2153 L, respectivamente. Estas producciones de leche acumuladas fueron menores a lo reportado en literatura para ganado Simmental en países templados y tropicales (Pantelic et al. 2009). Por ejemplo, Shevhuzhev et al. (2017) reportaron en una población Simmental doble propósito en Austria producciones de 3123, 4245 y 5261 kg de leche a PL150, PL210 y PL305, respectivamente. Un valor más similar fue encontrado en una población de Rumania con una producción de leche a 305 días de 4053 ± 1392 kg (Nistor etal. 2014). Por otro lado, Perisic et al. (2009) compilaron el desempeño productivo de la raza Simmental en 13 países europeos, en los cuales la producción de leche a 305 días también fue más alta y osciló entre 4459 y 6907 kg. La alta variabilidad de los valores reportados podría ser debido al uso de la raza en sistemas de producción de carne, leche o doble propósito. En Colombia, el uso de la raza Simmental es doble propósito, lo cual puede explicar parcialmente menores producciones al ser comparadas con países europeos, los cuales generalmente tienen sistemas de producción de leche especializados.
Las heredabilidades para PL60, PL150, PL210 y PL305 variaron desde 0,20 ± 0,17 a 0,27 ± 0,23; de 0,25 ± 0,25 a 0,52 ± 0,24; de 0,30 ± 0,28 a 0,35 ± 0,27, y de 0,20 ± 0,10 a 0,23 ± 0,10, respectivamente. Cobuci et al. (2011) estimaron heredabilidades similares en diferentes puntos de la primera lactancia en ganado Holstein, variando desde 0,22 a 0,32. No obstante, la comparación entre países debe ser analizada con precaución debido a diferencias en las bases de datos, metodologías utilizadas y definición de las características evaluadas. Las heredabilidades más altas reportadas aquí pueden ser debido a la ausencia de selección genética por parte de los productores colombianos. No obstante, estos resultados deben ser interpretados con precaución debido a que los errores de los parámetros también fueron altos, posiblemente por el bajo número de registros fenotípicos.
Por su parte, los valores más altos de la heredabilidad para la producción de leche en ganado Holstein fueron estimados alrededor de los 4 meses de lactancia (Haile-Mariam y Goddard 2008). Asimismo, Ojango et al. (2019) estimaron una heredabilidad de 0,41 para PL150 y fue la más alta en toda la lactancia. Estos resultados coinciden por lo encontrado en este estudio, en donde PL150 tuvo la heredabilidad más alta (0,52). Esto podría justificar la inclusión de PL150 en un programa de mejoramiento genético local. Sin embargo, esta decisión debe ser confirmada con análisis posteriores que incluyan mayor información fenotípica debido a que PL150 tuvo el mayor error estándar de las cuatro características evaluadas. Una mayor heredabilidad para PL150 respecto a PL60 en los modelos bivariados también se observó en una población de ganado Holstein, pero con heredabilidades más bajas de 0,12 y 0,07; respectivamente (Cho et al. 2016).
Aunque existieron marcadas diferencias de las heredabilidades entre modelos univariados y bivariados, las diferencias fueron reducidas al comparar modelos con y sin parentesco genómico (Tabla 1). La mayor discrepancia entre modelos con parentesco por pedigrí y genómico fue hallada para PL210 (5%) en el modelo univariado. No obstante, esta diferencia disminuyó cuando la característica fue evaluada a través del modelo bivariado con PL305. Las diferencias de las heredabilidades para producción de leche usando modelos basados en pedigrí e información genómica coincidieron con lo reportado por otros autores, los cuales tampoco encontraron diferencias superiores a un 5% (Jattawa et al. 2016; Petrini et al. 2016). Una mayor heredabilidad en modelos genómicos podría ser obtenida al aumentar el número de animales genotipados con registros y con un mayor parentesco con la población no genotipada, teniendo en cuenta que las estimaciones a partir del ssGBLUP están soportadas en la relación de parentesco entre familias y el muestreo mendeliano (Tiezzi y Malteca 2015).
TABLA 1 Heredabilidades para la producción de leche acumulada (PLA) en ganado Simmental de Colombia usando modelos con parentesco genómico y por pedigrí.
| PLA (días) | Modelo | Heredabilidades | |
|---|---|---|---|
| Pedigrí | Genómico | ||
| 60 | Univariado Bivariado con 305 | 0,27 ± 0,23 0,22 ± 0,17 | 0,26 ± 0,23 0,20 ± 0,17 |
| 150 | Univariado Bivariado con 305 | 0,27 ± 0,25 0,52 ± 0,24 | 0,25 ± 0,25 0,50 ± 0,24 |
| 210 | Univariado Bivariado con 305 | 0,35 ± 0,27 0,34 ± 0,20 | 0,30 ± 0,28 0,33 ± 0,18 |
| Univariado | 0,20 ± 0,10 | 0,20 ± 0,10 | |
| 305 | Bivariado con 60 Bivariado con 150 | 0,21 ± 0,10 0,20 ± 0,10 | 0,21 ± 0,10 0,20 ± 0,09 |
| Bivariado con 210 | 0,23 ± 0,10 | 0,22 ± 0,10 | |
Los modelos con parentesco genómico tuvieron heredabilidades más bajas y la mayoría de los errores estándar fueron similares entre modelos poligénicos y genómicos. Por su parte, Haile-Mariam et al. (2012) observaron una reducción en la varianza explicada por información genómica comparado con las estimaciones basadas en parentesco por pedigrí. Un problema inherente en la combinación de parentescos por pedigrí y genómicos es debido a las diferencias conceptuales en la definición de las poblaciones fundadoras (Meyer et al. 2018). En los modelos basados en el pedigrí por parentesco, los animales fundadores no asumen relación entre ellos. Sin embargo, la inclusión de parentesco genómico pudo relacionarlos y generar las diferencias observadas en los modelos para la estimación de varianzas y heredabilidades.
Las correlaciones genéticas entre las producciones de leche disminuyen en la medida en la que el tiempo entre las medidas incrementa (Ojango et al. 2019). Las correlaciones genéticas de PL60, PL150 y PL210 con PL305 estuvieron entre 0,64 y 0,97. La correlación más alta fue estimada entre PL150 y PL305 con un valor de 0,97 y la correlación más baja fue entre PL60 y PL305 con un valor de 0,64, indicando así un menor efecto pleiotrópico entre las producciones acumuladas más distantes en tiempo. Bignardi et al. (2012) y Gebreyohannes et al. (2016) también encontraron correlaciones genéticas más bajas entre producciones más distantes, pero en producciones de leche adyacentes fueron mayores a 0,7. Por lo tanto, la selección genética de PL60, PL150, PL210 o PL305 permitiría obtener un progreso genético indirecto importante para las otras tres producciones acumuladas de leche. No obstante, al igual que las heredabilidades, las correlaciones genéticas deben ser interpretadas con precaución porque sus precisiones dependen de los errores de las heredabilidades, los cuales fueron altos para las cuatro características.
Los valores de AIC y BIC (Tabla 2) fueron iguales en los modelos con y sin parentesco genómico para cada característica, indicando que la información poligénica y genómica tienen el mismo grado de adaptación a sus respectivas funciones de máxima verosimilitud (De Melo et al. 2007). Las diferencias encontradas en los modelos estuvieron dadas por el criterio de convergencia. Aunque todos los modelos tuvieron un criterio de convergencia menor a 1e-10, los modelos con la inclusión de parentesco genómico tuvieron una convergencia más estricta, variando entre 1-e13 y 1-e14. Por otro lado, los modelos que incluyeron parentesco por pedigrí tuvieron convergencias de 1-e12. Los valores más estrictos de convergencia alcanzados en modelos con parentesco genómico sugieren que estos modelos tuvieron una mejor adaptación a la función de verosimilitud y que podrían ser computacionalmente más eficientes frente a modelos poligénicos.
TABLA 2 AIC y BIC para modelos con parentesco por pedigrí y genómico utilizados en características para producción de leche acumulada (PLA) en ganado Simmental de Colombia.
| PLA (días) | Modelo | AIC | BIC | ||
|---|---|---|---|---|---|
| Pedigrí | Genómico | Pedigrí | Genómico | ||
| Univariado | 2843 | 2843 | 2839 | 2839 | |
| 60 | |||||
| Bivariado con 305 | 12998 | 12998 | 12986 | 12986 | |
| Univariado | 2626 | 2626 | 2622 | 2622 | |
| 150 | Bivariado con 305 | ||||
| 12827 | 12827 | 12815 | 12815 | ||
| Univariado | 2844 | 2845 | 2840 | 2841 | |
| 210 | Bivariado con 305 | ||||
| 13083 | 13083 | 13071 | 13072 | ||
| Univariado | 10587 | 10587 | 10583 | 10583 | |
| Bivariado con 60 | 12998 | 12998 | 12986 | 12986 | |
| 305 | Bivariado con 150 | ||||
| 12827 | 12827 | 12815 | 12815 | ||
| Bivariado con 210 | 13083 | 13083 | 13071 | 13072 |
Los valores iniciales de la varianza genética y residual influyen en el número de iteraciones requeridas para la estimación de componentes de varianza en modelos genéticos. En este estudio, la inclusión de información genómica pudo afectar la convergencia porque la estructura de la varianza de los animales genotipados pudo ser menos dominante diagonalmente que la estructura de la varianza estimada a partir del pedigrí (Calus y Veerkamp 2011; Koivula et al. 2015; Rolf et al. 2015). El mayor número de iteraciones pudo ser debido a los requerimientos extra que demanda la inversión de la matriz que contiene las diferencias entre el parentesco genómico y por pedigrí de animales genotipados.
Los errores de las estimaciones genómicas son más bajos cuando los animales están más relacionados porque permite mejorar la estimación del muestreo mendeliano (Clark et al. 2011; Gray et al. 2012). Esto es más importante para poblaciones con procesos permanentes de inmigración genética, lo cual es característico en países que carecen de evaluaciones genéticas (Pfibyl et al. 2012; Baba et al. 2016), que en el caso de Colombia puede tener un mayor impacto debido al uso masivo y continuo de toros norteamericanos y europeos en el programa nacional de inseminación artificial. Así pues, la inclusión del parentesco genómico de estos toros podría mejorar el ajuste de los parentescos debido a que contribuyen con más del 85% de la descendencia de ganado Simmental en Colombia.
CONCLUSION
La inclusión de información genómica a través del ssGBLUP en la población de ganado Simmental de Colombia no mejora la estimación de parámetros genéticos para características asociadas a producción de leche. El genotipado de animales locales y extranjeros con una mayor conectividad genética con la población no genotipada podrían mejorar notablemente las estimaciones de las heredabilidades. Asimismo, una mayor recolección de fenotipos para la producción de leche permitiría incrementar las precisiones y obtener un mayor beneficio de la información genómica. Finalmente, el beneficio de la selección genómica a través de la metodología del ssGBLUP es prácticamente nulo bajo las condiciones presentadas en este estudio.