Introducción
Recientemente se han evidenciado las profundas consecuencias de la resistencia bacteriana a los antibióticos en la salud humana y el impacto macroeconómico en términos de mortalidad (1,27 millones de muertes atribuibles a la resistencia antimicrobiana, para el año 2019) [1] y de morbilidad, con serias implicaciones en la fuerza productiva; además, se ha convertido en una de las mayores amenazas para la medicina moderna [2]. La aparición de infecciones no tratables, causadas por microorganismos multirresistentes, advierte la llegada de una era postantibiótica, donde no existirá tratamiento efectivo para combatir las enfermedades infecciosas [2,3]. Dentro de las principales causas de resistencia bacteriana se encuentra el uso excesivo e inadecuado de antibióticos, que trae como consecuencia la selección y la propagación de bacterias resistentes [3]. Tradicionalmente, los hospitales se han señalado como la fuente o reservorios de esta problemática; sin embargo, el alarmante aumento de bacterias resistentes ha permitido dilucidar otras fuentes provenientes de la comunidad y el ambiente [4], donde el agua ha cobrado gran importancia.
El ambiente acuático es el hábitat natural de un gran número de microorganismos, incluyendo bacterias resistentes a antibióticos, y es calificado como el principal receptor de antimicrobianos, bacterias resistentes y genes de resistencia a antibióticos (Antibiotic Resistance Genes, arg) provenientes de las actividades humanas [4]. Todos estos elementos son considerados “contaminantes emergentes”, dada su capacidad de amplificarse, difundirse y persistir en el medio ambiente acuático [5]. Esta contaminación del agua tiene implicaciones serias para la salud pública, relacionadas con la diseminación de la resistencia bacteriana y con la emergencia de nuevos mecanismos de resistencia. Los estudios realizados describen en general la presencia de estos contaminantes en el medio ambiente acuático, y si bien faltan estudios desde un enfoque epidemiológico que permitan medir o cuantificar el riesgo para la colonización o infección con bacterias resistentes a partir del contacto con aguas contaminadas, su presencia en estos escenarios ya supone un motivo de gran preocupación, al entender el papel del agua como fuente de adquisición de enfermedades infecciosas.
En esta revisión de tema, se brinda información del papel de los ambientes acuáticos en la diseminación de la resistencia bacteriana, las razones por las que son considerados entornos favorables para esta problemática y se describe el impacto para la salud pública. Esta temática es de gran importancia para el entendimiento de la aparición y la propagación de microorganismos resistentes no solo en espacios hospitalarios, sino también en otras fuentes o reservorios provenientes de la comunidad y el ambiente.
La información contenida en esta revisión proviene de fuentes bibliográficas encontradas en Medline-PubMed y Science Direct, incluyendo artículos de investigación y revisiones de revistas científicas indexadas. Así mismo, la información fue seleccionada con base en su calidad y aportes al conocimiento científico.
Resistencia bacteriana en ambientes acuáticos
El agua constituye un recurso necesario y vital para los seres humanos, pero también es el hábitat natural de un gran número y diversidad de microorganismos [6]. Los ambientes acuáticos son entornos favorables para la aparición y la diseminación de bacterias resistentes, y esto se debe a varias razones:
1. El ambiente acuático es el hábitat natural para un gran número y diversidad de grupos microorganismos. El agua presenta gran diversidad de microorganismos autóctonos y alóctonos, que pueden intercambiar genes de resistencia por transferencia horizontal de material genético y servir como reservorio de arg [7]. Varios elementos genéticos (plásmidos, secuencias de inserción, transposones, integrones, islas genómicas, prófagos) participan en la adquisición y la diseminación de genes, y en la recombinación genética entre bacterias del agua [8,9]. En estos elementos genéticos se encuentran genes de resistencia a familias de antimicrobianos, como: quinolonas, tetraciclinas y belactámicos, que pueden ser compartidos entre bacterias acuáticas y bacterias patógenas de humanas [10].
Dentro de los elementos reconocidos por su papel en la diseminación de la resistencia bacteriana en el agua están los integrones tipo i. Estudios sobre este elemento genético, llevados a cabo en aguas, principalmente residuales, muestran que en ambientes contaminados con antibióticos existe alta cantidad de arg [11,12] y mayor prevalencia del integrón tipo i, en comparación con otras clases de integrones [12]; por tanto, es un indicador de contaminación por antibióticos en las aguas [13]. Así mismo, se ha reportado una frecuencia importante de bacteriófagos portando arg en el agua, lo que evidencia el papel como vectores de arg que favorecen la transferencia de mecanismos de resistencia antimicrobiana en estos escenarios [14].
Como ejemplo de un microorganismo autóctono del ambiente acuático y reconocido por su papel en la diseminación de la resistencia antimicrobiana, se encuentra Aeromonas spp., que además de presentar intrínsecamente arg, porta gran diversidad de elementos genéticos móviles que posibilitan su capacidad para intercambiar genes de resistencia con otras especies bacterias, incluyendo patógenas como Escherichia coli o Enterobacter aerogenes[7].
2. Los ambientes acuáticos son una fuente natural de arg. Gran variedad de bacterias porta naturalmente arg como mecanismo de protección frente a compuestos antimicrobianos producidos por microrganismos competidores [7], es decir, tenían arg antes de la introducción de los antibióticos.
En la naturaleza, varios son los ejemplos del vínculo y la movilización de genes de resistencia entre bacterias ambientales y patógenas humanas. Uno de esos ejemplos es el origen del gen que codifica para la betalactamasa de espectro extendido blaCTX-M, de gran importancia en bacterias patógenas humanas, cuyo origen se relacionó con especies de Kluyvera spp., una bacteria ambiental [4].
Otros ejemplos incluyen genes de resistencia a quinolonas qnrA y carbapenémicos blaOXA-48, localizados en plásmidos y encontrados en aislados clínicos de Enterobacteriaceae, cuyo origen se describe en el cromosoma de bacterias ambientales como Shewanella algae y Vibrionaceae[15-17]; por lo tanto, los ecosistemas naturales se consideran fuente natural de arg.
3. El ambiente acuático es uno de los principales receptores de antimicrobianos, bacterias resistentes y genes de resistencia a los antibióticos. El agua es un recurso que se ve afectado por una amplia variedad de contaminantes, entre estos, los antimicrobianos, las bacterias resistentes y los arg provenientes de diferentes fuentes antropogénicas (véase Figura 1], los cuales contribuyen a la aparición y la diseminación de la resistencia bacteriana. La presencia de estos contaminantes emergentes en el ambiente acuático puede dar lugar a la selección de bacterias resistentes o a la transferencia horizontal de genes de resistencia entre bacterias [10].
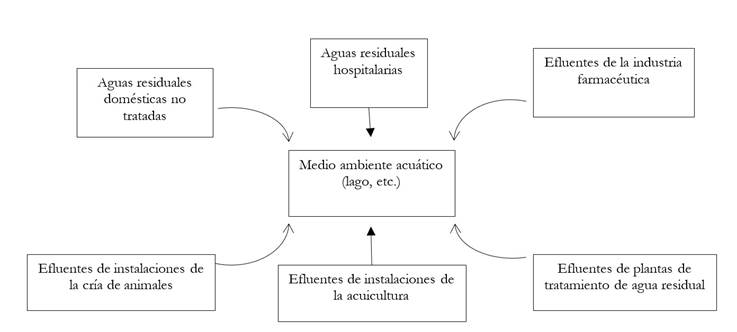
Fuente: Elaboración propia.
Figura 1 Fuentes de contaminación del medio ambiente acuático con antimicrobianos, bacterias resistentes y genes de resistencia a los antibióticos
Con respecto a la selección de bacterias resistentes en el ambiente acuático, experimentos de selección realizados en modelos bacterianos, como E. coli y Salmonella Typhimurium, muestran que a pesar de las bajas concentraciones de antimicrobianos en el agua en comparación con las utilizadas en el tratamiento para humanos y animales (humanos en μg/L y en ambiente ng/L), estas concentraciones son suficientes para la selección de bacterias resistentes [18,19]. No obstante, estudios recientes sugieren que los procesos selectivos bacterianos en el agua pueden ser más complejos. Por ejemplo, se ha visto que según el antimicrobiano evaluado, existe variabilidad en la magnitud del efecto selectivo, así como un posible efecto selectivo adictivo cuando varios antimicrobianos de la misma familia se encuentran en el ambiente acuático [20].
Por otra parte, los procesos de interacción entre comunidades microbianas también participan en la selección de bacterias resistentes en el ambiente. Se ha observado cómo la competencia bacteriana con y entre comunidades microbianas genera efectos variables en los procesos selectivos [20-23]. La interacción entre algunas comunidades microbianas naturales evitaría la selección de resistencia bacteriana en concentraciones selectivas mínimas de antimicrobianos [21], contrario a lo que se había detectado en ensayos in vivo en experimentos de una sola especie [19,24]. Este fenómeno podría ser explicado por un aumento del costo biológico de la resistencia y un efecto protector de algunas comunidades microbianas resistentes para el fenotipo susceptible [20,21].
A continuación se describen las fuentes de contaminación del agua con antimicrobianos, bacterias resistentes y genes de resistencia a los antibióticos (ARG), de acuerdo con la carga o la presión de selección que ejercen en el ambiente (véase Figura 1]:
1. Aguas residuales hospitalarias. Son altamente peligrosas, no solo por sus características infecciosas y tóxicas, sino también porque albergan gran cantidad de antibióticos y de bacterias patógenas multirresistentes, logrando alterar incluso la microbiota natural presente en ecosistemas acuáticos [25]. Estas aguas se describen como espacios favorables para el intercambio de genes de resistencia entre microorganismos clínicos patógenos y las bacterias ambientales, y ello es debido a la gran cantidad de antimicrobianos de amplio espectro y bacterias resistentes que son eliminados por pacientes colonizados e infectados [26].
El rol de estos efluentes y su impacto en la contaminación de los ambientes acuáticos, en comparación con otras fuentes de contaminación, vienen siendo estudiados, encontrándose resultados diversos. Por una parte, se describe gran variedad de tipos de arg en aguas residuales hospitalarias, incluso mayor a la que se presenta en otros sistemas de aguas residuales comunitarias [25], y se señala una abundancia alta de genes de resistencia transcripcionalmente activos, posiblemente relacionados con el nivel de uso de antibióticos en el hospital a lo largo del tiempo y con los residuos de antibióticos [27].
Sin embargo, por otro lado, también se ha descrito un impacto limitado en la diseminación de la resistencia antimicrobiana en sistemas de alcantarillado de los efluentes hospitalarios, en comparación con las plantas de tratamiento de agua residual de la comunidad [27], o un efecto variable del efluente hospitalario sobre la resistencia bacteriana de acuerdo con el microorganismo evaluado [28]. Lo anterior sugiere que si bien las aguas residuales hospitalarias no son el principal factor que contribuye al desarrollo y la persistencia de la resistencia bacteriana en el ambiente acuático, tienen un papel importante y es necesario establecer estrategias que contribuyan a su manejo adecuado [25].
2. Aguas residuales domésticas no tratadas. Las aguas residuales, al provenir del sistema de abastecimiento de agua de las poblaciones, después de haber sido modificada por diversos usos en actividades domésticas, industriales y comunitarias, pueden ser fuente de contaminación de cuerpos de agua con resistencia bacteriana.
Es posible detectar gran cantidad de microorganismos resistentes y antibióticos en las aguas residuales. Esto puede deberse al alto uso de antibióticos en humanos. Se estima que, a nivel mundial, el uso de antibióticos entre los años 2000 y 2010 aumentó en un 36 %, siendo más alto en países como Brasil, Rusia, India, China y Sudáfrica [29]. Estas cifras plantean graves preocupaciones para la salud pública y la salud ambiental, debido a que gran parte de estos antibióticos se eliminan por completo a través de las heces y la orina; su amplio uso en personas de la comunidad podría llevar a procesos de selección de bacterias patógenas que colonizan el intestino humano y, en consecuencia, detectar bacterias resistentes en aguas residuales [30,31].
3. Plantas de tratamiento de agua residual (PTAR). Las PTAR no fueron diseñadas para reducir la carga de antibióticos, bacterias resistentes en el agua y sus ARG asociados que llegan en las aguas residuales [10,30]. En cambio, se ha visto que cumplen un papel importante en la contaminación del ambiente acuático con resistencia bacteriana [10,30]. Y esto se debe a que constituyen uno de los principales receptores de resistencia bacteriana, provenientes de diferentes fuentes antropogénicas, como aguas residuales domésticas, residuos hospitalarios [5,32], e incluso vertidos de acuicultura, la escorrentía agrícola y ganadera, en países en vías de desarrollo, donde los sistemas canalización de agua residuales son precarios o inexistentes.
Además, en las ptar pueden concentrarse antibióticos y bacterias resistentes, lo que podría favorecer procesos de selección bacteriana o transferencia horizontal de genes de resistencia entre bacterias [10,33].
Por otra parte, los efluentes de las ptar están referenciados como una de las principales fuentes de dispersión de resistencia bacteriana a los antibióticos [33].
4. Industria farmacéutica. Los efluentes de la industria farmacéutica pueden ser una fuente puntual importante de antibióticos [34]. Se ha demostrado que las descargas de sus efluentes proporcionan condiciones en las que los antibióticos alcanzan concentraciones que son selectivas para el enriquecimiento de la resistencia [34]. Así mismo, se ha descrito cómo los residuos de antibióticos provenientes de la industria farmacéutica pueden alcanzar las aguas residuales, debido a la alta solubilidad de estos compuestos y, a menudo, a su pobre degradabilidad [35].
5. Vertidos de acuicultura y la escorrentía agrícola y ganadera. Los residuos de antimicrobianos, las bacterias resistentes y los arg que llegan al agua provenientes de la agricultura y la ganadería, e incluso de la acuicultura, se deben a la utilización de antimicrobianos en estas áreas, principalmente como promotores de crecimiento (antimicrobianos que mejoran el crecimiento animal o mejoran la eficiencia de la alimentación, a fin de incrementar la producción de derivados de animales) [10,36].
En animales productores de alimentos (ganado vacuno, cerdos, aves de corral y en la acuicultura) son utilizados más de 30 antimicrobianos diferentes [4]. Un estudio sobre estimaciones del consumo mundial de antimicrobianos en la veterinaria muestra cifras altas en países como China (45 %), Brasil (7,9 %), Estados Unidos (7,0 %), Tailandia (4,2 %) e India (2,2 %); además, predice que entre el 2017 y 2030, el consumo mundial de antimicrobianos veterinarios aumentara, pasando de 93 309 toneladas de antibióticos en el 2017, a 104 079 toneladas para el 2030 [37]. A pesar del avance en la reducción del uso de antibióticos en países como Alemania y China, se estima que el uso de antimicrobianos en animales productores de alimentos se incremente en todos los continentes [37].
Estos datos son relevantes, debido a que se ha considerado al ganado como una fuente importante de liberación, al medio ambiente, de antibióticos, bacterias resistentes o genes asociados [38]. Esta liberación se da principalmente con la utilización del estiércol proveniente de animales tratados con antimicrobianos como fertilizantes; asimismo, con el riego de cultivos con aguas contaminadas provenientes de granjas. Se estima que del 75 al 90 % de los antimicrobianos utilizados en los alimentos de los animales son excretados al medio ambiente, a través de la escorrentía agrícola [4]. A partir de allí, los antimicrobianos se mueven del campo a las aguas subterráneas y eventualmente entran en contacto con las aguas superficiales, mediante eventos como la lluvia [7].
En la acuicultura, los antimicrobianos se utilizan para la prevención y el tratamiento de las infecciones bacterianas en peces e invertebrados [39]. En los peces, la administración de los antimicrobianos se realiza con los alimentos. Los residuos de alimentos con antibióticos y las excreciones de los animales, que generalmente contienen trazas del antibiótico, se acumulan en los sedimentos y estaques, y por las corrientes de agua pueden ser diseminados [39]. En países como China y Chile hay un amplio uso de antimicrobianos en esta área [39,40].
Finalmente, el uso de antimicrobianos en la agricultura es escaso, y cuando son utilizados, su presencia en el ambiente puede estar influenciada por procesos de adsorción del suelo, dilución de la lluvia, la fotodegradación y la degradación microbiana [41].
Uno de los antibióticos más utilizados en la agricultura es la estreptomicina, la cual se emplea en el control de fuego bacteriano causado por Erwinia amylovora en manzanas y perales [41].
Presencia de bacterias resistentes y genes de resistencia a antibióticos en ambientes acuáticos: impacto en la salud pública y ambiental
Conocer el impacto de la diseminación, a través del agua, de la resistencia bacteriana para la salud pública y ambiental es motivo de gran preocupación. Dentro de los efectos importantes, o escenarios de riesgo para la salud pública y ambiental, se encuentra que la presencia de bacterias resistentes y ARG en el agua puede llevar a la aparición de nuevos mecanismos de resistencia, movilización y transferencia de genes de resistencia a patógenos humanos y a la diseminación de la resistencia bacteriana [42].
Durante la última década, varios estudios a nivel mundial sobre bacterias resistentes y arg en el medio ambiente acuático han considerado estos entornos como favorables para la aparición y la diseminación de la resistencia bacteriana [7]. Estos estudios se han realizado en afluentes y efluentes de ptar [43,44]; directamente en ríos, lagos y el mar [45]; en aguas residuales de hospitales [46]; en aguas residuales y sus anexos (alcantarillado, riachuelos o charcos); agua recreacional [47], y agua potable [48], donde han reportado, por ejemplo, la presencia de microorganismos ambientales asociados específicamente con fenómenos de resistencia a antibióticos [49], bacterias patógenas resistentes, ARG y elementos genéticos móviles vinculados con resistencia bacteriana [13,50].
La descripción de la presencia de bacterias residentes y ARG en las aguas ha sido fundamental para entender la relevancia y la participación de las bacterias ambientales en la diseminación y la transferencia de la resistencia a los antibióticos. Esto se suma a los hallazgos de bacterias patógenas resistentes como Pseudomonas aeruginosa, Acinectobacter, E. coli, y otras especies de la familia Enterobacteriaceae en cuerpos de aguas, microorganismos que incluso son multirresistentes, presentando resistencia a más de un antibiótico, como tetraciclinas, quinolonas, sulfonamidas y betalactámicos [51].
Hay que resaltar que la mayoría de las investigaciones sobre resistencia en el ambiente acuático se han realizado en Estados Unidos, China y Europa; sin embargo, en África y Latinoamérica, los estudios son escasos [52]. En América Latina y el Caribe, hay limitaciones, ya que la mayoría de estas investigaciones se concentra en países como Brasil, Chile, México, Colombia y Argentina, por lo cual los estudios sobre la situación de la resistencia bacteriana en ambientes acuáticos en la región podrían ser no representativos [52]. Así mismo, en Latinoamérica, pocos estudios cuantifican la presencia de genes de resistencia en el agua o analizan el resistoma bacteriano (total de arg, incluidos los que circulan en bacterias patógenas, productores de antibióticos y bacterias no patógenas) [50].
En Colombia, desde el año 2010 se han venido presentando avances sobre el tema. En su mayoría, los estudios se han enfocado en la detección y la degradación de antibióticos. Con respecto a la detección de bacterias resistentes y ARG, en los últimos años (2019-2021) se ha publicado con mayor frecuencia información al respecto [44,50-55]. Estos estudios han podido determinar la presencia de antibióticos, bacterias resistentes y genes de resistencia en diferentes fuentes de agua [53], y han demostrado la diseminación de las bacterias resistentes desde el hospital y la comunidad hacia el medio ambiente [54]. Esto, a su vez, muestra el impacto de la presión selectiva antibiótica en entornos ambientales y las consecuencias para la emergencia de nuevos mecanismos de resistencia y para el equilibrio ecológico, lo que representa una gran amenaza para la salud pública y ambiental.
A pesar de estos avances sobre el tema, aún quedan preguntas que requieren mayores estudios, sobre el impacto en la salud pública y ambiental de la presencia de bacterias resistentes y ARG en ambientes acuáticos (véase Figura 2].
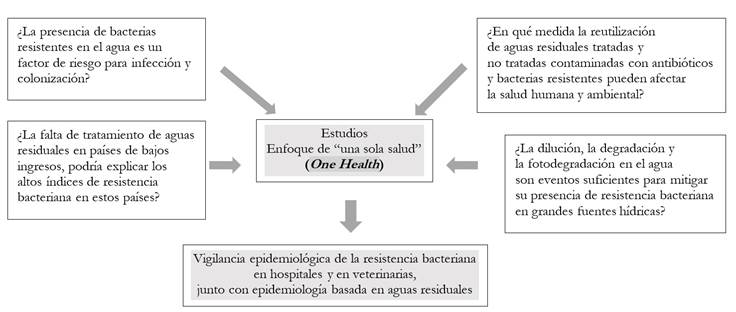
Figura 2 Preguntas que requieren mayores estudios sobre el impacto, en la salud pública y ambiental, de la presencia de bacterias resistentes y genes de resistencia a antibióticos en ambientes acuáticos.
En su mayoría, los estudios realizados a la fecha tienden a ser descriptivos sobre la presencia de estos contaminantes emergentes en aguas, pocos tienen un enfoque epidemiológico, longitudinal y de monitoreo contínuo, de tal forma que permitan establecer o medir el riesgo de colonización o infección con bacterias resistentes a partir el contacto con aguas contaminadas con estos contaminantes emergentes.
En los reportes o investigaciones que buscan evaluar la transmisión de bacterias resistentes a partir de agua contaminada con estos microorganismos y su impacto en la salud humana, uno de los modelos más estudiados es el caso de E. coli resistente a betalactámicos. Entre los estudios se resalta el de Dorado-García et al. 2019 [56], quienes evaluaron la similitud molecular de aislados de E. coli productores de betalactamasas de espectro extendido (BLEE) y AmpC provenientes de individuos de la población general con aislados procedentes de diferentes entornos, encontrando una mayor similitud entre los aislados derivados de ambientes clínicos humanos, aguas superficiales, aguas residuales y aves silvestres (índice de similitud 0,7-0,8), mientras que la similitud con los aislados resultantes del ganado o de alimentos era menor (índice de similitud 0,3-0,6) [56].
Otras investigaciones han evaluado los factores de riesgo asociados con la colonización por E. coli resistente a los antimicrobianos, señalando que las personas que consumen agua sin tratar, contaminada con esta bacteria, tienen un 26 % más de probabilidades de ser colonizadas por ella, que las personas que consumen agua no contaminada [57]. De igual forma, el riesgo de ser colonizados por E. coli productora de betalactamasas como blaCTX-M aumenta en personas en contacto constante con el agua [58], como fue evidenciado en población que practica deportes en agua, como los surfistas (rr = 4,09, ic del 95 %: 1,02-16,4), los cuales, por su profesión, tienen mayor riesgo de ingerir estos microrganismos directamente del agua de mar, ruta por la cual muchas personas podrían estar directamente expuestas [59-61]. Otros estudios epidemiológicos también sugieren que el contacto con agua recreativa en fuentes de agua dulce es un factor de riesgo no solo para la colonización, sino también para el desarrollo de infecciones del tracto urinario adquiridas en la comunidad y causadas por enterobacterias productoras de blee, como E. coli o Klebsiella pneumoniae en países de baja prevalencia (or = 2,1; ic del 95 %: 1,0-4,0) [62,63].
En última década, también se han reportado varios estudios en poblaciones colonizadas o infectadas por bacilos gramnegativos productores de carbapenamasas, como blaNDM-1 o blaIMI-2, sin antecedentes de hospitalización, al mismo tiempo que estos microorganismos son detectados en el agua del grifo, descargas de aguas residuales o ríos, con las que estas personas tienen contacto [48,64-66]. Algunos de estos estudios han utilizado métodos de tipificación molecular, que han permitido determinar relaciones genéticas entre aislamientos provenientes de personas infectadas, aislamientos de personas y animales colonizados, con aislados bacterianos provenientes de aguas recreacionales, aguas residuales, efluentes de ptar y ríos [63-65,67-70]. Estas relaciones genéticas de aislados clínicos y ambientales se han reportado en bacilos gramnegativos productores de blaNDM-1; también se han detectado entre aislados de K. pneumoniae, E. coli y otras enterobacterias portadoras de betalactamasas blaSHV-83 y blaOXA-10, blaCTX-M-15, blaVIM-1 y blaIMI-2 [64,65,69,71]. Aunque en estos estudios es difícil evidenciar la dirección de la transmisión desde el ambiente a los seres humanos, sí dan información sobre el riesgo potencial para la salud humana de la contaminación del agua a causa de la resistencia bacteriana [48], la posible propagación de microorganismos multidrogorresistentes a partir del agua y la importancia de la vigilancia de la resistencia bacteriana, junto con la incorporación de muestreos ambientales [66].
Otra de las preguntas sobre el impacto de la resistencia bacteriana en la salud ambiental está relacionada con el impacto de fenómenos naturales, como la dilución, la degradación y la fotodegradación, etc., en mitigar la presencia de antimicrobianos, bacterias resistentes y ARG en grandes fuentes hídricas. En el caso de antimicrobianos como betalactámicos, glicopéptidos y fluoroquinolonas, estos pueden en gran parte ser degradados, incluso en los procesos biológicos de las ptar, mientras que el trimetoprima y las lincosamidas parecen ser persistentes [72]. No obstante, algunos de los productos intermedios de la degradación de antimicrobianos pueden igualmente llevar a la coselección de resistencia bacteriana en el ambiente [73]. Por otro lado, fenómenos como la dilución en grandes fuentes hídricas pudiera tener efecto en las bacterias y la capacidad de realizar intercambio horizontal de genes de resistencia, dado que la probabilidad de intercambio genético aumenta cuando hay densidades celulares relativamente altas [74]. Sin embargo, esto no se ha demostrado completamente, porque se sugiere que si incluso las bacterias mueren, su adn puede quedar intacto en el fondo de lagos o arcilla, y ser capaz de transferirse a bacterias autóctonas del agua y estas, eventualmente, tener contacto con microbiota humana [75].
Otras preocupaciones o inquietudes sobre el impacto de la resistencia bacteriana en el medio ambiente acuático para la salud pública y ambiental están relacionadas, por un lado, con la reutilización de aguas residuales, y la aplicación de lodos o biosólidos provenientes de PTAR para cultivos, y cómo esta práctica puede llevar a su contaminación. Algunas investigaciones describen la translocación de bacterias resistentes y arg a cultivos alimentarios comestibles, cuando los cultivos son irrigados con aguas residuales tratadas o cuando son fertilizados con lodos provenientes de PTAR [76,77], y su impacto depende principalmente de la carga de arg introducida y de la intensidad del riego [20].
Por otro lado, la falta de tratamiento de aguas residuales puede aportar a la diseminación de la resistencia bacteriana. Aunque las PTAR convencionales no están diseñadas para reducir significativamente estos contaminantes emergentes, el riesgo es mucho mayor cuando las aguas residuales no son tratadas. En países de bajos y medianos ingresos existe una alta prevalencia de resistencia bacteriana, y de las aguas residuales generadas solo son tratadas entre el 8 y el 28 %. Además, los tratamientos terciarios son escasos, debido a limitaciones tecnológicas. En este contexto, la población podría estar más expuesta a estos contaminantes, sobre todo en áreas urbanas con altos niveles de pobreza y limitada infraestructura física de agua potable y saneamiento, donde conviven animales, agua y seres humanos, y pueden eventualmente contaminar las fuentes de agua dulce con aguas residuales [78].
Ante este panorama, es necesario no subestimar el riesgo para la salud humana que representa la resistencia bacteriana en el agua y destacar la importancia de fomentar más y mejores tratamientos de agua residual, para reducir en parte estos efectos [79]. Adicionalmente, muestra la importancia de realizar intervenciones en otros ámbitos de la vida social, debido a que la sola disminución del consumo de antibióticos parece no ser suficiente para reducir la resistencia global a los antimicrobianos, debido a la transmisibilidad de estos microorganismos [80].
Los hallazgos encontrados hasta el momento sobre el impacto para la salud pública y ambiental de la presencia de bacterias resistentes y ARG en ambientes acuáticos sugieren la importancia de realizar intervenciones empleando el enfoque de “una salud”, propuesto por la Organización Mundial de la Salud (OMS), que se define como un “esfuerzo conjunto de varias disciplinas que se unen para brindar soluciones para la salud humana, animal y ambiental” [81]. Así mismo, subrayan la necesidad de articular la vigilancia tradicional de la resistencia bacteriana enfocada a nivel humano y veterinario, con la vigilancia en el ambiente, utilizando metodologías como la epidemiología basada en aguas residuales [82].
La epidemiología basada en aguas residuales es un enfoque que se fundamenta principalmente en la extracción, la detección y el posterior análisis e interpretación de biomarcadores en aguas residuales, proporcionando un medio de recopilación de datos para estudios epidemiológicos, con el fin de evaluar condiciones de salud pública de una población y brindar información sobre salud de las comunidades [82,83]. Permite obtener información sobre diferentes factores relacionados con el estilo de vida, evaluar tendencias temporales o de respuesta a eventos de salud pública en el agua, y hacer seguimiento y vigilancia de enfermedades infecciosas [82,83]. Esta metodología se viene utilizando para apoyar la vigilancia y el seguimiento de enfermedades infecciosas o microrganismos patógenos, como en el caso de la pandemia por la COVID-19 y la detección de resistencia bacteriana (bacterias resistentes, antibióticos y ARG) en aguas residuales [82,83].
Con respecto al monitoreo de la resistencia bacteriana, la epidemiología en aguas residuales plantea la necesidad de monitorear aquellos genes clínicamente importantes, encontrados en elementos móviles y que confieran resistencia a antibióticos de alto consumo o a antibióticos que se han utilizado durante mucho tiempo, como tetraciclina y sulfonamidas. Este enfoque es de utilidad para detectar mecanismos de resistencia emergentes y determinar los patrones de resistencia y su extensión en la comunidades humanas y ambientales [82,83].
Son varios los ejemplos de estudios de vigilancia de resistencia bacteriana aplicando epidemiología basada en aguas residuales. Reinthaler et al.[84], entre los años 2000 y 2009, compararon los patrones de resistencia de cepas de E. coli aisladas de pacientes humanos con cepas aisladas de aguas residuales de ptar, encontrando un desarrollo paralelo de patrones de resistencia tanto en pacientes como en muestras ambientales, y un aumento de cepas de E. coli multirresistentes a lo largo del tiempo en muestras de ambas fuentes.
Otro estudio, realizado por Majlander et al.[85], se ocupó de la prevalencia y la abundancia de arg en aguas residuales de 2 hospitales durante 9 semanas del 2020, encontrando patrones de resistencia durante las semanas de seguimiento y perfiles de resistencia diferentes entre hospitales, información que fue útil para comprender la propagación de la resistencia a los antibióticos en los hospitales e identificar áreas potenciales de intervención.
Así mismo, otros estudios han detectado la presencia de ARG de importancia clínica en las aguas residuales, dentro de ellos genes de resistencia a carbapenémicos [50,51,86-88], arg para el tratamiento de tuberculosis [89], señalando un patrón de resistencia similar a los descrito en hospitales e incluso detectando ARG no hallados en hospitales [50].
Por otra parte, otras investigaciones han explorado la relación de la detección de resistencia bacteriana en aguas residuales con factores ambientales y humanos. Por ejemplo: se ha explorado cómo influye el uso de antibióticos en la población [90-92], el tamaño poblacional [90], la inmigración o los vuelos internacionales [91], el uso de suelo o agricultura [93], condiciones epidemiológicas y climatológicas [55,92], factores socioeconómicos, de salud y ambientales [91]. En general, todas estas investigaciones resaltan el potencial de la vigilancia de las aguas residuales para monitorear la resistencia a los antibióticos. Además, señalan la importancia de llevar a cabo la vigilancia basada en aguas residuales, a la par con la vigilancia tradicional, con el fin de responder aquellas preguntas sin resolver sobre el impacto que tiene la resistencia bacteriana en ambientes acuáticos sobre la salud pública [86].
Conclusión
En la actualidad, los ambientes acuáticos son fuentes y receptores principales de antimicrobianos, bacterias resistentes y arg provenientes de actividades antropogénicas, y tienen un papel importante en la diseminación y la amplificación de la resistencia antimicrobiana, con implicaciones serias para la salud pública.
El enfoque de “una salud”, propuesto por la oms, y la utilización de la epidemiología basada en aguas residuales se platean como estrategias que permiten tener una visión integral de la problemática, para de esta manera establecer acciones mancomunadas que permitan diseñar y aplicar programas y políticas que impacten la salud humana y ambiental.














