Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.24 n.2 Bogotá jun. 2004
Desarrollo y evaluación de una prueba de reacción en cadena de la polimerasa (PCR), utilizando la secuencia del gen hilA para diagnóstico de fiebre entérica por Salmonella spp.
Miryan Margot Sánchez 1, Nora María Cardona-Castro 2
1 Instituto Colombiano de Medicina Tropical y Universidad de Antioquia, Medellín, Colombia.
2
Instituto Colombiano de Medicina Tropical, Facultad de Medicina, Instituto de Ciencias de la Salud, CES, Medellín, Colombia.Institución donde se llevó a cabo el trabajo: Instituto Colombiano de Medicina Tropical.
El diagnóstico de fiebre entérica por Salmonella spp. se basa en el aislamiento de la bacteria en hemocultivos el cual consume tiempo, no siempre está disponible y tiene poca utilidad en pacientes con tratamiento antibiótico previo. Por consiguiente, se hace necesario el desarrollo de una prueba rápida, sensible y específica para el diagnóstico de fiebre entérica. Salmonella spp. utiliza el gen hilA (componente de la isla de patogenicidad I) para invadir células epiteliales y producir infección. Al usar la secuencia de este gen se diseñó una prueba de PCR para detectar la bacteria en sangre y se evaluó su sensibilidad, especificidad, valor predictivo positivo y valor predictivo negativo, utilizando la metodología prueba de una prueba. La prueba de oro fue el hemocultivo. Se estudiaron 34 individuos con sintomatología de fiebre entérica con aislamiento de Salmonella serotipo Typhi en hemocultivos; 35 individuos con sepsis por otros bacilos Gram negativos aislados de hemocultivo ( Klebsiella pneumoniae, 9; Serratia marcescens, 5; Escherichia coli, 4; Pseudomonas aeruginosa, 9; Providencia alcalifaciens, 4, y Enterobacter cloacae, 4) y 150 muestras de sangre de voluntarios asintomáticos. La sensibilidad, especificidad, valor pronóstico positivo y valor pronóstico negativo de la PCR fue del 100%. El número mínimo de UFC/ml que la PCR detecta en sangre es de 10.
Palabras clave: PCR, fiebre entérica, gen hilA, Salmonella spp.
Development and evaluation of a PCR method for diagnosis of Salmonella enteric fever, based on DNA sequences of the hilA gene
Typically, diagnosis of enteric fever due to Salmonella spp. is by bacterial isolation from blood culture; however, the blood culture method is slow, not always available, and not informative in patients with antibiotic treatment. Salmonella spp. uses the hilA gene (component of the pathogenicity island I) to invade epithelial cells and produce infection. Using the hilA gene sequence a PCR test was designed to detect Salmonella in blood samples. The sensitivity (S), specificity (SP), positive predictive value (PPV) and negative predictive value (NPV) of the PCR method were obtained by testing the blood samples from 34 patients with suspected of enteric fever. Presence of S. typhi was confirmed by blood culture. Blood samples were also tested from 35 patients with infections due to other non- Salmonella pathogens, again corroborated by blood culture ( Klebsiella pneumoniae, 9; Serratia marcescens, 5; Escherichia coli, 4; Pseudomonas aeruginosa, 9; Providencia alcalifaciens, 4; Enterobacter cloacae, 4). Control samples were obtained from 150 healthy volunteers. The S, SP, PPV and NPV for the PCR method were all 100%. The lowest number of colony forming units/ml detected by PCR in blood samples was 10.
Key words: PCR, enteric fever, hilA gene, Salmonella spp.
La fiebre entérica por Salmonella serotipo Typhi, es un problema de salud pública en países en desarrollo. Globalmente, se reportan más de 21 millones de casos, con más de 700.000 muertes por año (1). En nuestro medio, no se conoce la magnitud del problema, pues el diagnóstico de la fiebre entérica no siempre se apoya en el aislamiento bacteriano; el diagnóstico es eminentemente clínico (2). Sin embargo, es de esperarse que por las características geográficas, económicas y socioculturales de nuestro país, su incidencia y prevalencia sean de consideración, comparables a las encontradas en otros países en desarrollo en los cuales la fiebre entérica permanece endémica.
Las características de crecimiento de Salmonella spp. puede demandar varios días de incubación e identificación. La detección bacteriana por el hemocultivo puede estar influenciada por el medio de cultivo empleado, el número de bacterias circulantes, el tiempo de evolución de la infección, el volumen de sangre inoculado, la respuesta inmune del hospedero y la característica intracelular de esta bacteria (3). Las diferentes técnicas han sido utilizadas para el diagnóstico de fiebre entérica, incluso las de aislamiento de la bacteria como hemocultivo y cultivo de médula ósea y pruebas indirectas que detectan anticuerpos contra antígenos de la bacteria como la prueba de Widal, pruebas inmunoenzimáticas ELISA, dot blot y contrainmunoelectroforesis (4-6).
El hemocultivo es positivo en la primera semana de la infección, pero su sensibilidad está restringida al bajo número de bacterias que pueden causar una enfermedad grave, el cual puede ser de 10 UFC/ml (3). Los hemocultivos pueden detectar entre 40% y 45% de los casos y aun si no se ha suministrado tratamiento antibiótico, la tasa de detección no es mayor del 70% (3).
Las técnicas moleculares como la reacción en cadena de la polimerasa (PCR) detectan directamente el ADN del patógeno, lo cual permite un diagnóstico temprano de la enfermedad, con alta sensibilidad y especificidad (7).
El gen hilA ( hiperinvasive locus) se encuentra ubicado en la isla de patogenicidad I de Salmonella spp., la cual se encarga de la regulación del sistema de secreción tipo III que está relacionado con el proceso de invasión de la bacteria a las células epiteliales no fagocíticas del intestino delgado que son la puerta de entrada al sistema reticuloendotelial, a la sangre y a la médula ósea (8).
El diagnóstico de fiebre entérica por Salmonella sppl. en nuestro medio mejoraría de manera sustancial al tener una prueba que detecte la bacteria en forma rápida, sensible y específica, mediante la amplificación del gen hilA directamente de muestras de sangre.
Materiales y métodos
Población
La muestra para probar la sensibilidad (S), la especificidad (E), el valor pronóstico positivo (VPP) y el valor pronóstico negativo (VPN) de la PCR se calculó teniendo como base la prevalencia de salmonelosis, la cual es de 0,01% para la región de Urabá (datos locales del Hospital Antonio Roldán Betancur, Apartadó, Antioquia, 2000), con una confiabilidad del 90% y un error máximo de 0,05. Según el diseño muestral calculado para determinar S, E, VPP y VPN de la prueba (9,10), seleccionaron tres grupos de individuos, cada uno de ellos con características diferentes, a saber:
Grupo 1: muestras de sangre de 34 individuos de cualquier edad y sexo, con sintomatología de fiebre entérica y con aislamiento por hemocultivos de Salmonella serotipo Typhi.
Grupo 2: 35 individuos de cualquier edad y sexo con sepsis por otros bacilos Gram negativos aislados por hemocultivo ( Klebsiella pneumoniae, 9; Serratia marcescens, 5; Escherichia coli, 4; Pseudomonas aeruginosa, 9; Providencia alcalifaciens, 4, y Enterobacter cloacae, 4).
Grupo 3: 150 individuos voluntarios asintomáticos, de cualquier edad y sexo.
Todos los individuos del estudio firmaron un consentimiento informado que cumple con los lineamientos de la Resolución 008430 de 1993 del Ministerio de Salud de Colombia.
De cada individuo, en el momento de la admisión, se obtuvo una muestra de sangre en tubo con anticoagulante EDTA para realizar la PCR y se le tomó otra muestra para hemocultivo, de acuerdo con los métodos estándar (11).
Contaminación artificial de muestras
Para determinar el número de UFC/ml que la PCR puede detectar en sangre, se contaminaron 10 muestras de sangre de voluntarios sanos con inóculos de concentración conocida.
Se inoculó una colonia de S. Typhi S008 de un cultivo en agar nutritivo incubado a 37°C por 12-18 horas en 1 ml de caldo infusión de cerebrocorazón BHI (Becton Dickinson); se incubó a 37°C por 2 horas y, luego, se diluyó 1:10 en solución salina al 0,85%. Se leyó la concentración de la dilución en espectrofotómetro a 640 nm (densidad óptica, DO: 0,08 a 1) la cual fue la base para reproducir inóculos de concentración similar. Se realizaron diluciones seriadas del inóculo desde 1:10 a 1:10-8; el número de UFC/ml de cada dilución se determinó sembrando 100 µl en agar nutritivo e incubando por 12-18 horas a 37°C para realizar el recuento. Se inocularon 5 ml de sangre fresca con 1 ml de la dilución correspondiente a 5-50 UFC/ml.
Extracción del ADN bacteriano
El método para la extracción del ADN en muestras de los pacientes y en las muestras contaminadas artificialmente fue el de solución tamponada de lisis descrito por Haque et al. (12) al que se le introdujeron las modificaciones que se describen a continuación. Brevemente, se realizó la extracción de ADN a 1 ml de la sangre artificialmente contaminada y a 1 ml de la muestra del paciente. Cada muestra se centrifugó a 10.000 rpm por 5 minutos a temperatura ambiente para separar el plasma y el medio de cultivo, respectivamente. Se adicionó al sedimento un ml de solución tampón de lisis (0,2% Triton X 100 en tris HCL más EDTA, pH 8,0). La mezcla se aspiró suavemente varias veces para efectuar hemólisis, se centrifugó a 12.000 rpm a temperatura ambiente por 6 minutos; se descartó el sobrenadante y el procedimiento se repitió nuevamente. El sedimento se lavó una vez con agua destilada estéril y se centrifugó a 12.000 rpm a temperatura ambiente durante 1 minuto. El sobrenadante se descartó y el sedimento se resuspendió en 30 ml de agua destilada estéril; los tubos se sellaron, e pusieron en agua en ebullición por 20 minutos y se dejaron enfriar a emperatura ambiente antes de realizar la PCR.
Iniciadores
Los iniciadores se diseñaron teniendo en cuenta la secuencia del gen hilA (www.genome.ad.jp). La secuencia de los iniciadores es la siguiente:
Iniciador US: 5´-GCATGGATCCCCGCCGGCGA GATTGTG-3´
Iniciador DS: 5´-CGGAACGTTATTTGCGCCATGCTGAGGTAG-3´
El producto de amplificación correspondiente al gen hil A es de 854 bp.
Condiciones de amplificación
Para la PCR, se trabajó con una mezcla de amplificación de 50 ml de volumen total compuesta de 1,5 ml de cada iniciador a una concentración 20 mM, 10 ml de muestra de ADN, 3 µl de Taq polimerasa a una concentración de 0,5 U/µl (Promega Corp., Madison, WI, USA), 5 ml de solución tampón 10X (Promega), 6 ml de MgCl2 a una concentración de 20 mM, 0,1ml de cada dinucleótido a una concentración de 20 mM y 22,6 ml de agua destilada para completar el volumen de reacción.
Se utilizó un termociclador PTC 100 (M.J. Research, Inc., Peltier-effect cycling) para amplificar el ADN sometiendo la muestra al protocolo de amplicación ya descrito (12): ciclo 1, desnaturalización inicial a 94°C por 5 minutos; ciclo 2, paso 1 a 94 °C por 1 minuto; ciclo 2, paso 2 a una temperatura de 65 °C por 1 minuto; ciclo 2, paso 3, a 72 °C por 1 minuto; el ciclo dos se repite 30 veces; ciclo 3, extensión final, a 72°C por 10 minutos, finalización a 4 °C. Cada muestra fue amplificada por duplicado. En cada PCR se corrió un control positivo y dos controles negativos, uno inicial y otro final que no incluían ADN.
Detección de productos de amplificación
En un gel de agarosa al 1%, se fraccionaron por electroforesis12 µl del producto de amplificación a 100 V por 2 horas, que contenían 10 µl de bromuro de etidio (10 mg/ml). En las muestras positivas para Salmonella serotipo Typhi se observó una banda correspondiente a 854 bp.
Resultados
Grupo 1: de las 34 muestras positivas por cultivo para Salmonella serotipo Typhi, 34 fueron positivas para PCR.
Grupo 2: las 35 muestras contaminadas con otros bacilos Gram negativos fueron PCR negativas, no se observó ninguna banda, ni se presentaron reacciones inespecíficas.
Grupo 3: las 150 muestras provenientes de voluntarios sanos con hemocultivo negativo fueron PCR negativas. No se observó ninguna banda inespecífica.
Con base en estos datos, la PCR evaluada como prueba de diagnóstico, comparada con el hemocultivo (prueba de oro), mostró S, E, VPP y VPN del 100%, respectivamente. El número mínimo de UFC/ml que la PCR puede detectar fue de 10.
La
figura 1 muestra la banda de 854 bp en muestras de pacientes positivas para Salmonella serotipo Typhi.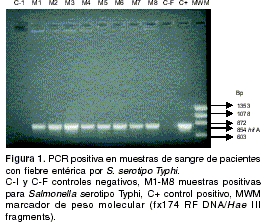
Discusión
La fiebre entérica por Salmonella serotipo Typhi es un problema de salud pública en países en desarrollo donde es endémica; su diagnóstico representa un problema para clínicos, laboratoristas y epidemiólogos. La reacción de Widal o seroaglutinación para el diagnóstico de fiebres entéricas ha sido evaluada como carente de sensibilidad y especificidad para garantizar un diagnóstico preciso (3).
Se han desarrollado pruebas serológicas para la detección de anticuerpos IgG e IgM como las pruebas de ELISA y dot blot, las cuales son una alternativa para usarlas como indicador indirecto de infección por Salmonella spp., especialmente cuando no se puede aislar la bacteria por tratamiento parcial con antibióticos, carencia de un laboratorio de microbiología y el estado tardío de la infección, entre otras (4,6,14).
La introducción de técnicas moleculares como la PCR para el diagnóstico de enfermedades infecciosas nos ofrece nuevas alternativas para aumentar la rapidez de detección de la infección con alta sensibilidad y especificidad (7).
Otros autores han desarrollado pruebas de PCR para diagnosticar la fiebre tifoidea con diversas alternativas en cuanto a la secuencia del ADN de Salmonella spp. que van a amplificar (12,15-17).
Song et al. (15) desarrollaron una PCR anidada para detección de Salmonella serotipo Typhi en muestras de sangre, utilizando iniciadores basados en el gen de la flagelina H1-d. Hashimoto et al. (16) desarrollaron una PCR anidada utilizando iniciadores basados en la secuencia de la región ViaB (antígeno Vi) de Salmonella serotipo Typhi. Haque et al. (12) desarrollaron una PCR anidada utilizando los iniciadores reportados por Song et al. y modificados por Frankel (17); sin embargo, la metodología de la PCR anidada requiere de dos procesos de amplificación, lo cual hace el procedimiento más largo y costoso. Otros investigadores han desarrollado pruebas de PCR para detectar Salmonella spp. en alimentos; Guo et al. (18) desarrollaron una PCR utilizando iniciadores que amplifican el gen hilA y que detecta Salmonella serotipo Montevideo en tomates infectados artificialmente, utilizando dos pares de iniciadores: el par denominado HILA1 produce un amplicón de 972 bp y da una banda inespecífica con Yersinia enterocolitica, y el par denominado HILA2 da un producto de amplificación de 497 bp. Los iniciadores que utilizamos en este trabajo fueron diseñados con el objetivo de detectar el gen hilA en serotipos de Salmonella de importancia clínica (13). El primer US 5´-3´ contiene un sitio de restricción BamHI y 17 nucleótidos y el primer DS 3´-5´ contiene un sitio de restricción Hindi y 21 nucleótidos que concuerdan perfectamente con la secuencia del gen hilA.
Estos iniciadores no amplificaron bandas inespecíficas en las muestras de pacientes infectadas con otras enterobacterias; además, han sido probados por otros autores con otras bacterias y no han amplificado secuencias inespecíficas (19).
La PCR en muestras de sangre propuesta en el presente trabajo detecta la secuencia del gen hilA que está presente en Salmonella serotipo Typhi, Typhimurium, Choleraesuis, Paratyphi A, Paratyphi B, Enteritidis y Pullorum (13); por lo tanto, puede ser útil para el diagnóstico de bacteriemias por estos serotipos de Salmonella spp. La sintomatología clínica del paciente y una PCR en muestras de sangre positivas para Salmonella spp. es una buena combinación para realizar un diagnóstico adecuado de fiebre entérica. La ventaja del método propuesto es que sólo se realiza una PCR, diferente de lo que implica una PCR anidada; la extracción del ADN es rápida; la sensibilidad y la especificidad óptimas, y su sensibilidad de detección de 10 UFC/ml aseguran la detección de bacteriemias con una baja concentración de microorganismos.
Hoorfar et al. sugieren la implementación de un control de amplificación interno para pruebas de PCR, indicado especialmente en pruebas de PCR no comerciales (7). La función primordial de este control interno de amplificación es el de asegurar que los resultados negativos de la prueba no estén dados por sustancias inhibitorias de la reacción, por mal funcionamiento de reactivos como la Taq polimerasa, errores de temperaturas en el equipo y otros factores externos que se verían reflejados en un resultado falso negativo. Este trabajo no incluye un control interno de amplificación pero, afortunadamente, todas las muestras que se procesaron de los pacientes y de las muestras inoculadas artificialmente con Salmonella, serotipo Typhi, fueron positivas, hecho que descarta la posibilidad de haber tenido un resultado falso negativo. Sin embargo, es de anotar que, en futuros trabajos se debe incluir el control de amplificación interno para mejorar el proceso de estandarización de esta prueba y brindar mayor confiabilidad diagnóstica.
El diagnóstico de fiebre entérica en nuestro medio mejorará de manera sustancial al tener una prueba que detecte Salmonella spp. en forma rápida sensible y específica, mediante la amplificación del gen hilA directamente de las muestras de sangre. Además, permitiría el diagnóstico precoz de casos de fiebre entérica por Salmonella spp. con disminución de las complicaciones y de la mortalidad producida por esta enfermedad.
Agradecimientos
Este proyecto fue financiado por Colciencias referencia 223-2000.
Correspondencia:
Nora M. Cardona, Instituto Colombiano de Medicina Tropical, Carrera 43ª N° 52-Sur-99, Sabaneta, Antioquia, Colombia.
Teléfono: 301 4300; fax: 301 4258
Recibido:12/02/04; aceptado: 27/05/04
Referencias
1. Thong K, Cheong Y, Puthucheary S, Koii CL, Pang T. Epidemiologic analysis of sporadic Salmonella Typhi isolates and those from outbreaks by pulsed-field gel electrophoresis. J Clin Microbiol 1994;32:1135-41. [ Links ]
2. Prada G. Tifoidea, flagelo del Tercer Mundo. Acta Med Colomb 1990;15:125-30. [ Links ]
3. Miller S, Pegues D. Salmonella species, including Salmonella Typhi. En: Mandell, Douglas and Bennetts, editors. Principles and practice of infectious diseases. 5th Edition. Philadelphia: Churchill Livingstone; 2000. p. 2344-63. [ Links ]
4. Cardona N, Agudelo P. Immunoenzymatic dot-blot test for the diagnosis of enteric fever caused by Salmonella Typhi in an endemic area. Clin Microb Infect 1998;4:64-8. [ Links ]
5. Gaviria MM, Cardona NM. Evaluation and comparison of different blood culture techniques for bacteriological isolation of Salmonella typhi and Brucella abortus. J Clin Microbiol 1995;33:868-71. [ Links ]
6. Cardona N, Gotuzzo E, Rodriguez M, Guerra H. Clinical application of a Dot Blot test for diagnosis of enteric fever due to Salmonella enterica serovar Typhi in patients with typhoid fever from Colombia and Peru. Clin Diag Lab Immun 2000;7:312-3. [ Links ]
7. Hoorfar J, Cook N, Malorny B, Wagner M, De Medici D, Abdulmawjood A et al. Making internal amplification control mandatory for diagnostic PCR. J Clin Microbiol 2003;41:5835. [ Links ]
8. Lee CA, Jones BD, Falkow S. Identification of a Salmonella typhimurium invasion locus by selection for hyperinvasive mutants. Proc Natl Acad Sci 1992;89: 1847-51. [ Links ]
9. Jekel JF, Elmore JG, Katz DL. Epidemiology, biostatistics, and preventive medicine. Philadelphia: W.B. Saunders Company, USA; 1996. p.87-91. [ Links ]
10. Reigelman RK, Hirsch RP. Cómo estudiar un estudio y probar una prueba: lectura crítica de la literatura médica. Bol Of Sanit Panam 1991;6:534-55. [ Links ]
11. Isenberg HD. Clinical microbiology procedures handbook. Washington, D.C.: American Society for Microbiology; 1992. p.5.19.5-5.19.6 [ Links ]
12. Haque A, Ahmed J, Qureshi J. Early detection of typhoid by polimerase chain reaction. Ann Saudi Med 1999;7:337-40. [ Links ]
13. Cardona-Castro N, Restrepo-Pineda E, Correa- Ochoa M. Detection of hilA gene sequences in serovars of Salmonella enterica subspecies Enterica. Mem Inst Oswaldo Cruz 2002;97:1153-6. [ Links ]
14. Carlsson HE, Lindberg AA, Hammarstorm S. Titration of antibodies to Salmonella O antigens by enzyme-linked immunosorbent assay. Infect Immun1972;6:703-8. [ Links ]
15. Song JA, Cho H, Park MY, Na DS, Moon HB, Pai CH. Detection of Salmonella Typhi in the blood of patients with typhoid fever by polymerase chain reaction. J Clin Microbiol 1993;31:1439-43. [ Links ]
16. Hashimoto Y, Itho Y, Fujinaga Y, Khan AQ, Sultana F, Miyake M et al. Development of nested PCR based on the ViaB sequence to detect Salmonella typhi. J Clin Microbiol 1995;33:775-7. [ Links ]
17. Frankel G. Detection of Salmonella typhi by PCR. J Clin Microbiol 1994;32:1415. [ Links ]
18. Guo X, Chen J, Beuchat LR, Brackett RE. PCR detection of Salmonella enterica serotype Montevideo in and on raw tomatoes using primers derived from hilA. Appl Environ Microbiol 2000;66:5248-52. [ Links ]
19. Pathmanathan SG, Cardona-Castro N, Sánchez-Jiménez MM, Correa-Ochoa MM, Puthucheary SD, Thong KL. Simple and rapid detection of Salmonella strains by direct PCR amplification of the hilA gene. J Med Microbiol 2003;52:773-6. [ Links ]














