Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.24 n.2 Bogotá jun. 2004
Genética de la preeclampsia:
una aproximación a los estudios de ligamiento genético
Nora Alejandra Zuluaga 1, Jorge Mauricio Cuartas 1, Juan Guillermo Londoño 2
1 Grupo de Genética Molecular, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.
2 Departamento de Obstetricia y Ginecología, Facultad de Medicina, Universidad de Antioquia, Medellín,
Colombia; centro asociado al Centro Latinoamericano de Perinatología/OPS.
La preeclampsia es considerada un problema de salud pública debido a su alta prevalencia. Muchas investigaciones coinciden en que su origen se relaciona con la interacción entre factores genéticos y ambientales. Por esta razón, múltiples estudios han explorado tales factores genéticos tratando de identificar regiones cromosómicas y genes candidatos cuyas variantes se relacionen con una mayor susceptibilidad a la enfermedad. Diversos estudios de asociación han identificado algunos genes de susceptibilidad a la preeclampsia, pero los resultados no se han replicado consistentemente en todas las poblaciones, quizá por su complejidad clínica y genética. El levantamiento de mapas de genes y regiones cromosómicas basado en análisis de ligamiento ha mostrado resultados interesantes con algunos marcadores en los cromosomas 2 y 4. En este sentido, hay muchas expectativas con respecto a los genes localizados en tales regiones candidatas, debido a que la identificación de los factores de riesgo genético podría ayudar al entendimiento de esta condición y en proveer claves para su prevención y tratamiento.
Palabras clave: preeclampsia, genética, mapa genético, análisis de ligamiento, cromosomas, susceptibilidad genética.
Genetics of preeclampsia: an approach to genetic linkage studies
Due to its high prevalence during pregnancies, preeclampsia is considered an important public health problem. Many investigators agree in that its expression is related to the interaction between genetic and environmental factors. Many studies have searched for genetic factors, attempting to identify chromosomal regions or candidate genes whose variants may be related to high preeclampsia susceptibility. Several studies have associated a number of susceptibility genes to preeclampsia, but the results have not been replicated consistently in all populations. Mapping of genes and chromosomal regions by linkage analysis has located potential markers on chromosomes 2 and 4. Identification of the genes located in these candidate regions will pinpoint the genetic risk factors, will lead to a better understanding of the syndrome, and will provide clues for its prevention and treatment.
Key words: preeclampsia, genetics, genetic mapping, linkage analysis, chromosomes, genetic susceptibility.
La preeclampsia es un complejo síndrome específico de la segunda mitad del embarazo (>20 semanas) que se caracteriza fisiopatológicamente por fenómenos principales como perfusión orgánica reducida, vasoespasmo y activación de la cascada de la coagulación, cuyo origen radica, al parecer, en alteraciones de la placentación. Las manifestaciones clínicas son, principalmente, hipertensión (140/90 mm Hg) y proteinuria (0,3 g en 24 horas), que desaparecen antes de la 12a semana posparto. Cuando la hipertensión y la proteinuria se suman a la presencia de convulsiones, constituyen la complicación de la preeclampsia, conocida como eclampsia (1).
Esta enfermedad hace parte del grupo de los síndromes hipertensivos del embarazo, pero se diferencia de ellos en varios aspectos (1,2); de ahí, la importancia de saber reconocer estas diferencias, para no incluir fenocopias en los estudios genéticos, lo cual podría alterar los resultados. Entre los otros síndromes hipertensivos del embarazo que debemos diferenciar de la preeclampsia, tenemos: 1) hipertensión crónica (hipertensión antes de la semana 20 de gestación o hipertensión diagnosticada por primera vez en el embarazo pero que no se resuelve posparto); 2) preeclampsia superpuesta a hipertensión crónica; 3) hipertensión gestacional (hipertensión diagnosticada por primera vez en el embarazo, sin proteinuria) (1).
La preeclampsia se presenta en 5% a 10% de los embarazos a nivel mundial (3). En general, los síndromes hipertensivos del embarazo son aún más frecuentes en los países en vía de desarrollo (15% en los hospitales universitarios) y causan entre el 30% y el 50% de la mortalidad materna en todo el mundo. En Colombia, la preeclampsia es la primera causa de mortalidad materna y, a su vez, es una de las primeras causas de mortalidad perinatal (4). Las grandes implicaciones en morbimortalidad y la alta prevalencia en nuestro medio hacen que esta enfermedad se constituya en un grave problema de salud pública.
En los últimos años se han estado realizando diversos estudios con el fin de determinar la etiología de la preeclampsia. Sin embargo, no ha sido posible explicar con exactitud cuál es la condición primaria que predispone al inicio de la cascada fisiopatológica desencadenante de esta enfermedad.
En cuanto al origen de la preeclampsia, múltiples investigaciones han demostrado que esta enfermedad se presenta con agregación familiar, lo cual sustenta la evidencia de que tiene un componente genético importante (5-10). Además, esto se demuestra por las altas tasas de concordancia entre gemelas monocigóticas, comparadas con las dicigóticas (11-13). Sin embargo, el hecho de que también se hayan observado gemelas discordantes sugiere que el fenotipo fetal, al igual que los factores ambientales, pueden ser importantes en la susceptibilidad a la preeclampsia (14,15).
El modo de herencia de la preeclampsia ha sido motivo de investigación y debate. Inicialmente, algunos investigadores propusieron que la susceptibilidad podría ser heredada por un gen único materno, autosómico recesivo (6,7,16), o por un gen dominante con penetrancia incompleta (8,17,18).
Posteriormente, se postuló que la susceptibilidad a la preeclampsia se condiciona por interacciones complejas entre dos o más genes maternos con factores ambientales, genotipos fetales y genotipos paternos (vía feto) (19-22). Es muy probable que la susceptibilidad se deba a la acción de varios genes que actúan en la madre y en el feto, modificados por factores ambientales (19-23). Esta heterogeneidad hace que la preeclampsia sea considerada actualmente como una enfermedad genéticamente compleja que no sigue un patrón de herencia mendeliano.
Al reconocer que la preeclampsia no está influenciada por un modelo genético materno exclusivo, se ha tratado de dilucidar el impacto de los genes fetales, encontrando que tanto madre como feto contribuyen igualmente al riesgo de preeclampsia; en este sentido, la contribución fetal estaría afectada por genes paternos.
En estudios posteriores se ha confirmado que el padre, producto de un embarazo con preeclampsia, o el padre de un embarazo previo con preeclampsia, tienen riesgo aumentado de tener un hijo producto de un embarazo con preeclampsia (20,24). La influencia paterna se confirmó recientemente en un estudio realizado en Utah, Estados Unidos (24), en el cual se encontró que los hombres y las mujeres productos de embarazos con preeclampsia tenían mayor probabilidad que los controles de tener un hijo que fuera producto de un embarazo complicado por preeclampsia. Específicamente, para los hombres se encontró una razón de posibilidades (OR) de 2,1 y para las mujeres de 3,3.
Como se puede apreciar, la genética de la preeclampsia está determinada por múltiples factores maternos, fetales y paternos a través del feto. Todo lo anterior hace aún más complejo su estudio. Por esta razón, hasta ahora los estudios en genética de la preeclampsia se han centrado más en el componente materno, como también porque existen ciertas dificultades que limitan un poco el abordaje de los otros factores, entre los cuales se encuentran el poco conocimiento sobre la interacción entre los genomas materno y fetal, el hecho de que los hombres no pueden ser exactamente evaluados para susceptibilidad y de que sus genotipos sólo pueden ser inferidos por la descendencia, la limitación en el número de generaciones en una genealogía porque las mujeres deben reproducirse antes de ser adscritas a un fenotipo, y las disparidades en cuanto a la circunscripción del fenotipo en los diferentes estudios (3).
Como se puede apreciar, muchos autores coinciden en que el origen de la preeclampsia está relacionado con la interacción entre factores tanto genéticos como ambientales (25-27).
Con relación a la fisiopatología, múltiples estudios han postulado gran cantidad de teorías, de las cuales vale la pena citar, en general, lo que se ha mencionado acerca de que la mala adaptación inmune y la alteración de la segunda oleada trofoblástica, con deficiencias en la remodelación vascular, producen placentación anormal e isquemia placentaria; esta isquemia origina estrés oxidativo, lo que lleva en últimas a disfunción endotelial y a las manifestaciones clínicas de la preeclampsia (26-28). En cuanto a esto, es bien reconocido que la predisposición genética influye acentuadamente en cada uno de los pasos que componen la cascada fisiopatológica de la preeclampsia (28). Por ello, se han realizado múltiples investigaciones para explorar tales factores genéticos, tratando de identificar regiones cromosómicas y genes candidatos, cuyas variantes estén relacionadas con una mayor susceptibilidad a la enfermedad.
Diversos estudios de asociación han relacionado algunos genes de susceptibilidad a preeclampsia (
cuadro 1). Sin embargo, los resultados no han sido replicados consistentemente en algunos de estos trabajos al estudiar diferentes poblaciones y genes aislados; esto se debe a la complejidad clínica y genética, características de tal enfermedad. Con relación a lo anterior, se considera específicamente que la preeclampsia es una enfermedad genéticamente compleja porque no sigue un patrón de herencia mendeliano y porque, además de presentar variabilidad fenotípica (expresividad variable, penetrancia incompleta y fenocopias), presenta variabilidad genotípica (heterogeneidad genética, pleiotropismo y epistasis) (ver glosario) (27,100-102). La heterogeneidad genética que caracteriza a las enfermedades complejas, las hace más difíciles en su abordaje por la diversidad de genes relacionados con su origen. Esto explica, en parte, el porqué de las limitaciones para replicar un estudio de asociación a un gen candidato; en este sentido, un gen que predisponga a preeclampsia en una población, puede no estar alterado en otra, y esto no significa que no esté asociado con la enfermedad, sino que en la segunda población pueden ser otros los genes que presentan las alteraciones. Además, la epistasis, que es la interacción entre los productos de los genes, complica aún más su estudio, generando modificaciones que exigen considerar siempre la influencia de otros genes asociados, adicionales al que se está estudiando.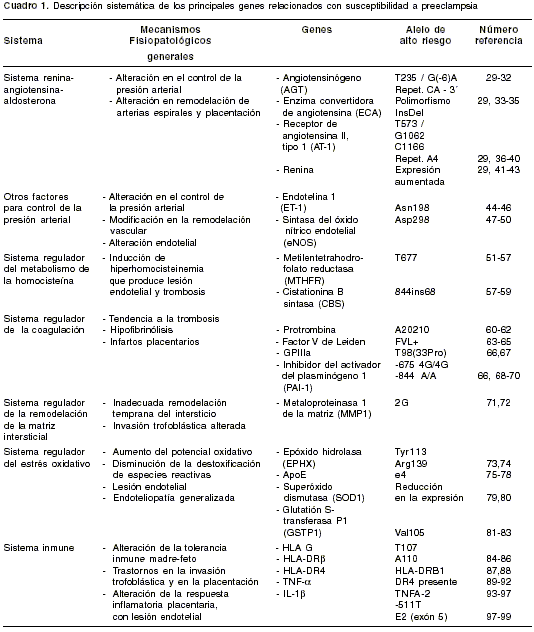
Con este referente, se han realizado estudios de ligamiento genético a través de los cuales se evalúa la transmisión de los genes de la enfermedad en conjunto con determinados marcadores genéticos. Por su proximidad a los genes en cuestión, dichos marcadores señalan regiones cromosómicas candidatas que facilitan el levantamiento del mapa de los posibles genes implicados en la preeclampsia (101-103). Una de las medidas más importantes en el análisis de ligamiento es el valor del LOD score, el cual expresa la probabilidad de que dos o más genes o loci (lugares genéticos) estén 'ligados' (cercanos entre sí). De acuerdo con el valor del LOD score (ver glosario) se puede interpretar si un determinado marcador está ligado o no al gen de la enfermedad, y así se puede determinar si la región en la que está ubicada el marcador contiene o no genes relacionados con la entidad en estudio.
Para comprender mejor la interpretación del valor del LOD score (101). En consideración a que la genética de las enfermedades complejas está condicionada por múltiples factores y a que su estudio no es igual al de las enfermedades con herencia mendeliana, Lander y Kruglyak propusieron ciertos rangos de los valores de LOD score para estimar la significancia de los hallazgos sugestivos de ligamiento en genética compleja, calificando estos rangos como indicadores de ligamiento sugestivo, significativo o altamente significativo (104) . Aunque tales rangos son un poco más estrictos, se acomodan mejor a la realidad de las enfermedades genéticamente complejas; de ahí que sea importante conocerlos y considerarlos en el momento de evaluar los resultados del análisis de ligamiento en enfermedades como la preeclampsia.
En consideración a la utilidad del análisis de ligamiento en el estudio de la genética de la preeclampsia, Wilton y colaboradores realizaron uno de los primeros estudios fundamentados en esta metodología (105); para esto, se basaron en los reportes previos que postulaban asociación con el HLA (90) y que también hipotetizaban sobre la propensión a la preeclampsia cuando la madre y el feto son homocigotos para un gen recesivo ligado a esta región (91-92). En su trabajo, específicamente trataron de comprobar tales postulados evaluando el ligamiento entre genes maternos de susceptibilidad y la región del HLA DRâ, para lo cual estudiaron 10 familias con múltiples casos de preeclampsia/eclampsia (n=29), asumiendo un modelo autosómico recesivo, diferentes grados de penetrancia y variabilidad fenotípica (casos moderados tomados como afectados o como normales). Sin embargo, no se encontró evidencia de ligamiento. De hecho, se hallaron LOD scores =-2 a è de 0,05, que por sí mismos excluyen ligamiento y sugieren que los loci de susceptibilidad a la preeclampsia deben estar por fuera de la región, como mínimo a 5-10 centimorgan (cM) del locus HLA DRß. Con base en esto, y teniendo en cuenta la poca evidencia de ligamiento entre el HLA DRß y algún locus de predisposición a la preeclampsia, consideraron que tal locus de susceptibilidad se ubicaría por fuera de dicha región. Como podemos apreciar, a pesar de los reportes previos de asociación, este estudio demostró ausencia de ligamiento, lo que indica que la región del HLA no es la única región involucrada en la susceptibilidad a la preeclampsia, dando muestra de la heterogeneidad genética propia de esta enfermedad. Consideramos que estos resultados deben interpretarse con cuidado puesto que, a pesar de que todos los casos tenían agregación familiar, aún así era una muestra pequeña (n=29), por lo cual no se puede descartar definitivamente que en esta región haya alguna variante genética relacionada directamente con el origen de la preeclampsia, o que se encuentre en desequilibrio de ligamiento con alguna otra variante cercana aún no identificada que influya directamente en la predisposición a esta enfermedad. Sin embargo, vale la pena resaltar que éste fue uno de los primeros estudios en reconocer la complejidad genética de la preeclampsia al postular que la región estudiada no era la única involucrada, sustentando así la heterogeneidad genética propia de la enfermedad.
En consideración a esta heterogeneidad genética, se han desarrollado posteriormente múltiples estudios en los que se han evaluado otras regiones, otros loci, otros genes. Es así como diferentes grupos de investigación comenzaron a evaluar la influencia del gen del angiotensinógeno en la preeclampsia. Para esto se partió de lo señalado por Ward et al. en 1993 (106), quienes basándose en reportes previos de asociación entre el alelo T235 del gen del angiotensinógeno y la hipertensión esencial, buscaron una correlación similar con preeclampsia, evaluando pacientes japonesas y americanas. Con sus estudios encontraron, efectivamente, asociación significativa entre el alelo T235 y la preeclampsia, en ambas poblaciones (106).
Basándose en este reporte de asociación, Arngrimsson y colaboradores tipificaron 22 familias islandesas con preeclampsia para un marcador en el gen del angiotensinógeno (107); con los datos realizaron análisis de ligamiento mediante el método de miembro afectado del pedigrí (108) y encontraron que había una proporción significativa de alelos compartidos en las mujeres afectadas, con lo cual dedujeron que entre dicho marcador del gen del angiotensinógeno y la preeclampsia había evidencia de ligamiento.
No obstante, este resultado no pudo ser replicado, y los estudios realizados por Morgan no mostraron evidencia a favor de estos hallazgos (109). Estudios posteriores han dado resultados variables, mostrando diferencias de acuerdo con el tipo de población estudiada (29-32). El hecho de no poder replicar estos resultados en otras poblaciones no se debe a que no exista correlación sino a que la heterogeneidad genética y el abordaje metodológico hacen que quizás en otras poblaciones sean otros genes los implicados en la predisposición a la preeclampsia que se presenta con agregación familiar, o que la metodología empleada no permita estudiar un fenotipo bien depurado. A pesar de la ausencia de replicabilidad de estos resultados, consideramos que estos postulados son plausibles debido a la ya bien demostrada influencia del angiotensinógeno y de todos los factores relacionados con el sistema renina-angiotensina-aldosterona (SRAA) sobre la regulación hemodinámica del embarazo, la adaptación fisiológica e, inclusive, algunos efectos sobre la placentación (29,43).
Posteriormente, en consideración a las correlaciones fisiopatológicas, el interés de los estudios se enfocó en el gen NOS3 que codifica para la óxido nítrico sintasa endotelial (eNOS) - ubicado en la región 7q36- el cual fue postulado como gen candidato, teniendo en cuenta la importancia del óxido nítrico en la fisiología del embarazo. En este sentido, Arngrimsson et al. (110), en 1997, evaluaron 148 pacientes islandesas y escocesas, para analizar tres marcadores microsatélites: dos de ellos flanqueantes al gen NOS3 (D7S505 y D7S483), y otro ubicado en el intrón 13 del mismo (NOS3i13) (ver glosario para comprender la nomenclatura de los marcadores). Debido a la complejidad de la genética de la preeclampsia, aplicaron métodos tanto paramétricos como no paramétricos del análisis de ligamiento (108,111,112); estos últimos se desarrollaron para detectar ligamiento sin tener que hacer presunciones acerca del modo de herencia. En el análisis paramétrico (18), el marcador flanqueante D7S505 mostró ligamiento sugestivo ( LOD score 3,36) (
cuadro 2). Con el análisis basado en modelos genéticos alternativos, propuestos por otros estudios (8,16) los LOD score máximos para este mismo marcador mostraron ligamiento significativo (2,54 y 4,03).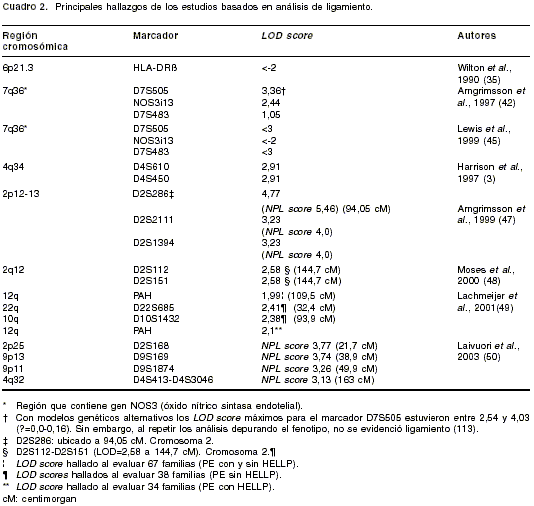
Con estos resultados, mediante métodos tanto paramétricos como no paramétricos, se confirmó el ligamiento entre el locus 7q36, que contiene el gen de la eNOS, y la preeclampsia. Específicamente, tales resultados indicaron que estos marcadores están estrechamente ligados a una variante genética que predispone a los síndromes hipertensivos asociados con el embarazo, de carácter familiar.
A nuestro modo de ver, llama la atención que el marcador D7S505, flanqueante al gen NOS3, tuvo un LOD score más significativo que el otro marcador ubicado dentro del gen mismo. Esto se debe quizá a que ciertos factores como heterogeneidad, fenocopias o modelos genéticos asumidos a priori, pudieron inflar los valores de recombinación, lo cual a su vez haría ver menor ligamiento entre este gen NOS3 y la variante génica relacionada con la preeclampsia de presentación familiar.
Ahora bien, si no fue esto lo que ocurrió también puede interpretarse que quizá el gen NOS3 no está involucrado directamente en la patogénesis de la enfermedad, sino que está en estrecha proximidad a una mutación en un gen que pudiera conferir alguna susceptibilidad a la preeclampsia. De todas formas, los resultados de este estudio nos muestran que es probable que exista un locus de susceptibilidad para la preeclampsia familiar en la región cromosómica 7q36, en la que se encuentra el gen NOS3.
Teniendo en cuenta lo anterior, en 1999, el grupo de Lewis (113) intentó repetir los hallazgos de ligamiento a esta región del gen NOS3, evaluando 104 pares de hermanas afectadas y 21 familias extendidas de Amsterdam y Cambridge. Al analizar los mismos marcadores estudiados por Arngrimsson (110) con métodos no paramétricos, no se encontró exceso de alelos compartidos entre las afectadas en ninguna de las dos poblaciones. Al evaluarlos con métodos paramétricos se encontró un LOD score = -2, que excluía ligamiento al marcador del intrón 13 del gen NOS3. En cuanto a los marcadores flanqueantes al gen, no se excluyó ligamiento en estas familias, encontrándose valores de LOD score menores de 3, algunos de ellos sugestivos de ligamento.
Los resultados de este trabajo no tuvieron la misma significancia que los del grupo anterior (110). Sin embargo, con algunos de estos resultados sugestivos de ligamiento, no se puede descartar la posibilidad de que en la región 7q36 exista alguna variante relacionada con el origen genético de la preeclampsia, que si no está en el gen NOS3, por lo menos sí esta muy cercana a él. Por estas razones, consideramos que dichos hallazgos sustentan la necesidad de realizar estudios adicionales para establecer cuál es el papel del gen NOS3 en la predisposición a la preeclampsia. Al analizar estos estudios sobre la región 7q36, se encuentra que hubo diferencias no sólo en las poblaciones estudiadas, sino también en la metodología y quizá en la depuración del fenotipo, factores que pueden alterar en cierta medida los resultados; de ahí, la importancia de unificar parámetros en el momento de diseñar estrategias metodológicas, y de basarse en los consensos sobre criterios diagnósticos para poder circunscribir estrictamente el fenotipo de la preeclampsia, y así tratar de evadir la variabilidad en los resultados, generada por las posibles fenocopias (preeclampsia vs. hipertensión gestacional).
Como podemos observar, en el abordaje de enfermedades complejas, el estudio de una única región cromosómica puede generar resultados limitados y posiblemente poco replicables por la heterogeneidad genética que las caracteriza. Este fenómeno motivó a los investigadores a adoptar la estrategia de barridos genómicos en los que, como su nombre lo indica, se evalúan múltiples marcadores a lo largo de todo el genoma, tratando de identificar aquellas regiones que se transmiten, en una familia afectada, junto con el locus o los loci de la enfermedad (100-102). Con este referente, diferentes grupos han venido desarrollando la estrategia de barridos genómicos como un abordaje clave en enfermedades genéticamente complejas para detectar loci de interés.
De acuerdo con esto, Harrison presentó un estudio en el que hizo un barrido en todo el genoma (3), con el fin de levantar mapas de genes de susceptibilidad a la preeclampsia mediante análisis de ligamiento. De esta manera, se evaluaron 15 grandes familias australianas, muy informativas, que contenían múltiples casos de preeclampsia, en las que se genotipificaron 90 marcadores distribuidos por todo el genoma. Se observó mayor informatividad en los del brazo largo del cromosoma 4 (D4S610 y D4S450), encontrando entre ellos un LOD score máximo de 2,91, resultado que aunque no establece ligamiento concluyente, sí muestra evidencia sugestiva de ligamiento, indicando que esta región del 4q podría contener un locus de susceptibilidad a la preeclampsia (
cuadro 2).Es importante resaltar que cerca a esta región candidata hay algunos genes que llaman la atención; entre ellos, los genes que codifican para la IL-2, el factor XI de la coagulación, el antígeno CD38 y el receptor tipo A de endotelina (3). Por lo que sabemos hasta ahora sobre la fisiopatología de la preeclampsia, estos genes podrían participar en alteraciones inmunológicas y de la coagulación, que llevarían a alteraciones en la placentación, o directamente a cambios hemodinámicos que modificarían la adaptación fisiológica al embarazo, desencadenando la cascada fisiopatológica que lleva a la preeclampsia (23,26-28,114).
Por otra parte, con la estrategia de barridos genómicos, el grupo de Arngrimsson (115) realizó otro estudio, mediante análisis de ligamiento, para el cual evaluaron 124 familias islandesas que contenían 343 mujeres con hipertensión gestacional, preeclampsia y eclampsia. En tales familias genotipificaron 440 marcadores microsatélites, distribuidos por todo el genoma, y analizaron los resultados asumiendo un modelo general (todas las pacientes como afectadas) y un modelo estricto (sólo preeclampsia y eclampsia como afectadas, sin incluir los casos de hipertensión gestacional).
Al aplicar el análisis de ligamiento ninguna región cromosómica alcanzó un LOD score>2, excepto la región 2p13. Este locus, bajo el criterio general, mostró un LOD score de 4,77 para el marcador D2S286. El LOD score según métodos no paramétricos ( NPL score) fue de 5,46, con el que se demostró ligamiento altamente significativo. Bajo el criterio estricto, se encontraron valores de LOD score un poco menores, pero aún así, también se demostró evidencia de ligamiento significativo. A nuestro modo de ver, con el modelo estricto (depuración de fenotipo), disminuye la significancia de los resultados de ligamiento, lo que se traduce en menor sensibilidad, pero mayor especificidad de los hallazgos. Además, en este estudio, al evaluar las familias más informativas y comparar sus haplotipos se encontró una pequeña región conservada en 2p13, con lo cual se dedujo que el gen de susceptibilidad está más probablemente entre los marcadores D2S292 y D2S329.
Como se puede apreciar de los estudios publicados hasta esa fecha, éste fue el que reportó resultados más significativos en cuanto al hallazgo de ligamiento, mostrando la región 2p13 como candidata para contener uno o varios genes relacionados con el origen de la preeclampsia. El estudio muestra una metodología estratégica por el adecuado número de marcadores usados para el barrido genómico y por las diferentes formas de abordar los modelos genéticos, en consideración a la heterogeneidad propia de la preeclampsia como enfermedad compleja. Además, se demostró de manera interesante cómo la densidad en la distribución de los marcadores circunscribe mejor la región en cuestión.
Al analizar estos resultados, vemos que el cromosoma 2 contiene una variante genética relacionada con los casos de preeclampsia que tienen un fuerte componente familiar. Sin embargo, dada la complejidad del fenotipo de esta enfermedad, tendremos que esperar a que el gen ubicado en esta región sea aislado para evaluar con confiabilidad el efecto del cromosoma 2. Muy probablemente, estén próximos a ser publicados múltiples estudios sobre esta región cromosómica, evaluando variantes en genes posiblemente relacionados con preeclampsia.
Continuando con los barridos genómicos, en el 2000, Moses y colaboradores (116) evaluaron 32 familias de Australia y Nueva Zelanda, que contenían 121 pacientes con preeclampsia grave, eclampsia e hipertensión arterial gestacional. En total, evaluaron 400 marcadores, aplicando los modelos de criterios diagnósticos, general y estricto (25). Con el criterio general, se encontró el pico de LOD score en el cromosoma 2 ( LOD score 2,58), específicamente en una ubicación cercana a la reportada previamente por Arngrimsson en 1999 (115) que había alcanzado el LOD score de 4,77 con el mismo análisis estadístico. Aunque los picos de LOD score en los dos estudios no se presentaron exactamente en la misma posición del cromosoma, están lo suficientemente cerca como para considerar un solo locus de riesgo que esté respondiendo por estas evidencias sugestivas y significativas de ligamiento.
Con el análisis basado en el criterio estricto, se mantiene la misma región como una de las más significativas, lo cual es consistente con la hipótesis de que un locus que influye en la preeclampsia/eclampsia se puede encontrar en esa región del brazo corto del cromosoma 2.
Los investigadores concluyeron que es probable que éste estudio y el islandés (115) hayan detectado el mismo locus en el cromosoma 2 y, por eso, propusieron que este debería ser designado como el locus PREG1 (preeclampsia/ eclampsia gene-1). En este barrido hubo evidencia sugestiva de ligamiento a 4q, pero no a 7q. Sin embargo, no se evaluaron los mismos marcadores de estudios previos por lo cual 7q no se puede excluir totalmente.
Desde nuestro punto de vista, aunque no se hayan encontrado exactamente las mismas localizaciones en el cromosoma 2, es muy importante este hallazgo, ya que, como se explicó al principio, estos marcadores muestran proximidad al gen o genes de riesgo. El hecho de que sus LOD scores sobresalgan frente a todos los demás marcadores del resto del genoma, en dos poblaciones diferentes, los hace muy significativos. Además, podríamos hipotetizar que por el alto valor de LOD score (4,77) encontrado en el estudio islandés (115), comparado con el mayor LOD score del último estudio, es más probable que el locus de interés esté más cercano a la pequeña región circunscrita en el estudio islandés.
Otro barrido genómico reportado, fue el desarrollado por Lachmeijer y colaboradores (117), en el 2001, quienes evaluaron 67 familias holandesas afectadas con preeclampsia, eclampsia y síndrome HELLP, complicación de la preeclampsia caracterizada por hemólisis, enzimas hepáticas elevadas y trombocitopenia, para lo cual estudiaron 293 marcadores distribuidos por todo el genoma. Entre los análisis iniciales se encontraron dos valores mayores de LOD score para marcadores que se superponían con otros marcadores mostrados en el estudio de Arngrimsson (115), en los cromosomas 3p y 15q. Al analizar los resultados de acuerdo con la presencia de síndrome HELLP, en familias sin HELLP, los resultados revelaron dos valores con evidencia sugestiva de ligamiento en las regiones 10q y 22q (cuadro 2). Al reevaluar las familias con HELLP, se encontró un LOD score en el cromosoma 12q que incrementó en las familias con HELLP y casi desapareció en las familias que no tenían el síndrome. Otro valor observado en un marcador en el cromosoma 11, en las familias con preeclampsia se traslapó con el segundo valor más alto en el estudio australiano (116). A pesar de la diversidad de resultados encontrados en este estudio en comparación con los estudios previos, los autores concluyeron que según este barrido genómico el síndrome HELLP podría tener una base genética diferente a la de la preeclampsia sin HELLP. Además, se encontraron otras regiones cromosómicas sugestivas de ligamiento superpuestas entre los diversos estudios (3p, 15q y 11).
Con estos resultados, consideramos que aunque los valores de LOD score no fueron tan altos como los de los cromosomas 2 y 4, son de todas formas importantes, puesto que se replicaron en diferentes poblaciones. Desde nuestro punto de vista, sería necesario continuar con estudios en estas regiones, aumentando el número de marcadores para dar una mejor resolución al mapa y así encontrar áreas bien circunscritas donde se puedan buscar genes candidatos posteriormente.
Por otra parte, Laivuori y colaboradores (118) presentaron el último barrido genómico en preeclampsia, para el cual tomaron una población fundadora finlandesa, cuyo fondo genético de preeclampsia podría mostrar heterogeneidad reducida (homogeneidad), facilitando el análisis genético. En su estudio, evaluaron 15 familias finlandesas con 63 pacientes (preeclampsia e hipertensión arterial gestacional), en las cuales se analizaron 435 marcadores, mediante los modelos diagnósticos general y estricto.
Cuando se usó el criterio general, se encontró ligamiento significativo a las regiones cromosómicas 2p25 y 9p13 (
cuadro 2). Además, se encontró evidencia sugestiva de ligamiento a las regiones 4q32 y 9p11. Cuando se usó el criterio estricto, los valores de NPL scores disminuyeron, al igual que en estudios previos, por el efecto de la depuración del fenotipo, como ya se explicó.Al comparar con los resultados de los barridos genómicos previos, este estudio presenta evidencia para un nuevo locus en 2p que por distancia genética no está ligado a los loci previamente reportados. Comparando los hallazgos con los estudios australiano (116) e islandés (115), es poco probable que los tres resultados de ligamiento detecten un mismo y único locus. En este sentido, aún queda difícil definir el número y la localización del gen o los genes de susceptibilidad a la preeclampsia ubicados en el cromosoma 2. Se formula la hipótesis de que tales resultados podrían deberse a la presencia de varios genes, con efectos complejos sobre la susceptibilidad a la preeclampsia. De manera alternativa podría considerarse que genes de una misma vía reguladora en la cascada de eventos fisiopatológicos podrían producir fenotipos similares con diferentes posiciones en el mapa genético (118).
El ligamiento sugestivo encontrado para la región 4q32 en este último barrido genómico se correlaciona con la misma localización que se encontró en los dos barridos realizados previamente en población australiana (3).
Como podemos observar, este último barrido genómico presentado por Laivuori et al. en el 2003 (118) mostró evidencias de ligamiento significativo a las regiones cromosómicas 2p25 y 9p13. El hallazgo de un locus en la región 2p25, diferente a los dos loci previamente reportados en otros barridos genómicos, genera la necesidad de investigaciones adicionales que complementen y aclaren estos hallazgos. También observamos que se replicaron los hallazgos en la región 4q32 en una población diferente, lo cual hace a esta región como otra de las promisorias en el mapa de genes relacionados con la preeclampsia.
Como puede apreciarse a lo largo de este texto, la región cromosómica candidata mejor caracterizada en cuanto a su relación con la preeclampsia es la 2p12-p25. Esta evidencia ha motivado el desarrollo de estudios específicos de asociación a loci ubicados en esta región. Uno de ellos es el trabajo publicado por Laasanen et al. en el 2003 (119), quienes realizaron un estudio de asociación en población finlandesa usando marcadores microsatélites de la región 2p13-p12 para encontrar loci potencialmente asociados con la preeclampsia y evaluar si la preeclampsia y la colestasis intrahepática comparten un solo locus de riesgo, considerando los reportes previos de asociación a esta última enfermedad. Este estudio incluyó 115 mujeres controles, 133 preeclámpticas y 57 mujeres colestásicas. El análisis de asociación alélica indicó que la preeclampsia y la colestasis obstétrica mostraron una asociación estadísticamente significativa con un alelo común del marcador D2S286, el cual estaba más frecuentemente en la colestasis intrahepática (0,42) y en la preeclampsia (0,37), cuando se compararon con el grupo control (0,28; p<0,05) (119). Estos hallazgos sugieren una posible relación genética entre la región 2p13-p12, la preeclampsia y la colestasis, indicando que podría contener un locus de riesgo común asociado con estas complicaciones obstétricas. Consideramos importante resaltar que el marcador D2S286, al cual se le encontró asociación en este último estudio, fue precisamente el que mostró mayores valores de LOD score (4,77) y NPL score (5,46) en el barrido genómico realizado por Arngrimsson en 1999 (115). De ahí que las expectativas estén puestas principalmente sobre esta región.
En conclusión, después de analizar todos estos estudios, podemos afirmar que se destacan los hallazgos relacionados con algunos marcadores del cromosoma 2 (2p12-p25; 2q12) y del brazo largo del cromosoma 4 (4q32) (
cuadro 2). También es importante citar las regiones 7q36, 9p11-13, que mostraron evidencia sugestiva de ligamiento, y que aún requieren ser replicadas. El cuadro 2 presenta un resumen de los resultados en análisis de ligamiento; como se puede apreciar allí, hay otras regiones también muy importantes. Esta variedad de resultados se explica por la heterogeneidad genética de la preeclampsia. Sin embargo, a pesar de las dificultades que conocemos por ser la preeclampsia una enfermedad compleja, se han logrado replicar algunos resultados en diferentes poblaciones. Como se analizó a lo largo del texto, cada estudio tiene sus logros y limitaciones. No obstante, teniendo en cuenta la significancia de los resultados se generan, entonces, muchas expectativas en cuanto a los genes ubicados en tales regiones candidatas que pudieran estar relacionados, de una u otra forma, con la fisiopatología de la preeclampsia, y que podrían estudiarse en aquellos casos con agregación familiar. De hecho, como lo mostró el estudio de Lassanen (119), ya nos estamos comenzando a acercar a los estudios de asociación partiendo de regiones candidatas. Aun así, debemos ser cuidadosos en el abordaje de estos estudios, por la complejidad genética de esta enfermedad.En consideración a los resultados significativos que relacionan la región 2p12-13, 2q12, hemos buscado en las bases de datos del genoma humano (NCBI- Genome Database) tratando de identificar genes ubicados en esas regiones que pudieran estar relacionados con la preeclampsia. Entre ellos podemos destacar el gen del TGF-á; el gen de la alfa aducina, una proteína asociada a la calmodulina, cuyas mutaciones generan cambios en la regulación de la presión arterial, en ratas (OMIM); el gen del receptor de takikinas 1, relacionado en parte con la neurocinina B, sustancia comprometida en la regulación vascular en preeclampsia (120-121); la anexina 4, una proteína anticoagulante placentaria (122); genes de moléculas de adhesión y muchas otras sustancias que podrían relacionarse de una u otra forma con la fisiopatología de la preeclampsia (23,26-28,114). Es muy probable que próximamente se publiquen estudios sobre genes ubicados en esta región y su asociación con la preeclampsia.
Consideraciones en torno al estudio de la genética de la preeclampsia
El estudio de los factores genéticos involucrados en el origen de la preeclampsia podría contribuir al esclarecimiento de su etiología, la cual, hasta el momento, no ha sido bien determinada. Considerando el lugar que ocupa la preeclampsia en la morbimortalidad materna y perinatal, sería ideal conocer a fondo los factores etiológicos y fisiopatológicos que la determinan para así poder hacer un abordaje integral, con el que se identifiquen exactamente los riesgos, se controlen específicamente las manifestaciones y se prevengan eficazmente las complicaciones. Adicional al conocimiento detallado del papel que cumplen los factores genéticos, sería fundamental también conocer qué tanto riesgo confieren los antecedentes familiares de preeclampsia, para así justificar un control prenatal cuidadoso con el que se puedan prevenir las complicaciones de una manera más eficiente.
De acuerdo con Wilson et al., la identificación de los factores de riesgo genéticos, o de una población con un riesgo excepcionalmente alto de preeclampsia, podría ayudar sustancialmente en el entendimiento de esta enfermedad y podría proporcionar claves para la prevención o el tratamiento de este importante problema de salud pública (28).
Para investigar sobre la genética de las enfermedades complejas, como es la preeclampsia, es pertinente considerar el efecto que tienen la heterogeneidad y la arquitectura genéticas de la enfermedad (el número de genes y la frecuencia de alelos que influyen en el rasgo) sobre la identificación de alelos de riesgo. Esto está críticamente determinado por el patrón de diversidad genética de la población a la cual pertenecen los pacientes (componente étnico, tiempo de fundación, grado de aislamiento genético y crecimiento demográfico), establecido como un resultado de su historia evolutiva.
Con respecto a esto, evaluar el potencial para el mapa genético en muestras que presenten un relativo aislamiento (posible homogeneidad de locus), podría ser ampliamente ventajoso. Una de las formas para garantizar la homogeneidad poblacional, puede ser evaluar poblaciones cuyos registros históricos indiquen que ellas podrían combinar dos fuentes de desequilibrio de ligamiento: 1) fundación reciente por un pequeño número de individuos y 2) mestizaje poblacional. Un ejemplo de esto es la población colombiana, y específicamente la antioqueña, en la que españoles inmigrantes fundaron el primer asentamiento no indígena durante los comienzos del siglo XVI y justo después de que se introdujeran individuos africanos como una consecuencia de las prácticas esclavistas. La llegada de inmigrantes condujo al establecimiento de una población mestiza cuya distribución estuvo fuertemente influenciada por la geografía y por la escabrosidad del terreno. Como consecuencia, el crecimiento demográfico de varias poblaciones colombianas ocurrió en relativo estado de aislamiento hasta las postrimerías del siglo XIX (123,124).
Estos hallazgos han contribuido a la determinación del poder de las poblaciones colombianas para realizar en ellas estudios de mapas genéticos (124,125), específicamente en enfermedades complejas, como son: diabetes mellitus, enfermedades neuropsiquiátricas (esquizofrenia, trastorno bipolar, hiperactividad, trastorno de Gilles de la Tourette, autismo, dislexia, trastorno obsesivo-compulsivo), resistencia/susceptibilidad a infecciones (leishmaniosis), etc. Precisamente, en la actualidad se están desarrollando estudios sobre la genética de tales enfermedades en población antioqueña. De la misma manera, investigar sobre la genética de la preeclampsia, analizando esta población en especial, podría resultar altamente informativo.
Adicionalmente, el hecho de que la preeclampsia tenga una alta prevalencia en nuestra población, hace más fácil la selección de una muestra para realizar estudios que arrojen resultados mucho más válidos. Como se ha mencionado antes, en cuanto al conocimiento de la genética de la preeclampsia hacen falta aún muchos más trabajos, que permitan replicar consistentemente los hallazgos previamente reportados, en ello es clave una población homogénea y con alta prevalencia, como la nuestra, que le confiera más poder a la muestra y más informatividad a los resultados.
Como conclusión final, sabemos hasta ahora que las regiones cromosómicas más significativas, en cuanto a ligamiento genético en preeclampsia, se ubican en los cromosomas 2 y 4. Reconocemos que existen múltiples dificultades en el abordaje de la genética de la preeclampsia, por la complejidad genética y el enfoque metodológico, que, de hecho, han limitado en parte estos estudios. Sin embargo, después de analizar todos estos reportes podemos concluir que se requiere seguir ahondando en este campo, de tal forma que se puedan aplicar metodologías estratégicas, entre ellas, la depuración del fenotipo (criterios de inclusión y exclusión), la evaluación de un número considerable de pacientes y familias que puedan conferir más poder a los resultados, la aplicación de métodos no paramétricos que permitan acercarnos aún más a la complejidad genética de la enfermedad, y considerar mejor la gran influencia de la heterogeneidad sobre la diversidad de resultados. Hasta el momento, tenemos ciertas ventajas como una alta prevalencia y una población con relativa homogeneidad genética. Este último factor hace que los estudios en nuestra población sean más poderosos, lo cual debe motivarnos a aprovechar tales ventajas para hacer estudios grandes que arrojen resultados significativos.
Las regiones cromosómicas identificadas hasta ahora son promisorias y en ellas están puestas todas las expectativas sobre los genes que puedan contener. Si aprovechamos el conocimiento de los condicionantes de esta genética compleja y la homogeneidad de nuestra población, se espera que a futuro podamos contribuir de manera importante en la determinación de los factores genéticos de la preeclampsia. Con esto, lo que se busca, en últimas, es entender mejor el origen y los mecanismos de la enfermedad, para así poder proporcionar claves en la prevención y el manejo de este importante problema de salud pública.
Glosario (101,102)
Alelos: formas alternativas de un gen o un marcador.
Análisis paramétrico: análisis de ligamiento que requiere un modelo genético predeterminado con variables que incluyen modo de herencia (autosómico dominante, recesivo, entre otros), penetrancia (completa e incompleta), frecuencias genotípicas, entre otras.
Análisis no paramétrico: análisis de ligamiento que no requiere presuponer un modelo de herencia específico. Se usa para el análisis de enfermedades genéticamente complejas, que no presentan herencia mendeliana.
Centimorgan (cM): unidad usada en los mapas de ligamiento, correspondiente a una distancia de separación entre dos genes que poseen una probabilidad de recombinar del 1%. En humanos, 1 cM equivale aproximadamente a 1 millón de pares de bases (1 Mb). Se usa como medida para expresar la posición de un marcador a partir del telómero del brazo corto del cromosoma.
Desequilibrio de ligamiento (asociación alélica): cuando alelos en dos loci distintos están en los gametos más frecuentemente de lo esperado, según las frecuencias alélicas conocidas y la fracción de recombinación entre los dos loci.
Epistasis: interacción entre los productos de dos genes, en la cual un gen interfiere la expresión fenotípica del otro.
Expresión génica: efecto del gen sobre el fenotipo; forma como el gen de una enfermedad se expresa por sí mismo.
Expresividad variable: extensión e intensidad variable de rasgos fenotípicos entre personas con un genotipo dado.
Fenocopia: cambio fenotípico inducido por factores ambientales, que remeda los efectos fenotípicos de una mutación.
Fracción de recombinación (è): para un par de loci dados, la proporción de meiosis en las cuales son separados por recombinación. Usualmente denotada como è. Los valores de è van de 0,0 a 0,5. A mayor distancia entre los loci, mayor fracción de recombinación.
Haplotipo: arreglo lineal y ordenado de alelos en un cromosoma; el análisis de haplotipos se usa para identificar eventos de recombinación.
Heterogeneidad: diferentes genotipos pueden producir fenotipos similares.
Heterogeneidad genética: cuando un trastorno tiene tipos diferentes de patrones de herencia; también tiende a interpretarse como heterogeneidad de locus.
Heterogeneidad de locus: cuando diferentes genes producen el mismo fenotipo clínico en diferentes familias.
Heterogeneidad alélica: cuando diferentes alelos (o mutaciones) en el mismo locus están presentes y resultan en el mismo fenotipo.
Intrón: región no codificante del gen.
Exón: región codificante del gen.
Ligamiento genético: tendencia de genes u otras secuencias de ADN en loci específicos, a ser heredados juntos como consecuencia de su proximidad física en un solo cromosoma. Dos genes están ligados cuando no son transmitidos independientemente a la descendencia. Mientras más ligados estén dos loci, es más probable que sean heredados juntos; al medir el grado de recombinación entre ellos se puede averiguar la distancia que los separa.
Locus (plural: loci): posición definida que tiene un gen o una secuencia en una molécula de ADN.
LOD score (del inglés, logarithm of odds): medida de la probabilidad de que dos o más genes ó loci estén 'ligados'.
La interpretación general de los valores de LOD score es la siguiente: LOD score<-2, evidencia en contra de ligamiento; LOD score entre -2 y 3, ligamiento probable, se requieren datos adicionales; LOD score=3, evidencia de ligamiento significativo. Según Lander y Kruglyak (1995) (104), en el caso particular de enfermedades genéticamente complejas el LOD score tiene ciertos criterios de significancia, así: ausencia de ligamiento ( LOD score 0-2,1), ligamiento sugestivo ( LOD score 2,2-3,5), ligamiento significativo ( LOD score 3,6-5,3), ligamiento altamente significativo ( LOD score>5,4).
Mapa genético: representación de la secuencia lineal de la información genética en cada cromosoma. Los mapas de ligamiento representan el orden de disposición de los genes y las fracciones de recombinación entre pares de ellos, que son proporcionales a la distancia física que los separa. Los mapas genéticos miden la cantidad de recombinación entre dos loci. Las unidades de mapa genético son cuantificadas en porcentaje de recombinación (è) o en centimorgan (cM) entre dos loci.
Marcador genético: ADN polimórfico de una sola localización genética, el cual es usado para el mapa genético. Segmentos de ADN cuya posición ya es conocida y que sirven como hitos para la localización de otras frecuencias u otros genes, en la estructuración de mapas genéticos. Los marcadores pueden ser genes o (más frecuentemente) secuencias no génicas.
NPL score: non parametric linkage score, medida de Lod score obtenida mediante métodos no paramétricos.
Nomenclatura de marcadores: por ejemplo, D4S450: D=ADN; 4=cromosoma 4; S=single (secuencia única); 450=# de referencia.
Método del miembro afectado del pedigrí (APM):
método de análisis de ligamiento no paramétrico que no requiere asumir un modo de herencia específico.
Penetrancia: frecuencia con la cual un genotipo se manifiesta por sí mismo en un fenotipo dado. La penetrancia es descrita como 'completa' o 'incompleta', de acuerdo con la frecuencia con que se expresa el genotipo en el fenotipo.
Pleiotropismo: cuando un gen produce múltiples efectos.
Recombinación ( crossing over): proceso complejo por el cual dos moléculas de ADN, una materna y otra paterna, intercambian segmentos recíprocamente. Ocurre en la profase meiótica. Proceso de intercambio de parte del material cromosómico entre cromosomas homólogos durante la meiosis, que produce nuevas combinaciones de la información genética
Correspondencia:
Nora Alejandra Zuluaga, Transversal 51B N° 64-B-85,
apartamento 1009, Medellín, Colombia.
Telefax: 510 60 95; beeper: 283 1515 (13876).
Recibido: 19/12/03; aceptado: 04/06/04
Referencias
1. National High Blood Pressure Education Program Working Group on High Blood Pressure in Pregnancy. Report of the National High Blood Pressure Education Program Working Group on High Blood Pressure in Pregnancy. Am J Obstet Gynecol 2001;183: S1-S22. [ Links ]
2. Cunninghan FG, MacDonald P, Gant NF, Leveno KJ, Gilstrap LC, Hankins GDV. Trastornos hipertensivos del embarazo. En: Cunninghan FG, MacDonald P, Gant NF, Leveno KJ, Gilstrap LC, Hankins DV, editores. William's Obstetricia. New York: McGraw-Hill; 2002. p.489-532. [ Links ]
3. Harrison GA, Humphrey KE, Jones N, Badenhop R, Guo G, Elakis G et al. A genomewide linkage study of preeclampsia/eclampsia reveals evidence for a candidate region on 4q. Am J Hum Genet 1997;60:1158-67. [ Links ]
4. SIVIGILA. Panorama de la mortalidad materna en Colombia, 1995-2000: logros, fracasos, compromisos y retos (parte II). Calendario epidemiológico. Informe Ejecutivo Semanal, Grupo de Vigilancia en Salud Pública. Semana Epidemiológica No. 38, 2001. [ Links ]
5. Chesley LC, Annitto JE, Cosgrove RA. The familial factor in toxemia of pregnancy. Obstet Gynecol 1968; 32:303-11. [ Links ]
6. Cooper DW, Liston WA. Genetic control of severe preeclampsia. J Med Genet 1979;16:409-16. [ Links ]
7. Sutherland A, Cooper DW, Howie PW, Liston WA, MacGillivray I. The incidence of severe pre-eclampsia amongst mothers and mothers-in-law of pre-eclamptics and controls. Br J Obstet Gynaecol 1981;88:785-91. [ Links ]
8. Arngrímsson R, Bjornsson S, Geirsson RT, Bjornsson H, Walker JJ, Snaedal G. Genetic and familial predisposition to eclampsia and preeclampsia in a defined population. Br J Obstet Gynaecol 1990;97:762-9. [ Links ]
9. Cincotta RB, Brennecke SP. Family history of preeclampsia as a predictor for pre-eclampsia in primigravidas. Int J Gynaecol Obstet 1998;60:23-7. [ Links ]
10. Mogren I, Hogberg U, Winkvist A, Stenlund H. Familial occurrence of preeclampsia. Epidemiology 1999;10:518-22. [ Links ]
11. Thornton JG, Macdonald AM. Twin mothers, pregnancy hypertension and pre-eclampsia. Br J Obstet Gynaecol 1999;106:570-5. [ Links ]
12. Ros HS, Lichtenstein P, Lipworth L, Cnattingius S. Genetic effects on the liability of developing preeclampsia and gestational hypertension. Am J Med Genet 2000;91:256-60. [ Links ]
13. O'Shaughnessy KM, Ferraro F, Fu B, Downing S, Morris NH. Identification of monozigotic twins that are concordant for preeclampsia. Am J Obstet Gynecol 2000;182:1156-7. [ Links ]
14. Thornton JG, Onwude JL. Pre-eclampsia: discordance among identical twins. BMJ 1991;303:1241-2. [ Links ]
15. Treloar SA, Cooper DW, Brennecke SP, Grehan MM, Martin NG. An Australian twin study of the genetic basis of preeclampsia and eclampsia. Am J Obstet Gynecol 2001;184:374-81. [ Links ]
16. Chesley LC, Cooper DW. Genetics of hypertension in pregnancy: possible single gene control of preeclampsia and eclampsia in the descendants of eclamptic women. Br J Obstet Gynaecol 1986;93:898-908. [ Links ]
17. Chesley LC. The genetics of preeclampsia. Hypertension Pregnancy 1993;12:7-10. [ Links ]
18. Arngrimsson R, Bjornsson H, Geirsson RT. Analysis of different inheritance patterns in preeclampsia/ clampsia syndrome. Hypertension Pregnancy 1995;14:27-38. [ Links ]
19. Walker JJ. Preeclampsia. Lancet 2000;356:1260-5. [ Links ]
20. Lie RT, Rasmussen S, Brunborg H, Gjessing HK, Lie-Nielsen E, Irgens LM. Fetal and maternal contributions to risk of preeclampsia: population based study. BMJ 1998;316:1343-7. [ Links ]
21. Liston WA, Kilpatrick DC. Is genetic susceptibility to preeclampsia conferred by homozygosity for the same single recessive gene in mother and fetus? Br J Obstet Gynaecol 1991;98:1079-86. [ Links ]
22. Kilpatrick DC. Influence of human leukocyte antigen and tumour necrosis factor genes on the development of pre-eclampsia. Hum Reprod Update 1999;5:94-102. [ Links ]
23. Lachmeijer A, Dekker GA, Pals G, Aarnoudse JG, Kate L, Arngrímsson R. Searching for preeclampsia genes: the current position. Eur J Obstet Gynecol Reprod Biol 2002;105:94-113. [ Links ]
24. Esplin MS, Fausett MB, Fraser A, Kerber R, Mineau G, Carrillo J, et al. Paternal and maternal components of the predisposition to preeclampsia. N Engl J Med 2001;344:867-72. [ Links ]
25. Morgan T, Ward K. New insights into the genetics of preeclampsia. Semin Perinatol 1999;23:14-23. [ Links ]
26. Dekker GA, Sibai BM. Etiology and pathogenesis of preeclampsia: Current concepts. Am J Obstet Gynecol 1998;179:1359-75. [ Links ]
27. Pridjian G, Puschett JB. Preeclampsia part 2: Experimental and genetics considerations. Obstet Gynecol Surv 2002;57:619-40. [ Links ]
28. Wilson ML, Goodwin TM, Pan VL, Ingles SA. Molecular Epidemiology of preeclampsia. Obstet Gynecol Surv 2003;58:39-66. [ Links ]
29. Boubaa I, Makrydimas G, Kalaitzidis R, Lolis DE, Siamopoulos KC, Georgiou L. Interaction between the polymorphisms of the renin-angiotensin system in preeclampsia. Eur J Obstet Gynecol Reprod Biol 2003; 110:8-11. [ Links ]
30. Levesque S, Moutquin JM, Lindsay C, Roy MC, Rousseau F. Implication of an AGT haplotype in a multigene association study with pregnancy hypertension. Hypertension 2004;43:71-8. [ Links ]
31. Kobashi G, Shido K, Hata A, Yamada H, Kato EH, Kanamori M, et al. Multivariate analysis of genetic and acquired factors; T235 variant of the angiotensinogen gene is a potent independent risk factor for preeclampsia. Semin Thromb Hemost 2001;27:143-7. [ Links ]
32. Arngrímsson R, Purandare S, Connor M, Walker JJ, Bjornsson S, Soubrier F, et al. Angiotensinogen: a candidate gene involved in preeclampsia? Nat Genet 1993;4:114-5. [ Links ]
33. Choi H, Kang JY, Yoon HS, Han SS, Whang CS, Moon IG, et al. Association of angiotensin-converting enzyme and angiotensinogen gene polymorphisms with preeclampsia. J Korean Med Sci 2004;19:253-7. [ Links ]
34. Gurdol F, Isbilen E, Yilmaz H, Isbir T, Dirican A. The association between preeclampsia and angiotensinconverting enzyme insertion/deletion polymorphism. Clin Chim Acta 2004;341:127-31. [ Links ]
35. Mello G, Parretti E, Gensini F, Sticchi E, Mecacci F, Scarselli G, et al. Maternal-fetal flow, negative events, and preeclampsia: role of ACE I/D polymorphism. Hypertension 2003;41:932-7. [ Links ]
36. Leung PS, Tsai SJ, Wallukat G, Leung TN, Lau TK. The upregulation of angiotensin II receptor AT(1) in human preeclamptic placenta. Mol Cell Endocrinol 2001; 26:95-102. [ Links ]
37. AbdAlla S, Lother H, Massiery A, Quitterer U. Increased AT(1) receptor heterodimers in preeclampsia mediate enhanced angiotensin II responsiveness. Nat Med 2001;7:1003-9. [ Links ]
38. Morgan L, Crawshaw S, Baker PN, Brookfield JF, Broughton F, Kalsheker N. Distortion of maternalfetal angiotensin II type 1 receptor allele transmission in pre-eclampsia. J Med Genet 1998;35:632-6. [ Links ]
39. Morgan L, Crawshaw S, Baker PN, Edwards R, Broughton Pipkin F, Kalsheker N. Functional and genetic studies of the angiotensin II type 1 receptor in pre-eclamptic and normotensive pregnant women. J Hypertens 1997;15:1389-96. [ Links ]
40. Rolfs A, Weber-Rolfs I, Regitz-Zagrosek V, Kallisch H, Riedel K, Fleck E. Genetic polymorphisms of the angiotensin II type 1 (AT1) receptor gene. Eur Heart J 1994;15:108-12. [ Links ]
41. Shah DM, Banu JM, Chirgwin JM, Tekmal RR. Reproductive tissue renin gene expression in preeclampsia. Hypertens Pregnancy 2000;19:341-51. [ Links ]
42. Arngrimsson R, Geirsson RT, Cooke A, Connor M, Bjornsson S, Walker JJ. Renin gene restriction fragment length polymorphisms do not show linkage with preeclampsia and eclampsia. Acta Obstet Gynecol Scand 1994;73:10-3. [ Links ]
43. Li C, Ansari R, Yu Z, Shah D. Definitive molecular evidence of renin-angiotensin system in human uterine decidual cells. Hypertension 2000;36:159-64. [ Links ]
44. Barden AE, Herbison CE, Beilin LJ, Michael CA, Walters BN, Van Bockxmeer FM. Association between the endothelin-1 gene Lys198Asn polymorphism blood pressure and plasma endothelin-1 levels in normal and pre-eclamptic pregnancy. J Hypertens 2001;19:1775-82. [ Links ]
45. Napolitano M, Miceli F, Calce A, Vacca A, Gulino A, Apa R, et al. Expression and relationship between endothelin-1 messenger ribonucleic acid (mRNA) and inducible/endothelial nitric oxide synthase mRNA isoforms from normal and preeclamptic placentas. J Clin Endocrinol Metab 2000;85:2318-23. [ Links ]
46. Singh HJ, Rahman A, Larmie ET, Nila A. Endothelinl in feto-placental tissues from normotensive pregnant women and women with pre-eclampsia. Acta Obstet Gynecol Scand 2001;80:99-103. [ Links ]
47. Hakli T, Romppanen EL, Hiltunen M, Helisalmi S, Punnonen K, Heinonen S. Endothelial nitric oxide synthase polymorphism in preeclampsia. J Soc Gynecol Investig 2003;10:154-7. [ Links ]
48. Yoshimura T, Yoshimura M, Tabata A, Shimasaki Y, Nakayama M, Miyamoto Y, et al. Association of the missense Glu298Asp variant of the endothelial nitric oxide synthase gene with severe preeclampsia. J Soc Gynecol Investig 2000;7:238-41. [ Links ]
49. Lade JA, Moses EK, Guo G, Wilton AN, Grehan M, Cooper DW, et al. The eNOS gene: a candidate for the preeclampsia susceptibility locus? Hypertens Pregnancy 1999;18:81-93. [ Links ]
50. Kobashi G, Yamada H, Ohta K, Kato E, Ebina Y, Fujimoto S. Endothelial nitric oxide synthase gene (NOS3) variant and hypertension in pregnancy. Am J Med Genet 2001;103:241-4. [ Links ]
51. Sohda S, Arinami T, Hamada H, Yamada N, Hamaguchi H, Kubo T. Methylenetetrahydrofolate reductase polymorphism and pre-eclampsia. J Med Genet 1997;34:525-6. [ Links ]
52. Murakami S, Matsubara N, Saitoh M, Miyakaw S, Shoji M, Kubo T. The relation between plasma homocysteine concentration and methylene-tetrahydrofolate reductase gene polymorphism in pregnant women. Obstet Gynaecol Res 2001;27:349-52. [ Links ]
53. Lachmeijer AM, Arngrimsson R, Bastiaans EJ, Pals G, ten Kate LP, de Vries JI, et al. Mutations in the gene for methylenetetrahydrofolate reductase, homocysteine levels, and vitamin status in women with a history of preeclampsia. Am J Obstet Gynecol 2001; 184:394-402. [ Links ]
54. Vefring H, Lie RT, ODegard R, Mansoor MA, Nilsen ST. Maternal and fetal variants of genetic thrombophilias and the risk of preeclampsia. Epidemiology 2004; 15:317-22. [ Links ]
55. Prasmusinto D, Skrablin S, Fimmers R, van der Ven K. Ethnic differences in the association of factor V Leiden mutation and the C677T methylenetetrahydrofolate reductase gene polymorphism with preeclampsia. Eur J Obstet Gynecol Reprod Biol 2004;112:162-9. [ Links ]
56. Zusterzeel PL, Visser W, Blom HJ, Peters WH, Heil SG, Steegers EA. Methylenetetrahydrofolate reductase polymorphisms in preeclampsia and the HELLP syndrome. Hypertens Pregnancy 2000;19:299-307. [ Links ]
57. Kim YJ, Williamson RA, Murray JC, Andrews J, Pietscher JJ, Peraud PJ, et al. Genetic susceptibility to preeclampsia: roles of cytosineto-thymine substitution at nucleotide 677 of the gene for methylenetetrahydrofolate reductase, 68-base pair insertion at nucleotide 844 of the gene for cystathionine betasynthase, and factor V Leiden mutation. Am J Obstet Gynecol 2001;184:1211-7. [ Links ]
58. Kraus JP, Oliveriusova J, Sokolova J, Kraus E, Vlcek C, de Franchis R, et al. The human cystathionine betasynthase (CBS) gene: complete sequence, alternative splicing, and polymorphisms. Genomics 1998;52:312-24. [ Links ]
59. Franco RF, Elion J, Lavinha J, Krishnamoorthy R, Tavella MH, Zago MA. Heterogeneous ethnic distribution of the 844ins68 in the cystathionine betasynthase gene. Hum Hered 1998;48:338-42. [ Links ]
60. Benedetto C, Marozio L, Salton L, Maula V, Chieppa G, Massobrio M. Factor V Leiden and factor II G20210A in preeclampsia and HELLP syndrome. Acta Obstet Gynecol Scand 2002;81:1095-100. [ Links ]
61. van Pampus MG, Wolf H, Koopman MM, van den Ende A, Buller HR, Reitsma PH. Prothrombin 20210 G: a mutation and Factor V Leiden mutation in women with a history of severe preeclampsia and (H)ELLP syndrome. Hypertens Pregnancy 2001;20:291-8. [ Links ]
62. Kupferminc MJ, Eldor A, Steinman N, Many A, Bar-Am A, Jaffa A, et al. Increased frequency of genetic thrombophilia in women with complications of pregnancy. N Engl J Med 1999;340:9-13. [ Links ]
63. Ament L. Factor V Leiden: a review of the literature. J Perinat Neonatal Nurs 2003;17:190-5. [ Links ]
64. Kosmas IP, Tatsioni A, Ioannidis JP. Association of Leiden mutation in factor V gene with hypertension in pregnancy and pre-eclampsia: a meta-analysis. J Hypertens 2003;21:1221-8. [ Links ]
65. Dudding TE, Attia J. The association between adverse pregnancy outcomes and maternal factor V Leiden genotype: a meta-analysis. Thromb Haemost 2004; 91:700-11. [ Links ]
66. Pegoraro RJ, Hira B, Rom L, Moodley J. Plasminogen activator inhibitor type 1 (PAI1) and platelet glycoprotein IIIa (GPIIIa) polymorphisms in Black South Africans with pre-eclampsia. Acta Obstet Gynecol Scand 2003; 82:313-7. [ Links ]
67. O'Shaughnessy KM, Fu B, Downing S, Morris NH. Thrombophilic polymorphisms in pre-eclampsia: altered frequency of the functional 98C>T polymorphism of glycoprotein IIIa. J Med Genet 2001;38:775-7. [ Links ]
68. Fabbro D, D'Elia AV, Spizzo R, Driul L, Barillari G, Di Loreto C, et al. Association between plasminogen activator inhibitor 1 gene polymorphisms and preeclampsia. Gynecol Obstet Invest 2003;56:17-22. [ Links ]
69. Glueck CJ, Kupferminc MJ, Fontaine RN, Wang P, Weksler BB, Eldor A. Genetic hypofibrinolysis in complicated pregnancies. Obstet Gynecol 2001;97:44-8. [ Links ]
70. Yamada N, Arinami T, Yamakawa-Kobayashi K, Watanabe H, Sohda S, et al. The 4G/5G polymorphism of the plasminogen activator inhibitor-1 gene is associated with severe preeclampsia. J Hum Genet 2000;45:138-41. [ Links ]
71. Gallery ED, Campbell S, Arkell J, Nguyen M, Jackson CJ. Preeclamptic decidual microvascular endothelial cells express lower levels of matrix metalloproteinase- 1 than normal. Microvasc Res 1999;57:340-6. [ Links ]
72. Jurajda M, Kankova K, Muzik J, Unzeitig V, Drabkova M, Izakovicova-Holla L, et al. Lack of an association of a single nucleotide polymorphism in the promoter of the matrix metalloproteinase-1 gene in Czech women with pregnancy-induced hypertension. Gynecol Obstet Invest 2001;52:124-7. [ Links ]
73. Laasanen J, Romppanen EL, Hiltunen M, Helisalmi S, Mannermaa A, Punnonen K, et al. Two exonic single nucleotide polymorphisms in the microsomal epoxide hydrolase gene are jointly associated with preeclampsia. Eur J Hum Genet 2002;10:569-73. [ Links ]
74. Zusterzeel PL, Peters WH, Visser W, Hermsen KJ, Roelofs HM, Steegers EA. A polymorphism in the gene for microsomal epoxide hydrolase is associated with pre-eclampsia. J Med Genet 2001;38:234-7. [ Links ]
75. Belo L, Gaffney D, Caslake M, Santos-Silva A, Pereira-Leite L, Quintanilha A, et al. Apolipoprotein E and cholesteryl ester transfer protein polymorphisms in normal and preeclamptic pregnancies. Eur J Obstet Gynecol Reprod Biol 2004;112:9-15. [ Links ]
76. Francoual J, Audibert F, Trioche P, Chalas J, Capel L, Lindenbaum A, et al. Is a polymorphism of the apolipoprotein E gene associated with preeclampsia? Hypertens Pregnancy 2002;21:127-33. [ Links ]
77. Francoual J, Audibert F, Claise C, Chalas J, Trioche P, Frydman R, et al. Implication of apolipoprotein E and the L-arginine-nitric oxide system in preeclampsia. Hypertens Pregnancy 1999;18:229-37. [ Links ]
78. Nagy B, Rigo J Jr, Fintor L, Karadi I, Toth T. Apolipoprotein E alleles in women with severe preeclampsia. J Clin Pathol 1998;51:324-5. [ Links ]
79. Wang Y, Walsh SW. Increased superoxide generation is associated with decreased superoxide dismutase activity and mRNA expression in placental trophoblast cells in pre-eclampsia. Placenta 2001;22:206-12. [ Links ]
80. Wang Y, Walsh SW. Antioxidant activities and mRNA expression of superoxide dismutase, catalase, and glutathione peroxidase in normal and preeclamptic placentas. J Soc Gynecol Investig 1996;3:179-84. [ Links ]
81. Ohta K, Kobashi G, Hata A, Yamada H, Minakami H, Fujimoto S, et al. Association between a variant of the glutathione S-transferase P1 gene (GSTP1) and hypertension in pregnancy in Japanese: interaction with parity, age, and genetic factors. Semin Thromb Hemost 2003;29:653-9. [ Links ]
82. Knapen MF, Peters WH, Mulder TP, Merkus HM, Jansen JB, Steegers EA. Plasma glutathione Stransferase Pi 1-1 measurements in the study of hemolysis in hypertensive disorders of pregnancy. Hypertens Pregnancy 1999;18:147-56. [ Links ]
83. Ohta K, Kobashi G, Hata A, Yamada H, Minakami H, Fujimoto S, et al. Association between a variant of the glutathione S-transferase P1 gene (GSTP1) and hypertension in pregnancy in Japanese: interaction with parity, age, and genetic factors. Semin Thromb Hemost 2003;29:653-9. [ Links ]
84. Hylenius S, Andersen AM, Melbye M, Hviid TV. Association between HLA-G genotype and risk of preeclampsia: a case-control study using family triads. Mol Hum Reprod 2004;10:237-46. [ Links ]
85. O'Brien M, McCarthy T, Jenkins D, Paul P, Dausset J, Carosella ED, Moreau P. Altered HLA-G transcription in pre-eclampsia is associated with allele specific inheritance: possible role of the HLA-G gene in susceptibility to the disease. Cell Mol Life Sci 2001; 58:1943-9. [ Links ]
86. O'Brien M, Dausset J, Carosella ED, Moreau P. Analysis of the role of HLA-G in preeclampsia. Hum Immunol 2000; 61:1126-31. [ Links ]
87. Takakuwa K, Honda K, Ishii K, Hataya I, Yasuda M, Tanaka K. Studies on the HLA-DRB1 genotypes in Japanese women with severe pre-eclampsia positive and negative for anticardiolipin antibody using a polymerase chain reaction-restriction fragment length polymorphism method. Hum Reprod 1999;14:2980-6. [ Links ]
88. de Luca Brunori I, Battini L, Simonelli M, Brunori E, Valentino V, Curcio M, et al. HLA-DR in couples associated with preeclampsia: background and updating by DNA sequencing. J Reprod Immunol 2003;59:235-43. [ Links ]
89. Kilpatrick DC. HLA-DR4 and pre-eclampsia. Hum Reprod 2000;15:2447. [ Links ]
90. Wilton AN, Cooper DW. Pre-eclampsia and HLA-DR4. Lancet 1990;336:323. [ Links ]
91. Kilpatrick DC, Gibson F, Livingston J, Liston WA. Pre-eclampsia is associated with HLA-DR4 sharing between mother and fetus. Tissue Antigens 1990; 35:178-81. [ Links ]
92. Kilpatrick DC, Liston WA, Gibson F, Livingstone J. Association between susceptibility to pre-eclampsia within families and HLA DR4. Lancet 1989;2:1063-5. [ Links ]
93. Heiskanen J, Romppanen EL, Hiltunen M, Iivonen S, Mannermaa A, Punnonen K, et al. Polymorphism in the tumor necrosis factor-alpha gene in women with preeclampsia. J Assist Reprod Genet 2002;19:220-3. [ Links ]
94. Lachmeijer AM, Crusius JB, Pals G, Dekker GA, Arngrimsson R, ten Kate LP. Polymorphisms in the tumor necrosis factor and lymphotoxin-alpha gene region and preeclampsia. Obstet Gynecol 2001;98:612-9. [ Links ]
95. Chen G, R Wilson, Wang SH, Zheng HZ, Walker JJ, McKillop JH. Tumour necrosis factor-alpha (TNFalpha) gene polymorphism and expression in preeclampsia. Clin Exp Immunol 1996;104:154-9. [ Links ]
96. Kilpatrick DC. Influence of human leukocyte antigen and tumour necrosis factor genes on the development of pre-eclampsia. Hum Reprod Update 1999;5:94-102. [ Links ]
97. Rinehart BK, Terrone DA, Lagoo-Deenadayalan S, Barber WH, Hale EA, Martin JN Jr, et al. Expression of the placental cytokines tumor necrosis factor alpha, interleukin 1beta, and interleukin 10 is increased in preeclampsia. Am J Obstet Gynecol 1999;181:915-20. [ Links ]
98. Lachmeijer AM, Nosti-Escanilla MP, Bastiaans EB, Pals G, Sandkuijl LA, Kostense PJ, et al. Linkage and association studies of IL1B and IL1RN gene polymorphisms in preeclampsia. Hypertens Pregnancy 2002;21:23-38. [ Links ]
99. Hefler LA, Tempfer CB, Gregg AR. Polymorphisms within the interleukin-1 beta gene cluster and preeclampsia. Obstet Gynecol 2001;97:664-8. [ Links ]
100. Altmüller J, Palmer LJ, Fischer G, Scherb H, Wjst M. Genomewide scans of complex human diseases: True linkage is hard to find. Am J Hum Genet 2001; 69:936-50. [ Links ]
101. Haines JL, Pericak-Vance MA, editors. Approaches to gene mapping in complex human diseases. New York: Wiley-Liss; 1998. [ Links ]
102. Strachan T, Read AP, editors. Human molecular genetics. 2nd. edition. New York: Wiley-Liss; 1999. [ Links ]
103. Griffiths AJF, Miller JH, Suzuki DT, Lewontin RC, Gelbart WM. Basic eukaryotic chromosome mapping. En: Griffiths AJF, Miller JH, Suzuki DT, Lewontin RC, Gelbart WM, editors. An introduction to genetic analysis. New York: WH Freeman; 2000. p.141-73. [ Links ]
104. Lander E, Kruglyak L. Genetic dissection of complex traits. Nat Genet 1995;11:241-7. [ Links ]
105. Wilton AN, Cooper DW, Brennecke SP, Bishop SM, Marshall P. Absence of close linkage between maternal genes for susceptibility to preeclampsia/eclampsia and HLA DRb. Lancet 1990;336:653-7. [ Links ]
106. Ward K, Hata A, Jeunemaitre X, Helin C, Nelson L, Namikawa C, et al. A molecular variant of angiotensinogen associated with preeclampsia. Nat Genet 1993;4:59-61. [ Links ]
107. Arngrímsson R, Purandare S, Connor M, Walker JJ, Bjornsson S, Soubrier F, et al. Angiotensinogen: a candidate gene involved in preeclampsia? Nat Genet 1993;4:115. [ Links ]
108. Weeks DE, Lange K. A multilocus extension of the affected-pedigree-member method of linkage analysis. Am J Hum Genet 1992; 50:859-68. [ Links ]
109. Morgan L, Baker P, Pipkin FB, Kalsheker N. Preeclampsia and the angiotensinogen gene. Br J Obstet Gynaecol 1995;102:489-90. [ Links ]
110.Arngrimsson R, Hayward C, Nadaud S, Baldursdottir A, Walker JJ, Liston WA, et al. Evidence for a familial pregnancy-induced hypertension locus in the eNOS-gene region. Am J Hum Genet 1997;61:354-62. [ Links ]
111. Spielman RS, McGinnis RE, Ewens WJ. Transmission disequilibrium test for linkage disequilibrium: the insuline gene region and IDDM. Am J Human Genet 1993;52:506-16. [ Links ]
112. Terwilliger JD. A powerful likelihood method for the analyses of linkage disequilibrium between trait loci and one or more polymorphic marker loci. Am J Hum Genet 1995;56:777-87. [ Links ]
113. Lewis I, Lachmeijer G, Downing S, Dekker G, Glazebrook C. Failure to detect linkage of preeclampsia of the NOS3 locus in chromosome 7q. Am J Hum Genet 1999;64:310-3. [ Links ]
114. Roberts JM, Lain KY. Recent insights into the pathogenesis of pre eclampsia. Placenta 2002;23:359- 72.
115. Arngrimsson R, Sigurdardottir S, Frigge ML, Bjarnadóttir RI, Jónsson T, Stefansson H, et al. A genome wide scan reveals a maternal susceptibility locus for pre eclampsia on chromosome 2p13. Hum Mol Genet 1999;8:1799-805. [ Links ]
116. Moses EK, Lade JA, Guo G, Wilton AN, Grehan M, Freed K, et al. A genome scan in families from Australia and New Zealand confirms the presence of a maternal susceptibility locus for pre-eclampsia, on chromosome 2. Am J Hum Genet 2000;67:1581-5. [ Links ]
117. Lachmeijer AM, Arngrímsson R, Bastiaans EJ, Frigge ML, Pals G, Sigurdardottir S, et al. A genomewide scan for preeclampsia in the Netherlands. Eur J Hum Genet 2001;9:758-64. [ Links ]
118. Laivuori H, Lahermo P, Ollikainen V, Widen E, Haiva- Mallinen L, Sundstrom H, et al. Susceptibility loci for preeclampsia on chromosomes 2p25 and 9p13 in Finnish families. Am J Hum Genet 2003;72:168-77. [ Links ]
119. Laasanen J, Hiltunen M, Romppanen EL, Punnonen K, Mannermaa A, Heinonen S. Microsatellite marker association at chromosome region 2p13 in Finnish patients with preeclampsia and obstetric cholestasis suggests a common risk locus. Eur J Hum Genet 2003;11:232-6. [ Links ]
120. Wareing M, Bhatti H, O'Hara M, Kenny L, Warren AY, Taggart MJ, et al. Vasoactive effects of neurokinin B on human blood vessels. Am J Obstet Gynecol 2003; 188:196-202. [ Links ]
121.Page NM, Lowry PJ. Is 'pre-eclampsia' simply a response to the side effects of a placental tachykinin? J Endocrinol 2000;167:355-61. [ Links ]
122.Konijnenberg A, Stokkers EW, van der Post JA, Schaap MC, Boer K, Bleker OP, et al. Extensive platelet activation in preeclampsia compared with normal pregnancy: enhanced expression of cell adhesion molecules. Am J Obstet Gynecol 1997;176: 461-9. [ Links ]
123.Carvajal-Carmona LG, Soto ID, Pineda D, Ortíz Barrientos D, Duque C, Ospina-Duque J, et al. Strong Amerindian/White sex bias and a possible sephardic contribution among the founder of a population in Northwest Colombia. Am J Hum Genet 2000;67:1287-95. [ Links ]
124.Mesa NR, Mondragon MC, Soto ID, Parra MV, Duque C, Ortiz-Barrientos D et al. Autosomal, mtDNA, and Y-chromosome diversity in Amerindians: pre- and post Columbian patterns of gene flow in South America. Am J Hum Genet 2000;67:1277-86. [ Links ]
125.Carvajal-Carmona LG, Ophoff R, Service S, Hartiala J, Molina L, Leon P e t al. Genetic demography of Antioquia (Colombia) and the Central Valley of Costa Rica. Hum Genet 2003;112:534-41. [ Links ]
.














