Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.24 n.3 Bogotá set. 2004
Evaluación de varias técnicas de extracción de ADN de
Cryptococcus spp. a partir de muestras ambientales
Alexandra Castañeda 1 , Juan McEwen 2,3 , Marylin Hidalgo 1 , Elizabeth Castañeda 1
1 Grupo de Microbiología, Instituto Nacional de Salud, Bogotá, D.C., Colombia.
2
Corporación para Investigaciones Biológicas, CIB, Medellín, Colombia.3
Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia.El género Cryptococcus comprende, al menos, 38 especies, pero sólo 3 se han informado como patógenas para el hombre y los animales: Cryptococcus laurentii, Cryptococcus albidus y Cryptococcus neoformans; esta última es la más frecuente. La infección se adquiere por la inhalación de los propágulos infectantes del medio ambiente. Los estudios del hábitat se han realizado con técnicas de extracción con soluciones tampón y cultivos en medios selectivos. El objetivo del trabajo fue evaluar varias técnicas de extracción del ADN de Cryptococcus spp . a partir de muestras ambientales. Como controles se emplearon aislamientos de C. neoformans, C. albidus, C. laurentii y Paracoccidiodes brasiliensis. Se emplearon vermiculitas, suelos contaminados en el laboratorio con 10 a 10 6 blastoconidias/g y muestras naturalmente colonizadas con C. neoformans. El ADN se extrajo con métodos físicos, químicos y con un estuche comercial, y se purificó usando bloques de agarosa y columnas de sílica. Para la amplificación con PCR se emplearon los iniciadores CN4-CN5 específicos para C. neoformans.
Sólo el estuche comercial permitió extraer y purificar el ADN de las muestras de suelos contaminados hasta una concentración de 10 blastoconidias/g de suelo y de una de las muestras naturalmente colonizadas. Con este trabajo se logró la extracción y amplificación de ADN de Cryptococcus spp. a partir de muestras ambientales lo cual constituye una herramienta importante para delimitar las áreas ecológicas de C. neoformans en nuestro país.
Palabras clave: Cryptococcus spp ., ecología, extracción de ADN, PCR, muestras ambientales.
Cryptococcus spp. DNA extraction from environmental samples
The genus Cryptococcus encompasses 38 species, but only 3 are associated with disease in humans and animals, Cryptococcus laurentii, Cryptococcus albidus and Cryptococcus neoformans. The last one is the most frequently reported. The disease is acquired by the inhalation of infectious propagules present in the environment. The habitat has been established using extraction techniques with buffer supplemented with antibiotics and plating in selective media. The aim of this work was to evaluate several DNA extraction techniques for Cryptocococus spp . from environmental samples. The control isolates were C. neoformans, C. albidus, C. laurentii and Paracoccidiodes brasiliensis. We also used vermiculita and soil samples contaminated with different yeast concentrations (10 to 10 6 cells/g) and samples naturally contaminated with C. neoformans. DNA was extracted with physical and chemical methods and with a commercial kit, and the DNA was purified with agarose blocks and silica columns. For the PCR amplification we used the CN4-CN5 primers, which are specific for C. neoformans. Only the commercial kit allowed DNA extraction and amplification from contaminated soil samples up to a concentration of 10 cells/g and from one sample naturally colonized. With this work we extracted and amplified DNA from Cryptococcus spp. from environmental samples with appropriate PCR specificity, it will be a tool to establish the ecological areas of C. neoformans in our country.
Key words: Cryptococcus spp ., ecology, DNA extraction, PCR, environmental samples.
La criptococosis es una micosis oportunista, cuyo agente etiológico son las tres variedades y el híbrido de la levadura capsulada Cryptococcus neoformans. El hombre se infecta por la inhalación de blastoconidias poco capsuladas (4-5 µm) o con las basidiosporas (3 µm ) de las diferentes variedades que se encuentran en el medio ambiente (1-4).
El hábitat de la var. grubii y neoformans se ha asociado desde 1950 con excrementos de aves, principalmente palomas (1), mientras que el de la var. gattii sólo a partir de 1990 se ha asociado con los detritos y material vegetal de diferentes árboles como Eucalyptus, acacias, ficus, oities y almendros (2,5-7). El hábitat de otras especies de Cryptococcus como Cryptococcus laurentii y Cryptococcus albidus se asocia con aire, vino, suelos, hojas, queso, flores, agua y frutas (1).
En los estudios realizados para determinar el hábitat de Cryptococcus sp. se han empleado dos procedimientos: la extracción con una solución tampón con antibióticos y su posterior siembra en medios selectivos, o la inoculación de estos extractos en animales de laboratorio (1). Ambos procedimientos requieren la viabilidad del microorganismo y una adecuada concentración en la muestra, lo que limita su sensibilidad (1).
Las técnicas que se emplean tradicionalmente en el estudio de bacterias del suelo son la extracción del ADN por congelación, maceración y su purificación en columnas de Sephadex G-200 (8) y la extracción directa por lisis con lisozima y la posterior congelación-maceración de la muestras, seguida por la extracción con sodio dodecilsulfato y fenol-cloroformo (9). Otras técnicas de extracción existentes incluyen ruptura física con perlas de vidrio y vórtex o con agitadores mecánicos (10,11), congelación de la muestra en nitrógenolíquido y su posterior maceración y sonicación y la lisis química con el empleo de fenol-cloroformo-alcohol isoamílico (10,11).
Es importante anotar que el paso siguiente a estos procedimientos son los métodos de purificación del ADN para eliminar los contaminantes que causan pérdida del ADN por degradación o por absorción, o que inhiben la Taq polimerasa empleada en la PCR (9). Para tal fin se han empleado bloques de agarosa, polivinilpirrilidona, bromuro de hexadecil-trimetilamonio (CTAB), columnas de sílica y membranas de Sephadex (9,12-14). Estos métodos se han empleado para el estudio de bacterias como Erwinia sp. (15), Desulforomas y Dehalococcocides (16) y hongos como Paracoccidioides brasiliensis (13), Histoplasma capsulatum (17 ) y Pneumocystis jiroveci (carinii) (18).
El objetivo del presente estudio fue evaluar varias técnicas de extracción y purificación de ADN de Cryptococcus spp . a partir de muestras ambientales, con el fin de utilizar la mejor de ellas como herramienta para delimitar el hábitat de este importante patógeno en nuestro país.
Materiales y métodos
Experimentos con las cepas control
Aislamientos control y ADN de P. brasiliensis. Se utilizaron 13 aislamientos de C. neoformans, discriminados de la siguiente manera: 2 aislamientos clínicos y 2 aislamientos ambientalesde C. neoformans, var. grubii serotipo A (NIH 68 A, WM-10, INS-551, INS-857), un aislamiento clínico de C. neoformans var. neoformans serotipo D (INS-20), 2 aislamientos clínicos y 2 ambientales de C. neoformans var. gattii serotipo B (NIH 112, NIH-4506, INS-453, INS-1112), 2 aislamientos clínicos y 2 ambientales de C. neoformans var. gattii serotipo C (NIH 191, NIH 18, INS-755, INS-682). También se empleó un aislamiento ambiental de C. laurentii (INS L-121) uno de C. albidus (INS A-13) y ADN de P. brasiliensis (como control negativo) · Extracción del ADN de cepas control. Se emplearon dos métodos de extracción por medio de ruptura física con perlas de vidrio de acuerdo con el protocolo descrito por Díez (13). Brevemente, las cepas de Cryptococcus sp. fueron sembradas en agar glucosado de Sabouraud durante 48 h, se agregó un tampón de extracción y una mezcla de fenol-cloroformo- alcohol isoamílico (25:24:1). Se adicionaron las perlas de vidrio (Sigma, grosor 0,4-0,6 mm) y se mezcló en vórtex por 30 minutos; se realizó una segunda extracción con fenol-cloroformo-alcohol isoamílico y el AND se precipitó con etanol absoluto y acetato de amonio; se lavó con etanol al 70%, se resuspendió en tampón TE y la concentración del ADN se midió por espectrofotometría en una absorbancia de 260 a 280 nm y se determinó su pureza teniendo en cuenta este valor; la pureza del ADN fue óptima cuando la relación entre la A 260/280 nm fue mayor de 1,8 (13). En el otro método de extracción se empleó nitrógeno líquido, según el método descrito por Meyer (19). El ADN extraído se amplificó mediante PCR con las condiciones descritas más adelante.
Material inerte
Un gramo de cada una de los tres tipos de vermiculita (Fumitoro Agroquímicos) disponibles en el mercado (fina, media y gruesa) se contaminó con 10 7 células por ml de la cepa control de C. neoformans var. gattii serotipo C (INS -755). Los experimentos semicuantitativos de contaminación se realizaron con concentraciones desde 10 hasta 1 x10 7 células por gramo de la cepa control, contaminando 1 g de vermiculita.
· Extracción y amplificación del ADN a partir de la vermiculita. Se probaron dos métodos de extracción: extracción con perlas de vidrio (13) y el método de extracción con nitrógeno líquido con posterior purificación con bloques de agarosa para eliminar los posibles inhibidores de la PCR presentes en la muestra. Brevemente, se preparó un molde para el bloque con una jeringa de tuberculina y se dispensaron en él 80 µl de ADN más 80 µl de agarosa de bajo punto de fusión. La mezcla se dejó solidificar a -20ºC y, luego, los bloques de agarosa se lavaron con el tampón TE durante 48 h, cambiando el tampón cada 24 h para eliminar los inhibidores de la PCR presentes en los suelos. Finalmente, los bloques se calentaron a 60ºC hasta su disolución y, a partir de los mismos, se tomó 1 µl para realizar la PCR. El ADN extraído se amplificó mediante PCR con las condiciones descritas más adelante.
Muestras ambientales
· Suelos contaminados en el laboratorio. Se emplearon suelos sin esterilizar, procedentes de un vivero y como inóculo se utilizó el aislamiento INS-755. La contaminación se controló con la enumeración de unidades formadoras de colonias por gramo (UFC/g) en los medios selectivos, agar ácido cafeico y agar semillas de Guizotia (20) con las concentraciones de 1x10 6 , 1x10 4 , 1x10 2 y 10 UFC/g.
· Métodos de extracción de ADN a partir de suelos contaminados en el laboratorio. Se emplearon cuatro métodos, como se muestra en el
cuadro 1, y para cada uno de ellos se contaminaron diferente número de muestras con la cepa control de C. neoformans var. gattii, serotipo C. El número de muestras fue diferente para cada uno de los métodos empleados ya que no se hicieron en paralelo; se hizo un ensayo después de otro para ver si se mejoraban los resultados de la extracción.
Cada uno de los métodos de extracción evaluados incluyó un método de purificación del AND diferente. Para los métodos de perlas de vidrio y nitrógeno líquido se realizó la purificación usando bloques de agarosa; para el método de agitación mecánica seguida de CTAB, se usaron columnas de sílica y el método del estuche comercial dispone de una membrana para este fin. Posteriormente, el ADN obtenido por los cuatro métodos anteriormente descritos se amplificó mediante PCR con las condiciones descritas posteriormente.
Suelos naturalmente colonizados
Se emplearon 17 muestras de suelos naturalmente colonizadas, las cuales fueron recolectadas a partir de detritos de almendros entre 1997 y 1999 y estaban guardadas a 4°C. Todas habían sido positivas por cultivo para C. neoformans var. gattii, serotipo C y se les había repetido el cultivo en el año 2001 (21). Para la extracción del ADN se siguieron dos de los procedimientos descritos, a saber con perlas de vidrio y purificación con bloques de agarosa para eliminar los posibles inhibidores de la PCR presentes en la muestra (13) y con el estuche comercial Ultra Clean soil DNA, de laboratorios MoBio (referencia 12800-50). El ADN extraído mediante los dos métodos anteriores se amplificó por PCR con las condiciones descritas a continuación.
Condiciones para la PCR. Se establecieron según la metodología descrita por Mitchell (22). La mezcla se realizó en el buffer tris (Perkin-Elmer) 10X (5 µl) pH 8,3, cloruro de magnesio (Perkin-Elmer) 25 mM (3 µl), acetato de magnesio 50 mM (3µl), mezcla de dideoxinucleótidos trifosfato (Promega) 100 mM (1 µl de cada uno), Taq polimerasa (Perkin Elmer) 5U (1 µl), los iniciadores CN4 (5' ATC ACC TTC CCA CTA ACA CAT T 3') y CN5 (5' GAA GGG CAT GCC TGT TTG AGA G 3') a una concentración de 10 pg/µl, 25 ng de AND procedente de cultivo ó 1µl del ADN extraído de las muestras, en con un volumen final de 50 µl. Los controles positivos empleados para la PCR fueron cepas control de C. neoformans, ya que los iniciadores CN4 y CN5 son informados en la literatura como específicos para esta especie (22). Los controles negativos fueron ADN de cepas control de otras especies de Cryptococcus como C. laurentii y C. albidus, de P. brasiliensis y de muestras de suelos no contaminados. El control negativo de la reacción contenía todos los reactivos empleados en la PCR, excepto el ADN. El perfil de amplificación usado fue el siguiente: 1 ciclo inicial a 95°C por 5 m, 30 ciclos 95°C x 30 ,58 °C x 30 s y 72°C x 30 s y 1 ciclo final a 72°C x 5 m. La visualización de los productos de la PCR se realizó mediante electroforesis horizontal en geles de agarosa al 2 % con 0,3 µg/ml de bromuro de etidio. El tamaño de la banda amplificada se estableció mediante comparación con un marcador de peso molecular de 100 pb (Promega) (22).
Sensibilidad de la PCR. La sensibilidad de la prueba se definió como la mínima cantidad de ADN detectada por los iniciadores empleando la técnica de PCR. Se determinó con diluciones del ADN de la cepa control C. neoformans INS-755 (desde 20 ng hasta 5 pg).
Resultados
Cepas control: extracción del ADN y sensibilidad de la PCR
Con el fin de identificar el mejor método para extracción de ADN, se realizó primero la extracción de varias cepas a partir de cultivos puros. Elpromedio de ng/µl obtenido para las 13 cepas control de C. neoformans, C. albidus y C. laurentii con el método de extracción con perlas de vidrio fue de 845 ng/µl (465ng/µl - 1070 ng/µl), mientras ue para el método de extracción con nitrógeno fue de 644 ng/µl (199 ng/µl - 937 ng/µl).
Para determinar la sensibilidad de la PCR se utilizó este ADN purificado. La mínima cantidad de AND detectada con los iniciadores CN4-CN5 fue de 50 pg de ADN (
Figura 1a).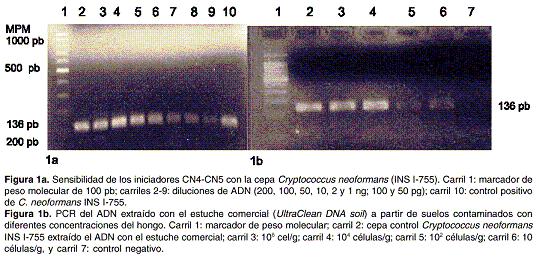
Extracción, purificación y amplificación del AND a partir de vermiculita
Como primer paso en el intento de estandarizar un protocolo de extracción a partir de muestras ambientales se procedió a contaminar vermiculita con células del hongo con el fin de verificar posteriormente si el ADN extraído a partir de ella era de buena calidad y qué cantidad se podía recuperar después del procedimiento. La vermiculita es un mineral laminar que tiene apariencia de mica. La única vermiculita contaminada con la cepa control que amplificó con los iniciadores CN4-CN5 fue la vermiculita fina; por tanto, fue escogida para los experimentos semi-cuantitativos de contaminación. Con el método de extracción con perlas de vidrio y purificación con bloques de agarosa se logró amplificar con la PCR únicamente la muestra contaminada con 1x10 6 células/g y con el método de nitrógeno líquido se logró amplificar hasta la muestra contaminada con 10 células/g.
Extracción, purificación y amplificación del ADN a partir de suelos contaminados en el laboratorio
Una vez lograda la extracción a partir de vermiculita, se procedió a contaminar suelos con diferentes concentraciones del hongo. Se obtuvieron 9.600 UFC/ml para las muestras de suelos contaminadas con 1x10 6 células/g y 1x10 4 células/g y 648 para las muestras contaminadas con 10 2 células/g. No se logró recuperar el hongo en las muestras contaminadas con 10 células/g. Estas muestras fueron luego extraídas usando diferentes métodos (cuadro 1).
a) Método de extracción con perlas de vidrio y purificación con bloques de agarosa. De las 18 muestras contaminadas con concentraciones desde 1x10 3 hasta 2,5 x 10 8 , no se logró amplificar el ADN a partir de ninguna de ellas empleando los iniciadores CN4 y CN5. De las 8 muestras contaminadas con 3x10 8 , se logró amplificar AND a partir de 1 muestra (12,5%) y de las 68 muestras contaminadas con 3,5x10 8 se logró amplificar el ADN de 33 de ellas (48,5%). Los resultados anteriores señalan que para utilizar este método de extracción se requiere una alta concentración de las levaduras en el suelo (3,5x10 8 células/g de suelo) para lograr amplificar el ADN con el empleo de la PCR.
b) Método de extracción con nitrógeno líquido y purificación con bloques de agarosa. De las 77 muestras de suelos contaminados con las diferentes concentraciones del hongo, se logró amplificar ADN a partir de 9 de ellas (11,7%) empleando los iniciadores CN4 y CN5. Sinembargo, no se observó relación entre la cantidad del inóculo con el que se contaminaron las muestras y la positividad de la PCR; de las 4 muestras contaminadas con 10 y 1x10 2 célulasl/g sólo amplificaron 2 (50%); de las 8 muestras contaminadas con 1x10 3 -1x10 5 células/g y de las 10 contaminadas con 3,5x10 8 células/g no amplificó ninguna; de las 18 muestras contaminadas con 1x10 6 -1x10 8 , amplificaron 3 (16%); de las 27 muestras contaminadas con 2,5x10 8 células/g, amplificaron 3 (11%), y de las 10 muestras contaminadas con 3x10 8 células/g sólo amplificó una (10%).
c) Método de extracción con "bead-beater", CTAB y purificación con sílica. De las 18 muestras de suelos contaminadas no se logró amplificar AND a partir de ninguna de ellas empleando la PCR con los iniciadores CN4 y CN5.
d) Método de extracción con el estuche comercial. De las 4 muestras de suelos contaminadas se logró amplificar el ADN a partir de todas utilizando los iniciadores CN4-CN5 mediante PCR (figura 1b).
Extracción, purificación y amplificación del ADN a partir de suelos naturalmente colonizados
Los resultados obtenidos a partir de suelos contaminados en el laboratorio indicaron que las mejores extracciones se obtenían utilizando perlas de vidrio y el estuche comercial. Por consiguiente, se emplearon únicamente estas dos técnicas al realizar la extracción a partir de suelos naturalmente contaminados.
a) Método de extracción con perlas de vidrio y purificación con bloques de agarosa. Se realizaron dos ensayos con 2 de las 17 muestras y no se obtuvo producto de amplificación.
b) Método de extracción con el estuche comercial. Se realizaron ensayos de extracción a partir de 7 de las 17 muestras, amplificando una de las muestras.
Discusión
El objetivo del presente estudio fue evaluar varias técnicas de extracción y purificación de ADN de Cryptococcus spp. a partir de muestras ambientales, como una alternativa para el método tradicional de recuperación del hongo con la siembra en medios selectivos, con el fin de utilizar la mejor de ellas como herramienta para delimitar el hábitat de este importante patógeno en nuestro país. Este método debería darnos la posibilidad de obtener ADN con una calidad y pureza suficientes para ser amplificado mediante una PCR. Para cumplir con este objetivo, se inició con una extracción del ADN del hongo a partir de cultivos; luego, se determinaron las condiciones para extraer el ADN a partir de un material inerte (vermiculita) contaminado con el hongo; postriormente, se evaluaron varias alternativas para la extracción a partir de suelos contaminados en el laboratorio y, finalmente, se trabajó con muestras de suelos naturalmente colonizadas con el hongo. Este proceso permitió establecer que el método de nitrógeno, que funciona con las cepas control, no es igualmente eficiente cuando se utiliz a en muestras de suelos contaminadas o naturalmente colonizadas. Además, este método no presentó una correlación entre la cantidad de muestra inoculada en los suelos y el AND amplifcado por PCR, aunque éste es el método recomendado por Meyer para la extracción del ADN de C. neoformans a partir de cultivo (19). La falta de correlación entre el inóculo inicial y una reacción positiva por PCR se puede explicar ya sea por variabilidad en la eficiencia de extracción del ADN o por la presencia de inhibidores de la PCR. Debido a la baja cantidad de ADN que usualmente se recupera a partir de suelos, fue imposible determinar la presencia de AND directamente con medición espectrofotométrica. Sin embargo, el hecho de que todos los suelos contaminados tenían el mismo origen y, por ende, la presencia de inhibidores ha debido ser similar en todas las muestras, sugiere un problema inherente al protocolo de extracción cuando se trata de suelos, el cual presenta gran variabilidad ya sea en la eficiencia de extracción de ADN o en el grado de pureza del ADN obtenido. Es importante anotar, además, que para realizar este método de extracción fue necesario partir de una muestra muy pequeña de suelo (30 mg a 200 mg), ya que una muestra de 1 g fue difícil de macerar completamente. Esto, por consiguiente, dificulta la obtención de buena cantidad de ADN con este método.
Teniendo en cuenta los anteriores resultados, fue necesario identificar nuevas estrategias para lograr una extracción eficiente a partir de estas muestras. El procedimiento de extracción del ADN con perlas de vidrio y vórtex ha sido empleado con éxito en aislamientos de Aspergillus fumigatus, Bacillus globigii, Fusarium y P. brasiliensis (13,23,24). Este. método no emplea enzimas lo cual disminuye el costo de la técnica, es fácil de realizar en el laboratorio y no requiere de una infraestructura especial. Sin embargo, cuando se empleó con las muestras de suelos contaminados con diferentes concentraciones del hongo (10-3,5x10 8 células/ g), la técnica fue poco sensible y requirió una alta concentración del inóculo en los suelos (3,5x10 8 UFC/g). En la literatura se han informado densidades de 30-60x10 6 blasto-conidias/g de C. neoformans en excrementos secos de palomas (25) y de 10 6 células/g en el polvo de casas de pacientes en África (1). Por consiguiente, la alta concentración del inóculo requerida en nuestro estudio es poco probable encontrarla en la naturaleza, y dado el caso que ocurriera, resultaría más económico realizar el protocolo tradicional de extracción a partir de ella y su posterior siembra en medios selectivos. Esto podría explicar el hecho que no se logró amplificar ADN a partir de las dos muestras ambientales analizadas con este método.
El método con el agitador mecánico, CTAB y sílica no funcionó con ninguna de las muestras contaminadas, sin embargo, en la literatura existen varios informes en que se emplea este protocolo de extracción para el ADN de bacterias a partir de muestras ambientales (9,11). En este caso, otra vez, es posible que hubieran quedado inhibidores de la PCR en las extracciones realizadas para lo cual habría que realizar pasos adicionales de limpieza del ADN. Los pasos adicionales de purificación aumentan los costos y el tiempo de trabajo y, por consiguiente, le restan la utilidad de este protocolo.
La extracción del ADN a partir de muestras ambientales con el estuche comercial ( UltraClean soil DNA) fue el método que permitió detectarlo en todas las muestras de suelos contaminados en el laboratorio hasta una concentración de 10 células de C. neoformans por gramo de suelo por PCR. Este estuche ha sido utilizado para la extracción de ADN de diferentes microorganismos a partir de muestras ambientales, entre ellas, bacterias como Desulforomonas y Dehalo-coccoides (16) detección de arqueas a partir de muestras de agua (26) y en estudios para la evaluación de la diversidad microbiana usada en la biorremediación (27). Hasta el momento no se han publicado estudios con ADN de hongos utilizando este método de extracción. Los resultados de este estudio señalan que la técnica funciona en hongos, lo cual representa una metodología muy útil para los estudios ecológicos.
Agradecimientos
Este proyecto fue cofinanciado por el Instituto Colombiano para el Desarrollo de la Ciencia y la Tecnología "Francisco José de Caldas" (Colciencias), código 2104-04-903-98, contrato367/99. A Sandra Huérfano por su colaboración en la elaboración de este trabajo y por sus comentarios al manuscrito. A los dos evaluadores por sus acertadas sugerencias y comentarios, los cuales enriquecieron el manuscrito.
Correspondencia:
Elizabeth Castañeda, Grupo de Microbiología, Instituto Nacional de Salud, Avenida calle 26 No. 51-60, Bogotá, D.C., Colombia.
Teléfono: (571) 220 7700, extensión 445
Recibido:14/04/04; aceptado: 07/09/04
Referencias
1. Casadevall A, Perfect JR. Cryptococcus neoformans. Washington, D.C.: American Society for Microbiology Press; 1998. [ Links ]
2. Ellis DH, Pfeiffer T. Natural habitat of Cryptococcus neoformans var. gattii. J Clin Microbiol 1990;28:1642-4. [ Links ]
3. Levitz SM. The ecology of Cryptococcus neoformans and the epidemiology of cryptococcosis. Rev Infect Dis 1991;13:1163-8.
4. Mitchell TG, Perfect JR. Cryptococcosis in the e a of AIDS 100 years after the discovery of Cryptococcus neoformans. Clin Microb Rev 1995;8:515-48.
5. Sorrel T. Cryptococcus neoformans variety gattii. Med Mycol 2001;39:155-68. [ Links ]
6. Lazera MS, Salmito MA, Londero AT, Trilles L, Nishikawa MM, Wanke B. Possible primary ecological niche of Cryptococcus neoformans. Med Mycol 2000;38: 379-83. [ Links ]
7. Callejas A, Ordóñez N, Rodríguez MC, Castañeda E. First isolation of Cryptococcus neoformans var. gattii, serotype C, from the environment in Colombia. Med Mycol 1998;36:341-4. [ Links ]
8. Tsai YL, Oson BH. Rapid method for separation of bacterial DNA from humic substance in sediments for polymerase chain reaction. Appl Environ Microbiol 1992; 58;2292-5. [ Links ]
9. Tsai YL, Olson BH. Rapid method for direct extraction of DNA from soil and sediments. Appl Environ Microbiol 1991;57:1070-4. [ Links ]
10. Left LG, Dana JR, McArthur JV, Shimkets LJ. Comparison of methods of DNA extraction from stream sediments. Appl Environ Microbiol 1995;61:1141-3. [ Links ]
11. Miller DN, Bryant JE, Madsen EL, Ghiorse WX. Evaluation of DNA extraction and purification procedures for soil and sediments samples. Appl Environ Microbiol 1999;65:4715-24. [ Links ]
12. Berthelet M, Whyte LG, Greer CW. Rapid, direct extraction of DNA from soils for PCR analysis using polivinylporrolidone spin columns. FEMS Microbiol Lett 1996;138:17-22. [ Links ]
13. Díez S, García EA, Pino PA, Botero S, Corredor GG, Peralta LA et al. PCR with Paracoccidioides brasiliensis specific primers: potential use in ecological studies. Rev Inst Med trop Sâo Paulo 1999;41:351-7. [ Links ]
14. Dijkman R, Jagers A, Kreps S, Collard JM, Mergeay M. Rapid method for purification of soil DNA for hybridization and PCR analysis. Microbiol Releases 1993; 2:29-34. [ Links ]
15. Llop P, Bonaterra A, Pañalver J, López MM. Development of a highly sensitive nested-PCR procedure using a single closed tube for detection of Erwinia amylovora in asymptomatic plant material. Appl Environ Microbiol 2000;66:2071-8. [ Links ]
16. Loeffler FE, Sun Q, Li J, Tiedje JM. 16S rRNA gene-based detection of tetrachloroethene-dechlorinating Desulfurormonas and Dehalococodides species. Appl Environ Microbiol 2000;66;1369-74. [ Links ]
17. Reid TM, Schafer MP. Direct detection of Histoplasma capsulatum in soil suspensions by two-stage PCR. Mol Cell Probes 1999;13:269-73. [ Links ]
18. Bartlett MS, Vermund SH, Jacobs R, Durant PJ, Shaw MM, Smith JW et al. Detection of Pneumocystis carinii DNA in air samples: likely environmental risk to susceptible persons. J Clin Microbiol 1997;35:2511-3. [ Links ]
19. Meyer W, Marszewska K, Amirmostofina M, Igreja RP, Hardtke C, Methling K et al. Molecular typing of global isolates of Cryptococcus neoformans var. neoformans by PCR- fingerprinting and RAPD. A pilot study to standardize techniques on which to base a detailed epidemiological survey. Electrophoresis 1999;20:1790-9. [ Links ]
20. Staib F. New concepts in the ocurrence and identification of Cryptococcus neoformans. Mycopathologia 1963;19: 143-5.
21. Castañeda A, Huérfano S, Rodríguez MC, Castañeda E. Recuperación de Cryptococcus neoformans var. gattii serotipo C a partir de detritos de almendros. Biomédica 2001;21:70-4. [ Links ]
22. Mitchell T, Freedman E, White T, Taylor JW. Unique oligonucleotides primers in PCR for identification of Cryptococcus neoformans. J Clin Microbiol 1994;32: 253-5. [ Links ]
23. Mohlehhoff P, Muller L, Gorushina A, Petersen K. Molecular approach to he characterization of fungal communities: methods for DNA extraction, PCR amplification and DGGE analysis of painted art objects. Fems Microbiol Lett 2001;195:169-73. [ Links ]
24. van Burik JAH, Screchise RW, White TC, Bowden RA, Myerson D. Comparison of six extraction techniques for isolation of DNA from filamentous fungi. Med Mycol 1998;36:299-303. [ Links ]
25. Littman M, Borok R. Relation of the pigeon to cryptococcosis: natural carries state, heat resistance and survival of Cryptococcus neoformans. Mycopathol Mycol Appl 1968;35:329-45. [ Links ]
26. Takai K, Horikoshi K. Rapid detection and quantification of members of the archael community by quantitative PCR using fluorogenic probes. Appl Environ Microbiol 2000;66:5066-72. [ Links ]
27. Dojka MA, Hugenholtz P, Haah SK, Pace NR. Microbial diversity in a hydrocarbon and chlorinated-solvent contaminated aquifer undergoing intrinsic bioremediation. Appl Environ Microbiol 1998;38:69-77. [ Links ]














