Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO  Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.25 no.1 Bogotá Mar. 2005
Evaluación de métodos fenotípicos y genotípicos para la detección de farmacorresistencia de Mycobacterium tuberculosis
Tania Bibiana Porras 1, Clara Inés León 1, Martha Inírida Guerrero 1, Anandi Martin 2,
FranÁoise Portaels 2, Juan Carlos Palomino 2
1 Grupo de Micobacterias, Instituto Nacional de Salud, Bogotá, D.C., Colombia.
2
Mycobacteriology Unit, Institute of Tropical Medicine, Antwerp, Belgium.Introducción.
La dificultad en el diagnóstico temprano y oportuno de la susceptibilidad a los medicamentos de Mycobacterium tuberculosis hace necesario el diseño y la evaluación de nuevas técnicas que superen la efectividad, reduzcan el costo y el tiempo que toman los métodos tradicionales.Objetivo. Evaluar y comparar dos metodologÍas fenotÍpicas y una genotípica, ÿtiles en la determinación rápida de susceptibilidad de M. tuberculosis, teniendo el método de las proporciones mÿltiples como referencia.
Materiales y métodos. Se incluyeron 21 cepas de M. tuberculosis para la evaluación de las metodologías fenotípicas de oxidación y reducción de Alamar azul (Maba) y el bromuro de 3-(4-5-dimetiletiazol-2-il)-2,5-difenil tetrazolio (MTT o Tema), frente a estreptomicina, isoniacida, rifampicina y etambutol. Mediante análisis ROC (receiver operative characteristic curves) se determinó el desempeño de las pruebas y se determinaron los valores pronósticos y el punto de corte para cada antibiótico. Además, mediante tablas de contingencia se determinó el desempeño y los valores predictivos de la metodología genotípica de PCR e hibridación reversa o rifoligotyping, versión 3, en la determinación de las cepas multirresistentes.
Resultados. Los perfiles de susceptibilidad por las pruebas colorimétricas se obtuvieron a los 7 días, con valores de área bajo la curva menores de 0,9, sensibilidad de 100% y especificidad mayor de 80%. La metodología de rifoligotyping resultó eficaz en la detección de mutirresistencia, con sensibilidad de 100% y especificidad de 93%.
Conclusión. Con los resultados obtenidos consideramos las pruebas Maba, Tema y rifoligotyping como alternativas rápidas y eficaces en la detección de farmacorresistencia de M. tuberculosis, aplicables a pacientes con alto riesgo de desarrollar tuberculosis multirresistente.
Palabras clave: Mycobacterium tuberculosis/efecto de drogas, Alamar azul, MTT, rifoligotyping, resistencia a mÿltiples drogas, tests de sensibilidad microbiana.
Phenotypic and genotypic methods for detecting multidrug resistance in Mycobacterium tuberculosis
Background. Expeditious charactization of drug susceptibility in Mycobacterium tuberculosis is difficult and, calls for the design and evaluation of faster, cheaper and more effective new techniques.
Objective. The aim of the current study was to compare one genotypic and two phenotypic methods for rapid susceptibility detection of M. tuberculosis.
Material and methods. Twenty-one M. tuberculosis strains were evaluated by phenotypic methodologies of oxidation and reduction of Alamar blue and MTT in the presence of streptomycin, isoniazid, rifampin and ethambutol. In all tests, the proportion method was applied as the comparison standard. By means of receiver operative characteristic (ROC) analysis, the performance, predictive values and threshold values for all drugs were determined. In addition, the performance of PCR and reverse line blot hybridization in establishing predictive values for sensitivlity and resistance were compared in contingency tables.
Results. The susceptibility patterns established by colorimetric techniques were obtained after seven days of incubation. The performances of these tests were excellent for all drugsó the areas under curves were >0.9, 100% of sensitivity (S) and specificity (E) >80%. The genotypic method of RFLP oligotyping detected multidrug resistance with S of 100% and E of 93%.
Conclusion. The results indicated that Alamar blue, MTT and RFLP methodologies are rapid and useful tools for characterizing multidrug resistance in M. tuberculosis, particularly for those patients with high risk of developing multidrug resistant tuberculosis.
Keywords: Mycobacterium tuberculosis, susceptibility testing, multidrug resistant, Alamar azul, MTT, rifoligotyping.
La infección con cepas resistentes y multirresistentes de Mycobacterium tuberculosis, dada la facilidad en la transmisión del bacilo además del impacto social, trae consigo elevados costos en cuanto a la implementación de nuevos esquemas terapéuticos. Por esta razón, es necesario detectar rápidamente estos casos para cortar oportunamente con la cadena de transmisión de estos bacilos (1).
La Organización Mundial de la Salud (OMS) calcula una prevalencia media de mutirresistencia global del 1% (2). Aparentemente, aunque esta cifra no es alarmante, existen regiones en las que se alcanzan prevalencias superiores al 10% (2). En Colombia, los estudios nacionales de resistencia inicial realizados en el Instituto Nacional de Salud han revelado prevalencias de multirresistencia inicial del 1,81% (IC95% 0,9 a 3,5) para 1991-1992, y de 1,5% (IC95% 0,9 a 2,4) para 1999-2000. Con estas cifras se concluye que en nuestro país la media de multirresistencia es similar a la global y aún no representa un grave problema de salud pública; sin embargo, por la falta de control y detección tardía, este porcentaje fácilmente puede incrementarse y convertirse en un grave problema de salud pública (3).
La actual metodología de referencia para la detección de susceptibilidad a los medicamentos es el método de las proporciones mÿltiples diseñado en 1961 (4); es costoso, laborioso y requiere un tiempo prolongado para emitir el resultado. Por ello, es indispensable implementar nuevas técnicas que reduzcan el tiempo y el costo para la detección de la susceptibilidad a los medicamentos. Ejemplo de ello es el sistema radiométrico Bactec que ofrece resultados en poco tiempo, pero sus mayores inconvenientes son el uso de material radioactivo y su elevado costo. El tubo indicador de crecimiento (Mycobacteria Growth Indicator Tube, MGIT) que usa un indicador fluorescente sensible a los cambios de presión de oxígeno es otra alternativa pero aÿn no está totalmente evaluado y registra altos porcentajes de contaminación dada la riqueza del medio y el uso de tubos con taparrosca. Este ÿltimo parámetro ha sido mejorado en el nuevo sistema BACTEC MGIT 960 (5,6).
Así mismo, las técnicas basadas en la difusión del antibiótico en placas de agar como el E-test ayudan en la determinación rápida de farmacorresistencia (7).
Los indicadores de viabilidad celular, como el alamar azulTM resazurin, permiten detectar crecimiento, viabilidad y susceptibilidad de M. tuberculosis a los medicamentos (8-10). Para detectar esta susceptibilidad de M. tuberculosis a los medicamentos se emplean microplacas con medio líquido, y de acuerdo con el indicador utilizado se conocen estas pruebas como Rema (resazurin microtiter assay) (11,12) y Maba (microdilution alamar blue assay) (13,14). Otro colorante usado es la sal de tetrazolio MTT (bromuro de 3(4,5-dimetiltizol-2-i)-2,5-difenil tetrazolio) ÿtil en ensayos de proliferación y citotoxicidad de células eucariotas (15,16). Su incorporación en cultivos líquidos de M. tuberculosis, realizados en microplacas, permite determinar rápidamente la susceptibilidad a los diferentes agentes antibacterianos (17-23).
El conocimiento de las bases genéticas de la resistencia de M. tuberculosis a los fármacos, en especial a rifampicina, ha sido la base del desarrollo de técnicas moleculares que amplifican por PCR la región determinante de resistencia a rifampicina localizada en el gen rpoB que codifica el blanco del fármaco. En ella se localiza cerca del 90% de las mutaciones que confieren resistencia a este medicamento (24,25).
Las técnicas moleculares como PCR-SSCP (single strand conformational polymorphism), determinación de secuencias y técnicas de hibridación, como INNO-LipA, PCR-PHL o rifoligotyping y PCR-ELISA ayudan a determinar en forma rápida y eficaz la susceptibilidad a rifampicina e isoniacida (26-32). La técnica basada en la hibridación reversa de productos amplificados rifoligotyping en su tercera versión incluye una mutación adicional a las incluidas en las versiones 1 y 2; Èsta se ha evaluado y ha resultado ÿtil en la determinación rápida de resistencia a la rifampicina, parámetro importante dado que este medicamento constituye la columna vertebral del esquema terapéutico antituberculoso.
El presente trabajo se realizó con el objetivo de evaluar dos técnicas fenotípicas basadas en la reducción de alamar azulTM y MTT (Maba y Tema) para la determinación de susceptibilidad de M. tuberculosis frente a agentes antibacterianos de primera línea, dada su facilidad en el montaje y lectura de los resultados, rapidez, bajo costo y a no requerir de equipos ni personal especializado y la técnica de PCR e hibridación reversa o rifoligotyping (PCR-PHL), versión 3, para la detección de mutaciones asociadas con la resistencia a rifampicina la cual es una metodología genotípica fácil de aplicar, rápida y poco costosa comparada con pruebas comerciales como la InnoLipA y menos laboriosa que la PCR- ELISA y la PCR-SSCP, usando como método de referencia la técnica de las proporciones múltiples.
Materiales y métodos
Cultivo de las cepas
Se incluyeron 20 cepas de M. tuberculosis y la cepa de referencia H37Rv enviadas por la profesora FranÁoise Portaels del International Reference Laboratory, Antwerp, Belgium; de acuerdo con los criterios establecidos por la Red Supranacional de Laboratorios, se tuvo en cuenta la inclusión de una proporción similar de cepas sensibles y resistentes a los cuatro medicamentos estudiados (comunicación personal, A. Laszlo).
Las cepas se sembraron nuevamente en medio Lwenstein-Jensen por 4 semanas; luego, se subcultivaron en caldo Middlebrook 7H9 enriquecido con ácido oleico, albÿmina, dextrosa y catalasa (OADC) (BBLô) durante 12 días, de los cuales 9 días fueron en agitación a 120 rpm a 37°C, con el objeto de obtener un inóculo homogéneo en fase logarítmica y en cantidad suficiente para realizar el montaje de todas las pruebas de susceptibilidad.
Pruebas de susceptibilidad Maba y Tema
Se utilizó una placa de poliestireno de 96 pozos (FalconÆ) para el montaje de las dos pruebas, para una misma cepa. Se añadieron 200 µl de agua destilada estéril a los pozos externos con el fin de evitar la evaporación del medio durante la incubación.
Se prepararon soluciones madre de 1 mg/ml en agua destilada estéril para isoniacida, etambutol y estreptomicina, y de 10 mg/ml en dimetilsulfóxido (DMSO) para la rifampicina. Posteriormente, se prepararon soluciones de trabajo de 32 µg/ml, 4 µg/ml, 128 µg/ml en medio Middlebrook 7H9 para la estreptomicina, la isoniacida y el etambutol, respectivamente, y de 8 µg/ml para la rifampicina.
Se dispuso una columna con 6 pozos para el ensayo con cada antibiótico en los que se adicionaron 100 µl de Middlebrook 7H9 y 100 µl de las soluciones de trabajo de cada fármaco. Directamente dentro de la placa se realizaron diluciones seriadas en base dos de cada medicamento para lo cual se tomaron 100 µl y se transfirieron al pozo siguiente de forma tal que en cada pozo quedó un volumen final de 100 µl con concentraciones finales de los antibióticos entre 0,25 y 8 µg/ml para la estreptomicina; 0,031 y 1 µg/ml para la isoniacida; 0,061 y 2 µg/ml para la rifampicina y 1 y 32 µg/ml para el etambutol. Posteriormente, se adicionaron 100 µl de inóculo consistente en una dilución 1:20 obtenida a partir de una suspensión bacteriana igual a la del tubo nÿmero 1 de la escala de McFarland.
Además, para cada prueba se dispuso un control de medio con 200 µl de Middlebrook 7H9 para comprobar su esterilidad; también, un control del inóculo diluido 1:100 para garantizar el crecimiento sólo del 1% de la población bacilar, así como cuatro controles de crecimiento en los que se adicionó el inóculo bacteriano sin antibiótico.
Se incubaron las placas a 37°C en ambiente hÿmedo. A los 5 días se evidenció el crecimiento por la incorporación de 32 µl de una solución de alamar azulTM (Trek Sistemas Diagnóstico Ltd., U.K) y Tween 80 al 20% para Maba, y 22 ml de una solución de MTT (SigmaÆ) de 5 mg/ml y Tween 80 al 20% para Tema. Luego, se incubaron nuevamente las placas por 24 horas y se observó el cambio de color de los colorantes. La reducción del alamar azulTM fue evidente por el cambio de azul a rosa y la de MTT por el cambio de amarillo a pÿrpura, indicativa Èsta de crecimiento bacteriano; en este caso, se añadió la solución de los colorantes a los demás pozos de la placa (controles de crecimiento y pozos con antibióticos) para la determinación posterior de la concentración inhibitoria mínima (CIM) (33).
Prueba de PCR e hibridación reversa o rifoligotyping, tercera versión
Las cepas se sembraron nuevamente en medio Lwenstein-Jensen durante 15 días y, luego, se suspendió la masa bacilar en solución tampón TE 1X (Tris-HCl, EDTA) para la recuperación del AND cromosómico. Para ello, se siguió la metodología descrita por Kremer et al. (28), usando lisozima, CTAB, extracción con cloroformo y alcohol isoamílico y, finalmente, precipitación con isopropanol. El ADN se reconstituyó en solución amortiguadora TE 0,1X.
Posteriormente, se amplificó por PCR una región de 156 pb del gen rpob, que incluyó la región determinante de resistencia a la rifampicina, con los iniciadores: TR10a (5¥-CGCCGCGATCAAGGA GT-3¥) y TR11a (5¥-ACGTCGCGGACCTCCA-3¥), este ÿltimo marcado con biotina en el extremo 5¥ (28).
Se realizó la hibridación del amplificado usando una solución de 10 µl del producto de la PCR y 150 µl de solución amortiguadora SSPE 2X y SDS al 0,1%. sta se llevo a cabo con secuencias mutadas y secuencias silvestres complementarias a la región determinante de resistencia a la rifampicina, adheridas a una membrana de nylon (Biodyne-C). Esta membrana incluye 5 oligonucleótidos sin mutación denominados Rif 1 a Rif 5 y seis oligos con las mutaciones más frecuentemente asociadas con resistencia a rifampicina, denominadas: Rif 2a, Rif 2b, Rif 4a, Rif 4b, Rif 5a, Rif 5b y Rif 6 (28) (cuadro 1).

La detección de la hibridación se evidenció por la incorporación inicial de conjugado de estreptavidina marcado con peroxidasa de RocheTM; luego, los reactivos de detección ECLô (peróxido de hidrógeno y luminol) (Amersham Pharmacia Biotech) y, finalmente, la señal de quimioluniniscencia se detectó por impresión de la misma en una película fotosensible de rayos X (FujiMedical).
Análisis de resultados
El método de las proporciones mÿltiples en su variante simplificada de Canetti, Rist y Grosett (4) se usó como prueba de oro para la evaluación y la comparación de las metodologías fenotípicas Maba y Tema. Una vez obtenidos los valores de CIM para cada medicamento, se determinó el rendimiento de las pruebas en términos de la sensibilidad, especificidad y área bajo la curva obtenidos con análisis con curvas ROC usando el programa MedCalc® versión 7.2.0.0 para Windows, así como el punto de corte y su valor p.
Se tuvieron en cuenta los valores del área bajo la curva (ABC) para la determinación de la eficiencia de las técnicas y fueron catalogadas como excelentes cuando dicho valor se encontró entre 1 y 0,9; buenas, entre 0,9 y 0,8; regulares, entre 0,8 y 0,7; malas, entre 0,7 y 0,6 e imperfectas, entre 0,6 y 0,5 (10,34).
Mediante tablas de contingencia se determinó el valor de la sensibilidad, la especificidad, los valores pronósticos de resistencia, sensibilidad y eficiencia, para el análisis de la metodología rifoligotyping.
Resultados
El perfil de susceptibilidad de las 21 cepas usadas en este estudio, se muestra en el
cuadro 2.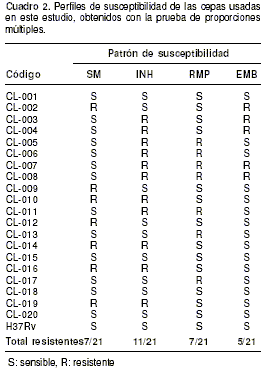
Pruebas de oxidorreducci
ón Maba y TemaEl tiempo total que se tardó en la prueba de referencia para obtenerse el resultado fue de 42 días, aproximadamente, mientras que las pruebas colorimétricas tomaron sólo 19 días, luego de haber tenido el cultivo de la cepa. En cuanto al montaje de la prueba como tal, las placas de Maba o Tema requirieron 10 minutos luego de haber preparado las soluciones de antibióticos, inóculo bacteriano y el caldo de cultivo. Del mismo modo, se necesitaron 24 minutos para el montaje de la prueba de proporciones mÿltiples luego de haberse preparado el inóculo y el medio con medicamentos y sin ellos.
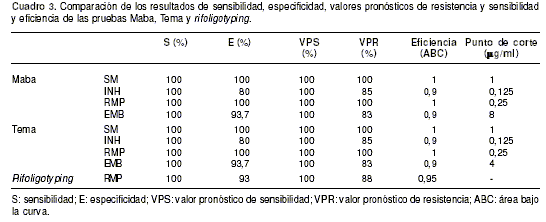
La CIM para cada antibiótico se obtuvo a los 7 días de incubación de las placas y mediante análisis por ROC se determinaron los valores de sensibilidad de 100% para las dos técnicas frente a los cuatro antibióticos. Así mismo, la especificidad fue de 100% para la estreptomicina y la rifampicina, y para la isoniacida y el etambutol de 80 y 93,7%, respectivamente.
El valor pronóstico de la sensibilidad mostrado por Maba y Tema fue de 100% para cada antibiótico, al igual que el valor pronóstico de resistencia frente a estreptomicina y rifampicina. La eficiencia de las pruebas, dada por los valores de ABC, fue superior al 0,9 por lo que se clasificaron como pruebas excelentes en la detección de resistencia comparadas con el método de referencia.
Además, mediante este mismo análisis se determinaron los puntos de corte o concentraciones críticas para cada fármaco (cuadro 3), que fueron iguales por las dos técnicas, para la estreptomicina, la isoniacida y la rifampicina. Para el etambutol se observó diferencia en una dilución, ya que por Maba el punto de corte fue de 8 µg/ml y por Tema fue de 4 µg/ml, diferencia que no se consideró significativa, segÿn el criterio del NCCLS (National Committee for Clinical Laboratory Standards) (33).
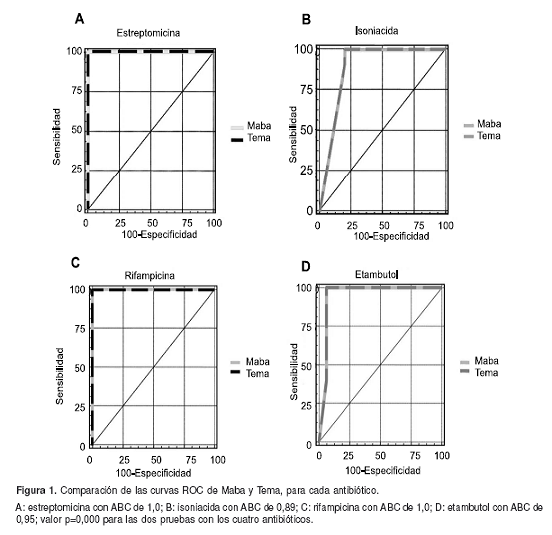
Además, se compararon las dos técnicas entre sí mediante el análisis comparativo de las curvas ROC con el objeto de establecer diferencias entre ellas, pero no se observó tal discrepancia para ninguno de los medicamentos incluidos. La figura 1 muestra el resultado de esta comparación.
Técnica de PCR e hibridación en reversa o rifoligotyping, versión tres
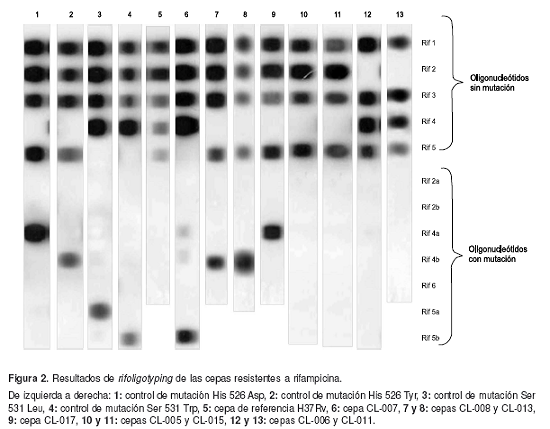
Luego del cultivo de las cepas, la técnica solo tomó tres días para la obtención del resultado. La técnica solamente identificó 12 de las 13 cepas sensibles, las cuales mostraron ausencia de hibridación con los oligonucleótidos mutados; se obtuvo un resultado discrepante para una cepa que, siendo sensible, mostró resistencia a la rifampicina por esta técnica. A las 7 cepas resistentes a la rifampicina se les detectó alguna mutación en el gen rpob por esta metodología.
Por lo anterior, el rifoligotyping detectó 8 cepas resistentes, de las cuales, cuatro reaccionaron con alguna de las secuencias mutadas, mientras que en las restantes no se observó hibridación con estas secuencias, aunque por la falta de esta señal con todas las secuencias salvajes, se pudieron catalogar como resistentes (figura 2). Las mutaciones detectadas en las cepas resistentes fueron Ser 531 Trp, His 526 Tyr e His 526 Asp.
Se analizaron los resultados obtenidos y la sensibilidad de esta metodología fue de 100%; la especificidad de 93%, y los valores pronósticos de sensibilidad y resistencia de 100 y 88%, respectivamente, con una eficiencia del 95,2% (
cuadro 3).Discusión
La determinación de la resistencia a fármacos de primera línea, en especial a la rifampicina y la isoniacida, ha sido la base del diseño de técnicas de susceptibilidad, dada la importancia que estos dos medicamentos tienen dentro del esquema de tratamiento, y sabiendo que las cepas resistentes a la rifampicina lo son generalmente a la isoniacida, lo que se conoce como multirresistencia; se destaca la detección de la resistencia a la rifampicina para predecir la multirresistencia.
El tiempo requerido para el montaje de las pruebas colorimétricas fue menor al empleado en la prueba de referencia, lo que se tradujo en una economía de más de la mitad de tiempo. Este hecho resulta fundamental al momento de detectar susceptibilidad a un gran nÿmero de cepas.
Los resultados de las pruebas Maba y Tema se obtuvieron a los 19 días; tiempo que comparado con los 42 días que toma el método de referencia, representó un ahorro significativo. Lo anterior se destaca ya que los pacientes, en los que fuere necesario ajustar e implementar un nuevo esquema, pueden verse beneficiados a partir del segundo mes de tratamiento.
Este tiempo de obtención del patrón de susceptibilidad fue menor al compararlo con los estudios en los que se ha obtenido a los 8, 10 o 14 días (8-10,13-23). Esto se logró gracias a que en nuestro estudio utilizamos un inoculo más concentrado que favoreció el crecimiento de las cepas en un menor tiempo.
La sensibilidad y especificidad mostradas por Maba y Tema fueron similares para los cuatro medicamentos. Se obtuvieron resultados excelentes para la rifampicina por las dos técnicas, razón por lo cual resultan igualmente bondadosas en la detección de multirresistencia de M. tuberculosis. La eficiencia, dada por el ABC, permitió catalogarlas como pruebas excelentes, ÿtiles en la detección eficaz de farmacorresistencia a medicamentos de primera línea. Al comparar este hallazgo con el reportado en estudios previos, encontramos que la eficiencia de las pruebas para la rifampicina y la estreptomicina fue superior, mientras que fueron similares para la isoniacida y el etambutol (10).
Los puntos de corte establecidos para cada antibiótico fueron similares a los reportados en estudios basados en la reducción de alamar azul como indicador de crecimiento (10). La diferencia en la concentración crítica establecida para el etambutol por las dos metodologías puede deberse a la dificultad que se tiene al estandarizar las pruebas de susceptibilidad a este fármaco, dada la naturaleza bacteriostática del medicamento (35).
La buena correlación de las pruebas Maba y Tema con el método de referencia es comparable con otros estudios en los que se han evaluado metodologías también realizadas en medio líquido como MGIT automatizado y MGIT manual, teniendo como referencia el método radiométrico Bactec 460 (36), en los que se reportan sensibilidades y especificidades similares para la isoniacida y la rifampicina, e incluso menores para la estreptomicina y el etambutol que las reportadas en nuestro estudio.
Destacamos algunas bondades de las pruebas fenotípicas como la rapidez en la emisión de los resultados y en el montaje de las mismas, traducidas en el significativo ahorro de tiempo comparado con el método de las proporciones mÿltiples; estos parámetros son importantes en la detección rápida y temprana de resistencia en aislamientos clínicos, lo cual tiene un impacto positivo en la comunidad derivado del tratamiento adecuado y oportuno de los pacientes. Además, porque se pueden incorporar diferentes antibióticos a los de primera línea empleados para el tratamiento de la tuberculosis, lo cual es ÿtil en el diagnóstico de susceptibilidad de otras micobacterias diferentes a M. tuberculosis, con importancia en pacientes coinfectados con VIH como Mycobacterium avium intracellulare, Mycobacterium fortuitum, Mycobacterium chelonae y otros.
De igual forma, la determinación de la concentración inhibitoria mínima resulta ÿtil para micobacterias con niveles intermedios de sensibilidad o "sensibilidad moderada" y en casos en los que se requiera ajustar las dosis terapéuticas de los medicamentos teniendo en cuenta las concentraciones séricas y la farmacocinética de cada uno, aspectos cruciales en el momento de tratar a los pacientes.
Así mismo, la no utilización de material radioactivo y el ahorro sustancial de reactivos dado el formato de microplacas utilizado constituyen ventajas frente a los métodos convencionales.
A pesar de que el rendimiento observado por las dos técnicas fue igual, tuvimos cierta dificultad al interpretar los resultados de Maba debido a que la tonalidad del colorante no fue exclusivamente azul o rosa, sino que se presentaron tonos violetas lo que constituye una desventaja de la técnica. Así mismo, la baja bioseguridad que se tiene al usar placas con medio líquido por la posible generación de aerosoles que aumenta el riesgo biológico (13,20,37), obligan a pensar a futuro que la implementación de estas técnicas se debe centralizar en los laboratorios que cuenten con la infraestructura y el volumen de muestras necesarios para emplear estas pruebas, por lo que no se justifica la implementación de ellas en muchos de los laboratorios con escaso volumen de muestras.
La determinación de la resistencia a la rifampicina como factor de predicción de multirresistencia fue el objeto de la metodología PCR HPL o rifoligotyping, tercera versión, en la que se detectó exactamente la mutación en las cepas resistentes a este fármaco (28,31,32).
A las cepas resistentes a la rifampicina incluidas en este estudio se les determinó por esta técnica alguna mutación asociada con resistencia a este medicamento. Dichas mutaciones, asociadas con altos niveles de concentración mínima inhibitoria, son las comÿnmente reportadas no sólo en Colombia sino a nivel mundial (24-27,29-32).
El hecho de que a cuatro cepas resistentes a la rifampicina no se les identificó la mutación exacta responsable de la resistencia, por ausencia de hibridación con las secuencias mutadas, no constituye un hallazgo exótico en esta prueba ya que estudios previos han reportado un nÿmero significativo de cepas resistentes que no hibridizan con las secuencias mutadas incluidas en la membrana de rifoligotyping (32). Lo anterior debido a la presencia de mutaciones diferentes a las incluidas en esta membrana o cambios que, a pesar de localizarse en el mismo codón, constituyen otro diferente a los incluidos, pero igualmente responsables de la resistencia a este medicamento (25,26,29,31,32). Cabe destacar la importancia que tendría en un estudio posterior, la inclusión de mutaciones propias de cepas colombianas como las reportadas previamente (30), ya que se podrían captar aquéllas que poseen otras mutaciones además de las incluidas en este estudio.
Llamó la atención el caso de una cepa (CL-015) sensible a rifampicina que por rifoligotyping fue resistente y mediante la metodología de INNOLipA obtuvo un resultado igual (no se muestran los datos). Al analizar el resultado de la prueba de oro se observó que se le calculó un porcentaje de 0,18% de bacilos mutantes resistentes y, gracias a la alta sensibilidad de la PCR, se pudo amplificar e identificar esta pequeña poblaciÓn. Se han reportado hallazgos similares en Colombia por Cohen et al. (30), quienes al usar la técnica de PCR-SSCP detectaron porcentajes entre 0,5 y 0,99% de bacilos resistentes en cepas catalogadas como sensibles por el método de referencia. Por esta razón, planteamos la posibilidad de considerar una cepa sensible a la rifampicina cuando no registre crecimiento alguno por el método de las proporciones mÿltiples o con una proporción crítica de 0% y no del 1%, como se usa actualmente.
Por lo anterior, destacamos el método molecular de rifoligotyping, versión tres, por su eficacia en la detección de multirresistencia, resaltando bondades como la rapidez, la sensibilidad, la especificidad, los altos valores pronósticos, el bajo riesgo biológico que significa el trabajo con material genético y no con bacterias viables, además de la posible inclusión de mutaciones propias de cepas colombianas. Así mismo, la futura aplicación directa en muestras clínicas reduciría significativamente el tiempo de diagnóstico de multirresistencia. Además, este tipo de pruebas genéticas son útiles en ensayos epidemiológicos que pueden darnos una idea de la transmisibilidad y circulación de clones de M. tuberculosis en una región determinada.
Igualmente, con este trabajo destacamos las metodologías fenotípicas Maba y Tema por su rapidez, facilidad en el montaje e interpretación de los resultados, bondades que las convierten en herramientas ÿtiles en la determinación rápida de farmacorresistencia en M. tuberculosis.
Teniendo en cuenta los resultados de este estudio, se sugiere para un estudio posterior la inclusión de aislamientos clínicos de pacientes colombianos, diagnosticados con tuberculosis, con el objeto de determinar la utilidad de las pruebas en la detección de resistencia en grupos con alto riesgo de desarrollar tuberculosis multirresistente.
Agradecimientos
A Carlos Arturo Hernández del Instituto Nacional de Salud por su acertada orientación en la escritura y presentación de este artículo. A Dick van Soolingen del Mycobacteria Reference Department, National Institute of Public Health and Environment, Bilthoven, The Netherlans, por la donación del estuche de rifoligotyping, versión 3.
Conflicto de intereses
Los autores manifiestan que no existe ningún conflicto de interés.
Financiación
El presente estudio se desarrolló gracias a la financiación del Instituto Nacional de Salud de Colombia, proyecto INS 39-2001, y al apoyo de Colciencias dentro del Programa de formación y capacitación de recursos humanos para la ciencia y la tecnología, convenio 24-2002. Además, gracias al soporte de la Comisión Europea, CE, Programa INCO Upgrade Diagno MDR-TB ICA4- CT 2001- 10087.
Correspondencia:
Clara Inés León, Avenida Calle 26 Nº 51-60, Bogotá, D.C., Colombia.
Teléfono: (571) 220 7700, extensión 436; fax: (571) 220 7700, extensión 255.
Recibido: 01/07/04; aceptado: 09/12/04
Referencias
1. Espinal MA. The global situation of MDR-TB. Tuberculosis 2003;83:44-51. [ Links ]
2. World Health Organization. Anti-tuberculosis drug resistance in the world. Report 2, prevalence and trends. Geneva: World Health Organization; 2000. [ Links ]
3. World Health Organization. Anti-tuberculosis drug resistance in the world. Report 3. Geneva: World Health Organization; 1999-2002. [ Links ]
4. Canetti G, Fox W, Khomenko A, Mathler HT, Menon NK, Mitchison DA et al. Advances in techniques of testing mycobacterial drug sensitivity, and the use of sensitivity tests in tuberculosis control programs. Bull Wld Hlth Org 1969;41:21-43. [ Links ]
5. Tortoli E, Benedetti M, Fontanelli A, Simonetti M. Evaluation of automated BACTEC MGIT 960 system for testing susceptibility of Mycobacterium tuberculosis to four major antituberculous drugs: comparison with the radiometric BACTEC 460TB method and the agar plate method of proportion. J Clin Microbiol 2002;40:607-10. [ Links ]
6. Scarparo C, Ricordi P, Ruggiero G, Piccoli P. Evaluation of the fully automated BACTEC MGIT 960 system for testing susceptibility of Mycobacterium tuberculosis to pyrazinamide, streptomycin, isoniazid, rifampin and ethambutol and comparison with the radiometric BACTEC 460TB method. J Clin Microbiol 2004;42:1109-14. [ Links ]
7. Hausdorfer J, Sompek E, Allerberg F, Dierich M, R¸sch-Gerdes S. E-test for susceptibility testing of Mycobacterium tuberculosis. Int J Tuberc Lung Dis 1998; 2:751-5. [ Links ]
8. Yajko DM, Madej JJ, Lancaster MV, Sanders CA, Cawthon VL, Gee B et al. Colorimetric method for determining MICs of antimicrobial agents for Mycobacterium tuberculosis. J Clin Microbiol 1995;33: 2324-7. [ Links ]
9. Palomino JC, Portaels F. Simple procedure for drug susceptibility testing of Mycobacterium tuberculosis using a commercial colorimetric assay. Eur J Clin Microbiol Infect Dis 1999;18:380-3. [ Links ]
10. Luna-Herrera J, MartÌnez-Cabrera G, Parra-Maldonado R, Enciso-Moreno JA, Torres-Lûpez J, Quesada-Pascual F et al. Use of received operating characteristics curves to assess the performance of a microdilution assay for determination of drug susceptibility of clinical isolates of Mycobacterium tuberculosis. Eur J Clin Microbiol Infect Dis 2003;22:21-7. [ Links ]
11. Palomino JC, Martin A, Camacho M, Guerra H, Swings J, Portaels F. Resazurin microtiter assay plate: simple and inexpensive method for detection of drug resistance in Mycobacterium tuberculosis. Antimicrob Agents Chemother 2002;46:2720-2. [ Links ]
12. Martin A, Camacho M, Portaels F, Palomino JC. Resazurin micotiter assay plate testing of Mycobacterium tuberculosis susceptibilities to secondline drugs: rapid, simple, and inexpensive method. Antimicrob Agents Chemother 2003;47:3616-9. [ Links ]
13. Collins LA, Franzblau SG. Microplate Alamar blue assay versus BACTEC 460 system for high-throughput screening of compounds against Mycobacterium tuberculosis and Mycobacterium avium. Antimicrob Agents Chemother 1997; 41: 1004-9. [ Links ]
14. Franzblau SG, Witzig RS, McLaughlin JC, Torres P, Madico G, Hern·ndez A et al. Rapid, low-technology MIC determination with clinical Mycobacterium tuberculosis isolates by using the microplate Alamar blue assay. J Clin Microbiol 1998;36:362-6. [ Links ]
15. Mosmann T. Rapid colorimetric assay for cellular growth and survival: application to proliferation and cytotoxicity assay. J Immunol Methods 1983;65:55-63. [ Links ]
16. Berridge MB, Tan AS, McCoy KD, Wang R. The biochemical and cellular basis of cell proliferation assay that the tetrazolium salts. Biochemica 1996;4:14-9. [ Links ]
17. Abate G, Mshana RN, Mirner H. Evaluation of a colorimetric assay based on 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl tetrazolium bromide (MTT) for rapid detection of rifampicin resistance in Mycobacterium tuberculosis. Int J Tuberc Dis 1998;2:1011-6. [ Links ]
18. Mshana RN, Tadesse G, Abate G, Mirner H. Use of 3-(4,5-dimethylthiazol-2-yl)-2,5-diphenyl tetrazolium bromide for rapid detection of Rifampin-resistant Mycobacterium tuberculosis. J Clinical Microbiol 1998; 36:1214-9. [ Links ]
19. Foongladda S, Roengsanthia D, Arjrattanakool W, Chuchottaworn C, Chaiprasert A, Franzblau SG. Rapid and simple MTT method for rifampicina and isoniazid susceptibility testing of Mycobacterium tuberculosis. Int J Tuberc Lung Dis 2002;6:1118-22. [ Links ]
20. Caviedes L, Delgado J, Gilman RH. Tetrazolium microplate assay as a rapid and inexpensive colorimetric method for determination of antibiotic susceptibility of Mycobacterium tuberculosis. J Clin Microbiol 2002;40: 1873-4. [ Links ]
21. Abate G, Aseffa A, Selassie A, Goshu S, Fekade B, Wolde-Meskal D et al. Direct colorimetric assay for rapid detection of rifampicin resistant Mycobacterium tuberculosis. J Clin Microbiol 2004;42:871-3. [ Links ]
22. Morcillo N, Di Giulio B, Testani B, Pontino M, Chirico C, Dolmann A. A microplate indicador based method for determining the susceptibility of multidrug resistant Mycobacterium tuberculosis to antimicrobial agents. Int J Tuberc Lung Dis 2004;8:253-9. [ Links ]
23. De Logu A, Uda P, Pellerano ML, Pusceddu MC, Saddi B, Schivo ML. Comparison of two rapid colorimetric methods for detection resistance of Mycobacterium tuberculosis to rifampin, isoniazid, and streptomycin in liquid medium. Eur J Clin Microbiol Infect Dis 2001;20:33-9. [ Links ]
24. Ramaswamy S, Musser JM. Molecular genetic basis of antimicrobial agent resistance in Mycobacterium tuberculosis: 1998 update. Tubercle Lung Dis 1998;79:3-29. [ Links ]
25. Musser JM. Antimicrobial agent resistance in mycobacteria: molecular genetic insights. Clin Microb Rev 1995;8:496-514. [ Links ]
26. Bobadilla del Valle M, Ponce de LeÛn A, Arenas- Huertero C. rpoB gene mutations in rifampin resistant Mycobacterium tuberculosis identified by polymerase chain reaction single stranded conformational polymorphism. Emerging Infect Dis 2001;7:1010-3. [ Links ]
27. Rossau R, Traore H, De Beenhouwer H, Mijis W, Jannes G, De Rijk P et al. Evaluation of the INNOLiPA Rif. TB assay, a reverse hybridization assay for the simultaneous detection of Mycobacterium tuberculosis complex and its resistance to rifampin. Antimicrobial Agents Chemother 1997;41:2093-8. [ Links ]
28. Kremer K, van Zetten M, van Embden J, Chouls L, van Soolingen D, Mycobacteria department, National Institute of Public Health and the Environment. PCR+reverse line blot hybridization (PHL) to detect rifampin resistance, laboratory manual. Bilthoven, The Netherlands: National Institute of Public Health and the Environment; 1997. [ Links ]
29. GarcÌa L, Alonso-Sanz M, Rebollo M, Tercero J. Mutations in the rpoB gene of rifampin resistant Mycobacterium tuberculosis isolates in Spain and their rapid detection by PCR enzyme linked immunosorbent assay. J Clin Microbiol 2001;39:1813-8. [ Links ]
30. Cohen IB. Caracterización de eventos involucrados en la resistencia a rifampicina de cepas colombianas de Mycobacterium tuberculosis (tesis). Bogotá, Colombia: Universidad Nacional de Colombia; 2002. [ Links ]
31. Guerrero MI, Suffys PN, Vanderborcht B, De Oliveira MM, Cohen IB, LeÛn CI. Comparación de métodos moleculares útiles en la detección rápida de Mycobacterium tuberculosis multirresistente (MTBMRD). Infectio 2001;5:203-12. [ Links ]
32. Morcillo N, Zumarraga M, Alito A, Dolmann A, Schouls L, Cataldi A et al. A low cost, home-made, reverse-line blot hybridization assay for rapid detection of rifampicine resistance in Mycobacterium tuberculosis. Int J Tuberc Lug Dis 2002;6:959-65. [ Links ]
33. Zweig MH, Campbell G. Receiver-operative characteristic (ROC) plots: a fundamental evaluation tool in clinical medicine. Clin Chem 1993;39:561-77. [ Links ]
34. National Committee for Clinical Laboratory Standards. Susceptibility testing for mycobacteria, nocardia and other aerobic actinomycetes. Tentative standard M24-T2. Wayne, PA: National Committee for Clinical Laboratory Standards; 2000. [ Links ]
35. Madison B, Robinson-Dunn B, George I, Gross W, Lipman H, Metchock B et al. Multicenter evaluation of ethambutol susceptibility testing of Mycobacterium tuberculosis by agar proportion and radiometric methods. J Clin Microbiol 2002;40:3973-9. [ Links ]
36. Ådjers-Kosketa K, Katila M. Susceptibility testing with the manual mycobacteria growth indicator tube (MGIT) and the MGIT 960 system provides rapid and reliable verification of multidrug resistant tuberculosis. J Clin Microbiol 2003;41:1235-9. [ Links ]
37. Lemus D, Martin A, Montoro E, Portaels F, Palomino JC. Rapid alternative method for detection of rifampicin resistance in Mycobacterium tuberculosis. J Antimicrob Chemother 2004;54:130-3. [ Links ]














