Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO  Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.25 no.1 Bogotá Mar. 2005
Producción y purificación de la proteína Core del virus de la hepatitis C en el sistema de expresión del baculovirus para ensayos biológicos
Ivonne Rubio 1, Alba Lucía Cómbita 2, Blanca Ortiz-Reyes 3, María-Cristina Navas 1
1 Grupo de Gastrohepatología, Universidad de Antioquia, Medellín, Colombia.
2
Grupo de Investigación en Biología del Cáncer, Instituto Nacional de Cancerología, Bogotá, D.C., Colombia.3
Grupo de Inmunología Celular e Inmunogenética, Universidad de Antioquia, Medellín, Colombia.
Introducción.
La infección por el virus de la hepatitis C (VHC) se caracteriza por la alta frecuencia de infección persistente. La capacidad del VHC para inducir alteraciones en la función de las células del sistema inmune, específicamente en las células dendríticas, parece ser una de las estrategias virales implicada en el establecimiento de la infección persistente. Esta estrategia parece estar mediada en gran parte por una de las proteínas estructurales codificada por el genoma del VHC, la proteína Core, unidad estructural de la cápside viral.Objetivo. Una de las limitantes para la evaluación de las propiedades de Core es la obtención y la purificación de la proteína nativa (191 aa). El objetivo de este estudio fue la producción y la purificación de la proteína Core completa en un sistema eucariote para evaluar las propiedades de la proteína en cultivos de células dendríticas humanas.
Resultados. En este estudio se logró la producción de la proteína Core completa en el sistema de expresión de baculovirus y su purificación por la técnica de separación por punto isoeléctrico y electroelución. La pureza de la proteína obtenida se confirmó por Western blot y tinción con plata. Estos análisis mostraron dos bandas únicas correspondientes a las isoformas p23 y p21 de la proteína Core, previamente descritas en la literatura.
Conclusiones. La proteína obtenida posee varias de las condiciones de la proteína Core nativa, como peso molecular, isoformas y localización subcelular. Los procedimientos descritos en este artículo son aplicables a proteínas asociadas a membranas producidas en sistemas de expresión eucarióticos.
Palabras clave: virus de la hepatitis C, proteína Core, Baculovirus, sistema de expresión, isoelectroenfoque, purificación.
Hepatitis C virus core protein production and purification in a baculovirus expression system for biological assays
Background. The hepatitis C virus (HCV) commonly causes persistent infection. One of the viral mechanisms in preventing viral clearance is the ability of HCV to induce functional alterations of the immune system cells, specifically of dendritic cells. Viral proteins producing the dendritic cell functional alterations have been identified as the HCV core protein, which makes up the capsid structural unit.
Objective. One of the limitations to evaluate core protein properties is the difficulty in obtaining and purifying the complete protein -consisting of 191 amino acids. The aim of the current study was to produce and purify the recombinant core protein in a eukaryotic system, and to evaluate its effect in human dendritic cell cultures.
Results. The core protein p23 isoform was expressed in a baculovirus system and purified using isoelectric point separation and electroelution. The purity of the core protein was confirmed by silver stain and Western blot. These analyses showed the presence of two bands that correspond to p23 and p21 isoforms of core protein as previously reported.
Conclusion. The expressed protein differed from naive core protein is terms of molecular weight, isoforms and subcellular localization. The procedures developed for core protein are applicable for expression of other membrane-associated proteins produced in eukaryotic systems.
Keywords: hepatitis C Virus, core protein, baculovirus, expression system, isoelectrofocusing, electroelution.
La infección por el virus de la hepatitis C (VHC) es considerada un problema de salud pública, teniendo en cuenta los 200 millones de casos estimados en el mundo y la alta frecuencia de hepatitis crónica asociada al VHC (1,2).
La infección persistente parece ser debida a diversas estrategias virales de evasión y modificación de la respuesta inmune, como la variación antigénica, la asociación de las partículas virales con b-lipoproteínas y la infección de células del sistema inmune como linfocitos B y células dendríticas (3-6).
Si se tiene en cuenta que la célula dendrítica es la célula presentadora de antígeno por excelencia, indispensable en la inducción y regulación de la respuesta inmune específica, su modificación inducida por el VHC podría favorecer el establecimiento de la persistencia viral. Diversos estudios han aportado argumentos a favor de esta hipótesis, en los cuales se ha demostrado in vivo e in vitro la infección por el VHC de células dendríticas y su modificación funcional (7-11). La principal proteína viral implicada en la inducción de dichas alteraciones funcionales es la proteína Core del VHC (12,13).
La proteína Core es la subunidad estructural de la cápside viral (14). El extremo C-terminal altamente hidrofóbico media la translocación de la poliproteína al retículo endoplasmático, donde es hidrolizada por una proteasa celular entre los residuos 191 y 192, dando origen a la isoforma p23 de la proteína (15). El procesamiento de p23, mediado por una proteasa celular dependiente de la membrana, genera la isoforma p21 con 173- 179 aminoácidos (aa), considerada la forma madura de Core (16). Luego del procesamiento enzimático, la p21 es liberada al citosol donde se autoensambla para formar las cápsides virales; también se puede encontrar formando complejos con la isoforma p23 de Core o asociada a acúmulos de lípidos (17,18).
El extremo N-terminal de Core, específicamente la región entre los aminoácidos 1 y 120, conforman un dominio altamente hidrofílico (19,20). El plegamiento y oligomerización de este dominio parece estar mediado por una región rica en triptófano ubicado entre los aminoácidos 82 y 102 (21,22). El extremo N-terminal contiene además varios epítopos B, en particular, un epítopo B conformacional inmunodominante ubicado entre los aminoácidos 20 y 45 (23-26).
La caracterización parcial de la estructura y de los dominios de la proteína Core ha permitido plantear su papel en la morfogénesis de la partícula viral, en la modulación de procesos como el ciclo celular, la apoptosis, la transformación celular, el metabolismo de lípidos y en la evasión de la respuesta inmune (12,27-30).
Debido a la ausencia de un sistema eficiente de replicación in vitro del VHC, ha sido necesaria la producción de proteína recombinante. Se han obtenido formas truncas de la proteína Core, con deleciones del extremo C-terminal, en sistemas de expresión como Escherichia coli (31); esta deleción incrementa la eficiencia de purificación (32). Aunque la proteína no posee todas las modificaciones postranscripcionales de la proteína nativa, se ha descrito la formación de estructuras similares a la nucleocápside (nucleocapsid-like particles) (31,33).
De otra parte, se ha intentado producir la proteína Core en diferentes sistemas eucarióticos como levaduras (34), células de insectos (35) y células de mamíferos (36,37), con relativo éxito. En el caso de la producción en células de insecto, los artículos publicados en la literatura corresponden a la expresión simultánea de las proteínas estructurales Core, E1 y E2 del VHC (35,38-40). La purificación de las isoformas de Core ha representado un obstáculo adicional en el estudio de sus propiedades, en particular, por ser una proteína unida a la membrana.
En este artículo se describe la construcción de un baculovirus recombinante, diseñado para expresar la proteína Core completa (191 aminoácidos) en células de insecto y la purificación de dicha proteína por punto isoeléctrico y electroelución. Estos sistemas combinados de purificación permitieron obtener la proteína recombinante Core del VHC, completa y funcional, similar a la proteína nativa.
Materiales y métodos
Células
Las líneas celulares Sf9, Sf21 (Spodoptera frugiperda) y H5 High five (Trichoplusia ni) (Invitrogen, Escocia) se cultivaron a 28°C en medio de Grace (MG) (Invitrogen), con suplemento del 10% de suero bovino fetal (SBF) (Gibco Life Technologies, Escocia) (MG-SBF). Para los experimentos de transfección, se utilizaron medios libres de proteínas, X-press (Invitrogen) y medio de Grace modificado (MGLP) (Invitrogen).
Anticuerpos
Se utilizó el anticuerpo monoclonal humano B12.F8, gentilmente donado por el Dr. Mario Mondelli (Istituto di Clinica delle Malattie Infettive, IRCCS Policlinico San Matteo, Universidad de Pavia, Italia). Este anticuerpo reconoce los residuos 27 a 59 del extremo amino-terminal de la proteína Core del VHC (24). También se utilizó el anticuerpo de oveja anti-IgG humana marcada con peroxidasa (Amersham-Pharmacia, Uppsala, Suecia) en los ensayos de Western blot.
Clonación de la secuencia Core en el sistema baculovirus
Para la construcción de baculovirus recombinantes, la secuencia Core del VHC se amplificó a partir del plásmido pSFV1-Core (41), con los iniciadores Bac-Core5´ (5´CAT CGA GGA TCC GTG GCA CCA TGA GCA CGA ATC CTA AAC C 3´) y Bac-Core3´ (3´CAA GCT AAG CTT CAC TTG GGC TGA AGC GGG CAC GGT C 5´). Las condiciones de amplificación fueron: 94°C, 5 minutos, 25 ciclos a 94°C 30 segundos, 50°C 30 segundos, 72°C, 90 segundos y 72°C, 7 minutos. La secuencia fue clonada en el plásmido pBluescript SK+ (Stratagene, La Jolla, Estados Unidos), previa digestión (Promega Corp., Madison, Estados Unidos) y purificación (Qiagen, Hilden, Alemania).
Los clones recombinantes obtenidos por transformación de células competentes E. coli DH5a con pBS-Core, fueron seleccionados según el análisis de restricción. Se obtuvo la secuencia del extremo 3´ del fragmento clonado a partir de pBS-Core, utilizando el estuche 7-Deaza-dGTP Cy5/Cy5.5 Dye primer cycle sequencing (Amersham, Buckinghamshire, Reino Unido) y el iniciador para la secuencia de M13 (5´GTA AAA C GAC GGC CAG T) (Promega). El producto de amplificación se analizó usando el secuenciador SEQ4*4 (Amersham).
El fragmento fue subclonado en el plásmido de transferencia pBlueBac4.5/V5-His (Invitrogen) para generar una proteína de fusión con 6 histidinas en el extremo carboxiterminal (pBB4.5-Core/His).
Para la generación de baculovirus recombinantes, se cotransfectaron 2x105 células de insecto Sf9 con 4 µg de plásmido de transferencia pBB4.5- Core/His y 0,5 µg de ADN de Bac-N-Blue por la técnica de liposoma catiónico (Cellfectin Invitrogen) en medio Xpress (Invitrogen). Luego de la cotransfección, las células Sf9 se cultivaron con MG con suplemento al 10% de SBF (Gibco) y6 se incubaron a 28°C por 72 horas.
El sobrenadante del cultivo se conservó a 4°C, protegido de la luz. Las partículas virales presentes en el sobrenadante se amplificaron por infección de células Sf9 en fase logarítmica de crecimiento, cultivadas en MG con suplemento al 10% de SBF. Como control negativo se incubaron células Sf9 en MGLP, en las condiciones anteriormente mencionadas.
Aislamiento del clon recombinante
Se llevó a cabo un ensayo de dilución límite a partir del sobrenadante de cotransfección, para el aislamiento de un clon recombinante baculovirus para la proteína Core. Se incubaron durante 10 días diluciones seriadas sobre monocapas de células Sf9 en placas de cultivo de 48 pozos, con observación microscópica diaria. A partir de los botones de las células infectadas correspondientes a cada dilución, se realizó la extracción de ADN con la técnica de fenol:cloroformo:alcohol isoamílico (25:24:1), para posterior detección del genoma viral por PCR.
Se amplificó la región que flanquea el gen de la polihedrina con los iniciadores sentido (5TTT ACT GTT TTC GTA ACA GTT TTG 3´) y antisentido (5CAA CAA CGC ACA GAA TCT AGC 3´) con las siguientes condiciones: 94°C, 2 minutos, 30 ciclos 94°C 1 minuto, 55°C 2 minutos y 72°C 3 minutos y 72°C por 7 minutos, según recomendaciones de Invitrogen.
Luego de la identificación del clon recombinante, el título viral se amplificó por infecciones sucesivas de cultivos de células H5. Las partículas recombinantes se conservaron a 4°C, protegidas de la luz.
Cinética de producción de la proteína Core en células H5 infectadas
Se infectaron monocapas de células H5 en fase logarítmica de crecimiento con las partículas recombinantes. Los botones celulares obtenidos a las 24, 48, 72 y 96 horas después de la infección se conservaron a -20°C, para posterior análisis por la técnica de Western blot.
Producción en masa de la proteína recombinante
Luego de identificar el punto de mayor expresión de la proteína recombinante Core, se realizó la infección de monocapas de células H5. Luego de 72 a 96 horas de incubación, las células fueron sometidas a centrifugación (500g durante 10 minutos). Los botones de células infectadas y sobrenadantes se conservaron a –20°C y 4°C, respectivamente.
Obtención de proteínas
Los botones de las células infectadas o las células control (2x106) se resuspendieron en 100 µL/106 células de tampón RIPA (Tris-HCl 50mM pH 7,5, NaCl 150 mM, Nonidet P40 1%, deoxicolato de sodio 0,5%, SDS 0,1%) e inhibidores de proteasas (10 µl/ml fenilmetilsulfonilfluoruro 100mM, 10 µg/ ml leupeptina y 10 mg/ml aprotinina (Sigma, San Luis, Estados Unidos), y se incubaron durante 15 minutos en hielo. Los lisados celulares se sometieron a centrifugación (14.000g, 15 minutos), y se obtuvieron dos fracciones, proteínas citosólicas (sobrenadante) y asociadas a membrana (botón); estas fracciones se utilizaron en los ensayos de Western blot.
Para los ensayos de purificación, la fracción de proteínas se obtuvo utilizando el tampón CHAPS 2 mM en solución tamponada de fosfato salino (PBS), (3-[(3-colamidopropil) dimetilamonio]-1- propanosulfonato) (Sigma), con suplemento de inhibidores de proteasas, como se describió anteriormente. Los botones de células se homogenizaron en hielo con 3 ciclos de 15 segundos (Biospec Products, Inc Bartlesville, Estados Unidos). Posteriormente, se sometieron a centrifugación (14.000g, 20 minutos), previa incubación en hielo durante 20 minutos. La fracción de proteínas asociadas a membrana (botón) fue sometida a las técnicas de isoelectroenfoque y electroelución.
Análisis de proteínas por electroforesis en gel de poliacrilamida (SDS-PAGE)
Una alícuota de 30 µg de proteínas citosólicas o proteínas de membrana obtenidas de células control o células infectadas se sometió a electroforesis en gel de poliacrilamida-dodecil sultafo de sodio (SDS-PAGE) al 12%, en condiciones reductoras y desnaturalizantes, durante 2 horas a 100 voltios.
Detección de la proteína recombinante por Western blot
Las proteínas se transfirieron a una membrana Hybond-polivinildifluoruro (PVDF) (Amersham), en cámara semiseca (Biorad) durante 30 minutos a 0,25 miliamperios. La membrana fue sometida a bloqueo en una solución de PBS, Tween 20 (0,05%) y leche descremada (5%) durante 16 horas y, posteriormente, incubada con el anticuerpo B12.F8 y anti-IgG humana de ratón conjugada con peroxidasa (Amersham, Reino Unido). La detección de las proteínas se realizó utilizando un estuche de quimioluminiscencia (ECLTM, Amersham).
Separación de proteínas por punto isoeléctrico
Se utilizó el sistema de rotofor (Biorad, Richmond, Estados Unidos) para separar las proteínas por punto isoeléctrico (42). La fracción correspondiente a las proteínas asociadas con membrana se preparó así: 2,5 ml de muestra, 1 ml de anfolitos con rango de pH de 3 a 10, 3 ml de glicerol estéril y 11,5 ml de agua miliq estéril. Las condiciones del rotofor fueron las siguientes: 12 watios durante 3 ½ horas a 4°C. Se separaron 20 fracciones por punto isoeléctrico, aisladas por succión al vacío. Posteriormente, se analizó el pH de cada fracción y la distribución de las proteínas por peso molecular mediante electroforesis, según se describió previamente (43). Mediante la técnica de Western blot se identificó la fracción en que estaba presente la proteína Core.
Purificación de la proteína Core por Electroelución
Las proteínas presentes en las fracciones obtenidas por isoelectroenfoque, correspondientes a las fracciones con pH entre 6,9 y 8,2, fueron sometidas a electroforesis en gel preparativo de SDS-PAGE al 15% (100 voltios, 18 horas, 4°C). Los geles se tiñeron con azul de Coomassie al 0,05%.
Teniendo en cuenta el peso molecular de las isoformas p23 y p21 de la proteína Core, se escogieron para electroelución 6 bandas entre los marcadores correspondiente a 20 kd y 25 kd, comenzando por la banda ubicada al mismo nivel del marcador 20 kd. Las bandas seleccionadas se cortaron y guardaron en tubos Eppendorf.
Las bandas de gel fueron sometidas a electroelución en las siguientes condiciones: 10 miliamperios por tubo, 5 horas a 4°C (sistema electroeluidor, BioRad). Las fracciones electroeluidas, ubicadas entre la membrana de diálisis de poro 10 kd y el filtro contenedor de gel, se dializaron con agua ultrapura libre de endotoxina, durante 12 horas en agitación constante a 4°C y se precipitaron con acetona (2 ½ volúmenes) durante 12 horas a –20°C. Las proteínas electroeluidas se analizaron por electroforesis y posterior coloración con nitrato de plata y ensayo de Western blot.
La cuantificación de la proteína Core purificada se hizo por densitometría de un ensayo de Western blot, comparando la densidad de la banda de 1 µg de la proteína Core trunca (120 aminoácidos, gentilmente donada por Laboratorios Biomérieux, Marcy LEtoile, Francia), con la intensidad de la banda de la fracción de proteína Core completa purificada.
Cuantificación de proteínas totales
Las proteínas se cuantificaron por el método del ácido bicinconínico (BCA) utilizando un estuche comercial (Pierce). El valor de absorbancia se determinó a 562 nm. Para el cálculo de la concentración de las muestras se sembraron 5 estándares de concentración conocida (2.000 a 150 mg/ml). El cálculo de la concentración de las muestras se hizo por regresión lineal.
Resultados
Amplificación y clonación de la secuencia Core del VHC en el vector de transferencia de baculovirus pBlueBac4.5/V5-His
La secuencia que codifica para la proteína Core se amplificó a partir del constructo pSFV1-Core, como se indicó en la metodología. El análisis de la electroforesis de agarosa al 2% permitió identificar una banda de 600 pb, aproximadamente. La clonación del producto de amplificación en el plásmido pBluescript (pBS-Core) y la subclonación en pBueBac4.5/V5-His (pBB4.5-Core/His) se confirmó por digestión con las enzimas HindIII y BamHI. El sentido de clonación de la secuencia Core en el vector pBueBac4.5/V5-His se determinó por digestión con las enzimas KpnI y NhoI (no se muestra el dato).
Análisis de secuencia del marco de lectura de proteína recombinante Core 6 his
El análisis de la secuencia a partir del constructo pBS-Core confirmó la conservación del marco de lectura de Core. Al comparar la secuencia clonada con las secuencias de Core correspondientes a aislamientos de VHC (HCJ238799, HPCK1R1, HPCK1R2 y HPCK1S2) registradas en el GenBank, se encontró un cambio de nucleótido en la posición 476 de la secuencia Core (posición 817 del genoma viral), lo que posiblemente se tradujo en un cambio de un aminoácido hidrofílico a uno hidrofóbico (ácido glutámico a glicina) (no se muestra el dato). Esta mutación no parece estar presente en otros aislamientos, teniendo en cuenta las secuencias publicadas en el GenBank/ EMBL a la fecha.
Detección de la producción de la proteína en células cotransfectadas
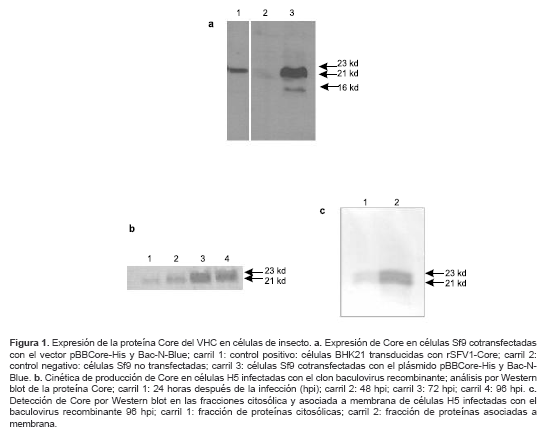
La producción de la proteína Core en células Sf9 cotransfectadas se confirmó por la técnica de Western blot. El ensayo reveló tres bandas de proteína reconocidas por el anticuerpo B12.F8, con pesos moleculares de 23, 21 y 16 kd, correspondientes a las isoformas de la proteína Core, previamente descritas (17) (figura 1a).
Aislamiento del clon recombinante
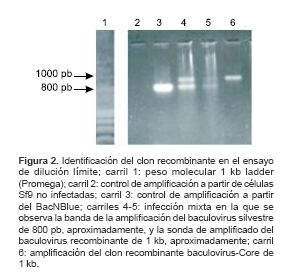
De las 32 diluciones evaluadas, sólo en la dilución 18 se logró amplificar una banda de 1 kb, aproximadamente, que corresponde al baculovirus recombinante. De otra parte, en la dilución 31 se logró amplificar una banda de 839 pb que corresponde al baculovirus silvestre. Mientras que, en el resto de las diluciones, se obtuvieron poblaciones mixtas de virus silvestre y virus recombinante, es decir, bandas de amplificaciones de 1.000 y 840 pb (figura 2).
Cinética de producción de Core
La expresión de la proteína Core se demostró 24 horas después de la infección en los cultivos de células H5, con máximo nivel de producción 96 horas después de la infección (figura 1b). Este resultado es similar al tiempo de producción de las proteínas recombinantes en células H5, previamente descrito por otros autores (44).
La proteína Core del VHC se encuentra principalmente asociada con proteínas de membrana en lisados de células H5.
El análisis por la técnica de Western blot de las fracciones de proteínas citosólicas y proteínas de membrana, obtenidas por lisis de células infectadas con CHAPS, demostró la presencia de Core en ambas fracciones. Sin embargo, la cantidad de proteína recombinante presente en la fracción de proteínas de membrana es netamente mayor que en la fracción de proteínas citosólicas (figura 1c).
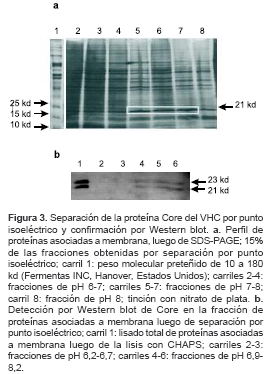
La proteína Core del VHC se ubicó en un rango de pH entre 6,9 y 8,2. La separación de proteínas por punto isoeléctrico permitió comprobar el enriquecimiento de las fracciones proteicas en diferentes rangos de pH, según lo observado luego de la tinción del gel con nitrato de plata (figura 3a). El análisis por Western blot permitió identificar la proteína Core en las fracciones de pH 6,9 a 8,2 (figura 3b), que corresponden a fracciones con menor carga proteica, lo que facilita la purificación a partir de gel.
Purificación de la proteína Core por electroelución a partir de gel
Las fracciones proteicas aisladas por electroelución se sembraron en dos geles, bajo las mismas condiciones y se sometieron a SDSPAGE al 15% y posterior tinción con plata o análisis por Western blot.
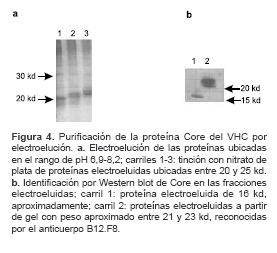
El análisis por tinción con plata reveló la presencia de una o dos bandas de proteína, como máximo, por fracción (figura 4a). El análisis por Western blot demostró que la banda número 2, a partir del peso molecular de 20 kd corresponde a la proteína Core (figura 4b). En esta fracción se detectó principalmente la isoforma Core p23. Según la cuantificación por densitometría, se obtuvieron 300 ng de proteína recombinante a partir de 0,8 mg de proteína total (no se muestra el dato).
Discusión
En este artículo se describe la construcción de un baculovirus recombinante para la proteína Core completa del VHC, la expresión en células de insecto y la purificación de la proteína recombinante.
El sistema de producción de proteínas en baculovirus tiene dos ventajas sobre los otros sistemas eucariotes: 1) las proteínas recombinantes son procesadas, modificadas y se ubican en la fracción subcelular donde normalmente se localizan luego de ser sintetizadas por la célula blanco, por lo que se espera una proteína recombinante funcional, similar a la proteína nativa, en la mayoría de los casos, y 2) el alto nivel de expresión de la proteína recombinante, que puede corresponder hasta el 20% del total de las proteínas celulares expresadas (45-47).
En este estudio, se detectaron las tres isoformas de Core del VHC en la fracción de proteínas asociadas con membranas celulares, luego de infectar células de insecto con el baculovirus recombinante; esta localización corrobora lo descrito previamente en la literatura (36).
Como se mencionó anteriormente, los dominios hidrofóbicos de las isoformas p23 y p21 permiten la asociación de Core a la membrana del retículo endoplasmático y a acúmulos de lípidos. Esta propiedad es importante en el procesamiento mediado por proteasas de membrana, en la correcta morfogénesis y en la gemación de las partículas virales de novo. Además, se ha descrito que la estabilidad de la proteína Core depende de los dominios hidrofóbicos, posiblemente por la asociación con acúmulos de lípidos que contienen triacilglicerol (15,17,18).
La purificación de proteínas asociadas con la membrana representa un obstáculo importante en el estudio de este tipo de proteínas. Existen diversas técnicas, aunque con resultados menos satisfactorios que en el caso de purificación de proteínas solubles. Los métodos preparativos de purificación de proteínas en condiciones nativas incluyen el uso de detergentes que permiten la obtención de la proteína a partir de las membranas, y la separación por métodos que no alteren la proteína, como isoelectroenfoque, cromatografía de columna y gradientes de sacarosa (48).
En este estudio, la purificación de la proteína Core obtenida en el sistema de expresión de baculovirus, se intentó inicialmente por afinidad a columna de níquel; teniendo en cuenta que la subclonación se realizó en el plásmido pBlueBac4.5/V5-His para generar una proteína de fusión Core-His con ubicación de las histidinas en el extremo C-terminal de la proteína. Sin embargo, los diferentes ensayos realizados con esta técnica no permitieron obtener la proteína de interés.
Este resultado puede deberse a diferentes factores: en primer lugar, estos ensayos se realizaron con el detergente Nonidet P40 (tampón RIPA) que no es el detergente ideal para la obtención de proteínas asociadas a membrana. En segundo lugar, la ubicación de las histidinas en el extremo C-terminal de la proteína pudo haber limitado la obtención de la proteína de interés, puesto que sólo la isoforma p23 correspondería a la proteína de fusión; sin embargo, la formación de complejos entre las isoformas p23, p21 y p16 (49), teóricamente podría permitir la purificación de estos complejos, usando la columna en mención.
Por último, es posible que la cola de histidinas no esté accesible al níquel en la columna, debido al plegamiento de la proteína Core.
Posteriormente, se realizaron ensayos de purificación por inmunoafinidad a la columna de sephadex-proteína A, utilizando como anticuerpo de captura el anticuerpo B12.F8. La cantidad de proteína Core purificada fue escasa debido probablemente a problemas de saturación de la columna. Dado que este anticuerpo corresponde al sobrenadante de la línea celular de hibridoma humano B12.F8 en el cual se encuentra presente gran cantidad de proteínas, se hace difícil la determinación exacta de la concentración de anticuerpo y la saturación correcta de la columna (24).
Finalmente, la obtención de las proteínas asociadas a membrana se logró utilizando el tampón CHAPS, por su capacidad de liberar las proteínas asociadas a membranas, ya que es un compuesto anfifílico. La purificación de la proteína Core a partir de proteínas asociadas a membrana se realizó por combinación de las técnicas de separación de proteínas por isoelectroenfoque (42) y electroelución (50).
En un primer experimento, se identificó el rango de pH en el que se encontraba ubicada la proteína Core que correspondió al rango de pH entre neutro y básico. Este resultado difirió del estudio publicado por Yvon et al. en el que se describe la ubicación de la proteína Core en fracciones de pH ácido (51); esta discrepancia puede deberse a la presencia de la cola de 6 histidinas en el extremo C-terminal, que puede favorecer la migración de la proteína hacia el cátodo. Aunque esta afirmación es aplicable a la isoforma p23, no se debe descartar la posibilidad de la formación de complejos entre p23, p21 y p16, y, por tanto, la posibilidad de encontrar las tres isoformas de Core en la fracción purificada (49).
Una ventaja de la técnica de purificación de proteínas por punto isoeléctrico es la eliminación de otras proteínas con un peso molecular similar a la proteína de interés pero con punto isoeléctrico diferente. Como se describió previamente, gran cantidad de proteínas se ubicaron principalmente en el rango de pH ácido, y menor cantidad en el rango de pH neutro-básico, lo que permitió mejor separación de las proteínas con un peso molecular entre 20 y 25 kd.
El análisis por tinción con nitrato de plata de la fracción de proteínas de membrana obtenidas de células no infectadas, luego de separación por punto isoeléctrico, reveló la ausencia de proteínas en las fracciones del rango de pH en mención (no se muestra el dato). Sin embargo, teniendo en cuenta el límite de detección de la técnica de tinción con nitrato de plata (0,5 a 8 ng), no se puede descartar la presencia de proteínas en esta fracción.
Los ensayos de electroelución permitieron obtener la proteína Core recombinante pura, con predominancia de la isoforma p23 con relación a la isoforma p21.
No obstante, este método de purificación también tiene algunas desventajas, entre las que se encuentran la cantidad de proteína purificada obtenida, aproximadamente, 300 ng de proteína Core a partir de 5x108 células H5, y la manipulación prolongada de la proteína durante los procedimientos, haciendo factible su contaminación.
Considerando que la proteína Core obtenida posee varias de las condiciones de la proteína nativa, como peso molecular, isoformas y localización subcelular, consideramos que la proteína producida por este sistema es similar a la proteína producida durante la infección natural (52). Además, la proteína fue reconocida por el anticuerpo B12.F8 que reconoce un epítopo B conformacional en el dominio N-terminal de la proteína Core del VHC. Este anticuerpo ha sido utilizado en ensayos de detección por inmunofluorescencia de la proteína Core en células BHK-21 y células HepG2 transducidas con partículas recombinantes del virus Semliki Forest para la proteína Core (rSFVCore) (41).
Los procedimientos descritos en este artículo son aplicables a proteínas asociadas a membranas producidas en sistemas de expresión eucarióticos.
Agradecimientos
Los autores agradecen a Andrés Arias, Gloria Inés Sánchez y María Teresa Rugeles, investigadores de los grupos de Inmunodeficiencias, Infección y Cáncer e Inmunovirología-Biogénesis de la Universidad de Antioquia, respectivamente, por la asesoría técnica y la disponibilidad de equipos y laboratorio.
Conflicto de intereses
Los autores del artículo declaramos no tener vinculación laboral con ningún laboratorio farmacéutico o empresa interesada en obtener resultados positivos o negativos de la investigación.
Financiación
Este trabajo fue financiado por el Instituto Colombiano para el Desarrollo de la Ciencia y la Tecnología "Francisco José de Caldas" Colciencias (proyecto 1115-05-11076), por la Fundación para la Promoción de la Investigación y la Tecnología del Banco de la República y el Comité para el Desarrollo de la Investigación, CODI, de la Universidad de Antioquia.
Correspondencia:
María Cristina Navas, Carrera 51D No. 62-29, Facultad de
Medicina, Universidad de Antioquia, Medellín, Colombia.
Teléfono: (574) 210 6076; fax (574) 210 6047
Recibido: 11/06/04; aceptado: 13/12/04
Referencias
1. World Health Organization. Hepatitis C-Global prevalence (update). Wkly Epidemiol Rec 2000;75:18-9. [ Links ]
2. Alberti A, Chemello L, Benvegnu L. Natural history of hepatitis C. J Hepatol 1999;31:17-24. [ Links ]
3. Toyoda H, Fukuda Y, Nakano I, Katano Y, Takayama T, Kumada T et al. Quasispecies nature of hepatitis C virus (HCV) in patients with chronic hepatitis C with mixed HCV subtypes. J Med Virol 1998;54:80-5. [ Links ]
4. Monazahian M, Kippenberger S, Muller A, Seitz H, Bohme I, Grethe S et al. Binding of human lipoproteins (low, very low, high density lipoproteins) to recombinant envelope proteins of hepatitis C virus. Med Microbiol Immunol 2000;188:177-84. [ Links ]
5. Gale M Jr, Blakely CM, Kwieciszewski B, Tan SL, Dossett M, Tang NM et al. Control of PKR protein kinase by hepatitis C virus nonstructural 5A protein: molecular mechanisms of kinase regulation. Mol Cell Biol 1998;18:5208-18. [ Links ]
6. Thimme R, Bukh J, Spangenberg HC, Wieland S, Pemberton J, Steiger C et al. Viral and immunological determinants of hepatitis C virus clearance, persistence, and disease. Proc Natl Acad Sci USA 2002;99:15661-8. [ Links ]
7. Goutagny N, Fatmi A, Ledinghen VD, Penin F, Couzigou P, Inchauspe G et al. Evidence of viral replication in circulating dendritic cells during hepatitis C virus infection. J Infect Dis 2003;187:1951. [ Links ]
8. Kanto T, Hayashi N, Takehara T, Tatsumi T, Kuzushita N, Ito A et al. Impaired allostimulatory capacity of peripheral blood dendritic cells recovered from hepatitis C virus-infected individuals. J Immunol 1999;68:4776-84. [ Links ]
9. Auffermann-Gretzinger S, Keeffe E, Levy S. Impaired dendritic cell maturation in patients with chronic, but not resolved, hepatitis C virus infection. Blood 2001;15: 3171-6. [ Links ]
10. Bain C, Fatmi A, Zoulim F, Zarski JP, Trepo C, Inchauspe G. Impaired allostimulatory function of dendritic cells in chronic hepatitis C infection. Gastroenterology 2001;120:512-24. [ Links ]
11. Navas MC, Fuchs A, Schvoerer E, Bohbot A, Aubertin AM, Stoll-Keller F. Dendritic cell susceptibility to hepatitis C virus genotype 1 infection. J Med Virol 2002;67:152-61. [ Links ]
12. Lee CH, Choi YH, Yang SH, Lee CW, Ha SJ, Sung YC. Hepatitis C virus core protein inhibits interleukin 12 and nitric oxide production from activated macrophages. Virology 2001;279:271-9. [ Links ]
13. Kim HS, Lee JK, Yang IH, Ahn JK, Oh YI, Kim CJ et al. Identification of hepatitis C virus core domain inducing suppression of allostimulatory capacity of dendritic cells. Arch Pharm Res 2002;25:364-9. [ Links ]
14. Grakoui A, Wychowski C, Lin C, Feinstone SM, Rice CM. Expression and identification of hepatitis C virus polyprotein cleavage products. J Virol 1993;67:1385-95. [ Links ]
15. McLauchlan J, Lemberg MK, Hope G, Martoglio B. Intramembrane proteolysis promotes trafficking of hepatitis C virus core protein to lipid droplets. EMBO J 2002;21:3980-8. [ Links ]
16. Weihofen A, Binns K, Lemberg MK, Ashman K, Martoglio B. Identification of signal peptide peptidase, a presenilin-type aspartic protease. Science 2002;296: 2215-8. [ Links ]
17. Yasui K, Wakita T, Tsukiyama-Kohara K, Funahashi SI, Ichikawa M, Kajita T et al. The native form and maturation process of hepatitis C virus Core protein. J Virol 1998;72:6048-55. [ Links ]
18. Hope RG, Murphy DJ, McLauchlan J. The domains required to direct core proteins of hepatitis C virus and GB virus-B to lipid droplets share common features with plant oleosin proteins. J Biol Chem 2002;277:4261- 70. [ Links ]
19. Ravaggi A, Natoli G, Primi D, Albertini A, Levrero M, Cariani E. Intracellular localization of full-length and truncated hepatitis C virus core protein expressed in mammalian cells. J Hepatol 1994;20:833-6. [ Links ]
20. Li D, Takyar ST, Lott WB, Gowans EJ. Amino acids 1-20 of the hepatitis C virus (HCV) core protein specifically inhibit HCV IRES-dependent translation in HepG2 cells, and inhibit both HCV IRES- and cap-dependent translation in HuH7 and CV-1 cells. J Gen Virol 2003;84: 815-25. [ Links ]
21. Nolandt O, Kern V, Muller H, Pfaff E, Theilmann L, Welker R et al. Analysis of hepatitis C virus core protein interaction domains. J. Gen Virol 1997;78:1331-40. [ Links ]
22. Kunkel M, Watowich SJ. Biophysical characterization of hepatitis C virus core protein: implications for interactions within the virus and host. FEBS Lett 2004; 557:174-80. [ Links ]
23. Jolivet-Reynaud C, Dalbon P, Viola F, Yvon S, Paranhos-Baccala G, Piga N et al. HCV core immunodominant region analysis using mouse monoclonal antibodies and human sera: characterization of major epitopes useful for antigen detection. J Med Virol 1998;56:300-9. [ Links ]
24. Cerino A, Boender P, La Monica N, Rosa C, Habets W, Mondelli MU. A human monoclonal antibody specific for the N terminus of the hepatitis C virus nucleocapsid protein. J Immunol 1993;151:7005-15. [ Links ]
25. Siemoneit K, da Silva Cardoso M, Wolpl A, Koerner K, Subanek B. Isolation and epitope characterization of human monoclonal antibodies to hepatitis C virus core antigen. Hybridoma 1994;13:9-13. [ Links ]
26. Menez R, Bossus M, Muller BH, Sibai G, Dalbon P, Ducancel F et al. Crystal structure of a hydrophobic immunodominant antigenic site on hepatitis C virus core protein complexed to monoclonal antibody 19D9D6. J Immunol 2003;170:1917-24. [ Links ]
27. Yamanaka T, Kodama T, Doi T. Subcellular localization of HCV core protein regulates its ability for p53 activation and p21 suppression. Biochem Biophys Res Commun 2002;294:528-34. [ Links ]
28. Dolganiuc A, Kodys K, Kopasz A, Marshall C, Do T, Romics L Jr et al. Hepatitis C virus core and nonstructural protein 3 proteins induce pro- and antiinflammatory cytokines and inhibit dendritic cell differentiation. J Immunol 2003;170:5615-24. [ Links ]
29. Sarobe P, Lasarte JJ, Casares N, Lopez-Diaz de Cerio A, Baixeras E, Labarga P et al. Abnormal priming of CD4(+) T cells by dendritic cells expressing hepatitis C virus core and E1 proteins. J Virol 2002;76:5062-70. [ Links ]
30. Yao ZQ, Nguyen DT, Hiotellis AI, Hahn YS. Hepatitis C virus core protein inhibits human T lymphocyte responses by a complement-dependent regulatory pathway. J Immunol 2001;167:5264-72. [ Links ]
31. Lorenzo LJ, Duenas-Carrera S, Falcon V, Acosta- Rivero N, Gonzalez E, de la Rosa MC et al. Assembly of truncated HCV core antigen into virus-like particles in Escherichia coli. Biochem Biophys Res Commun 2001;281:962-5. [ Links ]
32. Hitomi Y, McDonnell WM, Baker JR Jr, Askari FK. High efficiency prokaryotic expression and purification of a portion of the hepatitis C core protein and analysis of the immune response to recombinant protein in BALB/c mice. Viral Immunol 1995;8:109-19. [ Links ]
33. Kunkel M, Lorinczi M, Rijnbrand R, Lemon SM, Watowich SJ. Self-assembly of nucleocapsid-like particles from recombinant hepatitis C virus core protein. J Virol 2001;75:2119-29. [ Links ]
34. Acosta-Rivero N, Aguilar JC, Musacchio A, Falcon V, Vina A, de la Rosa MC et al. Characterization of the HCV core virus-like particles produced in the methylotrophic yeast Pichia pastoris. Biochem Biophys Res Commun 2001;287:122-5. [ Links ]
35. Baumert TF, Ito S, Wong DT, Liang TJ. Hepatitis C virus structural proteins assemble into viruslike particles in insect cells. J Virol 1998;72:3827-36. [ Links ]
36. Blanchard E, Brand D, Trassard S, Goudeau A, Roingeard P. Hepatitis C virus-like particle morphogenesis. J Virol 2002;76:4073-9. [ Links ]
37. Shimizu YK, Feinstone SM, Kohara M, Purcell RH, Yoshikura H. Hepatitis C virus: detection of intracellular virus particles by electron microscopy. Hepatology 1996;23:205-9. [ Links ]
38. Xiang J, Wunschmann S, George SL, Klinzman D, Schmidt WN et al. Recombinant hepatitis C virus-like particles expressed by baculovirus: utility in cell-binding and antibody detection assays. J Med Virol 2002;68: 537-43. [ Links ]
39. Clayton RF, Owsianka A, Aitken J, Graham S, Bhella D et al. Analysis of antigenicity and topology of E2 glycoprotein present on recombinant hepatitis C virus-like particles. J Virol 2002;76:7672-82. [ Links ]
40. Zhao W, Liao GY, Jiang YJ, Jiang SD. No requirement of HCV 5NCR for HCV-like particles assembly in insect cells. World J Gastroenterol 2003;9:2226-31. [ Links ]
41. Henao LF, Yepes JO, Alvarez CM, Balcazar N, Navas MC. Expresión de la proteína verde fluorescente y proteína Core del Virus de la hepatitis C en la línea de hepatoma HepG2 mediante el sistema de expresión del Semliki Forest Virus. Act Biol 2004;26:23-9. [ Links ]
42. Kang X, Frey DD. Chromatofocusing of peptides and proteins using linear pH gradients formed on strong ion-exchange adsorbents. Biotechnol Bioeng 2004;87: 376-87. [ Links ]
43. Burnette WN. Western blotting: electrophoretic transfer of proteins from sodium dodecyl sulfatepolyacrylamide gels to unmodified nitrocellulose and radiographic detection with antibody and radioiodinated protein A. Anal Biochem 1981;112:195-203. [ Links ]
44. Bonning BC, Hoover K, Duffey S, Hammock BD. Production of polyhedra of the Autographa californica nuclear polyhedrosis virus using the Sf21 and Tn5B1-4 cell lines and comparison with host-derived polyhedra by bioassay. J Invertebr Pathol 1995;66:224-30. [ Links ]
45. Luckow VA. Baculovirus systems for the expression of human gene products. Curr Opin Biotechnol 1993;4: 564-72. [ Links ]
46. Patterson RM, Selkirk JK, Merrick BA. Baculovirus and insect cell gene expression: review of baculovirus biotechnology. Environ Health Perspect 1995;103:756-9. [ Links ]
47. Marchal I, Jarvis DL, Cacan R, Verbert A. Glycoproteins from insect cells: sialylated or not? Biol Chem 2001;382:151-9. [ Links ]
48. Kashino Y. Separation methods in the analysis of protein membrane complexes. J Chromatogr B Analyt Technol Biomed Life Sci 2003;797:191-216. [ Links ]
49. Harada S, WatanabeY, Takeuchi K, Suzuki T, Katayama T, Takebe Y, et al. Expression of processed core protein of hepatitis C virus in mammalian cells. J Virol 1991;24:43-51. [ Links ]
50. Stralfors P, Belfrage P. Electrophoretic elution of proteins from polyacrylamide gel slices. Anal Biochem 1983;128:7-10. [ Links ]
51. Yvon S, Rolland D, Charrier JP, Jolivet M. An alternative for purification of low soluble recombinant hepatitis C virus core protein: preparative twodimensional electrophoresis. Electrophoresis 1998;19: 1300-5. [ Links ]
52. Yamanaka T, Uchida M, Doi T. Innate form of HCV core protein plays an important role in the localization and the function of HCV core protein. Biochem Biophys Res Commun 2002;294:521-7
. [ Links ]













