Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO  Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.25 n.1 Bogotá mar. 2005
Análisis por LSSP-PCR de la variabilidad genética de
Trypanosoma cruzi en sangre y órganos de ratones
Ana María Mejía, Omar Triana
Grupo de Chagas, Instituto de Biología, Universidad de Antioquia, Medellín, Colombia
Introducción.
La enfermedad de Chagas, causada por Trypanosoma cruzi, presenta un curso clínico variable que oscila desde casos asintomáticos a casos crónicos. T. cruzi tiene una estructura clonal y las cepas infectivas son a menudo multiclonales. La variabilidad genética de T. cruzi puede ser un determinante para el tropismo diferencial a tejidos y, consecuentemente, para las formas clínicas de la enfermedad.Objetivo. Caracterizar genéticamente los parásitos de sangre y órganos de ratones infectados con dos cepas colombianas de T. cruzi.
Materiales y métodos. Se infectaron ratones con dos cepas colombianas de T. cruzi con el fin de determinar la infección en sangre y órganos. Para esto, se evaluó la sensibilidad de tres marcadores moleculares diferentes, y se determinó la variabilidad genética de los clones por la técnica de reacción en cadena de la polimerasa de baja astringencia con un único iniciador específico (LSSP-PCR), utilizando el marcador del ADN del cinetoplasto (kADN). Los perfiles de bandas obtenidos con la LSSP-PCR se analizaron por el método de neighbor-joining .
Resultados y conclusiones. Nuestros resultados confirmaron la presencia de los dos grupos de T. cruzi en las cepas y el carácter policlonal de éstas. El marcador más sensible fue el kADN y el órgano más afectado, el corazón. Se encontraron diferencias genéticas entre los clones presentes en la sangre y los órganos de los ratones infectados. En conclusión, estos resultados apoyan el uso de la LSSP-PCR para el entendimiento de la epidemiología de la enfermedad de Chagas.
Palabras clave: Trypanosoma cruzi, variación genética, LSSP-PCR, ADN del cinetoplasto, Enfermedad de Chaga
Genetic variability of Trypanosoma cruzi in blood and organs of infected mice determined by LSSP-PCR
Background. Chagas disease, caused by Trypanosoma cruzi, has a variable clinical course, ranging from asymptomatic infection to chronic disease. Trypanosoma cruzi has a clonal population structure, although infecting strains are often multiclonal. Genetic variability of T cruzi may be one of the determinant factors in differential tissue tropism and consequently of the clinical forms of the disease.
Objective. To examine this possibility, mice were infected with two Colombian T. cruzi strains to determine the distribution of genetic variants in blood and organs. The sensitivity of three molecular markers was evaluated, and the genetic variability of the clones was determined.
The latter was attained by means of low-stringency single specific primer polymerase chain reaction (LSSP-PCR) using a kinetoplast DNA (kDNA) marker. The kDNA signatures obtained with the LSSP-PCR were analyzed by neighbor-joining.
Results and conclusion. The presence of the two lineages of T. cruzi was confirmed, as well as the multiclonal character of the two strains. The most sensitive marker was the kDNA. The most affected organ was the heart, which showed the greatest number of positive results with the three markers. Genetic differences were noted between the clones from the blood and organs of infected mice. These results support the value of the LSSP-PCR technique for the study of the molecular epidemiology of Chagas disease.
Keywords: Trypanosoma cruzi, genetic variability, LSSP-PCR, kinetoplast DNA, Chagas disease.
La enfermedad de Chagas, causada por el Trypanosoma cruzi, afecta a 17 millones de personas de diferentes regiones de Centro y Suramérica. Esta situación ha convertido la enfermedad en un problema de salud pública en muchos países. En Colombia afecta cerca del 5% de la población y, aproximadamente, el 11% está en riesgo de contraer la infección (1).
Aunque la enfermedad de Chagas se reporta desde hace casi 100 años (2), poco se conoce de su patogénesis. El curso clínico es muy variable; se presenta desde casos asintomáticos hasta casos crónicos graves que pueden involucrar problemas cardiacos, gastrointestinales o ambos (3). La gravedad y los síntomas de la enfermedad varían en las distintas regiones donde se presenta y se ha sugerido que dicha variabilidad puede ser el resultado de factores genéticos tanto del hospedero como del parásito (4).
T. cruzi es considerado como un organismo diploide, aunque su reproducción es básicamente clonal (5,6); su interacción sexual se restringe a algunas poblaciones selváticas (7). Mediante ensayos con isoenzimas y microsatélites se encontró que las cepas de este protozoo son frecuentemente multiclonales (6,8) y, posiblemente, los pacientes y los vectores se infectan al menos con dos clones genéticamente diferentes (9,10).
Desde principios del siglo pasado se ha postulado que la distribución diferencial de T. cruzi en los tejidos puede influir en la patogénesis de la enfermedad (11). Diferentes estudios han logrado determinar algunas asociaciones epidemiológicas de la enfermedad en las diferentes regiones. Así, mientras las manifestaciones gastrointestinales son raras en Centroamérica, México, Colombia y Venezuela (12), en Brasil, estas manifestaciones tienen mayor incidencia con o sin patología cardiaca asociada (13). Sin embargo, en estudios recientes se ha podido establecer que las poblaciones multiclonales que infectan un individuo, tienen clones con tropismo específico a diferentes tejidos y es exactamente la distribución de estos clones lo que puede influir o determinar el curso clínico de cada paciente (14). También se ha observado que los clones predominantes en las cepas pueden ser los responsables del tropismo preferencial hacia órganos y tejidos específicos, y esto podría contribuir a diferentes patrones de manifestación clínico-patológica en un área geográfica determinada (15), lo que se conoce como el "modelo histotrópico clonal" de la enfermedad de Chagas (16).
En búsqueda de un mejor entendimiento de la epidemiología molecular y de la variabilidad clínica de la enfermedad de Chagas, se ha propuesto estudiar la variabilidad genética del parásito directamente de los tejidos (17,18). Para realizar estos estudios, tanto de las cepas en cultivo como en tejidos, se ha sugerido la técnica de la reacción en cadena de la polimerasa de baja astringencia con un único iniciador específico (LSSP-PCR) que permite traducir el polimorfismo intraespecífico de una secuencia de nucleótidos en perfiles electroforéticos únicos y reproducibles. Los cambios en una base pueden alterar el patrón multibandas generado por la LSSP-PCR y producir nuevos perfiles que son diagnósticos de alteraciones específicas (9,17,18). Esta técnica ha sido validada por comparación de secuencias del marcador analizado (19-21).
Teniendo en cuenta que en Colombia también se reportó una estructura clonal para las cepas de T. cruzi (22,23), el objetivo del presente trabajo fue evaluar la variabilidad genética de dos cepas colombianas de T. cruzi en la sangre y los diferentes órganos de ratones, durante el curso de la infección, mediante el uso de la LSSP-PCR. Este aspecto es de gran importancia para el entendimiento de la diversidad de las manifestaciones clínicas de la enfermedad de Chagas.
Materiales y métodos Parásitos
Se utilizaron dos cepas colombianas de T. cruzi pertenecientes al grupo I: Cas15 y Mg8. La primera se aisló del vector Rhodnius prolixus silvestre del departamento de Casanare en el 2000. Esta cepa se ha mantenido en el Laboratorio de Chagas de la Universidad de Antioquia por repiques sucesivos cada siete días en medio de cultivo líquido LIT a 28°C (24) y por infección a ratones BALB/c. La segunda se aisló del vector Triatoma dimidiata silvestre del departamento del Magdalena en el 2003, fecha en la cual se llevó a cabo este estudio. Además, se utilizaron las cepas Y de Brasil y Af1 de Colombia, pertenecientes al grupo T. cruzi II, como control en algunos de los experimentos.
Infección de los ratones
Cada cepa fue evaluada en 9 ratones machos BALB/c de 20 días de edad. Los ratones se inocularon intraperitonealmente con 12x106 tripomastigotes, obtenidos del medio de cultivo. A los ocho días después de la inoculación se evaluó la infección por el método del microhematocrito (25) y se obtuvo una gota de sangre de la cola de cada animal para ser analizada por PCR (26). Este procedimiento se repitió cada 8 días hasta llegar al día 32 después de la infección. A los 16, 24 y 32 días posteriores a la infección se sacrificaron tres ratones infectados con cada una de las cepas y se les extrajo el corazón, el hígado, el bazo y el intestino delgado para estudiar la presencia del parásito en cada uno de ellos. Como control negativo se utilizó un ratón macho BALB/c no infectado. El sacrificio de los ratones se llevó a cabo por el método de inhalación de CO2, el cual es aprobado por el Comité de Ética para experimentación con animales de la Universidad de Antioquia.
Extracción del ADN
El ADN de los epimastigotes de las cepas en cultivo y de las manchas de sangre se obtuvo por los métodos de salting out (27) y Chelex® (26), respectivamente. El ADN de los órganos se obtuvo por extracción con fenol-cloroformo (28), previa homogeneización en solución de digestión (SDS 10%, NaCl 5 M, EDTA 0,5 M, Tris-HCl 1 M pH 7,5) y proteinasa K (100 µg/ml).
Detección del parásito por PCR
Para determinar si el parásito se encontraba presente en la sangre y en los diferentes órganos, se utilizaron tres marcadores diferentes: el AND del cinetoplasto (kADN), el espaciador intergénico de los genes miniexón y una secuencia satélite del ADN nuclear de T. cruzi (Sat-DNA).
Para amplificar el kADN se utilizaron los oligos S35 (5- AATAATGTACGGGGAGATGCATGA-3) y S36 (5-GGGTTCGATTGGGGTTGGTGT-3), los cuales amplifican un fragmento de 330 pb correspondiente a la región variable de los minicírculos (29). La PCR se llevó a cabo en un volumen final de 50 µL de reacción que contenía 1 µl de ADN molde, 50 mM de KCl, 10 mM Tris-HCl, 0,1% Tritón X-100, 1,5 mM de MgCl2, 10 pmol de cada iniciador, 200 µM de dNTPs y 2,5 unidades de Taq polimerasa (Fermentas). Los ciclos de amplificación se llevaron a cabo a una temperatura inicial de 94°C por 3 minutos, seguida de 35 ciclos de 94°C por 45 segundos, 63°C por 45 segundos y 72°C por 45 segundos, y un ciclo final de 72°C por 10 minutos.
Para amplificar el espaciador intergénico de los genes miniexón, se hizo una PCR múltiple, utilizando los oligos TC1 (5-GTGTCCGCCACCTCCT-TCGGGCC-3), TC2 (5-CCTGCAGGCACACGTGTGTGTG- 3) y TCC (5-CCCCCCTCCCAGGCCACACTG- 3), los cuales amplifican un fragmento de 300 pb para el grupo T. cruzi II y un fragmento de 350 pb para el grupo T. cruzi I (30). La PCR se hizo en un volumen final de 25 µl que contenían 2,5 µl de ADN molde, 50 mM KCl, 10 mM Tris-HCl, 0,1% Tritón X-100, 1,5 mM MgCl2,12,5 pmol de cada iniciador, 200 mM dNTPs y 0,625 unidades de Taq polimerasa (Fermentas). Los ciclos de amplificación se llevaron a cabo a una temperatura inicial de 94°C por 3 minutos, seguida de 27 ciclos de 94°C por 30 segundos, 55°C por 30 segundos, 72°C por 30 segundos y un ciclo final de 72°C por 10 minutos. Además, se hizo una PCR con las mismas condiciones anteriores, utilizando solamente los oligos TCC y TC1, para determinar la presencia del grupo T. cruzi II en las muestras.
Para amplificar el Sat-DNA se utilizaron los oligos TcZI (5- GAGCTCTTGCCCACACGGGTGCT-3) y TcZII (5-CCTCCAAGCAGCGGATAGTTCAGG-3), los cuales amplifican un fragmento de 188 pb (31). La PCR se llevó a cabo en un volumen final de 50 mL de reacción que contenía 1 ml de ADN molde, 50 mM de KCl, 10 mM Tris-HCl, 0,1% Tritón X-100, 1,5 mM de MgCl2, 20 pmol de cada iniciador, 200 µM de dNTPs y 2,5 unidades de Taq polimerasa (Fermentas). Los ciclos de amplificación se llevaron a cabo a una temperatura inicial de 94°C por 3 minutos, seguida de 45 ciclos de 94°C por 45 segundos, 60°C por 1 minuto y 72°C por 1 minuto, y un ciclo final de 72°C por 10 minutos.
Los productos amplificados se analizaron en geles de agarosa al 1% teñidos con bromuro de etidio y se visualizaron con luz ultravioleta (28).
Análisis por LSSP-PCR de la región variable del kADN
El amplificado de 330 pb correspondiente a la región variable del kADN se cortó y se purificó de los geles de agarosa de bajo punto de fusión al 1,5%. El producto purificado se diluyó 10 veces en agua bidestilada. Como molde para la reacción de LSSP-PCR (17) se empleó 1 µl de la dilución. La reacción se llevó a cabo en un volumen final de 25 µl, utilizando 50 mM de KCl, 10 mM Tris-HCl, 0,1% Tritón X-100, 1,5 mM de MgCl2, 120 pmoles del iniciador S35, 200 µM de cada dNTP y 4 unidades de Taq polimerasa (Fermentas). Los ciclos de amplificación se llevaron a cabo a una temperatura inicial de 94°C por 3 minutos, seguida de 35 ciclos de 94°C por 45 segundos, 30°C por 45 segundos y 72°C por 45 segundos y un ciclo final a 72°C por 10 minutos (19). Los productos amplificados se analizaron en geles de poliacrilamida al 6% (28), teñidos con nitrato de plata (9). Este procedimiento se practicó por duplicado con el fin de evaluar la reproducibilidad de la técnica.
Análisis de los resultados
Los perfiles de bandas obtenidos con la LSSPPCR para la banda de 330 pb de las muestras de sangre y de los órganos de cada uno de los ratones infectados con las dos cepas y para cada una de las cepas en cultivo se analizaron cualitativamente por observación de los geles y con el programa de manejo de imágenes ImageJ (32). Este programa genera electroferogramas, en los cuales la altura del pico indica la presencia de la banda y el ancho, su intensidad. Con lo anterior se construyó una matriz de ceros y unos, según la ausencia o presencia de la banda, respectivamente. Estos datos se analizaron con el programa PAUP 4.0, el cual genera dendrogramas por el método de neighbor-joining (33). Este análisis se hizo para cada grupo de ratones tomando todas las muestras en conjunto. Además, y para lograr una mejor comparación de los resultados, se hizo un análisis independiente de los patrones multibandas obtenidos de sangre, corazón, hígado, bazo e intestino para cada ratón. Del mismo modo, para determinar la variabilidad del parásito durante el transcurso de la infección, se analizaron los patrones obtenidos de las muestras de sangre y órganos correspondientes a un ratón de cada uno de los tiempos estudiados (16, 24 y 32 días posteriores a la infección).
Resultados
Detección del parásito por microhematocrito
Para la cepa Cas15 se observaron parásitos en la sangre a los 8 días después de la infección en sólo 3 de los 9 ratones infectados, mientras que en los otros momentos posteriores a la infección la presencia del parásito en todos los ratones fue positiva. Por su parte, los 9 ratones infectados con la cepa Mg8 fueron positivos para la presencia del parásito en todos los momentos analizados posteriores a la infección.
Detección del parásito por PCR
Para la detección del parásito en sangre se observó una mayor sensibilidad de la técnica de PCR del marcador kADN (98% de muestras positivas) con respecto al microhematocrito (88,7% de muestras positivas). A su vez, para ambas cepas, el marcador kADN mostró mayor sensibilidad con el 88% de muestras positivas, en comparación a lo obtenido para el gen miniexón (45,5%) y el Sat-DNA (13%). En las dos cepas, el órgano que se encontró con mayor número de resultados positivos para los tres marcadores estudiados fue el corazón, mientras que el intestino delgado fue positivo en muy pocas muestras (cuadros 1 y cuadro 2, figura 1).

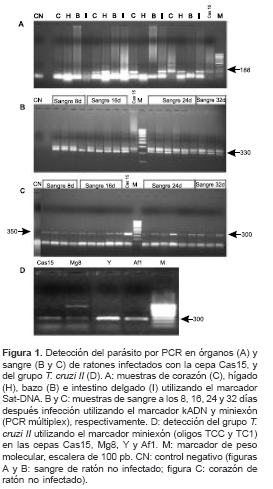

Es importante resaltar que al amplificar el espaciador intergénico de los genes miniexón, se encontró en las muestras de sangre la presencia de ambos grupos de T. cruzi, mientras que en las cepas en cultivo y en los órganos sólo estaba presente el grupo I (
figura 1C). Estos resultados sugerían la presencia de ambos grupos de T. cruzi en las dos cepas, lo cual se confirmó con una PCR que utilizó solamente los oligos TC1 y TCC, específicos para el grupo II (figura 1D). Esta demostración llevó a evaluar los órganos con estos mismos oligos; se encontró infección con dicho grupo en algunos de los corazones (no se muestran los datos).Perfiles electroforéticos del kADN por LSSP-PCR
En los ratones infectados con la cepa Cas15 se encontró que los parásitos presentaban perfiles electroforéticos diferentes entre las muestras de sangre a los 8 y 16 días, mientras que a los 24 y 32 días, estos perfiles fueron más homogéneos.
Los perfiles obtenidos para cada uno de los órganos del mismo ratón se encontraron muy semejantes entre sí y, a la vez, diferentes a los obtenidos en sangre (
figura 2).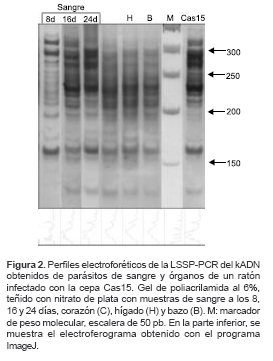
Por el contrario, los ratones infectados con la cepa Mg8 presentaron perfiles electroforéticos más similares entre las muestras de sangre y órganos a los diferentes tiempos analizados (no se muestran los datos).
Es importante resaltar que los duplicados de estos ensayos fueron reproducibles.
Análisis de los perfiles electroforéticos del kADN
Al igual que lo observado en el análisis cualitativo y de los electroferogramas de los geles, el dendrograma que se obtuvo para los ratones infectados con la cepa Cas15, muestra una cercanía genética entre las poblaciones de T. cruzi que se encuentran en los diferentes órganos. A su vez, los parásitos de las muestras de sangre a los 8 (sangre A) y 16 días (sangre B) son más heterogéneos que los de 24 (sangre C) y 32 días (sangre D). Además, se observó que las poblaciones de parásitos presentes en los órganos son diferentes a las que se encuentran en sangre (
figura 3A).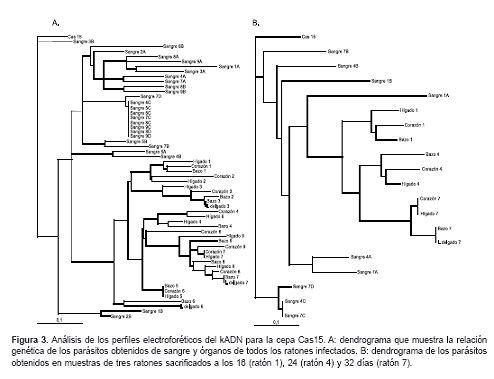
Cuando el análisis se hizo teniendo en cuenta un ratón para cada uno de los tiempos posteriores a la infección (16, 24 y 32 días), se observó claramente la presencia de tres subgrupos, cada uno correspondiente a los órganos de un mismo ratón. Como en el análisis anterior, se observó una clara división entre los clones presentes en sangre y en órganos (
figura 3B).Finalmente, se pudo ver que en los dendrogramas obtenidos para cada uno de los ratones existe la misma tendencia observada en los análisis anteriores. Sin embargo, para las muestras de sangre se observaron dos relaciones con respecto a los órganos. En la primera, que está en la mayoría, las poblaciones parasitarias presentes en sangre a los 8 días después de la infección, son más parecidas genéticamente a las que se encuentran en los órganos estudiados (
figura 4A); y en la segunda, se observa un mayor parecido entre las poblaciones que se encuentran en la sangre con las recolectadas el mismo día a partir de los órganos (figura 4B).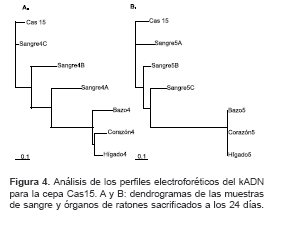
Los dendrogramas obtenidos para las muestras de la cepa Mg8 revelan la misma tendencia que los obtenidos para la cepa Cas15; sin embargo, se observó una mayor similitud entre las poblaciones presentes en la sangre y en los órganos para todos los tiempos analizados (
figura 5). Además, las poblaciones que se encontraron en los órganos fueron similares a la presentes en sangre a los 8 días después de la infección para todos los casos.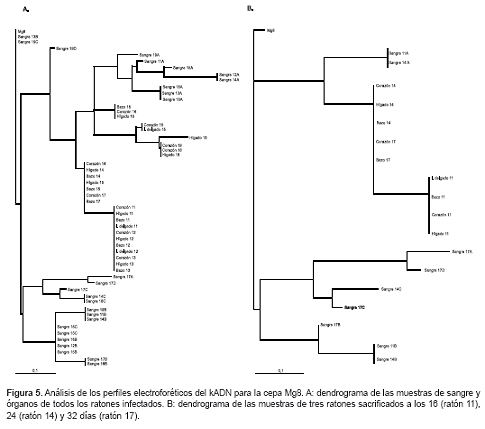
Discusión
La enfermedad de Chagas es considerada como un problema de salud pública en muchos países por la gran cantidad de personas afectadas y la variedad de manifestaciones clínicas con que se presenta. Como una contribución para el entendimiento de la epidemiología y la clínica de la enfermedad, durante años se ha tratado de encontrar mejores técnicas y marcadores para detectar T. cruzi en sangre y en los diferentes órganos infectados (9,17). Sin embargo, la mayoría de estos estudios se han llevado a cabo utilizando clones del parásito, lo que permite entender varios aspectos de la enfermedad, pero no representa la historia natural de la infección (17).
En este trabajo se utilizaron dos cepas colombianas de T. cruzi pertenecientes al grupo I, con el fin de detectar el parásito en diferentes órganos y muestras de sangre de ratones infectados, para una posterior caracterización genética de las poblaciones presentes en cada una de estas muestras.
En estudios previos se ha establecido un tropismo preferencial de las cepas de T. cruzi colombianas por el músculo cardiaco (34). Nuestro estudio apoya tales reportes, al ser el corazón el órgano con mayor número de resultados positivos, por lo que se puede inferir una mayor cantidad de parásitos presentes en este órgano. Sin embargo, la presencia del parásito en otros órganos, como el intestino, no podría descartarse teniendo en cuenta la gran cantidad de inhibidores de la PCR presentes en él. Por lo tanto, además de la PCR se deberían realizar estudios histopatológicos para verificar la presencia del parásito en los órganos. Es importante resaltar la necesidad de utilizar diferentes marcadores específicos para detectar el parásito en las diferentes muestras, lo que genera resultados más confiables. En general, los resultados obtenidos mostraron mayor sensibilidad al utilizar la PCR con el marcador kADN, por lo cual se seleccionó este marcador para realizar los estudios de variabilidad genética por LSSP-PCR. Con este marcador se observó la presencia de dos bandas entre 330 y 400 pb para la cepa Cas 15. Para determinar si estas dos bandas correspondían a los dos grupos de T. cruz se practicó la LSSP-PCR de ellas por separado, y se encontraron perfiles idénticos por lo que se concluyó que estas bandas correspondían al kADN y que la banda de mayor peso molecular podría ser el resultado de un artefacto de la PCR.
La heterogeneidad encontrada para los patrones de las diferentes muestras sugiere un carácter policlonal de las cepas utilizadas, y fue más evidente en la cepa Cas15. Además, la mayor heterogeneidad en los perfiles de la LSSP-PCR para el kADN encontrada en los parásitos presentes en las muestras de sangre, podría explicarse por la presencia de ambos grupos de T. cruzi en ésta y de sólo el grupo T. cruzi I en los órganos, como se evidenció al utilizar el espaciador intergénico de los genes miniexón. Por otra parte, las divergencias encontradas entre las dos cepas . pueden ser el resultado de diferencias en el origen biológico, ubicación geográfica y tiempo de mantenimiento en el laboratorio.
El análisis de los dendrogramas nos lleva a proponer dos dinámicas diferentes para explicar el comportamiento de las poblaciones parasitarias. La primera podría ser el resultado de una migración de la mayoría de los clones que están en la sangre hacia los órganos, por lo cual la sangre a los 8 días es tan parecida en contenido de clones a lo que se encuentra en los órganos, lugar donde se multiplican y pueden volver a circular. La sumatoria de los clones provenientes de la progenie de los nidos de parásitos más aquéllos que permanecieron en circulación determinarían que, durante el desarrollo de la infección a los tiempos subsiguientes, la sangre presente poblaciones parasitarias diferentes respecto a los órganos individuales. La segunda tendencia podría darse como resultado de que sólo algunos de los clones que están en la sangre llegan a infectar los órganos, razón por la cual los clones hallados en sangre a los 8 días de la infección son más diferentes que los de los órganos. Algunos de estos clones pueden ser eliminados por el sistema inmune del hospedero, a medida que pasa el tiempo y los que se encuentran en los órganos, al entrar en recirculación, hacen que sean más parecidos a la sangre que se recolecta el mismo día de los órganos, lo cual explica la mayor similitud entre éstos. Estas dos dinámicas podrían estar influenciadas por las características genéticas del hospedero, lo cual se ve reforzado al observar en los dendrogramas agrupaciones de los órganos de cada ratón.
Algunos autores han sugerido la circulación de los dos grupos de T. cruzi en iguales proporciones en la naturaleza y su asociación a diferentes hospederos, con un porcentaje muy bajo de infecciones mixtas (35). Sin embargo, nuestros resultados con el espaciador intergénico de los genes miniexón, aunque mostraron sólo el grupo T. cruzi I para las cepas, revelaron la presencia de los dos grupos en sangre. Este resultado sugirió que los dos grupos deben estar presentes en las cepas, lo cual fue confirmado por PCR con los oligos específicos para el grupo T. cruzi II, demostrándose una mezcla de los dos linajes con predominancia del grupo I. Es importante que en futuros estudios de caracterización de grupos de T. cruzi se utilicen por separado los oligos del gen miniexón para evitar falsos resultados debido a la presencia de un grupo predominante, comotambién los oligos TC1, TC2, TC3, TR y ME paraidentificar la presencia de los diferentes subgrupos T. cruzi II en los órganos negativos (36).
Además, la circulación de los dos grupos en lanaturaleza ha sido corroborada en heces de los vectores recolectados en el campo, donde se ha observado la presencia de ambos grupos por PCR (no se muestran los datos). Así, la posibilidad de que ambos grupos presenten una distribución diferencial a tejidos o asociación con diferentes hospederos debería confirmarse con otros estudios.
Es importante destacar que los clones presentes en las cepas no mostraron una distribución diferencial a tejidos, pero existe una diferencia entre los que se encuentran en sangre y en los diferentes órganos, lo que sugiere que los clones tienen diferente comportamiento biológico. Sin embargo, la distribución diferencial a tejidos se podría estudiar mediante la clonación de cada una de las cepas, aspecto que permitiría un mejor esclarecimiento de la epidemiología y la variabilidad clínica de la enfermedad de Chagas. Finalmente, nuestros resultados validan el uso de la técnica de la LSSP-PCR para comparar las poblaciones en la infección natural de esta enfermedad.
Agradecimientos
Ana María Mejía contó con el apoyo del CODI – Universidad de Antioquia en el programa joven investigador.
Conflicto de intereses
Los autores declaramos que no existe ningún conflicto de intereses.
Financiación
Este proyecto fue financiado por el programa de sostenibilidad 2001-2003 del CODI, Universidad de Antioquia.
Correspondencia:
Omar Triana, Calle 67 No. 53-108, Instituto de Biología,
Universidad de Antioquia, Medellín, Colombia.
otriana@matematicas.udea.edu.co
Recibido: 17/08/04; aceptado: 21/01/05
Referencias
1. Moncayo A. Chagas disease: current epidemiological trends after the interruption of vectorial and transfusional transmission in the southern cone countries. Mem Inst Oswaldo Cruz 2003;98:577-91. [ Links ]
2. Chagas C. Nova Tripanozomiase humana: estudos sobre a morfologia e o ciclo evolutivo do Schizotrypanum cruzi n. gen., n. sp., agente etiológico de nova entidade mórbida do homen. Mem Inst Oswaldo Cruz 1909;1:159-218. [ Links ]
3. Tanowitz HB, Kirchhoff LV, Simon D, Morris AS, Weiss LM, Wittner M. Chagas disease. Clin Microbiol Rev 1992;5:400-19. [ Links ]
4. Macedo AM, Pena SDJ. Genetic variability of Trypanosoma cruzi: implications for the pathogenesis of Chagas disease. Parasitol Today 1998;14:119-24. [ Links ]
5. Tibayrenc M, Ward P, Moya A, Ayala F. Natural populations of Trypanosoma cruzi, the agent of Chagas disease, have a complex multiclonal structure. Proc Natl Acad Sci USA 1986;83:115-9. [ Links ]
6. Tibayrenc M, Ayala F. Isozyme variability in Trypanosoma cruzi, the agent of Chagas disease: genetical, taxonomical, and epidemiological significance. Evolution 1988;42:277-92. [ Links ]
7. Gaunt MW, Yeo M, Frame IA, Stothard JR, Carrasco HJ, Taylor MC et al. Mechanism of genetic exchange in American trypanosomes. Nature 2003;421:936-9. [ Links ]
8. Oliveira RP, Broude NF, Macedo AM, Cantor CR, Smith CL, Pena SDJ. Probing the genetic population structure of Trypanosoma cruzi with polymorphic microsatellites. Proc Natl Acad Sci USA 1998;95:3776-80. [ Links ]
9. Andrade LO, Machado CRS, Chiari E, Pena SDJ, Macedo AM. Differential tissue distribution of diverse clones of Trypanosoma cruzi in infected mice. Mol Biochem Parasitol 1999;100:163-72. [ Links ]
10. Pinto AS, Lana M, Britto C, Bastrenta B, Tibayrenc M. Experimental Trypanosoma cruzi biclonal infection in Triatoma infestans: detection of distinct clonal genotypes using kinetoplast DNA probes. Int J Parasitol 2000;30:843-8. [ Links ]
11. Vianna G. Contribuição para o estudo da anatomia patológica da moléstia de Chagas. Mem Inst Oswaldo Cruz 1911;3:276-93. [ Links ]
12. Kirchhoff LV. American trypanosomiasis (Chagas disease). Gastroenterol Clin North Am 1996;25:517-33. [ Links ]
13. Dias JCP. Epidemiology of Chagas disease. En: Wendel S, Brener Z, Camargo MS, Rassi A, editors. Chagas disease (American Trypanosomiasis): its impact on transfusion and clinical medicine. São Paulo: ISBT Brazil; 1992. p.49-80. [ Links ]
14. Andrade SG, Magalhães JB. Biodemes and zymodemes of Trypanosoma cruzi strains: correlations with clinical data and experimental pathology. Rev Soc Bras Med Trop 1997;30:27-35. [ Links ]
15. Campos RF, Gonçalves MS, dos Reis EAG, dos Reis MG, Andrade SG. Comparative analysis by polymerase chain reaction amplified minicircles of kinetoplast DNA of a stable strain of Trypanosoma cruzi from São Felipe, Bahia, its clones and subclones: possibility of predominance of a principal clone in this area. Mem Inst Oswaldo Cruz 1999;94:23-9. [ Links ]
16. Macedo AM, Oliveira RP, Pena SDJ. Chagas disease: role of parasite genetic variation in pathogenesis. Exp Rev Mol Med (en línea) 2002 Mar [consultado 2004 Mar 1]. Disponible en: http://www-ermm.cbcu.cam.ac.uk/02004118h.htm. [ Links ]
17. Vago AR, Macedo AM, Oliveira R, Andrade LO, Chiari E, Galvão LMC et al. Kinetoplast DNA signatures of Trypanosoma cruzi strains obtained directly from infected tissues. Am J Pathol 1996;149:2153-9. [ Links ]
18. Vago AR, Andrade LO, Leite AA, Reis D, Macedo AM, Adad SJ et al. Genetic characterization of Trypanosoma cruzi directly from tissues of patients with chronic Chagas disease: differential distribution of genetic types into diverse organs. Am J Pathol 2000;156:1805-9. [ Links ]
19. Pena SDJ, Barreto G, Vago AR, De Marco L, Reinach F, Dias-Neto E et al. Sequence-specific "gene signatures" can be obtained by PCR with single specific primers at low stringency. Proc Natl Acad Sci USA 1994;91:1946-9. [ Links ]
20. Arboleda S. Relación genética entre cepas colombianas de Trypanosoma cruzi por análisis de secuencia de la región intergénica del gen Spliced-Leader RNA. Tesis de grado. Instituto de biología. Universidad de Antioquia 2004, [ Links ]
21. Cadavid M. Relación genética entre cepas colombianas de Trypanosoma cruzi por análisis con LSSP-PCR de la región intergénica del gen Spliced-Leader RNA. Tesis de grado. Instituto de biología. Universidad de Antioquia 200420. [ Links ]
22. Márquez E, Arcos-Burgos M, Triana O, Moreno J, Jaramillo N. Clonal population structure of Colombian sylvatic Trypanosoma cruzi. J Parasitol 1998;84:1143-9. [ Links ]
23. Triana O, Jaramillo N, Moreno J. Genetic variability of Colombian populations of Trypanosoma cruzi and Trypanosoma rangeli. Biol Res 1999;32:1-10. [ Links ]
24. Camargo EP. Growth and differentiation in Trypanosoma cruzi. Origin of metacyclic trypanosomes in liquid media. Rev Inst Med Trop São Paulo 1964;6:93-100. [ Links ]
25. Guhl F, Nicholls S. Manual de procedimientos para el diagnóstico de la enfermedad de Chagas. Primera edición. Santafé de Bogotá: Universidad de los Andes; 2001. p.22. [ Links ]
26. Walsh PS, Metzger DA, Higuchi R. Chelex® 100 as a medium for simple extraction of DNA for PCR-Based typing from forensic material. Biotechniques 1991;10:506-13. [ Links ]
27. Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Research 1988;16:12-5. [ Links ]
28. Sambrook J, Fritsch EF, Maniatis T. Molecular cloning. A laboratory manual. Second edition. Cold Spring: Harbor Laboratory Press; 1989. [ Links ]
29. Sturm N, Degrave W, Morel C, Simpson L. Sensitive detection and schizodeme classification of Trypanosoma cruzi cells by amplification of kinetoplast minicircle DNA sequences. Use in diagnosis of Chagas disease. Mol Biochem Parasitol 1989;33:205-14. [ Links ]
30. Fernandes O, Souto RP, Castro JA, Pereira JB, Fernandes NC, Junqueira ACV et al. Brazilian isolates of Trypanosoma cruzi from humans and triatomines classified into two lineages using mini-exon and ribosomal RNA sequences. Am J Trop Med Hyg 1998;58:807-11. [ Links ]
31. Schijman AG, Vigliano C, Burgos J, Favaloro R, Perrone S, Languens R et al. Early diagnosis of recurrence of Trypanosoma cruzi infection by polymerase chain reaction after heart transplantation of a chronic Chagas heart disease patient. J Heart Lung Transplant 2000;19:1114-7. [ Links ]
32. Rasband W. ImageJ 1.29x. National Institute of Health, USA. [consultado 2003 sep 1]. Disponible en: http://rsb.info.nih.gov/ij/
33. Sworfford DL. PAUP* phylogenetic analysis using parsimony (*and others methods). Version 4.0. Sunderland, Massachusetts Sinauer Associates; 2000. [ Links ]
34. Devera R, Fernandes O, Coura JR. Should Trypanosoma cruzi be called "cruzi" complex? A review of the parasite diversity and the potencial of selecting population after in vitro culturing and mice infection. Mem Inst Oswaldo Cruz 2003;98:1-12. [ Links ]
35. Fernandes O, Mangia RH, Lisboa CV, Pinho AP, Morel CM, Zingales B et al. The complexity of the sylvatic cycle of Trypanosoma cruzi in Rio de Janeiro state (Brazil) revealed by the non-transcribed spacer of the mini-exon gene. Parasitology 1999;118:161-6. [ Links ]
36. Fernández O, Santos SS, Cupolillo E, Mendonça B, Derre R, Junqueira CV et al. A mini-exon multiplex polymerase chain reaction to distinguish the major groups of Trypanosoma cruzi and T. rangeli in the Brazilian Amazon. Trans R Soc Trop Med Hyg 2001;95:97-9. [ Links ]














