Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO  Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.25 no.2 Bogotá June 2005
Expresión específica de los genes de la respuesta inflamatoria
en subpoblaciones de macrófagos
Vera María Ripoll 1,2, David Hume 2, Marta Raquel Fontanilla 1,3
1 Facultad de Ciencias, Universidad Nacional de Colombia, Bogotá, D. C., Colombia.
2
Macrophage Laboratory, Institute for Molecular Bioscience, the University of Queensland, Australia.3
Instituto de Biología Molecular, Universidad El Bosque, Bogotá, D. C., Colombia.Como brazo efectivo de la respuesta inmune, los macrófagos reconocen, fagocitan y destruyen patógenos potenciales. También, coordinan respuestas adicionales del hospedero mediante la síntesis de un amplio rango de mediadores de la inflamación que, en últimas, activan la respuesta inmune adaptativa y establecen la inmunidad protectora. Aunque son componentes claves del sistema de defensa de los mamíferos, el resultado de su actividad no siempre es beneficioso para el hospedero. El papel central jugado en la enfermedad hace que su regulación se convierta en un blanco importante en la prevención, el control y la curación de los procesos inflamatorios. De hecho, los genes de expresión restringida en macrófagos pueden ser puntos cruciales de la intervención terapéutica. Este trabajo revisa el uso de los microarreglos de cADN para la comparación de los genes de la inflamación que se expresan de diferente forma en dos poblaciones de macrófagos, derivados de la médula ósea y macrófagos peritoneales obtenidos después de inducir una inflamación con tioglicolato, con genes expresados en fibroblastos primarios aislados de embrión y células esplénicas no adherentes. La comparación de los perfiles de expresión indica que los genes de la inflamación de los macrófagos están asociados con categorías funcionales esperadas como la degradación lisosómica, la fagocitosis, la defensa del hospedero y de la homeostasis. La información revisada por este estudio contribuye a entender la biología de los macrófagos.
Palabras clave: macrófagos, perfil de la expresión génica, análisis de secuencia en orden de oligonucleótidos.
Specific expression of inflammatory genes in macrophage subpopulations
Macrophages serve as an effective component of innate immunity in their ability to recognize, engulf and kill potential pathogens. They also coordinate additional host responses by synthesizing a range of inflammatory mediators that can activate the adaptive immune response and establish protective immunity. Although they are a key component of mammalian defense system, macrophage activity is not always beneficial to the host. The centrality of macrophages in disease processes makes macrophage regulation a major target in the prevention, control and cure of inflammatory processes. Consequently, macrophage-restricted genes may be crucial targets for therapeutic intervention. A review is presented of the use of large-scale cDNA microarrays to compare macrophage inflammatory genes differentially expressed in two distinct macrophages populations bone marrow derived macrophages (bmm) and inflammatory thioglycolate-elicited peritoneal macrophages (tepm) to non-macrophage cell populations consisting of primary embryonic fibroblast and spleen non-adherent cells. Expression profiles indicate that macrophage inflammatory genes are associated with expected functional categories, such as lysosomal degradation, phagocytosis, host defense and homeostasis.
Key words: macrophages, gene expresion profiling, oligonucleotide array sequence analysis.
Las enfermedades caracterizadas por inflamación grave son causa importante de mortalidad y morbilidad en los humanos. Comúnmente, la respuesta inflamatoria forma parte de los mecanismos normales de vigilancia y defensa empleados por el hospedero para destruir y eliminar sustancias extrañas procedentes, en su mayoría, de microorganismos. Sin embargo, las respuestas inflamatorias pueden tornarse excesivas o persistentes debido a la acumulación de células activadas y sus productos de secreción (1).
Los macrófagos son uno de los principales tipos celulares involucrados en la inflamación (2); su capacidad de reconocer, fagocitar y destruir patógenos los hace parte esencial de la denominado inmunidad innata. Ésta es la primera línea de defensa del organismo contra invasores y su principal función es controlar la infección. En el caso de resultar ineficientes en la eliminación de patógenos, los macrófagos tienen la habilidad de activar otras células del sistema inmune como los linfocitos, formándose así el puente entre la inmunidad innata y la adquirida, esta última, provista no sólo de medios más versátiles de defensa, especificidad y diversidad sino de memoria en caso de una reinfección (3).
A pesar de ser esenciales en la iniciación de la respuesta inmune, la función de los macrófagos puede resultar tan benéfica como perjudicial para el organismo. La presencia y la proliferación excesiva de estas células en los procesos inflamatorios crónicos y, particularmente, su participación en un diverso número de alteraciones como el síndrome inflamatorio sistémico, la meningitis, la fiebre reumática y la diabetes, entre otras, ha sido reportada ampliamente (1).
La investigación y el estudio continuo de los mecanismos normales y patológicos que generan y regulan las respuestas inflamatorias son cruciales en la prevención, el control y la curación de enfermedades en humanos. Avances significativos en la comprensión de los eventos moleculares que se llevan a cabo en los macrófagos como respuesta al reconocimiento de productos bacterianos, lipopolisacáridos (LPS) y peptidoglicanos han sido posibles gracias a la identificación y la caracterización de receptores, las moléculas de señalización y los mediadores de la inflamación involucrados no sólo en éste, sino también en otros procesos inflamatorios. Sin embargo, la respuesta inflamatoria es compleja y existen muchos eventos no se han elucidado hasta el momento (1).
La expresión alterada de genes juega un papel importante en el desarrollo de las enfermedades inflamatorias. Con el surgimiento de la tecnología de microarreglos de ADNc, se ha hecho posible la identificación simultánea de cientos de genes expresados en diferentes tipos de células o de estas últimas en diversas condiciones (3). El uso de esta herramienta en la comparación de los perfiles de expresión de genes en células inflamatorias, como los macrófagos, bajo diferentes estímulos, ha permitido el descubrimiento de genes diferencialmente expresados asociados con estados inflamatorios en enfermedades como el asma, la artritis reumatoidea, las alergias y la autoinmunidad (4,5).
Este manuscrito revisa el papel de los macrófagos en la immunidad, haciendo énfasis en el uso de la tecnología de microarreglos de ADNc como herramienta para comprender su función. Al mismo tiempo, discute resultados preliminares de la identificación de los genes de la inflamación específicamente expresados en diversas poblaciones de macrófagos activados.
Microarreglos e identificación de genes inflamatorios
La expresión simultánea de genes en células, en tejidos o ambos se puede analizar con microarreglos de ADN, los cuales están conformados por secuencias blanco de ADN arregladas según coordenadas x, y, z en un soporte sólido de vidrio con silicona sobre el que se inmovilizan químicamente. Las secuencias genes de la respuesta inflamatoria en macrófagos inmobilizadas corresponden a un gen blanco determinado y, generalmente, son ADNc obtenido por retrotranscripción a partir de mARN u oligonucleótidos sintéticos (6).
El uso de microarreglos de ADNc para el análisis de los patrones de expresión de genes en células de mamíferos se ha venido incrementando desde los primeros estudios de la respuesta de fibroblastos a la estimulación con suero (7). Esta herramienta se ha utilizado en la comparación de diferentes especímenes provenientes de muestras clínicas y, particularmente, de tejidos inflamados afectados por algún tipo de enfermedad (7,8). Los datos obtenidos han permitido la identificación de genes involucrados en estos procesos, lo cual demuestra que el uso de microarreglos es útil para el entendimiento de los mecanismos que subyacen diversas enfermedades (5).
La utilización de la tecnología de microarreglos en macrófagos se ha centrado en investigar la respuesta de estas células a la presencia de citocinas como IFNg, IL-10 (9,10) y de bacterias o sus productos, principalmente, LPS (9-11). Todos estos estudios coinciden en plantear la existencia de grupos de genes cuya expresión es regulada en la respuesta a estímulos específicos.
Regulación de la maquinaria transcripcional de macrófagos por LPS y productos bacterianos
Quizá una de las respuestas exacerbadas más comunes y ampliamente estudiadas es la de los macrófagos a los productos o residuos bacterianos. Aunque, generalmente, el antígeno es eliminado sin la producción de una respuesta inflamatoria con características clínicas detectables, la promueve una cantidad y localización inusuales. Las infecciones bacterianas sistémicas generan la producción incontrolada de citocinas proinflamatorias por parte del endotelio y los leucocitos. Éstos son los responsables directos de la coagulación intravascular diseminada, la falla orgánica generalizada y la muerte. La activación de los macrófagos por LPS implica su unión a CD14 y al receptor tipo toll 4 (TLR4), cuya señalización intracelular se lleva a cabo por medio del complejo de cinasas asociado al receptor de interleucina 1 (IL-1). Lo anterior conduce a la activación del factor nuclear kappa N (NF-kB) y la transcripción de genes de respuesta, entre los cuales se encuentran la IL-1ab, la IL-6, la IL-12, la IL-18, el factor de necrosis tumoral alfa (FNTa) y las moléculas de adhesión (12,13).
En términos de la septicemia, las actividades redundantes de IL-1, IL-6 y FNTa han sido las más estudiadas (14,15). Últimamente, se ha incrementado el interés sobre las citocinas multifuncionales como la IL-12 (proinflamatoria) y la IL-10 (antinflamatoria) (16, 17). Aunque ha sido posible la identificación de nuevos intermediarios de señalización con el desarrollo de animales knockout, todavía quedan por esclarecer varios procesos y, lo más importante, una terapia efectiva (18). De ahí, la importancia de la búsqueda de métodos para la identificación a gran escala de los genes involucrados en el proceso inflamatorio.
La vigilancia y el estudio con microarreglos de la influencia del lipolisacárido (LPS) en la inducción o supresión de los transcritos en los macrófagos revelan que la expresión de casi todos se encuentra modificada de alguna manera. Hasta el momento, se han documentado más de 500 genes inducidos por LPS (10,11,19,20) empleando, en su mayoría, células en cultivo. Todos estos estudios coinciden en concluir que más del 25% de los genes totales de los macrófagos exhiben alteraciones en su patrón de expresión y muestran una regulación positiva o negativa. El número de genes suprimidos supera el de genes inducidos y, al mismo tiempo, se plantea la existencia de grupos de genes cuya expresión es regulada de manera coordinada en respuesta a estímulos específicos. La gran mayoría de estos genes inducibles están involucrados en la defensa y en la activación de la repuesta inmune (
cuadro 1). El espectro completo de los genes inducidos por LPS está siendo analizado y la búsqueda de marcadores de activación así como de genes posibles candidatos para ser blanco de nuevos medicamentos es uno de los principales objetivos.
Además de la respuesta a LPS, el cambio global en la maquinaria transcripcional de los macrófagos se ha establecido en presencia de otros productos bacterianos como petidoglicano, ácido lipoteicoico y péptidos murámicos (21,22). Igualmente, se ha , investigado el programa de expresión de genes inducidos en macrófagos por bacterias (Escherichia coli, Salmonella typhimurium), levaduras (Candida albicans) y virus (influenza) (23-25). Lo encontrado en estos tres últimos estudios sugiere que existen respuestas comunes de estas células a los tres tipos de patógenos. Estas repuestas se caracterizan por la rápida regulación negativa de los transcritos relacionados con la fagocitosis, el reconocimiento de patógenos, la inducción transitoria de los genes relacionados con la producción de citocinas y quimiocinas proinflamatorias, el procesamiento y la presentación de antígenos y la migración a tejidos (24). Lo anterior sugiere que la respuesta innata parece estar programada para reaccionar de la misma manera a diversos patógenos.
Genes regulados por interferones
El estudio de los perfiles de expresión de genes inducidos por interferones (IFN) indica que los genes pertenecientes a la categoría funcional de señalización intracelular (por ejemplo, MYD88, JUN, RELA, MAP2K1, MAP3K8) son los más estimulados. Les siguen los que contribuyen en la defensa y la inmunomodulación (quimiocinas: MIG, EBI1, SCYA2, 5, IL-8; moléculas de adhesión: ICAM1, SELL, CD47). Los factores de transcripción hacen parte importante de las moléculas inducidas, la mayoría de los cuales son activadores (IRF1-7, HIF1a , MPB1) y no supresores de dichos procesos (9).
Los estudios de los perfiles de expresión de los genes en respuesta a IFN han permitido elucidar nuevas funciones y comprender los fenotipos celulares inducidos por este grupo de moléculas. La identificación de genes, como el CD47 y el MYD88, ha sido importante en el esclarecimiento de las diferentes vías de señalización. Se ha demostrado que ambos, el interferón (IFN) y el IFNa/ß, pueden emplear vías alternas a la dependiente de Stat-1. Lo anterior tiene relevancia fisiológica por su contribución en la respuesta antiviral in vivo así como en la regulación de la migración celular y la regulación de proteínas de importancia inmunológica (26,27).
Genes regulados por IL-10
A continuación se discuten algunos aspectos de los perfiles de expresión de los macrófagos obtenidos en presencia de moléculas anti-inflamatorias como la IL-10. Esta citocina, secretada por las células T y los macrófagos, es capaz de bloquear o de reducir la producción de moléculas proinflamatorias (TNFa, IL-6, IL-12 prostaglandinas) y, como consecuencia, la respuesta inflamatoria en sí (28). Muchos de los mecanismos involucrados en estos procesos de inhibición no han sido esclarecidos; sin embargo, existen indicios que demuestran que el efecto de la IL-10 se puede atribuir a la modulación del factor de transcripción NFkB y de cinasas asociadas con mitógenos (MAPK) (29,30), de la estabilidad del mRNA (31) y de la eficiencia de los procesos de traducción (30). El empleo de microarreglos ha sido importante en la confirmación de algunas de estas hipótesis (10,32).
El uso de microarreglos en la búsqueda de nuevas moléculas marcadoras en los macrófagos ha entrado en una era bastante compleja; la selección y la interpretación de los datos así como el grado de confianza y su posibilidad de presentación en el modelo in vivo son motivo de discusión (33).
Determinacion de perfiles transcripcionales en diferentes poblaciones de macrófagos mediante microarreglos
La detección de los cambios globales en la expresión de los genes en macrófagos estimulados en comparación con no estimulados, mediante microarreglos de cADN, ha permitido la identificación de los genes asociados con la inflamación. De este modo, se ha progresado en la comprensión de este tipo de respuestas y han surgido posibilidades de medicamentos que aún se están validando. Sin embargo, la mayoría de estos estudios, así como los reportados en la literatura, han empleado células en cultivo (macrófagos derivados de médula ósea o líneas celulares). Los experimentos enfocados a la validación de los datos de microarreglos de la respuesta de macrófagos al ADN bacteriano han revelado que los grupos de genes que aparecen superinducidos en las células en cultivo, se encuentran expresados en niveles muy bajos en el sistema in vivo (manuscrito en preparación). Por esta razón, en un estudio reciente algunos de nosotros comparamos los perfiles de expresión entre poblaciones de macrófagos in vivo e in vitro con el fin de identificar los genes de la inflamación específicamente expresados en los macrófagos activados in vivo (34).
En el trabajo de Ripoll (34), se investigó el programa transcripcional de macrófagos bajo condiciones normales de cultivo (macrófagos aislados de médula ósea - bone marrow macrophages, BMM) y fisiológicas inflamatorias (macrófagos peritoneales inducidos con tioglicolato - thioglycolate-elicited peritoneal macrophages, TEPM) La población de BMM se generó in vitro por medio de la diferenciación de las células progenitoras de la médula ósea con el factor de estimulador de colonias (CSF). La población TEPM está conformada por macrófagos de respuesta inflamatoria, aislados del peritoneo de ratón luego de su estímulo con un agente inespecífico (caldo de tioglicolato). La expresión de estas dos poblaciones se comparó con la exhibida por las células del sistema inmune adquirido (linfocitos) y por fibroblastos. Se aisló el ARN total de todas las muestras y el producto de su retrotranscripción usado como sonda de hibridación de 15.000 cADN presente en chips de los National Institutes on Aging. Las comparaciones directas entre todas las muestras se llevaron a cabo obviando el uso de ARNA de referencia (ARN total de 17,5 dpc de embriones de ratón) que han demostrado no representar un gran grupo de genes expresados por los macrófagos en respuesta a los estímulos inflamatorios (19).
El
cuadro 2 presenta el número de genes inducidos que se encontraron en cada grupo celular en un experimento determinado y, consistentemente, durante todas las comparaciones.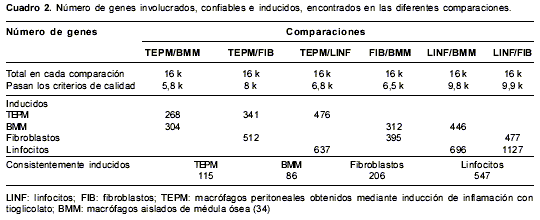
Luego de identificar los genes específicos para cada uno de los tipos celulares estudiados, los genes inducidos consistentemente fueron analizados y se determinó su expresión común o restringida. De 115 genes inducidos en TEPM, cerca del 77% (88 genes) se expresaron de manera restringida; el 13 % (15 genes) se expresó igualmente en BMM, y sólo 6% (7 genes) y 4% (5 genes) en fibroblastos y linfocitos, respectiva-mente. Aproximadamente, en BMM, el 71% (61 genes) estaban restringidos, el 17,3 % (15 genes) compartidos con TEPM, 10,5% (9 genes) con fibroblastos, y 1,2% (1 gen) con linfocitos.
Entre los genes compartidos por TEPM y BMM se encuentran los marcadores clásicos de macrófagos (receptores del complemento y de inmunoglobulinas, lectinas y ferritina); entre BMM y fibroblastos, los genes modulados por las condiciones de cultivo in vitro (ciclo celular, mitosis y replicación). Es importante anotar que el número de genes (527) inducidos y restringidos en los linfocitos es significativamente superior a todas las poblaciones analizadas, quizá debido a que se trata de una población heterogénea de células (34).
Para tener una idea más clara de la maquinaria transcripcional de cada una de las poblaciones celulares estudiadas, se llevó a cabo un análisis de agrupamiento funcional empleando la ontogenia simplificada del programa GeneSpring v5.1. Las categorías funcionales caracterizadas para cada población indican que los genes expresados en BMM están relacionados con los procesos de adhesión al plástico de las cajas de cultivo que se presentan en estas células.
Se sabe que este proceso activa las moléculas involucradas en el citoesqueleto (principal categoría funcional caracterizada), y que generan un fenotipo propio que se caracteriza por una morfología en la cual las células toman una forma alargada y estrellada semejante a la de las neuronas. La extensión de pseudópodos típica de estas células y la relación de la maquinaria del citoesqueleto con la movilización de los compartimentos endosómicos para la formación de fagosomas en el caso de la eventual presencia de patógenos, describe los procesos conocidos que acompañan la función de los macrófagos (19,35).
Otros grupos funcionales de genes involucrados en la respuesta inmune, como los relacionados con la producción de diferentes tipos de oxígeno (estrés oxidativo), también se encontraron en BMM. Igualmente, los grupos funcionales correspondientes al ciclo y a la proliferación celular hicieron parte de los genes inducidos como consecuencia del cultivo (
figura 1A).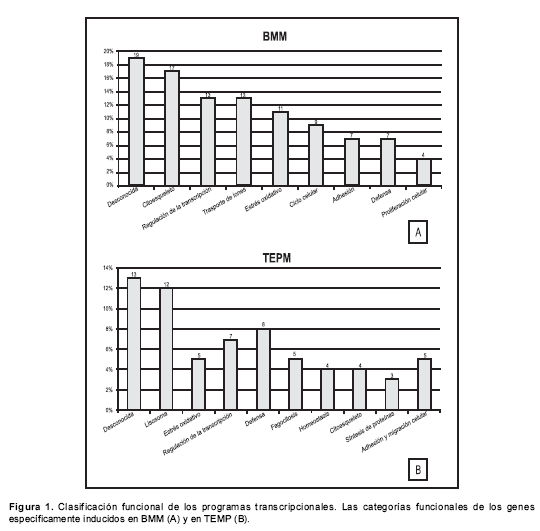
La principal categoría funcional encontrada en TEPM estuvo constituida por todo el repertorio de enzimas involucradas en la degradación de patógenos (
figura 1B). La expresión de estas moléculas se ha informado ampliamente para el caso de los macrófagos activados en la presencia de LPS, secuencias de ADN ricas en guanina y citosina u otros componentes bacterianos (19,20). Bajo estas condiciones de inflamación estas células son también capaces de sintetizar y liberar moléculas relacionadas con los procesos de destrucción de patógenos mediante los diferentes tipos de oxígeno, citocinas y compuestos involucrados en la presentación de antígenos descritos bajo las categorías de estrés oxidativo y defensa, respectivamente.La capacidad fagocítica de los macrófagos de la inflamación, también, se incrementa con el fin de habilitarlos para la ingestión de partículas que superan su tamaño (36). Los componentes del fagosoma correlacionados con el grupo funcional genes de la respuesta inflamatoria en macrófagos de fagocitosis se identificaron como inducidos en TEPM. Un número menos importante de genes encontrados forman parte de la categoría de homeostasis, citoesqueleto y migración celular, procesos que acompañan la función de estas células en la respuesta inflamatoria (37).
Las dos poblaciones de macrófagos que se estudiaron, compartieron una baja proporción de genes; la mayoría fueron marcadores extensamente caracterizados como los receptores para el complemento, inmunoglobulinas, lectinas y algunos componentes del fagosoma como la ferritina y la hemoglobina. Las principales diferencias encontradas en los perfiles de expresión de BMM y TEPM, se originaron en las condiciones de activación presentes en la respuesta inflamatoria. Sin embargo, otras variables que aportaron distinciones a estas dos poblaciones son las provenientes del efecto del cultivo in vitro de BMM y la adición del factor de crecimiento CSF-1, el cual se sabe que es capaz de inducir o reprimir genes de manera específica (38).
Se encuentran aún sin responder las preguntas de si el modelo BMM es el adecuado para el estudio de los macrófagos en reposo o en condiciones inflamatorias y si es aplicable en la extrapolación de los resultados al sistema in vivo. Otros estudios llevados a cabo por nosotros han revelado que muchos de los genes que parecen estar regulados positivamente en BMM, no se expresan en la misma proporción en las poblaciones de macrófagos in vivo (39).
Vale la pena resaltar que los datos del perfil de expresión de TEPM, obtenidos con microarreglos, se validaron con RT- PCR cuantitativa. Con base en los resultados de la validación, se seleccionaron genes que se clasificaron en cuatro grupos. El primer grupo está conformado por aquellos genes cuya expresión estuvo totalmente restringida a TEPM. En este grupo se encuentran los genes SEPP1, CTSD, PSAP y APOBEC1, todos ellos relacionados con el metabolismo. El segundo grupo corresponde a aquellos genes que, aunque son altamente expresados en TEPM, estaban presentes -aunque en baja cantidad- en fibroblastos (TIMP-2, LITAF y MFGE-8). Es interesante notar la función reguladora común de estos tres genes: el primero se relaciona con la actividad inhibidora de la destrucción de la matriz extracelular; el segundo con la represión de la expresión del TNFa (40), y el último con el reconocimiento y la fagocitosis de células apoptóticas con el fin de evitar la liberación de moléculas inmunogénicas (41).
El tercer grupo de genes estaba constituido por dos secuencias sin caracterizar (NIA, RIKEN), los cuales, al igual que los otros grupos mencionados anteriormente, presentan una alta expresión en TEPM aunque se encuentran presentes en los linfocitos.
Al analizar los perfiles de expresión de los 10 genes validados para TEPM cuando los BMM se trataron con LPS, se encontró que la mayoría de ellos son reprimidos. Este hallazgo sugiere y, a la vez, confirma no sólo la existencia de los diferentes estados del proceso de activación de los macrófagos sino también la diversidad de la respuesta inflamatoria. Es bien sabido que los macrófagos peritoneales de la respuesta inflamatoria no se encuentran completamente activados y que el fenotipo transcripcional de esta población debe diferir de aquélla estimulada con agentes bacterianos específicos como LPS. Lo que aún no está claro son los mecanismos por los cuales los macrófagos regulan sus estados de activación, ni la forma como las diversas poblaciones se interrelacionan y encuentran un equilibrio para controlar su función (42).
Al evaluar la utilidad de la información que se acaba de presentar, no se debe olvidar que los macrófagos se caracterizan por su alto grado de heterogeneidad entre y dentro de los diversos tejidos del organismo. Lo anterior promueve la formación de fenotipos funcionales y antigénicos, los cuales difieren en la producción de citocinas, la respuesta a los estímulos inmunomoduladores y destrucción de patógenos (43). Esta diversidad en las poblaciones de macrófagos dificulta aún más su estudio y la selección de un modelo particular para la caracterización de su papel en condiciones de salud o enfermedad.
Agradecimientos
Los autores agradecen a Carlos Andrés Chiriboga y a María Mercedes Posada su ayuda en la elaboración de este manuscrito.
Conflicto de interés
Los autores manifiestan no incurrir en ningún conflicto de interés durante la realización de esta revisión.
Financiación
Por parte del Macrophage Laboratory, Institute for Molecular Bioscience, the University of Queensland, Australia; el Posgrado Interfacultades de Microbiología, Universidad Nacional de Colombia; el Departamento de Farmacia, Facultad de Ciencias, Universidad Nacional y el Instituto de Biología Molecular, Universidad El Bosque.
Correspondencia:
Vera Ripoll, Macrophage Laboratory, Institute for Molecular Bioscience and CRC for Chronic Inflammatory Diseases, QLD Bioscience Precinct, 306 Carmody Road, The University of Queensland, St. Lucia, Brisbane Qld. 4072, Australia.
Phone: (61+ 7) 334 62075 y (61+ 7) 334 62351
Recibido: 08/07/04; aceptado: 14/04/05
Referencias
1. Gallin JI, Snyderman R. Inflammation: basic principles and clinical correlates. Third edition. Philadelphia, PA: Lippincott Williams & Wilkins; 1999 .p.1-5, 35-48. [ Links ]
2. Duffield JS. The inflammatory macrophage: a story of Jekyll and Hyde. Clin Sci 2003;104:27-38. [ Links ]
3. Doherty NS. Inflammation: mechanisms and therapeutic. Boston, MA: Basel, Birkhauser Verlag; 1995. p.1-21. [ Links ]
4. Mutch DM, Berger A, Mansourian R, Rytz A, Roberts MA. Microarray data analysis: a practical approach for selecting differentially expressed genes. Genome Biol 2001;2:1-26. [ Links ]
5. Heller RA, Schena M, Chai A, Shalon D, Bedilion T, Gilmore J et al. Discovery and analysis of inflammatory disease-related genes using cDNA microarrays. Proc Natl Acad Sci USA 1997;94:2150-5. [ Links ]
6. Lodish H, Berk A, Matsudaira P, Kaiser CA, Krieger M, Scout MP et al, editors. Molecular cell biology. Fifth edition. New York, NY: W. H. Freeman and Company; 2003. p.385-6. [ Links ]
7. Thornton S, Sowders D, Aronow B, Witte DP, Brunner HI, Giannini EH et al. DNA microarray analysis reveals novel gene expression profiles in collagen-induced arthritis. Clin Immunol 2002;105:155-68. [ Links ]
8. Yuyama N, Davies DE, Akaiwa M, Matsui K, Hamasaki Y, Suminami Y et al. Analysis of novel disease-related genes in bronchial asthma. Cytokine 2002;19:287-96. [ Links ]
9. Ehrt S, Schnappinger D, Bekiranov S, Drenkow J, Shi S, Gingeras TR et al. Reprogramming of the macrophage transcriptome in response to interferon-gamma and Mycobacterium tuberculosis: signaling roles of nitric oxide synthase-2 and phagocyte oxidase. J Exp Med 2001;194:1123-40. [ Links ]
10. Lang R, Patel D, Morris JJ, Rutschman RL, Murray PJ. Shaping gene expression in activated and resting primary macrophages by IL-10. J Immunol 2002;169: 2253-63. [ Links ]
11. Ravasi T, Wells C, Forest A, Underhill DM, Wainwright BJ, Aderem A et al. Generation of diversity in the innate immune system: macrophage heterogeneity arises from gene-autonomous transcriptional probability of individual inducible genes. J Immunol 2002;168:44-50. [ Links ]
12. Sweet MJ, Leung BP, Kang D, Sogaard M, Schulz K, Trajkovic V et al. A novel pathway regulating lipopolysaccharide-induced shock by ST2/T1 via inhibition of Toll-like receptor 4 expression. J Immunol 2001;166:6633-9. [ Links ]
13. Sweet MJ, Hume DA. Endotoxin signal transduction in macrophages. J Leukoc Biol 1996;60:8-26. [ Links ]
14. Beutler B, Cerami A. The biology of cachectin/TNF-a primary mediator of the host response. Annu Rev Immunol 1989;7:625-55. [ Links ]
15. Dinarello CA. The role of interleukin-1 in host responses to infectious diseases. Infect Agents Dis 1992;1:227-36. [ Links ]
16. Trinchieri G. Interleukin-12: a proinflammatory cytokine with immunoregulatory functions that bridge innate resistance and antigen-specific adaptive immunity. Annu Rev Immunol 1995;13:251-76. [ Links ]
17. Moore KW, O'Garra A, de Waal Malefyt R, Vieira P, Mosmann TR. Interleukin-10. Annu Rev Immunol 1993; 11:165-90. [ Links ]
18. Guha M, Mackman N. LPS induction of gene expression in human monocytes. Cell Signal 2001;13:85-94. [ Links ]
19. Wells CA, Ravasi T, Sultana R, Yagi K, Carninci P, Bono H et al. Continued discovery of transcriptional units expressed in cells of the mouse mononuclear phagocyte lineage. Genome Res 2003;13:1360-5. [ Links ]
20. Hume DA, Ross IL, Himes SR, Sasmono RT, Wells CA, Ravasi T. The mononuclear phagocyte system revisited. J Leukoc Biol 2002;72:621-7. [ Links ]
21. Hamerman JA, Aderem A. Functional transitions in macrophages during in vivo infection with Mycobacterium bovis bacillus Calmette-Guerin. J Immunol 2001;167:2227-33. [ Links ]
22. Nau GJ, Richmond JF, Schlesinger A, Jennings EG, Lander ES, Young RA. Human macrophage activation programs induced by bacterial pathogens. Proc Natl Acad Sci USA 2002;99:1503-8. [ Links ]
23. Boldrick JC, Alizadeh AA, Diehn M, Dudoit S, Liu CL, Belcher CE et al. Stereotyped and specific gene expression programs in human innate immune responses to bacteria. Proc Natl Acad Sci USA 2002;99: 972-7. [ Links ]
24. Ricciardi-Castagnoli P, Granucci F. Interpretation of the complexity of innate immune responses by functional genomics. Nat Rev Immunol 2002;2:881-9. [ Links ]
25. Rosenberger CM, Scott MG, Gold MR, Hancock RE, Finlay BB. Salmonella typhimurium infection and lipopolysaccharide stimulation induce similar changes in macrophage gene expression. J Immunol 2000;164: 5894-904. [ Links ]
26. Gil MP, Bohn E, O'Guin AK, Ramana CV, Levine B, Stark GR et al. Biologic consequences of Stat1-independent IFN signaling. Proc Natl Acad Sci USA 2001;98:6680-5. [ Links ]
27. de Veer MJ, Holko M, Frevel M, Walker E, Der S, Paranjape JM et al. Functional classification of interferon-stimulated genes identified using microarrays. J Leukoc Biol 2001;69:912-20. [ Links ]
28. Donnelly RP, Dickensheets H, Finbloom DS. The interleukin-10 signal transduction pathway and regulation of gene expression in mononuclear phagocytes. J Interferon Cytokine Res 1999;9:563-73. [ Links ]
29. Schottelius AJ, Mayo MW, Sartor RB, Baldwin AS Jr. Interleukin-10 signaling blocks inhibitor of kappaB kinase activity and nuclear factor kappaB DNA binding. J Biol Chem 1999;274:31868-74. [ Links ]
30. Kontoyiannis D, Kotlyarov A, Carballo E, Alexopoulou L, Blackshear PJ, Gaestel M et al. Interleukin-10 targets p38 MAPK to modulate ARE-dependent TNF mRNA translation and limit intestinal pathology. Embo J 2001;20:3760-70. [ Links ]
31. Bogdan C, Paik J, Vodovotz Y, Nathan C. Contrasting mechanisms for suppression of macrophage cytokine release by transforming growth factor-beta and interleukin-10. J Biol Chem 1992;267: 23301-8. [ Links ]
32. Kuwata H, Watanabe Y, Miyoshi H, Yamamoto M, Kaisho T, Takeda K et al. IL-10-inducible Bcl-3 negatively regulates LPS-induced TNFa production in macrophages. Blood 2003;102:4123-9. [ Links ]
33. Mosser DM. The many faces of macrophage activation. J Leukoc Biol 2003;73:209-12. [ Links ]
34. Ripoll V. Identificación de genes inflamatorios expresados específicamente en macrófagos usando microarreglos de cDNA (tesis). Bogotá: Universidad Nacional de Colombia; 2003. [ Links ]
35. Wells CA, Ravasi T, Faulkner GM, Carninci P, Okazaki Y, Hayashizaki Y et al. Genetic control of the innate immune response. BMC Immunol 2003;4:5. [ Links ]
36. Cox D, Lee DJ, Dale BM, Calafat J, Greenberg S. A Rab11-containing rapidly recycling compartment in macrophages that promotes phagocytosis. Proc Natl Acad Sci USA 2000;97:680-5. [ Links ]
37. Shurety W, Merino-Trigo A, Brown D, Hume DA, Stow JL. Localization and post-Golgi trafficking of tumor necrosis factor- alpha in macrophages. J Interferon Cytokine Res 2000;20:427-38. [ Links ]
38. Sweet MJ, Hume DA. CSF-1 as a regulator of macrophage activation and immune responses. Arch Immunol Ther Exp 2003;51:169-77. [ Links ]
39. Sestre DP, Beasley SJ, Sweet MJ, Fowles LF, Cronau SL, Stacey KJ et al. Bacterial/CpG DNA down-modulates colony stimulating factor-1 receptor surface expression on murine bone marrow-derived macro-phages with concomitant growth arrest and factor-independent survival. J Immunol 1999;163:6541-50. [ Links ]
40. Myokay F, Takashiba S, Lebo R, Amar S. A novel lipopolysaccharide-induced transcription factor regulating tumor necrosis factor alpha gene expression: molecular cloning, sequencing, characterization, and chromosomal assignment. Proc Natl Acad Sci USA 1999; 96:4518-23. [ Links ]
41. Hanayama R, Tanaka M, Miwa K, Shinohara A, Iwamatsu A, Nagata S. Identification of a factor that links apoptotic cells to phagocytes. Nature 2002;417: 182-7. [ Links ]
42. Gordon S. The role of the macrophage in immune regulation. Res Immunol 1998;149:685-8. [ Links ]
43. Rutherford MS, Witsell A, Schook LB. Mechanisms generating functionally heterogeneous macrophages: chaos revisited. J Leukoc Biol 1993;53:602-18. [ Links ]














