Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO  Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.25 n.2 Bogotá jun. 2005
Validación de la PCR en la detección de parásitos de Leishmania (Viannia) spp. En Lutzomyia (Diptera: Psychodidae) como herramienta en la definición de especies vectores
Erika Santamaría 1, Nubia Ponce 1, Concepción Puerta 2, Cristina Ferro 1
1 Laboratorio de Entomología, Subdirección de Investigación, Instituto Nacional de Salud, Bogotá. D. C., Colombia.
2
Laboratorio de Parasitología Molecular, Departamento de Microbiología, Facultad de Ciencias,Pontificia Universidad Javeriana, Bogotá, D. C., Colombia.
Lugar donde se realizó el trabajo: Laboratorio de Entomología, Instituto Nacional de Salud, Bogotá, D. C., Colombia.
Introducción. En leishmaniasis se acepta que la reacción en cadena de la polimerasa (PCR) ha simplificado el proceso de incriminación vectorial. Sin embargo, pocas veces se ha determinado la sensibilidad y la especificidad de cada PCR en la detección y la identificación del parásito en los flebótomos.
Objetivo. Evaluar la aplicabilidad de la reacción en cadena de la polimerasa (PCR), basada en los iniciadores B1 y B2, en la detección e identificación de parásitos de Leishmania (Viannia) en insectos vectores enteros sin disecar.
Metodología. Se determinó la sensibilidad y la especificidad de la PCR empleando diluciones de cultivo de parásitos de Leishmania spp. Se estableció el número máximo de hembras de Lutzomyia que pueden ser procesadas a la vez sin disminuir la sensibilidad de la PCR, procesando el ADN de grupos de una a cinco hembras de Lutzomyia en presencia del ADN de las diluciones de cultivo de parásitos. Además, se comparó en grupos de flebótomos infectados experimentalmente, la sensibilidad de esta PCR en la detección de infección por Leishmania (Viannia) frente al método de búsqueda de flagelados por disección del insecto y examen microscópico.
Resultados. La PCR detectó desde un parásito de Leishmania (Viannia) y permitió el procesamiento de hasta tres insectos enteros sin alterar la sensibilidad. Los porcentajes de infección experimental detectados con las dos técnicas fueron similares, 33,3% con la PCR y 30% con el examen microscópico. Además, se confirmó que los iniciadores B1 y B2 son específicos para especies del subgénero Leishmania (Viannia).
Conclusión. Los resultados obtenidos demuestran la sensibilidad y la especificidad de esta PCR y permiten recomendar su uso en la determinación de infección natural con parásitos de Leishmania (Viannia) en poblaciones silvestres de flebótomos.
Palabras clave: Lutzomyia, Leishmania (Viannia), PCR, sensibilidad, especificidad.
Validation of PCR as a tool for the detection of Leishmania (Vianna) spp. parasites in the Lutzomyia (Diptera: Psychodidae) vector
Introduction. The applicability of the polymerase chain reaction (PCR) was evaluated for the detection and identification of parasites in sand fly vectors and thereby precluding the necessity of dissecting them. DNA was extracted from individual, laboratory infected sand flies, and subjected to PCR amplification using specific B1 and B2 primers for parasites of the Leishmania (Viannia) subgenus.
Methodology. The sensitivity and specificity of the PCR primers were defined by means of serial dilutions of a Leishmania culture. Pooled samples of 1 to 5 sandflies were examined in association with the parasite dilutions to determine the point at which sensitivity became reduced.
Experimentally infected sand flies were used to compare the sensitivity of the PCR with sand fly dissection and searching for flagellated parasites by microscopic examination.
Results. As few as a single parasite was detected, and the sensitivity remained unaltered up to 3 female sand flies per pool. Detection rates were 33% for the traditional technique and 33.3% for PCR. The B1 and B2 primers were confirmed as specific for Leishmania (Viannia) parasites.
Conclusion. The demonstrably high sensitivity and specificity of PCR warrant the use of PCR in assessing natural infection rates of Leishmania (Viannia) in field populations of sand fly vectors.
Key words: Leishmania (Viannia), PCR, Lutzomyia , phlebotomine sand flies.
En Colombia, se han registrado hasta el momento 133 especies del género Lutzomyia (1-4), de las cuales seis especies: L. evansi, L. longipalpis, L. trapidoi, L. hartmanni, L. spinicrassa y L. umbratilis son consideradas como vectores (1), ya que cumplen con los criterios de incriminación sugeridos por Killick-Kendrick (5) como son: abundancia en el lugar de transmisión, antropofilia, competencia vectorial y aislamiento de la misma especie de Leishmania encontrada en pacientes.
Recientemente, la correlación estadística entre la abundancia y la distribución de los flebótomos y la distribución y la incidencia de los casos se constituye en otro criterio para sumar evidencia epidemiológica en vectores sospechosos (6-8); en esta categoría están incluidas especies como L. longiflocosa, L. gomezi, L. quasitownsendi, L. youngi, L. columbiana, L. torvida, L. yuilli y L. panamensis, en las cuales el único criterio que falta por comprobar es el hallazgo e identificación en ejemplares silvestres de la misma especie de parásito aislada de pacientes en cada zona específica.
El método empleado tradicionalmente para valorar la infección de los flebótomos consiste en aislar y caracterizar parásitos de Leishmania a partir de vectores provenientes de zonas endémicas. Este método se desarrolla sin apoyo de la biología molecular y, para ello, se realiza un congelamiento progresivo de los ejemplares recolectados para mantener al parásito vivo, seguido de la disección del tracto digestivo de cada hembra y la búsqueda de flagelados al microscopio.
En el caso de lograr visualizar los flagelados, se transfiere el tracto digestivo a un medio de cultivo o se inocula en animales de laboratorio para la caracterización posterior del parásito (9,10). Este es un procedimiento dispendioso que involucra la disección de miles de hembras, ya que en el Nuevo Mundo los porcentajes de infección son muy bajos (11). Además, el aislamiento y el cultivo de los parásitos a partir de los intestinos infectados del vector se ve afectado frecuentemente por la contaminación, lo cual obstaculiza la identificación de la especie (12).
En las últimas dos décadas, la PCR se ha constituido en una técnica alternativa, altamente sensible y específica, que ha permitido identificar diferentes especies de Leishmania a partir de muestras de pacientes (13,14), reservorios (15,16) y vectores (17,18).
Teniendo en cuenta que en el control de las leishmaniasis es de vital importancia conocer la especie o especies de Lutzomyia involucradas en la transmisión en cada foco específico, para diseñar programas de control basados en la biología y el comportamiento de estas especies; el objetivo general del presente estudio fue valorar el uso de la PCR basada en los iniciadores B1 y B2, para la detección de parásitos de Leishmania en grupos de insectos del género Lutzomyia sin disecar.
Materiales y métodos
Parásitos
Para los ensayos de sensibilidad y como controles positivos y de especificidad se trabajó con formas promastigotes de las siguientes cepas de referencia del Laboratorio de Parasitología del Instituto Nacional de Salud de Colombia: Leishmania (Viannia) panamensis (MHOM/CO/87/CL-412), L. (V.) braziliensis (MHOM/CO/86/CL- 250), L. (Leishmania) mexicana amazonensis (MHOM/ME/94/CL-856) y L. (L.) chagasi (MHOM/CO/84/CL-044B). Así mismo, se utilizaron formas epimastigotas de cepas de referencia de Trypanosoma cruzi (MHOM/CO/92/FCh) y T. rangeli (MHOM/CO/99/99/5048). Los parásitos se cultivaron en masa según lo descrito por Duque et al. (19) a una concentración promedio de 1 x 106 parásitos por ml.
Vectores
Se trabajó con grupos de hembras de L. ovallesi, L. serrana y L. longipalpis, provenientes de colonias del Laboratorio de Entomología del INS, establecidas y mantenidas según la metodología de Modi y Tesh (20) y Ferro et al. (21).
Extracción del ADN
El ADN de los insectos se obtuvo de acuerdo con el protocolo estandarizado por Cabrera et al. (22), el cual consiste en la maceración de los insectos individuales o en grupos almacenados en alcohol al 70%, seguido de centrifugación a 10.000 rpm por 3 minutos, eliminación del sobrenadante, adición de 100 ml de Chelex 100® (Bio Rad) al 5%, incubación a 56°C por 30 min, agitación en vórtex por 10 segundos, incubación a temperatura de ebullición por 8 minutos, agitación en vórtex por 10 segundos y centrifugación a 10.000 rpm por 3 minutos. El sobrenadante así obtenido fue recuperado y almacenado a -20°C hasta ser usado como plantilla para la PCR.
Para la extracción del ADN de los parásitos, los cultivos se lavaron en solución salina; posteriormente, los parásitos se recolectaron por centrifugación, se eliminó el sobrenadante, se adicionaron 100 ml de Chelex 100® y se siguieron los demás pasos descritos para la obtención del ADN de insectos.
Amplificación del ADN
Se eligieron los iniciadores B1 (5'-GGG GTT GGT GTA ATA TAG TGG-3') y B2 (5'-CTA ATT GTG CAC GGG GAG G-3'), diseñados a partir de la región conservada de los minicírculos de ADN del cinetoplasto de parásitos de Leishmania y reportados como específicos en la detección de las especies del subgénero Viannia produciendo una banda de aproximadamente 750 pb (23). Dos microlitros (2 µl) del ADN extraído en cada ensayo se amplificaron en una mezcla de reacción que contenía 1,5 mM de MgCl, 100 pmol de cada iniciador, 0,2 mM de cada dNTP, 1X de tampón A (KCl 50 mM, 10 mM Tris-HCl) y 2,5 unidades de Taq polimerasa (Promega) en un volumen final de 50 µl. Como control positivo de la reacción se utilizó ADN desnudo de L. panamensis; como control negativo de la reacción se utilizó solución salina en reemplazo del ADN, y como control de especificidad se utilizó ADN de T. cruzi. Así mismo, las muestras negativas se evaluaron para la presencia de inhibidores de la reacción de PCR mediante la adición de ADN de L. (V.) panamensis, seguido de la prueba de PCR.
La amplificación se hizo en un termociclador Perkin-Elmer 2400® con la siguiente rutina térmica: predesnaturación a 94°C por 2 minutos, 35 ciclos con el siguiente perfil: denaturación a 93°C por 30 segundos, anillaje a 67, 5°C por 1 minuto y extensión a 72°C por 1 minuto, seguidos de una extensión final a 72°C durante 7 minutos. Los productos de amplificación obtenidos se mantuvieron a 4°C hasta su análisis.
Análisis de los productos de amplificación
Seis microlitros (6 µl) de las muestras de ADN amplificadas se analizaron mediante electroforesis horizontal en geles de agarosa (NuSieve: SeaKem, 3:1) al 2%, utilizando TBE 1X ( 89 mM Tris HCl, 90 mM de ácido bórico y 20 mM de EDTA, pH 8,3) como tampón de corrido. Los geles se tiñeron con 0,1 ug/ml de bromuro de etidio (Sigma). Como control del corrido electroforético se utilizó el marcador de peso molecular ADN 100 pb (Promega). Los productos se visualizaron en un transiluminador de luz ultravioleta.
Determinación de la sensibilidad de la PCR
Para determinar la concentración mínima de ADN de parásito que puede detectar la PCR basada en los iniciadores B1 y B2, se utilizaron cultivos de promastigotes de L.(V.) panamensis. Después de recolectar los parásitos por centrifugación, se lavaron tres veces en solución salina, se determinó su concentración en cámara de Newbauer y se realizaron siete diluciones seriadas en base 10 conteniendo un estimado de 106, 105, 104, 103 102, 10 y 1 parásitos, en un volumen final de 100 µl de solución salina. Estas diluciones se sometieron a los procesos de extracción de ADN, amplificación y análisis descritos previamente.
Determinación de la especificidad de la PCR
La especificidad de los iniciadores B1 y B2 se determinó aplicando el mismo protocolo al ADN desnudo de L. (V.) panamensis y L. (V.) braziliensis y al ADN de especies de Leishmania del subgénero Leishmania como L. (L.) mexicana amazonensis y L. (L.) chagasi y de otras dos especies de la familia Trypanosomatidae: T. cruzi y T. rangeli.
Validación de la PCR en flebótomos infectados experimentalmente con Leishmania
El primer objetivo en la validación de la PCR en flebótomos infectados experimentalmente con Leishmania fue probar que la PCR basada en los iniciadores B1 y B2 permitía detectar parásitos de Leishmania en hembras de Lutzomyia enteras sin disecar. Para ello, se infectaron experimental-mente con L.(V.) panamensis un grupo de hembras de 2 a 4 días de edad de la especie L. serrana, la cual es susceptible a la infección con L. (V.) panamensis (datos sin publicar, Laboratorio de Entomología, INS). Para la infección, estos insectos fueron expuestos al hocico de un hámster anestesiado e inoculado 2 meses antes con promastigotes de L. (V.) panamensis (24,25). Entre 6 y 8 días después de la alimentación sobre el hámster infectado, las hembras se preservaron individualmente en alcohol al 70% y se almacenaron a temperatura ambiente. Posteriormente, se realizó la extracción del ADN, seguida de la reacción de PCR y la visualización de los productos de amplificación, tal como se describe previamente.
El segundo objetivo fue comparar el método tradicional frente a la PCR en la detección de infección por Leishmania en grupos de flebótomos infectados de manera experimental. Para esto, se infectó con L. (V.) panamensis un grupo de 100 hembras, aproximadamente, de 2 a 4 días de edad de L. longipalpis, la cual es susceptible a la infección con L. (V.) panamensis (datos sin publicar, Laboratorio de Entomología, INS).
La infección se realizó siguiendo el método descrito por Tesh y Modi (26), el cual consistió en exponer los insectos a un recipiente con membrana que contenía una mezcla de cultivo de promastigotes de Leishmania a una concentración de 1,19 x 107 parásitos/ml y eritrocitos humanos lavados en una proporción de 2:1. Las hembras alimentadas se mantuvieron a 24°C, 80% de humedad relativa y a una dieta de agua y solución azucarada. Entre 7 y 8 días después de la comida infectiva, las hembras que sobrevivieron se dividieron aleatoriamente en dos grupos de 30 hembras cada uno. En el primer grupo, el porcentaje de infección se estableció por el método tradicional, para lo cual se realizó la disección del intestino de cada hembra en una gota de solución salina y la presencia de promastigotes se detectó bajo el microscopio a 40X. En el segundo grupo, las hembras se almacenaron individualmente, sin disecar, en alcohol al 70% y se procesaron por PCR de acuerdo con el protocolo descrito, determinando así el porcentaje de infección experimental en este grupo.
Determinación del número de ejemplares de Lutzomyia que pueden ser procesados a la vez sin disminuir la sensibilidad
Para determinar el número máximo de hembras de Lutzomyia que podían ser procesadas sin disminuir la sensibilidad de la PCR, se realizaron extracciones del ADN de grupos de 1, 2, 3, 4 y 5 hembras de L. ovallesi. El ADN de cada grupo se amplificó en presencia del ADN extraído de cada una de las diluciones de L. (V.) panamensis, que contenían teóricamente desde 106 hasta 1 parásito. Como control negativo se usó ADN extraído de 1 hembra de L. ovallesi sin adición de ADN de parásitos. Los productos de amplificación de cada grupo de flebótomos se visualizaron y se compararon entre sí.
Resultados
En cuanto a la sensibilidad de la PCR basada en los iniciadores B1 y B2, en el rango de concentraciones ensayadas, el producto de amplificación característico de 750 pb para el subgénero Leishmania (Viannia) se observó en todas las diluciones, incluso, en la que contenía un estimado de 1 parásito (
figura 1).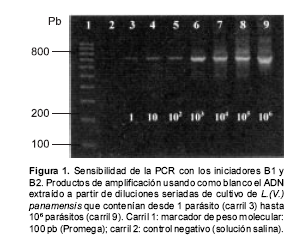
En cuanto a la especificidad de esta PCR, los iniciadores para el subgénero Leishmania (Viannia) B1 y B2 resultaron específicos para las especies de Leishmania pertenecientes a este subgénero, tal y como se había reportado por sus autores (23) y no se observaron productos de amplificación en especies del subgénero Leishmania (Leishmania), ni en especies del género Trypanosoma.
En la infección experimental de hembras de L. serrana enteras sin disecar, se obtuvo el producto de amplificación esperado en 8 de las 13 hembras que sobrevivieron al día séptimo y octavo después de la infección, lo cual representa un porcentaje de infección de 61,5%. De esta forma, se pudo comprobar que la PCR, bajo las condiciones descritas, puede detectar parásitos que se encuentran dentro del intestino del vector y que no es necesario disecar el ejemplar y que se puede procesar únicamente el intestino.
En la comparación de los dos métodos de detección de la infección por Leishmania en grupos de hembras experimentalmente infectadas de L. longipalpis, se observó que con la disección de cada hembra y la visualización de los flagelados al microscopio, se detectó la infección por Leishmania en 9 de 30 hembras, lo cual corresponde a un porcentaje de infección de 30% y empleando la PCR, basada en los iniciadores B1 y B2, se obtuvo la banda de amplificación esperada en 10 de 30 hembras procesadas, lo cual corresponde a un porcentaje de infección de 33,3%.
Respecto al número máximo de hembras de Lutzomyia que pueden ser procesadas al tiempo sin disminuir la sensibilidad de la técnica, se observó que la sensibilidad de la PCR en la detección de parásitos de Leishmania (Viannia) se mantiene cuando se procesan grupos de 1, 2 y hasta 3 hembras de Lutzomyia. Es decir, que al procesar hasta 3 hembras en el mismo tubo se puede detectar el ADN de 1 parásito en adelante. Por su parte, cuando se ensayó con el ADN extraído de 4 y 5 hembras, el número mínimo de parásitos detectado fue de 10 y 100 parásitos, respectivamente (
cuadro 1).
Discusión
La PCR basada en los iniciadores B1 y B2 se ha utilizado con éxito en la detección de parásitos de Leishmania (Viannia) en diferentes estudios epidemiológicos, especialmente, para el diagnóstico de leishmaniasis en pacientes (27,13). En el presente estudio se valoró el potencial de esta técnica en la detección de infección por Leishmania (Viannia) en flebótomos, con el fin de aplicar esta PCR en futuros estudios de infección natural en poblaciones silvestres de estos insectos.
En cuanto a la sensibilidad de la PCR empleando extracción con Chelex 100® y los iniciadores B1 y B2, teóricamente y según lo reportado por De Bruijn y Barker (23), el protocolo puede detectar hasta menos de un parásito e, incluso, lo correspondiente a 1.000 minicírculos del cinetoplasto de un parásito sería suficiente como plantilla para la amplificación. En este trabajo, el ADN extraído de una dilución con un estimado de un parásito de L. (V.) panamensis fue suficiente para obtener el producto de amplificación esperado, lo que confirma que la técnica es altamente sensible. En cuanto a la especificidad, los iniciadores B1 y B2 resultaron específicos para las especies del subgénero Viannia y no se observó ningún producto de amplificación en especies del subgénero Leishmania ni en otros cinetoplástidos como T. cruzi o T. rangeli.
Por otra parte, se valoró la eficiencia de la PCR en la determinación de porcentajes de infección experimental en flebótomos, comparada con la técnica tradicional. En este experimento, se observó que los porcentajes de infección obtenidos por disección y examen al microscopio comparado con la PCR son muy cercanos. El único reporte de un experimento similar lo presenta Michalsky et al. (28). Estos investigadores realizaron infecciones experimentales con tres combinaciones Lutzomyia-Leishmania: L. longipalpis con L. chagasi, L. migonei con L. amazonensis y L. migonei con L. braziliensis. El porcentaje de infección obtenido por disección y examen al microscopio para cada combinación fue de 70%, 40% y 50%, respectivamente, y el porcentaje de infección obtenido aplicando la PCR fue de 87%, 33% y 40%, respectivamente.
En el presente estudio se obtuvieron porcentajes de infección del 31,4%, empleando la técnica convencional y del 33,3% con la PCR. Esto indica que la PCR es tan eficiente como la disección y visualización de los flagelados al microscopio y que puede aplicarse con seguridad en estudios epidemiológicos de infección natural con Leishmania en poblaciones silvestres de flebótomos. Los bajos porcentajes de infección experimental obtenidos en nuestro estudio pueden atribuirse a que en la naturaleza L. longipalpis no es vector de L. (V.) panamensis, en cambio cuando se utiliza para la infección una combinación vector-parásito que se presente en la naturaleza la susceptibilidad del insecto al parásito es mayor y los porcentajes de infección experimental son mayores (29,30).
Otro paso en la valoración de la técnica fue medir la sensibilidad de la PCR en la detección de parásitos de Leishmania (Viannia) al adicionar a las diluciones de parásitos, ADN de uno a cinco insectos, encontrando que el ADN de hasta tres insectos no interfería en la sensibilidad. Es bien conocido que la presencia de ADN no específico de las células del insecto y otras sustancias presentes pueden ejercer un efecto de inhibición sobre la actividad de la Taq polimerasa y afectar la sensibilidad de la PCR (31).
Por otro lado, la capacidad de detección de los iniciadores puede disminuir debido a una dificultad en el acceso al ADN de la red de minicírculos del cinetoplasto (32). Por esta razón, en la mayoría de estudios recientes sobre infección natural con Leishmania en poblaciones de flebótomos, se diseca primero el ejemplar y se extrae su tracto digestivo y en caso de presentar flagelados se emplea la PCR para verificar que los flagelados son de Leishmania y clasificarlos taxonómica-mente (8,17,18), evitando así la inhibición por exceso de ADN no específico del insecto.
En otros estudios (32, 33) se han procesado para búsqueda de infección natural con Leishmania, hembras enteras de flebótomos sin disecar y de manera individual. Ambas situaciones aumentan la dificultad, la duración y el costo del proceso, debido a que en los estudios de infección natural en insectos, generalmente, se procesan cientos o miles de ejemplares.
Específicamente con los iniciadores B1 y B2 (23), no se había medido antes la cantidad de ejemplares que podían ser procesados al tiempo sin disminuir la sensibilidad de la técnica. Solamente existen reportes de Michalsky et al. (28), quienes procesaron flebótomos individuales enteros sin disección y aplicando esta PCR, obtuvieron la banda esperada. Así mismo, Torrez et al. (34) procesaron grupos de hasta 25 hembras y detectaron infección en el 2,2% de los grupos. Dado que en el presente estudio el ADN de 4 y 5 hembras procesadas al tiempo interfirió con la sensibilidad de la PCR, en el trabajo mencionado de Torrez et al. (34) no se puede descartar la posibilidad de que se haya presentado inhibición en algunos de los grupos procesados por la gran cantidad de hembras analizadas al tiempo.
La ventaja de la PCR aquí validada es que permite la detección e identificación del parásito en grupos de hasta tres hembras de Lutzomyia sin disecar, lo que implica una considerable reducción de tiempo y esfuerzo cuando se compara con la disección manual de cada hembra y su examen microscópico y, principalmente, facilita la identificación del parásito ya que el aislamiento de los flagelados a partir de intestinos infectados era pocas veces exitoso.
Entre las limitaciones de esta PCR frente a la técnica de disección están la imposibilidad de obtener cultivos del parásito y el no poder determinar la ubicación de la infección en el intestino del vector. Otra limitación es el número de hembras que pueden ser procesadas a la vez manteniendo una alta sensibilidad. El protocolo aquí descrito ofrece un nivel de detección de 1 parásito cuando se procesan hasta 3 hembras enteras de Lutzomyia. Sin embargo, los investigadores interesados en aplicar esta técnica, pueden elegir el límite de detección de 10 o 100 parásitos y procesar a la vez 4 o 5 hembras, teniendo en cuenta que, generalmente, una especie de Lutzomyia considerada como vector permitirá el desarrollo de los parásitos en un número superior a 100.
Es posible que al comparar otros protocolos de extracción y purificación del ADN, empleando una PCR anidada o hibridación con sondas de ADN específicas para el subgénero L. (V.) Viannia, se puedan procesar simultáneamente grupos más numerosos de hembras. No obstante, si se tiene en cuenta que es frecuente una alta diversidad de especies de Lutzomyia en los focos de transmisión de Leishmania y que es necesario identificar la especie por características externas e internas, consideramos que 3 hembras es un número adecuado cuando se pretende determinar infección natural con Leishmania. En los focos en los cuales predomina con un alto porcentaje de abundancia una sola especie de Lutzomyia es más sencillo procesar grupos más numerosos.
La técnica de PCR descrita en este trabajo es utilizada cuando se desea definir la especie o especies de Lutzomyia que presentan infección con parásitos de Leishmania (Viannia). Sin embargo, debido a que en el Nuevo Mundo los porcentajes de infección natural con Leishmania en los vectores son muy bajos (entre el 0,1 y el 1,9%) (11), se asume que al resultar positivo para Leishmania un grupo de tres hembras, la probabilidad de que más de una hembra estuviera infectada es muy baja. Para el cálculo exacto del porcentaje de infección natural lo más adecuado sería extraer el ADN de las hembras individualmente y mezclar el ADN de varias hembras en grupos; posteriormente, procesar estos grupos por PCR y al obtener un grupo positivo, correr de nuevo el ADN de cada hembra perteneciente a ese grupo (35).
Se concluye que la PCR con los iniciadores B1 y B2, bajo las condiciones descritas, fue un método sensible y específico en la detección de parásitos de Leishmania (Viannia) en flebótomos y puede recomendarse su uso en la determinación de infección natural en poblaciones silvestres de especies de Lutzomyia spp. como herramienta en la incriminación de especies vectores.
Conflicto de interés
No hay conflicto de interés.
Agradecimientos
A Martha Ayala y Marleny Montilla del Laboratorio de Parasitología del Instituto Nacional de Salud por el suministro de cepas de referencia de Leishmania spp. y Trypanosoma spp, y a Marco Fidel Suárez por su ayuda en el mantenimiento de las colonias de Lutzomyia spp.
Financiación
Instituto Nacional de Salud y Colciencias, proyecto código 2104-04-12677.
Correspondencia:
Cristina Ferro, Laboratorio de Entomología, Instituto Nacional de Salud, Avenida Calle 26 N. 51-60, Bogotá, D.C., Colombia.
telefax: 2200923, Bogotá, Colombia. mferro@ins.gov.co.
Recibido: 13/10/04; aceptado: 28/03/05
Referencias
1. Montoya J, Ferro C. Flebótomos (Diptera: Psychodidae) de Colombia. En: Amat G, Andrade MG, Fernández F, editores. Insectos de Colombia. Volumen II. Colección Jorge Alvarez Lleras No. 13. Academia Colombiana de Ciencias Exactas, Físicas y Naturales. Santafé de Bogotá: Centro Editorial Javeriano; 1999. p.211-45. [ Links ]
2. Wolff M, Galati EA. Description of Pintomyia limafalcaoae and Pintomyia antioquiensis, two new species of phlebotomine sand fly (Diptera, Psychodidae) from the Colombian Andes. Mem Inst Oswaldo Cruz 2002;97:317-24. [ Links ]
3. Barreto M, Burbano ME, Young DG. Description of Lutzomyia (Trichophoromyia) pabloi n. sp. and the female of L. howardi (Diptera: Psychodidae) from Colombia. J Med Entomol 2002;39:601-4. [ Links ]
4. Bejarano EE, Duque P, Vélez ID. Taxonomy and distribution of the series pia of the Lutzomyia verrucarum group (Diptera: Psychodidae), with a description of Lutzomyia emberai n. sp. J Med Entomol 2004;41:833-41. [ Links ]
5. Killick-Kendrick R. Phlebotomus vectors of the leishmaniasis: a review. Med Vet Entomol 1990;4:1-24. [ Links ]
6. Davies CR, Llanos-Cuentas EA, Campos P, Monge J, Villaseca P, Dye C. Cutaneous leishmaniasis in the Peruvian Andes: risk factors identified from a village cohort study. Am J Trop Med Hyg 1997;56:85-95. [ Links ]
7. Dye C. The analysis of parasite transmission by bloodsucking insects. Annu Rev Entomol 1992;37:1-19. [ Links ]
8. Muñoz G. The sandfly vectors and epidemiology of cutaneous leishmaniasis in the Landazuri focus, Colombia (tesis). London: University of London; 1998. [ Links ]
9. Young DG, Morales A, Kreutzer RD, Alexander JB, Corredor A, Tesh RB et al. Isolations of Leishmania braziliensis (Kinetoplastida: Trypanosomatidae) from cryopreseved Colombian sand flies (Diptera: Psychodidae). J Med Entomol 1987;24:587-9. [ Links ]
10. Kreutzer RD, Corredor A, Grimaldi G Jr, Grogl M, Rowton ED, Young DG et al. Characterization of Leishmania colombiensis sp.n. (Kinetoplastida: Trypanosomatidae), a new parasite infecting humans, animals, and phlebotomine sand flies in Colombia and Panama. Am J Trop Med Hyg 1991;44:662-75. [ Links ]
11. Travi BL, Montoya J, Solarte Y, Lozano L, Jaramillo C. Leishmaniasis in Colombia. I. Studies on the phlebotomine fauna associated with endemic foci in the Pacific Coast region. Am J Trop Med Hyg 1988;39: 261-6. [ Links ]
12. Rowton ED, De Mata M, Rizzo N, Porter CH, Navin TR. Isolation of Leishmania braziliensis from Lutzomyia ovallesi (Diptera: Psychodidae) in Guatemala. Am J Trop Med Hyg 1992;46:465-8. [ Links ]
13. Isaza DM, Arboleda M, Restrepo M, McCann SH, Barker DC. Validation of the polymerase chain reaction for the diagnosis of human cutaneous leishmaniasis in north-west Colombia. Trans R Soc Trop Med Hyg 2002; 96(Suppl.1):165-8. [ Links ]
14. Volpini AC, Passos VM, Oliveira GC, Romanha AJ. PCR-RFLP to identify Leishmania (Viannia) braziliensis and L. (Leishmania) amazonensis causing American cutaneous leishmaniasis. Acta Trop 2004;90:31-7. [ Links ]
15. Reithinger R, Lambson BE, Barker DC, Davies CR. Use of PCR to detect Leishmania (Viannia) spp. in dog blood and bone marrow. J Clin Microbiol 2000; 38:748-51. [ Links ]
16. Alexander B, Lozano C, Barker DC, McCann SH, Adler GH. Detection of Leishmania (Viannia) braziliensis complex in wild mammals from Colombian coffee plantations by PCR and DNA hybridization. Acta Trop 1998;69:41-50. [ Links ]
17. Feliciangeli MD, Rodriguez N, Bravo A, Arias F, Guzman B. Vectors of cutaneous leishmaniasis in north-central Venezuela. Med Vet Entomol 1994;8:317-24. [ Links ]
18. Rodríguez N, Aguilar CM, Barrios A, Barker DC. Detection of Leishmania braziliensis in naturally infected individual sand flies by the polymerase chain reaction. Trans R Soc Trop Med Hyg 1999;93:47-9. [ Links ]
19. Duque S, Peláez D, Corredor A. Normas para cultivo in vitro de parásitos de la familia trypanosomatidae. Manual de procedimientos. Bogotá: Instituto Nacional de Salud; 1993. [ Links ]
20. Modi GB, Tesh RB. A simple technique for mass rearing Lutzomyia longipalpis and Phlebotomus papatasi (Diptera: Psychodidae) in the laboratory. J Med Entomol 1983;20:568-9. [ Links ]
21. Ferro C, Cárdenas E, Corredor D, Morales A, Munstermann LE. Life cycle and fecundity analysis of Lutzomyia shannoni (Dyar) (Diptera: Psychodidae). Mem Inst Oswaldo Cruz 1998;93:195-9. [ Links ]
22. Cabrera OL, Munstermann LE, Cárdenas R, Gutiérrez R, Ferro C. Definición de las condiciones de temperatura y almacenamiento adecuadas en la detección de ADN de Leishmania por PCR en flebotominos. Biomédica 2002;22:296-302. [ Links ]
23. De Bruijn MH, Barker DC. Diagnosis of New World leishmaniasis: specific detection of species of the Leishmania braziliensis complex by amplification of kinetoplast DNA. Acta Trop 1992;52:45-58. [ Links ]
24. Osorio Y, Melby PC, Pirmez C, Chandrasekar B,Guarin N, Travi BL. The site of cutaneous infection influences the immunological response and clinical outcome of hamsters infected with Leishmania panamensis. Parasite Immunol 2003;25:139-48. [ Links ]
25. Travi BL, Osorio Y, Melby PC, Chandrasekar B, Arteaga L, Saravia NG. Gender is a major determinant of the clinical evolution and immune response in hamsters infected with Leishmania spp. Infect Immun 2002;70:2288-96. [ Links ]
26. Tesh RB, Modi GB. A simple method for experimental infection of phlebotomine sand flies with Leishmania. Am J Trop Med Hyg 1984;33:41-6. [ Links ]
27. De Bruijn MH, Labrada LA, Smyth AJ, Santrich C, Barker DC. A comparative study of diagnosis by the polymerase chain reaction and by current clinical methods using biopses from Colombian patients with suspected leishmaniasis. Trop Med Parasitol 1993;44: 201-7. [ Links ]
28. Michalsky EM, Fortes-Dias CL, Pimienta PF, Secundino NF, Dias ES. Assessment of PCR in the detection of Leishmania spp. in experimentally infected individual phlebotomine sandflies (Diptera: Psychodidae: Phlebotominae). Rev Inst Med Trop Sao Paulo 2002;44: 255-9. [ Links ]
29. Santamaría E, Castillo M, Cárdenas R, Bello F, Ayala M, Ferro C. Transmisión experimental de Leishmania braziliensis a hámster por picadura de Lutzomyia longiflocosa (Diptera: Psychodidae) provenientes de un foco endémico en la zona cafetera colombiana. Medicas UIS 1998;12:279-84. [ Links ]
30. Santamaría E, Castillo M, Cárdenas R, Bello F, Ayala M, Ferro C. Competencia vectorial de las especies de Lutzomyia del grupo verrucarum (Diptera: Psychodidae) en un foco endémico de Leishmania braziliensis en Reventones, Cundinamarca. Biomédica 1999;19:115-26. [ Links ]
31. Rodgers MR, Popper SJ, Wirth DF. Amplification of kinetoplast DNA as a tool in the detection and diagnosis of Leishmania. Exp Parasitol 1990;71:267-75. [ Links ]
32. Aransay AM, Scoulica E, Tselentis Y. Detection and identification of Leishmania DNA within naturally infected sand flies by seminested PCR on minicircle kinetoplastic DNA. Appl Environ Microbiol 2000;66:1933-8. [ Links ]
33. Mukherjee S, Hassan MQ, Gosh A, Gosh KN, Bhattacharya A, Adhya S. Leishmania DNA in Phlebotomus and Sergentomyia species during a kala-azar epidemic. Am J Trop Med Hyg 1997;57:423-5. [ Links ]
34. Torrez M, López M, Le Pont F, Martinez E, Muñoz M, Hervas D et al. Lutzomyia nuñeztovari anglesi (Diptera: Psychodidae) as a probable vector of Leishmania braziliensis in the Yungas, Bolivia. Acta Trop 1998;71:311-6. [ Links ]
35. Pérez JE, Ogusuku E, Inga R, López M, Monje J, Paz L et al. Natural Leishmania infection of Lutzomyia spp. in Peru. Trans Roy Soc Trop Med Hyg 1994;88: 161-4. [ Links ]














