Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO  Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.25 no.4 Bogotá Dec. 2005
Diversidad genética de Plasmodium falciparum y sus
implicaciones en la epidemiología de la malaria
Judy Natalia Jiménez 1, 2, Carlos Enrique Muskus 1, Iván Darío Vélez 1
1 Programa de Estudio y Control de Enfermedades Tropicales (PECET), Medellín, Colombia.
2
Escuela de Bacteriología y Laboratorio Clínico, Universidad de Antioquia, Medellín, Colombia.La diversidad genética le confiere a Plasmodium falciparum la capacidad de evadir la respuesta inmune del hospedero y producir variantes resistentes a medicamentos y a vacunas, aspectos que juegan un papel importante en el establecimiento de medidas de control contra la malaria.
Diferentes autores han documentado la existencia de diversas cepas o clones de P. falciparum, cuya diversidad genética se ha confirmado a través de distintos ensayos de PCR (reacción en cadena de la polimerasa). Numerosas investigaciones realizadas en poblaciones con diferente grado de transmisión de malaria han mostrado la relación existente entre la estructura de la población de P. falciparum y la epidemiología de la enfermedad. En este artículo se describen las fases del ciclo de vida en las que los eventos de recombinación originan la diversidad genética de P. falciparum, se revisan los estudios realizados sobre este aspecto en regiones con diferentes grados de endemicidad, así como sobre sus implicaciones en la adquisición de inmunidad y en el desarrollo de medidas de control.
Palabras clave: malaria, Plasmodium falciparum, diversidad genética, epidemiología, inmunidad.
Genetic diversity of Plasmodium falciparum and its implications in the epidemiology of malaria
Genetic diversity provides Plasmodium falciparum with the potential capacity of avoiding the immune response, and possibly supporting the selection of drug or vaccine resistant parasites.
These genetic characters play key roles in the selection of appropriate malaria control measures.
Diverse clones of Plasmodium falciparum, often denoted as strains, has been documented, and the degree of genetic diversity supported by several kinds of PCR (polymerase chain reaction) assays. Many studies in different endemic regions with differences in their level of disease transmission have clarified the interactions between the parasite populations and malaria epidemiology. This paper describes recombination events of the malaria parasite life cycle that originate such genetic diversity in P. falciparum, reviewing different studies on this aspect and its implications in the immunity and development of control measures in regions with different degrees of endemicity.
Key words: malaria, Plasmodium falciparum, genetic diversity, epidemiology, immunity.
La malaria es causada por parásitos protozoos del género Plasmodium, los cuales son transmitidos de un individuo a otro por la picadura de la hembra de mosquitos del género Anopheles.
De las cuatro especies de Plasmodium que afectan al hombre, P. falciparum se considera la especie más virulenta al ser responsable de la mayoría de muertes y de las formas graves de la enfermedad.
La malaria continúa siendo uno de los principales problemas de salud pública a nivel mundial, a pesar de la gran cantidad de estrategias de control dirigidas contra el parásito y contra el vector. En la actualidad, más de 3.200 millones de personas se encuentran en riesgo de adquirir la enfermedad y se estima que la incidencia annual de casos clínicos en el mundo es de 350 a 500 millones (1).
La complejidad y la diversidad genéticas de Plasmodium son en gran parte responsables del éxito de la supervivencia de este parásito en la historia evolutiva, así como del fracaso de las medidas empleadas con el objetivo de erradicarlo. Entre otras ventajas, la diversidad genética le confiere a Plasmodium la capacidad para evadir la respuesta inmune del hospedero y producir variantes resistentes a medicamentos y a vacunas, características estas relevantes para el control de la enfermedad.
La diversidad genética de Plasmodium se origina en primera instancia en su poderosa habilidad para mutar; sin embargo, el mecanismo principal que genera y mantiene esta diversidad radica en la existencia de una fase sexual obligatoria del protozoo, en la cual se presenta la recombinación genética, y que da lugar a nuevos genotipos del parásito en la mayoría de las oportunidades. Este hecho contrasta con lo que ocurre en otros protozoos como Leishmania, Trypanosoma y Giardia, que presentan una fase sexual opcional en su ciclo de vida (2).
En P. falciparum el grado de diversidad se pudo demostrar, inicialmente, empleando marcadores bioquímicos en el estudio de poblaciones naturales del parásito. Posteriormente, el desarrollo de métodos moleculares basados en ADN, como la reacción en cadena de la polimerasa (PCR), permitió precisar diferencias genéticas dentro de la especie.
Hasta la fecha, las investigaciones realizadas sugieren que la diversidad genética de las poblaciones de P. falciparum varía de acuerdo con las características epidemiológicas de la región (3-5). En consecuencia, para la comprensión de los mecanismos involucrados en la adquisición de inmunidad a la malaria, y para el desarrollo de medidas de control efectivas en las diferentes regiones con transmisión de la enfermedad, se requiere, entre otros aspectos, conocer la estructura genética de P. falciparum.
A continuación se hace una breve descripción de las fases del ciclo de vida que dan origen a la diversidad genética de P. falciparum, se revisan los estudios realizados sobre este aspecto en regiones con diferentes grados de endemicidad, así como sobre sus implicaciones en la adquisición de inmunidad y en el desarrollo de medidas de control.
Ciclo de vida de Plasmodium
Plasmodium es un protozoo con un ciclo de vida bastante complejo que comprende una fase asexual o esquizogonia en el hospedero humano, y una breve fase sexual obligatoria en el mosquito vector.
En el humano, la infección comienza con la picadura de la hembra del género Anopheles infectada, la cual introduce las formas del parásito denominadas esporozoítos, que circulan por corto tiempo e invaden las células hepáticas para formar los esquizontes tisulares. Estos lisan los hepatocitos y liberan miles de merozoítos (10.000 a 30.000 merozoítos) que invaden los glóbulos rojos. Dentro del glóbulo rojo, los merozoítos maduran a los estadios de anillo, trofozoíto y esquizonte eritrocítico. El esquizonte maduro rompe el eritrocito y libera merozoítos que posteriormente invadirán otros glóbulos rojos.
Algunos de estos merozoítos sufren un proceso de diferenciación en formas sexuales denominadas microgametocitos y macrogametocitos, que son los estadios infectantes para el mosquito vector. Una vez el parásito entra al intestino medio de Anopheles, ocurre la maduración y la fertilización de gametos dando como resultado la formación del cigoto, el cual se convierte en una forma móvil, denominada ooquineto, que posteriormente se transforma en ooquiste. Esta última forma parasitaria contiene el producto meiótico y mitótico de un único cigoto y da lugar a los esporozoítos que migran a las glándulas salivares del mosquito listas para infectar al humano en una nueva ingesta sanguínea (6).
Recombinación genética en el ciclo de vida de Plasmodium
La recombinación genética es el principal mecanismo para generar formas de un organismo con nuevos genotipos. En los eucariotes, la recombinación se presenta principalmente en la meiosis, y en proporción menor en la mitosis.
Durante la mayor parte de su ciclo de vida, la cual transcurre en el humano , Plasmodium presenta estadios biológicos haploides y la replicación ocurre mediante división mitótica (7). En el mosquito vector, el cigoto formado por la unión de los gametos representa la única fase diploide de su ciclo de vida, y en este estadio se presenta la meiosis, la cual es de importancia crítica para la generación de diversidad, ya que la recombinación genética trae como consecuencia la redistribución de genes ubicados en diferentes cromosomas, así como de eventos de entrecruzamiento entre genes ligados en el mismo cromosoma y, más raramente, dentro del mismo gen (8).
Evidencias de la recombinación genética
Los experimentos sobre recombinación genética en Plasmodium partieron del supuesto de que en el ciclo de vida del parásito se producen en igual proporción gametos macho y hembra que sufren apareamiento aleatorio, es decir, que los eventos de fertilización entre clones iguales y entre clones diferentes se presentan en igual número, y dan como resultado 50% de cigotos híbridos.
En 1978, Beale et al. demostraron por primera vez la recombinación genética en modelos animales usando Plasmodium de roedores (9). Posteriormente, en 1987, Walliker et al. realizaron cruces experimentales con P. falciparum consistentes en alimentar mosquitos con una mezcla de 2 clones del parásito y en infectar hospederos vertebrados con los esporozoítos resultantes (10). Cuando examinaron las formas sanguíneas del parásito demostraron la presencia de formas recombinantes. Más recientemente, en 1993, Ranford–Cartwright et al. evaluaron la constitución genética de ooquistes de P. falciparum, empleando los genes MSP-1 y MSP- 2 provenientes de mosquitos alimentados con una mezcla de 2 clones diferentes y pudieron demostrar formas homo y heterocigotas del parásito en este estadio (11).
Recombinación genética en la transmisión de la malaria
La diversidad generada por la recombinación genética de P. falciparum puede presentar variaciones de acuerdo con las características de la transmisión de la malaria en una región. En primer lugar, un mosquito vector puede ingerir sangre infectada con gametocitos pertenecientes a un solo clon (principalmente en zonas de baja transmisión); en este caso, los eventos de fertilización se presentan entre gametocitos genéticamente idénticos y, por consiguiente, originan cigotos (diploides) homocigóticos en todos los loci. La recombinación durante la meiosis de homocigóticos no tiene mayores consecuencias genéticas, ya que da lugar a productos haploides que tienen el mismo genotipo del clon original (autoentrecruzamiento) (1,12). De otro lado, como lo han demostrado estudios realizados en regiones con alto grado de transmisión de malaria, un individuo puede estar infectado con múltiples clones de P. falciparum y, por ende, el mosquito Anopheles puede ingerir gametocitos pertenecientes a clones genéticamente distintos (3,12-18). Este hecho puede conducir a la formación de cigotos heterocigotos, en los cuales la recombinación meiótica desemboca en la producción de nuevas combinaciones de genes dentro de la progenie haploide (esporozoitos) (10,11,19) (
figura 1).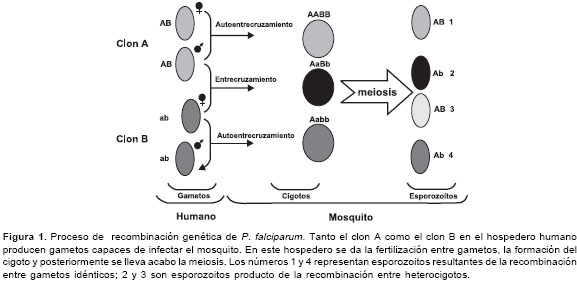
Estructura de población de P. falciparum: clonal o panmítica
En organismos como P. falciparum que presentan una forma de reproducción sexual, la principal característica para determinar la estructura de población es calcular la frecuencia de recombinación en las poblaciones naturales.
Existen dos caminos para calcular la frecuencia de entrecruzamiento en una población. El primero es calcular el desequilibrio de ligamiento (LD), un método indirecto, que permite medir las asociaciones entre loci; así por ejemplo, se determina que una población está en equilibrio de ligamiento cuando alelos de genes no ligados se presentan en combinaciones aleatorias en los parásitos, es decir, la tasa de entrecruzamientos es alta y se espera un estado de panmixia. Por el contrario, cuando en una población se encuentra una sobrerrepresentación de alelos comunes de genes no ligados se habla de desequilibrio de ligamiento, lo cual podría ser indicativo de clonalidad a causa de altos niveles de entrecruzamiento de gametos genéticamente idénticos (20).
El segundo método para calcular la frecuencia de entrecruzamiento es más directo y consiste en clasificar los cigotos en formas homocigotas y heterocigotas y comparar las frecuencias encontradas con las esperadas en una población en equilibrio de Hardy-Weinberg (entrecruzamiento aleatorio).
Así, en una población clonal se esperaría una tasa alta de homocigotos y en una población panmítica una de heterocigotos.
Ambos métodos se han empleado en el estudio de la estructura de población de P. falciparum y los resultados han causado gran controversia. Algunos autores se inclinan hacia una estructura clonal (4, 20-22), es decir cuando la recombinación genética ocurre raramente y los organismos con genotipo idéntico son la norma. Otros autores, en cambio, sugieren panmixia (10,23-26), en la cual los apareamientos aleatorios y la recombinación genética aseguran que la mayoría de los genotipos sean distintos.
La hipótesis de clonalidad en protozoarios fue enunciada por primera vez por Tibayrenc en 1990 (20), basándose en varios criterios que posteriormente fueron sustentados por otros autores, a saber:
i. sobrerrepresentación de genotipos en la población (4);
ii. ausencia de genotipos recombinantes (22), y
iii. alta tasa de desequilibrio de ligamiento (20,21).
Por otra parte, la estructura de población panmítica se sustenta en las siguientes observaciones:
- para que el ciclo de vida Plasmodium sea completado, la fase sexual es obligatoria y no opcional como ocurre en otros protozoos;
- los modelos experimentales han demostrado la presencia de formas heterocigotas, lo que sugiere la presencia de recombinación genética (10), y
- en estudios de campo realizados en varias regiones del mundo con diferentes niveles de transmisión, las frecuencias alélicas encontradas no difieren de las esperadas en una recombinación aleatoria (23-26).
Parte del origen de esta controversia puede radicar en que la mayoría de estudios sobre estructura de población de P. falciparum han usado dos o tres loci que codifican antígenos que se exponen al sistema inmune, por lo cual están bajo fuerte presión de selección (27,28). Este aspecto puede modificar sustancialmente las frecuencias alélicas sobre generaciones y la producción de frecuencias genotípicas; de esta manera, la interpretación de la estructura de poblaciones por medio de datos derivados de estos loci es compleja, ya que no es claro si las relaciones observadas reflejan la historia de la población o son el resultado de la selección natural (29).
Hasta la fecha, los resultados disponibles sobre la estructura poblacional no son consistentes; incluso una investigación realizada con marcadores moleculares no sometidos a una fuerte presión de selección, como los microsatélites, muestra resultados que sugieren que P. falciparum presenta una estructura de población diferente, que va desde la clonal hasta la panmítica dependiendo de la intensidad de transmisión de la enfermedad en el lugar estudiado (4,29 ). De esta manera, un estado de clonalidad en P. falciparum podría esperarse en regiones de baja endemicidad, donde un único clon es responsable de los casos de la enfermedad y, en consecuencia, la recombinación en el mosquito vector no de lugar a nuevas combinaciones del parásito. De otro lado, en regiones de alta transmisión se espera una gran diversidad genética en la población y un estado de panmixia debido a que las infecciones con múltiples clones y el entrecruzamiento entre gametos diferentes son frecuentes (29,30).
Así mismo, las diferencias en la estructura de población de P. falciparum pueden también reflejar la historia del parásito en las zonas de transmisión de malaria; es decir, la estructura de población clonal encontrada en zonas de baja transmisión se explicaría por la reciente colonización del parásito en estas regiones, pues no ha transcurrido el tiempo suficiente para generar nuevas combinaciones genéticas.
Por su parte, la estructura panmítica en zonas de alta transmisión se explicaría por una presencia mucho más prolongada del parásito en estas zonas, dando así lugar a la posibilidad de una mayor diversidad (29) (cuadro 1).

Estudio de la diversidad genética en P. falciparum
Muchos autores han documentado la existencia de diferentes cepas o clones de P. falciparum; Garnham, en 1966 (31), diferenció las cepas de P. falciparum de acuerdo a características clínicas, susceptibilidad en el vector e inmunidad cruzada. Posteriormente, en 1973 (23), Carter et al. realizaron los primeros estudios bioquímicos para diferenciar cepas de P. falciparum por medio de perfiles isoenzimáticos, método que fue reemplazado por electroforesis de proteínas de dos dimensiones, con la cual las formas alélicas de al menos 20 proteínas de P. falciparum pudieron ser diferenciadas por su punto isoeléctrico y peso molecular (32,33). En 1985, McBride et al. (34) emplearon inmunofluorescencia para diferenciar variantes antigénicas en muestras de campo.
En la década de los 90, la identificación y secuenciamiento de genes de Plasmodium condujeron al uso extensivo de metodologías basadas en ADN; particularmente, la reacción en cadena de la polimerasa (PCR) se ha convertido en el método de elección para estudios de diversidad genética en muestras recolectadas en el campo, debido principalmente a la alta sensibilidad y especificidad para amplificar marcadores genéticos, a su capacidad de revelar polimorfismos de longitud y a la posibilidad de usar muestras pequeñas de sangre o mosquitos sin la necesidad de realizar cultivos de parásitos, los cuales pueden conducir a una selección de clones.
Entre las secuencias polimórficas que han sido de utilidad como marcadores genéticos de las poblaciones de P. falciparum se encuentran los genes de las proteínas de superficie del merozoíto 1 y 2 (MSP-1) (MSP-2) (35,36) y el gen de la proteína rica en glutamato (GLURP) (37). Para MSP-1 y MSP-2 se han descrito dos tipos de polimorfismo, en secuencia y en tamaño, mientras que para GLURP sólo se ha observado polimorfismo en tamaño. Las variaciones de los productos de amplificación en cada uno de estos loci pueden distinguirse fácilmente por medio de electroforesis en gel de agarosa. A continuación se hace una breve descripción de MSP-1, MSP-2 y GLURP.
Gen de la proteína de superficie del merozoíto 1 (MSP-1)
El gen MSP-1 es de copia única y está localizado en el cromosoma 9 (38). Codifica uno de los antígenos más estudiados de la fase eritrocítica del parásito y se ha señalado como un potencial candidato para vacuna. La comparación de secuencias de nucleótidos de MSP-1 en diferentes clones de laboratorio ha permitido identificar 17 bloques o dominios: 7 altamente variables, 5 conservados y 5 semiconservados. Los bloques variables (bloques 2, 4, 6, 8, 10, 14, 16) presentan dos alelos denominados MAD20 y K1 (39); una excepción la presenta el bloque 2, el cual cuenta con un alelo adicional denominado RO33 (40) (figura 2). Los alelos MAD20 y K1 presentan diferentes unidades de secuencias repetidas mientras que el alelo RO33 consiste de una secuencia única.
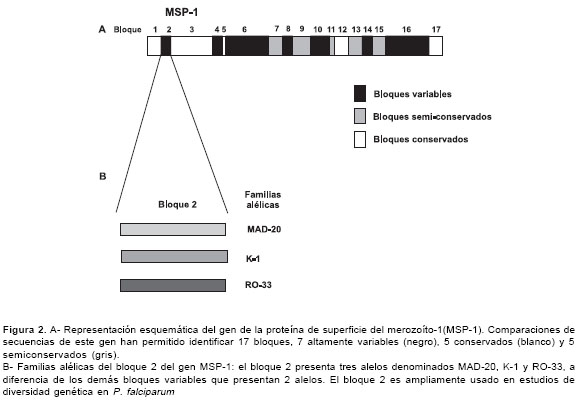
El nombre de estos alelos de MSP-1 se deriva de la línea de parásitos en la cual fueron observados por primera vez (41). Los bloques variables, y principalmente el bloque 2, son ampliamente usados en estudios de diversidad genética de P. falciparum.
Gen de la proteína de superficie del merozoíto 2 (MSP-2)
Este es un gen de copia única localizado en el cromosoma 2 (38) que codifica una proteína de superficie también considerada candidata para vacuna. Para este gen se han descrito secuencias polimórficas tanto en cepas mantenidas en el laboratorio como en cepas silvestres, y de éstas, la más utilizada en estudios sobre diversidad genética es la secuencia polimórfica denominada bloque 3 o bloque central. En sus extremos, el bloque 3 contiene secuencias conservadas no repetidas, las cuales permiten clasificar al gen MSP-2 en dos familias alélicas denominadas FC27 (FC) e IC/3D7 (IC), que derivan igualmente su nombre de las líneas de parásitos en las cuales fueron inicialmente observadas (42). Adicionalmente, este bloque presenta una región central compuesta por secuencias repetidas polimórficas, las cuales caracterizan los alelos dentro de cada familia alélica (
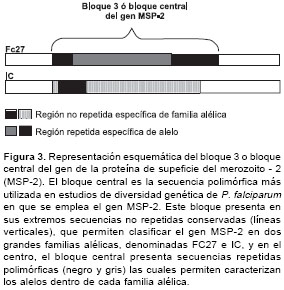
Gen de la proteína rica en glutamato (GLURP)
Localizado en el cromosoma 10 (38), este gen codifica una proteína inmunodominante rica en glutamato. Presenta una región central conservada no repetida, rodeada por dos regiones repetidas. Una de estas regiones repetidas, denominada R II, de aproximadamente 60 pb, varía entre las diferentes cepas de parásitos, por lo cual es ampliamente utilizada en los estudios de diversidad genética de P. falciparum (37) (figura 4).

Determinación de la multiplicidad de infección por P. falciparum
Inicialmente se sugirió que la infección simultánea en un individuo por múltiples clones de P. falciparum (multiplicidad de infección) se debía al proceso conocido como super infección, es decir, cuando los clones de parásito presentes en el hospedero humano provenían de múltiples picaduras causadas por diferentes mosquitos infectados (43). Hace poco tiempo, además, se demostró que la multiplicidad de infección puede provenir de un solo inóculo del mosquito compuesto por varios clones del parásito (44). Las primeras investigaciones que mostraron que el humano o el mosquito vector podían estar simultáneamente infectados con una mezcla del parásito haploide, se llevaron a cabo con antígenos, enzimas y marcadores de sensibilidad a medicamentos. En 1973, Carter et al. (23) demostraron que pacientes de Gambia infectados con P. falciparum presentaban dos alelos de las enzimas glucosa fosfato isomerasa (GPI) y de la lactacto deshidrogenasa (LDH), las cuales son codificadas por genes de copia única.
Más tarde, trabajos de clonación realizados por Rosario en 1981 y Graves et al. en 1984 revelaron clones enzimáticamente distintos en muestras provenientes de pacientes (45,46). Así mismo, en 1984 en Tailandia, Thaithong et al. demostraron por medio de marcadores de sensibilidad a medicamentos, la presencia en un paciente de 7 clones genéticamente diferentes (47).
En la actualidad, el método más eficiente para diferenciar los clones de P. falciparum es el de marcadores genéticos, y para calcular el número de clones presentes en una infección se han utilizado varios procedimientos, desde los que se basan en el simple conteo de los diferentes alelos presentes en un solo locus del parásito (13,23,48), hasta los modelos matemáticos o biológicos que estiman el genotipo y la distribución de los clones por individuo partiendo de la información simultánea de varios loci e incorporando las frecuencias de los genotipos en la población (22,43,49)
Diversidad genética en P. falciparum y su relación con la endemicidad
El término endemicidad se refiere en general al grado de transmisión de la malaria en un área o una comunidad dada. De acuerdo con MacDonald (50), la endemicidad de la malaria puede clasificarse en tres tipos: 1) malaria estable, cuando la transmisión de la malaria es alta y no presenta fluctuaciones marcadas en el tiempo, a pesar de que ocurran cambios estacionales; 2) malaria inestable, cuando existen marcadas fluctuaciones en la transmisión de la malaria de un año a otro, y 3) malaria epidémica, considerada como un extremo de la malaria inestable, que se presenta cuando se da un incremento en la tasa normal de transmisión de la malaria en determinada región.
Investigaciones realizadas en zonas de transmisión estable han encontrado que existe una diversidad genética de P. falciparum considerablemente alta en estas regiones, que tanto el humano como el insecto vector frecuentemente están infectados con una mezcla de más de un clon del parásito (multiplicidad de infección), y que la composición de alelos de P. falciparum circulantes cambia en el tiempo (3,13-17,24,48,51,52).
Por otra parte, en zonas con transmisión inestable se han realizado pocos estudios, la mayoría de ellos en Sudán (3,53,54). Hasta la fecha, los resultados obtenidos no son consistentes; en algunos estudios se ha encontrado una diversidad genética alta en las poblaciones de P. falciparum mientras que en otros se ha detectado poca diversidad (3,18,55-61). En zonas con transmisión inestable, generalmente los individuos están infectados por un número limitado de clones (18, 57,58), y los estudios longitudinales llevados a cabo sugieren que el repertorio de alelos se mantiene en el tiempo (51,61) (cuadro 2).

Diversidad genética y multiplicidad de infección
En regiones con transmisión estable de malaria la principal causa generadora de niveles altos de diversidad genética y multiplicidad de infección, probablemente es la alta tasa de inoculación entomológica, definida como el número de picaduras infectivas por persona por año (62).
Una tasa de inoculación entomológica alta implica continuas inoculaciones infectivas y una constante generación de nuevos genotipos del parásito, producto de la tasa de recombinación alta en el mosquito vector.
En este sentido, un estudio realizado por Babiker et al. en 1997 (3), que comparaba las infecciones por P. falciparum en dos regiones de África con diferente endemicidad, mostró una asociación entre la tasa de inoculación entomológica y el número de genotipos en las personas. Sin embargo, esta relación no se observó claramente en Papúa Nueva Guinea, donde se encontró un número limitado de clones diferente a pesar de tener una tasa de inoculación entomológica alta (4). Estas discrepancias podrían deberse tanto a diferencias en el comportamiento del mosquito vector como del hospedero humano, las cuales tienen gran efecto en la dinámica de transmisión del parásito. Con respecto al primer componente, el grado de antropofília del vector tiene implicaciones en la carga clonal del hospedero humano. En Papua Nueva Guinea, Anopheles farauti, el principal vector, presenta un rango muy amplio en el índice de sangre humana, es decir en el grado de antropofilia, variando entre 0,09 y 0,83 (63), situación que difiere de la encontrada en África tropical, donde el mosquito vector Anopheles gambiae, se caracteriza por su eficiencia vectorial y su fuerte tendencia a alimentarse en humanos (64).
Con respecto al componente humano, existen varios factores que regulan la carga clonal, entre los que se encuentran, el secuestro que sufre P. falciparum en la microcirculación, la inmunidad específica de cepa, la cual puede limitar el número de clones encontrados en niños mayores y adultos, y la inhibición de una especie por otra en el caso de infecciones mixtas (65).
Dinámica de las infecciones
Los estudios realizados en regiones con diferente intensidad de transmisión de malaria muestran que en individuos provenientes de regiones con transmisión estable, los alelos de varios genes de P. falciparum fluctúan en el tiempo, mientras que en las personas residentes en zonas con transmisión inestable, los alelos se mantienen a través del período de seguimiento (51).
En 1997, Farnert et al. evaluaron en Tanzania muestras de sangre de niños asintomáticos infectados con P. falciparum durante un período de dos semanas y establecieron que los clones del parásito encontrados los días 1 y 3 eran similares entre sí, pero diferentes de los observados los días 2 y 4 (52). La explicación más probable para este hallazgo es que la población de parásitos estaba siendo secuestrada en días alternativos.
Adicionalmente, las investigaciones han mostrado que las frecuencias de alelos de un gen varían de acuerdo con la región geográfica (25,66,67). En 1997, Conway et al. mostraron que la frecuencia de familias alélicas del gen MSP-1 difería en varios países; en el sureste de Asia y en el este africano, la familia alélica denominada Wellcomelike tuvo una frecuencia de entre 25% y 50%, pero presentó una frecuencia menor a 1% en el oeste de África y en Brasil (68). Así mismo, en 1991, Scherf et al. encontraron grandes diferencias en la frecuencia con la que se presentaba el alelo RO33 del gen MSP-1 en Brasil y en Senegal, siendo en Brasil mucho más alta (69). En regiones con transmisión inestable como Honduras (18) y Colombia (58) se ha encontrado una distribución de alelos del gen MSP-1 en la que predomina el alelo MAD-20 sobre el K-1; sin embargo, en regiones de transmisión alta, como Tanzania y Senegal, esta distribución es invertida (69,70).
En general, los hallazgos en las diferentes regiones estudiadas permiten concluir que la diversidad genética, la multiplicidad de infección y la dinámica en el tiempo de las infecciones por P. falciparum se ven afectadas por el grado de endemicidad de la malaria en cada región.
Implicaciones de la diversidad genética de P. falciparum en la adquisición de inmunidad
La inmunidad protectora contra la malaria causada por P. falciparum se adquiere lentamente, luego de varios años de infecciones repetidas con el parásito, y mantenerla implica la exposición permanente a mosquitos infectados.
Las infecciones experimentales en humanos realizadas por Jeffery en 1966 (71) mostraron que la inmunidad producida contra una sola cepa de P. falciparum es ineficiente ante el reto con una cepa heteróloga. Por ello, en zonas de transmisión estable, el desarrollo de una inmunidad efectiva contra el parásito requiere de infecciones repetidas con los diferentes clones de P. falciparum que circulan en la región, de ahí que el desarrollo de la inmunidad sea un proceso lento (72).
Desde una definición epidemiológica, la inmunidad en malaria tiene varios componentes: una inmunidad clínica, que, según se cree, se desarrolla durante la infancia y permite mantener una carga importante de parásitos sin presentar síntomas de la enfermedad (73), y una inmunidad anti-parásito, la cual se adquiere lentamente y conduce a una marcada disminución en la densidad del parásito en el individuo infectado (13,74).
En 1935, Sergent y Parrot (75) acuñaron el término premunición para referirse a la protección contra la superinfección, es decir la infección por nuevos clones, como resultado de una infección existente, y denominaron semi-inmunidad a la inmunidad adquirida como consecuencia de continuas exposiciones, y que confiere la capacidad de controlar las infecciones, manteniendo la parasitemia crónica en bajos niveles (76-78).
Inmunidad en malaria, multiplicidad de la infección y edad del individuo
Una serie de estudios epidemiológicos, realizados en zonas de alta endemicidad como Senegal, Tanzania y Papúa Nueva Guinea (13,14,79,80), confirmaron la importancia de la multiplicidad de la infección en la adquisición de inmunidad en malaria, al encontrar que:
- la multiplicidad de la infección dentro de un hospedero parece depender no solo de la exposición sino también de la edad;
- la multiplicidad puede alcanzar niveles altos, por ejemplo, se han detectado en un mismo individuo hasta nueve clones diferentes de P. falciparum, y
- la multiplicidad de infección se relaciona positivamente con protección contra episodios clínicos de malaria no complicada.
A medida que los niños adquieren inmunidad, el promedio de infecciones múltiples incrementa, lo cual puede deberse a que la duración de las infecciones aumenta. En infantes (6 a 12 meses de edad), la defensa contra la infección por P. falciparum se basa principalmente en la fiebre y en la actividad de citocinas con fuerte efecto antiparasitario (81). Esta estrategia tiende a resolver las infecciones más que a permitir que ellas continúen en bajas densidades, por lo cual las infecciones son generalmente de corta duración. Sin embargo, con la edad, el niño desarrolla mecanismos que permiten la persistencia de infecciones crónicas asintomáticas con bajas densidades parasitarias; en consecuencia, la duración de las infecciones incrementa y la multiplicidad de las infecciones pasa a estar menos correlacionada con la densidad parasitaria (82).
En niños mayores, la alta multiplicidad de la infección es característica de la infección crónica asintomática y del bajo nivel de parasitemia, y por consiguiente, del estado inmune (76).
En adultos se ha observado que la multiplicidad de la infección es menor en comparación con lo encontrado en niños mayores y adolescentes (10,78). Este hallazgo parece relacionarse con el hecho de que estos individuos presentan densidades parasitarias muy bajas, que posiblemente se encuentran por debajo de los límites de detección de la PCR, o han adquirido suficiente inmunidad frente al repertorio de cepas circulantes (76).
Inmunidad y endemicidad de la malaria
En regiones con transmisión estable de malaria, la mayoría de los estudios realizados han encontrado que los pacientes con malaria clínica tienen menor promedio de infecciones múltiples en comparación con los asintomáticos de la misma edad y de la misma comunidad. Los trabajos longitudinales de Al-Yaman et al., en 1997(80), y de Beck et al., en 1997 (14), muestran una verdadera asociación entre la presencia de múltiples infecciones y la protección contra la enfermedad.
Por otra parte, las observaciones sobre la adquisición de inmunidad en regiones con transmisión inestable son paradójicas; por un lado, se ha considerado por tradición que los individuos que viven en estas regiones no logran desarrollar una inmunidad significativa frente a la enfermedad (50). En los estudios realizados, la presencia de malaria clínica en un individuo no confiere ningún tipo de protección contra la enfermedad en la siguiente estación de transmisión (83-85). Sin embargo, una significativa proporción de la población en estas regiones mantiene infecciones asintomáticas con títulos altos de anticuerpos antimalaria asociados con la estación de transmisión (83-86), y se ha obtenido confirmación parasitológica en individuos con microscopía negativa persistente y PCR positiva (84,87).
De acuerdo con el modelo de protección contra la superinfección (premunición), una posible explicación para la falta de una completa inmunidad a la malaria en regiones con transmisión inestable podría basarse en el hecho de que las infecciones crónicas en estas personas presentan una baja multiplicidad de la infección (la mayoría contiene probablemente un solo clon) (53). En este sentido, las infecciones crónicas son pobremente protectoras, ya que inducen una respuesta inmune de corta duración contra un número relativamente pequeño de epítopes variables del parásito. Sin embargo, tales infecciones podrían proteger contra la superinfección por parásitos de similitudes antigénicas con los ya experimentados.
No obstante, la verdadera causa de la relativa reducción en la incidencia de la malaria observada en adultos de zonas con transmisión inestable sigue siendo desconocida y difícil de explicar completamente con la teoría de la premunición en malaria.
Implicaciones de la diversidad genética de P. falciparum para el control de la malaria
Las diferencias en el grado de diversidad genética de las poblaciones de P. falciparum en las regiones con transmisión de malaria pueden influir en la eficacia de medidas de control como los medicamentos y las vacunas.
La recombinación genética de Plasmodium en el vector puede dar lugar a que un parásito presente combinaciones de genes que confieren resistencia a uno o varios medicamentos, pero igualmente puede disociar tales combinaciones. Este último aspecto se presenta sólo cuando el número promedio de clones por individuo es menor de 1,5, ya que tanto la presión del medicamento como la frecuencia de genes que confieren diferentes tipos de resistencia son bajas (88).
Los trabajos de modelación realizados por Curtis et al. en 1986 indican que la gran cantidad de recombinaciones entre los loci que codifican la resistencia a medicamentos podría conducir a una tasa lenta en la expansión de la resistencia a múltiples medicamentos, aspecto que respalda las recomendaciones internacionales de emplear combinaciones de medicamentos en lugar de usar uno solo, dado que la mutación de un solo locus por la presión de un medicamento es más frecuente que las mutaciones de dos locus bajo la presión de dos medicamentos (89). No obstante, algunas evidencias sugieren que la expansión de resistencia de P. falciparum a múltiples medicamentos se correlaciona con el grado de endemicidad de la malaria, progresando más rápido en áreas de alta transmisión y más lento en regiones de baja transmisión (90). A medida que aumenta el grado de endemicidad aumenta el número de clones en el hospedero y, por consiguiente, incrementa el número de mutantes resistentes a medicamentos. En consecuencia, la selección efectiva de parásitos resistentes a medicamentos es mayor en las regiones con altos niveles de transmisión de malaria (88).
La esperanza de una alternativa para el control de la malaria reside en el desarrollo de vacunas Hasta el momento, la mayoría de las moléculas consideradas candidatas a vacunas son proteínas localizadas en la superficie del parásito, y no se sabe si el alto polimorfismo que presentan estas moléculas podría ser un obstáculo para el desarrollo de vacunas; se constituiría en un problema indudable si la respuesta inmune protectora se orienta primordialmente contra dominios polimórficos, ya que la recombinación entre loci que codifican antígenos polimórficos le ofrece al parásito una amplia oportunidad para desarrollar formas resistentes a la respuesta inmune inducida por estas vacunas, de ahí la dificultad de desarrollar una vacuna efectiva contra la enfermedad (70).
Agradecimientos
Los autores desean agradecer a las doctoras Ángela Restrepo y Amanda Maestre por las correcciones al manuscrito.
Conflicto de intereses
Ninguno de los autores tiene conflicto de intereses personal o financiero con otras personas u organizaciones que pudiera haber influido indebidamente en los datos presentados en esta revisión.
Financiación
Este trabajo se realizó gracias al apoyo financiero de la Universidad de Antioquia y al programa Jóvenes investigadores de Colciencias, que financió a Judy Natalia Jiménez Q bajo el convenio de pasantía PS-0-140.
Correspondencia:
Carlos Muskus López, PECET, Universidad de Antioquia,
Calle 62 No. 52-59, Medellín, Colombia. A.A.1226.
Teléfono: 2 10 65 07 Fax: 2 10 65 11
Recibido: 19/05/05; aceptado: 31/08/05
Referencias
1. Korenromp El. Malaria incidence estimates at country level for the year2004 – proposed estimates and draft report. In: for the Roll Back Malaria Monitoring and Evaluation Reference Group & MERG Task Force on Malaria Morbidity. Geneva: World Health Organization; 2004. [ Links ]
2. Walliker D, Babiker H, Ranford- Cartwright L. The genetic structure of malaria parasite populations. In: Irwin W. Sherman, editor. Malaria: parasite biology, pathogenesis, and protection. Washington D.C: ASM Press; 1998. p.235-52. [ Links ]
3. Babiker HA, Lines J, Hill WG, Walliker D. Population structure of Plasmodium falciparum in villages with different malaria endemicity in east Africa. Am J Trop Med Hyg 1997;56:141-7. [ Links ]
4. Paul RE, Packer MJ, Walmsley M, Lagog M, Ranford-Cartwright LC, Paru R et al. Mating patterns in malaria parasite populations of Papua New Guinea. Science 1995;269:1709-11. [ Links ]
5. Marques PX, Saúte F, Pinto VV, Cardoso S, Pinto J, Alonso PL et al. Plasmodium species mixed infections in two areas of Manhiça District, Mozambique. Int J Biol Sci 2005;1:96-102. [ Links ]
6. Fujioka H, Aikawa M. The malaria parasite and lifecycle. In: Wahlgren M, Perlmann P, editors. Malaria molecular and clinical aspects. Amsterdam: Harwood Academic Publishers; 1999. p.19-55. [ Links ]
7. Walliker D, Carter R, Sanderson A. Genetic studies on Plasmodium chabaudi: recombination between enzyme markers. Parasitology 1975;70:19-24. [ Links ]
8. Sinden RE, Hartley RH. Identification of meiotic division of malarial parasites. J Protozool 1985;32:742-4. [ Links ]
9. Beale G, Carter R, Walliker D. Genetics. En: Killick-Kendrick R, Peters W, editors. Rodent Malaria. London: Academic Press; 1978. p.213-45. [ Links ]
10. Walliker D, Quakyi IA, Wellems TE, McCutchan TF, Szarfman A, London WT et al. Genetic analysis of the human malaria parasite Plasmodium falciparum. cience 1987;236:1661-6. [ Links ]
11. Ranford–Cartwright LC, Balfe P, Carter R, Walliker D. Frequency of cross- fertilization in the human malaria parasite Plasmodium falciparum. Parasitology 1993;107:11-8. [ Links ]
12. Babiker HA, Ranford-Cartwright LC, Walliker D. Genetic structure and dynamics of Plasmodium falciparum infections in the Kilombero region of Tanzania. Trans R Soc Trop Med Hyg 1999;93:11-4. [ Links ]
13. Ntoumi F, Contamin H, Rogier C, Bonnefoy S, Trape JF, Mercereau-Puijalon O. Age-dependent carriage of multiple Plasmodium falciparum merozoite surface antigen-2 alleles in asymptomatic malaria infections. Am J Trop Med Hyg 1995;52:81-8. [ Links ]
14. Beck HP, Felger I, Huber W, Steinger S, Smith T, Weiss N et al. Analysis of multiple Plasmodium falciparum infections in Tanzanian children during the Phase III Trial of the malaria vaccine SPf66. J Infect Dis 1997;175:921-6. [ Links ]
15. Kyes S, Craig AG, Marsh K, Newbold CI. Plasmodium falciparum: a method for the amplification of S antigens and its application to laboratory and field samples. Exp Parasitol 1993;77:473-83. [ Links ]
16. Zwetyenga J, Rogier C, Tall A, Fontenille D, Snounou G, Trape JF et al. No influence of age on infection complexity and allelic distribution on Plasmodium falciparum infections in Ndiop, a Senegalese village with seasonal, mesoendemic malaria. Am J Trop Med Hyg 1998;59:726-35. [ Links ]
17. Paul RE, Hackford I, Brockman A, Muller-Graf C, Price R, Luxemburger C et al. Transmission intensity and Plasmodium falciparum diversity on the northwestern border of Thailand. Am J Trop Med Hyg 1998;58:195-203. [ Links ]
18. Haddad D, Snounou G, Mattei D, Enamorado IG, Figueroa J, Stahl S et al. Limited genetic diversity of Plasmodium falciparum in field isolates from Honduras. Am J Trop Med Hyg 1999;60:30-4. [ Links ]
19. Wellems TE, Panton LJ, Gluzman IY, do Rosario VE, Gwadz RW, Walker-Jonah A et al. Chloroquine resistance is not linked to MDR-like genes in a Plasmodium falciparum cross. Nature 1990;345; 253-5. [ Links ]
20. Tibayrenc M, Kjellberg F, Ayala FJ. A clonal theory of parasitic protozoa: The population structure of Entamoeba, Giardia, Leishmania, Naegleria, Plasmodium, Trichomonas and Tripanosoma and their medical and taxonomical consequences. Proc Natl Acad Sci USA 1990;87:2414-8. [ Links ]
21. Abderrazak SB, Oury B, Lal AA, Bosseno MF, Force-Barge P, Dujardin JP et al. Plasmodium falciparum: population genetic analysis by multilocus enzyme electrophoresis and other molecular markers. Exp Parasitol 1999;92:232-8. [ Links ]
22. Babiker HA, Ranford-Cartwright LC, Currie D, Charlwood JD, Billingsley P, Teuscher T et al. Random mating in a natural population of the malaria parasite Plasmodium falciparum. Parasitology 1994;109:413-21. [ Links ]
23. Carter R, McGregor IA. Enzyme variation in Plasmodium falciparum in The Gambia. Trans R Soc Trop Med Hyg 1973;67:830-7. [ Links ]
24. Conway DJ, McBride JS. Population genetics of Plasmodium falciparum within a malaria hyperendemic area. Parasitology 1991;103:7-16. [ Links ]
25. Creasey A, Fenton B, Walker A, Thaithong S, Oliveira S, Mutambu S et al. Genetic diversity of Plasmodium falciparum shows geographical variation. Am J Trop Med Hyg 1990;42:403-13. [ Links ]
26. Babiker HA, Creasey AM, Fenton B, Bayoumi RA, Arnot DE, Walliker D. Genetic diversity of Plasmodium falciparum in a village in eastern Sudan: 1. Diversity of enzymes, 2D-PAGE proteins and antigens. Trans R Soc Trop Med Hyg 1991;85:572-7. [ Links ]
27. Hughes AL. Positive selection and interallelic recombination at the merozoite surface antigen-1 (MSA-1) locus of Plasmodium falciparum. Mol Biol Evol 1992;9:381-93. [ Links ]
28. Hughes MK, Hughes AL. Natural selection on Plasmodium surface proteins. Mol Biochem Parasitol 1995;71:99-113. [ Links ]
29. Anderson TJ, Haubold B, Williams JT, Estrada- Franco JG, Richardson L, Mollinedo R et al. Microsatellite markers reveal a spectrum of population structures in the malaria parasite Plasmodium falciparum. Mol Biol Evol 2000;17:1467-82. [ Links ]
30. Machado RL, Povoa MM, Calvosa VS, Ferreira MU, Rossit AR, dos Santos EJ et al. Genetic structure of Plasmodium falciparum populations in the Brazilian Amazon region. J Infect Dis 2004;90:1547-55. [ Links ]
31. Garnham P. Malaria parasites and other haemosporidia. Oxford: Blackwell Scientific Publications Ltd; 1966. [ Links ]
32. Tait A. Analysis of protein variation in Plasmodium falciparum by two-dimensional electrophoresis. Mol Biochem Parasitol 1981;2:205-18. [ Links ]
33. Fenton B, Walker A, Walliker D. Protein variation in clones of Plasmodium falciparum detected by twodimensional electrophoresis. Mol Biochem Parasitol 1985;16:173-83. [ Links ]
34. McBride JS, Newbold CI, Anand R. Polymorphism of a high molecular weight schizont antigen of the human malaria parasite Plasmodium falciparum. J Exp Med 1985;161:160-80. [ Links ]
35. Kimura E, Mattei D, Di Santi SM, Scherf A. Genetic diversity in the major merozoite surface antigen of Plasmodium falciparum: high prevalence of a third polymorphic form detected in strains derived from malaria patients. Gene 1990;91:57-62. [ Links ]
36. Fenton B, Clark JT, Khan CM, Robinson JV, Walliker D, Ridley R et al. Structural and antigenic polymorphism of the 35-to-48 kilodalton merozoite surface antigen (MSA-2) of the malaria parasite Plasmodium falciparum. Mol Cell Biol 1991;11:963-71. [ Links ]
37. Borre MB, Dziegiel M, Hogh B, Petersen E, Rieneck K, Riley E et al. Primary structure and localization of a conserved immunogenic Plasmodium falciparum glutamate-rich protein (GLURP) expressed in both the preerythrocytic and erithrocytic stages of the vertebrate life-cycle. Mol Biochem Parasitol 1991;49:119-31. [ Links ]
38. Triglia T, Wellems TE, Kemp DJ. Toward a high resolution map of the Plasmodium falciparum genome. Parasitol Today 1992;8:225-9. [ Links ]
39. Tanabe K, Mackay M, Goman M, Scaiffe JG. Allelic dimorphism in a surface antigen gene of the malaria parasite Plasmodium falciparum. J Mol Biol 1987; 195:273-87. [ Links ]
40. Certa U, Rotmann D, Matile H, Reber-Liske R. A naturally occurring gene encoding the major surface antigen precursor p190 of Plasmodium falciparum lacks tripeptide repeats. EMBO J 1987;6:4137-42. [ Links ]
41. Miller LH, Roberts T, Shahabuddin M, McCutchan TF. Analysis of sequence diversity in the Plasmodium falciparum merozoite surface protein-1 (MSP-1). Mol Biochem Parasitol 1993;59:1-14. [ Links ]
42. Smythe JA, Peterson MG, Coppel RL, Saul AJ, Kemp DJ, Anders RF. Structural diversity in the 45-kilodalton merozoite surface antigen of Plasmodium falciparum. Mol Biochem Parasitol 1990;39:227-34. [ Links ]
43. Hill WG, Babiker HA. Estimation of number of malaria clones in blood samples. Proc Biol Sci 1995;262:249-57. [ Links ]
44. Druilhe P, Daubersies P, Patarapotikul J, Gentil C, Chene L, Chongsuphajaisiddhi T et al. A primary malarial infection is composed of a very wide range of genetically diverse but related parasites. J Clin Invest 1998;101:2008-16. [ Links ]
45. Rosario V. Cloning of naturally occurring mixed infections of malaria parasites. Science 1981;212:1037-8. [ Links ]
46. Graves PM, Carter R, Keystone JS, Seeley DC Jr. Drug sensitivity and isoenzyme type in cloned lines of Plasmodium falciparum. Am J Trop Med Hyg 1984; 33:212-9. [ Links ]
47. Thaithong S, Beale GH, Fenton B, McBride J, Rosario V, Walker A et al. Clonal diversity in a single isolate of the malaria parasite Plasmodium falciparum. Trans R Soc Trop Med Hyg 1984;78:242-5. [ Links ]
48. Conway D, Greenwood BM, McBride JS. The epidemiology of multiple-clon Plasmodium falciparum infection in The Gambia. Parasitology 1991;103:1-6. [ Links ]
49. Martinelli A, Hunt P, Cheesman SJ, Carter R. Amplified fragment length polymorphism measures proportions of malaria parasites carrying specific alleles in complex genetic mixtures. Mol Biochem Parasitol 2004;136:117-22. [ Links ]
50. McDonald G. The epidemiology and control of malaria. London: Oxford University Press; 1957. [ Links ]
51. Daubersies P, Sallenave-Sales S, Magne S, Trape JF, Contamin H, Fandeur T et al. Rapid turnover of Plasmodium falciparum populations in asymptomatic individuals living in a high transmission area. Am J Trop Med Hyg 1996;54:18-26. [ Links ]
52. Farnet A, Snounou G, Rooth I, Bjorkman A. Daily dynamics of Plasmodium falciparum subpopulations in asymptomatic children in a holoendemic area. Am J Trop Med Hyg 1997;56:538-47. [ Links ]
53. Hamad AA, El Hassan IM, El Khalifa AA, Ahmed GI, Abdelrahim SA, Theander TG et al. Chronic Plasmodium falciparum infections in an area of low intensity malaria transmission in the Sudan. Parasitology 2000;120:447-56. [ Links ]
54. Babiker HA, Satti G, Walliker D. Genetic changes in the population of Plasmodium falciparum in a Sudanese village over a three-year period. Am J Trop Med Hyg 1995;53:7-15. [ Links ]
55. Hijar G, Padilla C, Marquino W, Falconi E, Montoya Y. Genetic polymorphism of Plasmodium falciparum isolates from Loreto, Peru. Trans R Soc Trop Med Hyg. 2002;96:137-40. [ Links ]
56. Urdaneta L, Lal A, Barnabe C, Oury B, Goldman I, Ayala FJ et al. Evidence for clonal propagation in natural isolates of Plasmodium falciparum from Venezuela. Proc Natl Acad Sci U S A 2001;98:6725-9. [ Links ]
57. Ariey F, Chalvet W, Hommel D, Peneau C, Hulin A, Mercereau-Puijalon O et al. Plasmodium falciparum parasites in French Guiana : limited genetic diversity and high selfing rate. Am J Trop Med Hyg 1999;61:978-85. [ Links ]
58. Montoya L, Maestre A, Carmona J, Lopes D, Do Rosario V, Blair S. Plasmodium falciparum: diversity studies of isolates from two Colombian regions with different endemicity. Exp Parasitol 2003;104:14-9. [ Links ]
59. Gómez D, Chaparro J, Rubiano C, Rojas MO, Wasserman M. Genetic diversity of Plasmodium falciparum field samples from an isolated Colombian village. Am J Trop Med Hyg 2002;67:611-6. [ Links ]
60. Snewin V, Herrera M, Sánchez G, Artur Scherf, Langsley G, Herrera S. Polymorphism of the alleles of merozoite surface antigens MSA 1 and MSA 2 in Plasmodium falciparum wild isolates from Colombia. Mol Biochem Parasitol 1991;49:265-76. [ Links ]
61. Sallenave-Sales S, Ferreira-da-Cruz MF, Faria CP, Cerruti C Jr, Daniel-Ribeiro CT, Zalis MG. Plasmodium falciparum: limited genetic diversity of MSP- 2 in isolates circulating in Brazilian endemic areas. Exp Parasitol 2003;103:127-35. [ Links ]
62. Beier JC, Oster CN, Onyango FK, Bales JD, Sherwood JA, Perkins PV et al. Plasmodium falciparum incidence relative to entomological inoculation rates at a site proposed for testing malaria vaccines in western Kenya. Am J Trop Med Hyg 1994;50:529-36. [ Links ]
63. Charlwood JD. Blood feeding and resulting behavior in the Anopheles punctulatus Dontiz complex (Diptera: Culicidea) from coastal Papua New Guinea. Bull Entomol Res 1985;75:463-75. [ Links ]
64. Charlwood JD. Density independent feeding success in malaria vectors in Tanzania. Bull Entomol Res 1995;85:29-35. [ Links ]
65. Richie TL. Interactions between malaria parasites infecting the same vertebrate host. Parasitology 1988;96:607-39. [ Links ]
66. Ranjit MR, Das A, Das BP, Das BN, Dash BP, Chhotray GP. Distribution of Plasmodium falciparum genotypes in clinically mild and severe malaria cases in Orissa, India. Trans R Soc Trop Med Hyg 2005;99:389-95. [ Links ]
67. Ferreira MU, Kaneko O, Kimura M, Liu Q, Kawamoto F, Tanabe K. Allelic diversity at the merozoite surface Protein-1 (MSP-1) locus in natural Plasmodium falciparum populations: A brief overview. Mem Inst Oswaldo Cruz 1998;93:631-8. [ Links ]
68. Conway DJ. Natural selection on polymorphic malaria antigens and the search for a vaccine. Parasitol Today 1997;13:26-9. [ Links ]
69. Scherf A, Mattei D, Sarthou JL. Multiple infections and unusual distribution of block 2 of the MSA1 gene of Plasmodium falciparum detected in West African clinical isolates by polymerase chain reaction analysis. Mol Biochem Parasitol 1991;44:297-9. [ Links ]
70. Babiker HA, Walliker D. Current view on the population structure of Plasmodium falciparum: implications for control. Parasitol Today 1997;13:262-7. [ Links ]
71. Jeffery GM. Epidemiological significance of repeated infections with homologous and heterologous strains and species of Plasmodium. Bull World Health Organ 1966;36:873-82. [ Links ]
72. Day KP, Marsh K. Naturally acquired immunity to Plasmodium falciparum. Immunol Today 1991;12:68-71. [ Links ]
73. Tomson GJ. Immunity in malaria. Trans R Soc Trop Med Hyg 1933;26:483-503.
74. Trape JF, Rogier C, Konate L, Diagne N, Bouganall H, Canque B et al. The Dielmo project: a longitudinal study of natural malaria infection and the mechanisms of protective immunity in a community living in a holoendemic area of Senegal. Am J Trop Med Hyg 1994;51:123-37. [ Links ]
75. Sergent E, Parrott L. L´immunité, la prémonition et la résistance innée. Arch Inst Pasteur Alger 1935;13:279-319. [ Links ]
76. Smith T, Felfer I, Tanner M, Beck, HP. The epidemiology of multiple Plasmodium falciparum infections. Premuition in Plasmodium falciparum infection: insights from the epidemiology of multiple infections. Trans R Soc Trop Med Hyg 1999;93:59-64. [ Links ]
77. Ross R. The prevention of malaria. London: Jhon Murray; 1910. [ Links ]
78. McGregor I. The development and maintenance of immunity to malaria in highly endemic areas. Clinics in Tropical Medicine and Communicable Disease 1986;1:29-53. [ Links ]
79. Contamin H, Fandeur T, Bonnefoy S, Skouri F, Ntoumi F, Mercereau-Puijalon O. PCR typing of field isolates of Plasmodium falciparum. J Clin Microbiol 1995;33:944-51. [ Links ]
80. Al-Yaman F, Genton B, Reeder JC, Mokela D, Anders RF, Alpers MP. Humoral response to defined Plasmodium falciparum antigens in cerebral and uncomplicated malaria and their relationship to parasite genotype. Am J Trop Med Hyg 1997;54:430-5. [ Links ]
81. Kwiatkowski D. Cytokines and anti-disease immunity to malaria. Res Immunol 1991;142:707-12. [ Links ]
82. Smith T, Beck H.P, Kitua A, Mwankusye S, Felger I, Fraser-Hurt N et al. The epidemiology of multiple Plasmodium falciparum infections. Age dependence of the multiplicity of Plasmodium falciparum infections and of other malariological indices in an area of high endemicity. Trans R Soc Trop Med Hyg 1999;93:15-20. [ Links ]
83. El-Hassan I, Hviid L, Jakobsen P, Giha H, Satti GM, Arnot D et al. High proportion of sub-clinical Plasmodium falciparum infections in an area of seasonal and unstable malaria in Sudan. Trans R Soc Trop Med Hyg 1995;53:78-83. [ Links ]
84. Roper C, Richardson W, El Hassan I, Hviid L, Giha H, Satti G et al. Seasonal changes in the Plasmodium falciparum populations in individuals and their relationship to clinical malaria: a longitudinal study in a Sudanese village. Parasitology 1998;116:501-10. [ Links ]
85. Giha HA, Rosthoj S, Dodoo D, Hviid L, Satii GM, Scheike T et al. The epidemiology of febrile malaria episodes in an area of unstable and seasonal transmission. Trans R Soc Trop Med Hyg 2000;94:645-51. [ Links ]
86. Cavanagh DR, Elhassan IM, Roper C, Robinson VJ, Giha H, Holder A et al. A longitudinal study of type-specific antibody responses to Plasmodium falciparum merozoite surface protein-1 in an area of unstable malaria in Sudan. J Immunol 1998;161:347-59. [ Links ]
87. Roper C, Elhassam IM, Hviid L, Giha H, Richardson W, Babiker H et al. Detection of very low level Plasmodium falciparum infections using the nested polymerase chain reaction and a reassessment of the epidemiology of unstable malaria in Sudan. Am J Trop Med Hyg 1996;54:325-31. [ Links ]
88. Mackinnon MJ, Hastings IM. The evolution of multiple drug resistance in malaria parasites. Trans R Soc Trop Med Hyg 1998;92:188-95. [ Links ]
89. Curtis C, Otoo L. A simple model for the buildup of resistance to mixtures of anti-malarial drugs. Trans R Soc Trop Med Hyg 1986;80:889-92. [ Links ]
90. Guiguemde TR, Aouba A, Ouedraogo JB, Lamizana L. Ten-year surveillance of drug-resistant malaria in Burkina Faso (1982-1991). Am J Trop Med Hyg 1994;50:699-704. [ Links ]














