Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.26 no.1 Bogotá Mar. 2006
Genotipificación de los genes
msp1 (bloque 2) y dhfr (codón108) de Plasmodium falciparum en muestras de campo recolectadas en cuatro localidades endémicas de ColombiaÁngela Patricia Guerra 1, Angélica Knudson 2, Rubén Santiago Nicholls 2, John Alexander Galindo 1, Zaava Ravid 1, Sonia Rahirant 1, Nidia Duarte 1, Jacqueline Chaparro-Olaya 1, Moisés Wasserman 3
1 Laboratorio de Bioquímica y Biología Celular, Instituto Nacional de Salud, Bogotá, D.C., Colombia.
2
Laboratorio de Parasitología, Instituto Nacional de Salud, Bogotá, D.C., Colombia.3
Laboratorio de Investigaciones Básicas en Bioquímica (LIBBIQ), Facultad de Ciencias, Universidad Nacional de Colombia, Bogotá, D.C., Colombia.Introducción. Plasmodium falciparum es un parásito altamente polimórfico, lo cual le permite evadir la respuesta inmune del hospedero, diseminar la resistencia a medicamentos y favorecer la transmisión.
Objetivos. Analizar la diversidad genética de las poblaciones de P. falciparum en muestras de cuatro zonas endémicas de malaria en Colombia.
Materiales y métodos. Se incluyeron muestras de sangre recolectadas en papel de filtro de 123 pacientes con malaria no complicada por P. falciparum durante los años 2002 a 2004; la genotipificación se realizó mediante reacción en cadena de la polimerasa con iniciadores específicos para los marcadores moleculares de la región polimórfica del bloque 2 del gen msp1 y del codón 108 de dhfr.
Resultados. En el bloque 2 del gen msp1 se detectó MAD20 en 95,9% (118/123; IC 95%: 90,8 a 98,7), K1 en 6,5% (8/123; IC 95%: 2,8 a 12,4) y RO33 en 2,4% (3/123; IC 95%: 0,5 a 6,9) de las muestras. Para el gen dhfr, el alotipo mutante N108 se detectó en todas las muestras analizadas y el alotipo T108 en 3,2% (4/123; IC 95%: 0,9 a 8,1); el alotipo silvestre S108 se encontró en 34,1% (42/123; IC 95%: 25,8 a 43,2). Al combinar los resultados de ambos genes, el 61,8% (76/123; IC 95%: 52,6 a 70,4) de las muestras correspondieron a infecciones simples y el 38,2% (47/123; IC 95%: 29,6 a 47,4) a infecciones mixtas, siendo MAD20/N108-S108 la combinación más frecuente entre estas últimas (30,1%).
Conclusiones. Las infecciones simples, o sea, la presencia de un solo alelo en cada uno de los genes, predominaron en las muestras estudiadas; las poblaciones de parásitos analizadas fueron muy homogéneas en su composición genética.
Palabras clave: Plasmodium falciparum, PCR, variación (genética), polimorfismo (genética), Colombia.
Genotyping of the Plasmodium falciparum msp1 (block 2) and dhfr (codon 108) genes in field samples collected in four endemic Colombian localities
Introduction. Plasmodium falciparum is a highly polymorphic parasite, which allows it to evade the host´s immune response, spread drug resistance and favours transmission. Objectives. To analyse the genetic diversity of P. falciparum populations in samples from four endemic localities in Colombia.
Materials and methods. 123 blood samples were collected on filter paper from patients with non-complicated P. falciparum malaria during 2002 to 2004. The samples were genotyped using polymerase chain reaction with specific primers for the polymorphic region of block 2 of the msp1 gene and the 108 codon of the dhfr gene.
Results. In msp1 block 2, 95.9% (118/123; 95% CI: 90.8-98.7) of the samples harboured MAD20; 6.5% K1 (8/123; 95% CI: 2.8-12.4) and 2.4% RO33 (3/123; 95% CI: 0.5-6.9). For the dhfr gene the mutant allele N 108 was found in all the samples amplified, T 108 in 3.2 % and the wild type S 108 in 34.1%. Taking together all the results from both genes, 61.8% (76/123; 95% CI: 52.6-70.4) of the samples were simple infections and 38.2% (47/123; 95% CI: 29.6-47.4) were mixed infections. MAD20/N108-S108 (30.1%) was the most frequent combination among the latter.
Conclusions. Simple infections, i.e, a single allelic type in each one of the genes studied, prevailed among the circulating parasite populations. In this study the genetic composition of P. falciparum parasite populations was very homogeneous.
Keywords: Plasmodium falciparum, PCR, variation (Genetics), polymorphism (Genetics), Colombia.
La malaria es la infección parasitaria más importante en el mundo y es considerada como el reto más grande que enfrentan los países del tercer mundo en el campo de salud pública. Se estima que cada año se presentan 300 millones de casos clínicos y entre 0,7 y 2,7 millones de muertes (1,2). Aunque hay cuatro especies del género Plasmodium que infectan a los seres humanos, casi todas las muertes y gran proporción de la morbilidad se deben a Plasmodium falciparum (3,4). En Colombia, la malaria representa un importante problema de salud pública en el 85% del territorio nacional y se estima que entre 18 y 24 millones de personas habitan zonas con riesgo de transmisión (5). De acuerdo con el Ministerio de la Protección Social, en Colombia se presentan aproximadamente entre 150.000 y 200.000 casos de malaria al año (6).
Las poblaciones naturales de P. falciparum son extraordinariamente diversas, lo cual favorece la transmisión de la infección, el aumento del riesgo de desarrollar patología grave, ya que permite que el parásito evada la respuesta inmune del hospedero, y la dispersión de resistencia a los medicamentos antimaláricos (7). Esta gran diversidad genética de las poblaciones de parásitos circulantes parece tener relación directa con la multiplicidad de la infección, la dinámica de las infecciones en el tiempo, la endemicidad de la malaria y la adquisición natural de inmunidad contra la enfermedad (8). Por consiguiente, conocer la estructura y dinámica de las poblaciones circulantes de parásitos en una región es importante, ya que puede facilitar la elección de estrategias de control más adecuadas (8).
El gen que codifica para la proteína 1 de superficie del merozoíto ( msp1) ha sido ampliamente utilizado para determinar la diversidad genética y geográfica de poblaciones de P. falciparum (9,10). La secuencia de msp1 ha sido dividida en 17 bloques de acuerdo con los niveles de divergencia interalélica, y la mayoría de la variación encontrada es dimórfica. Las secuencias pueden ser agrupadas en dos familias alélicas denominadas K1 y MAD20, las cuales contienen repeticiones de tripéptidos degenerados. El bloque 2 representa una excepción de ese dimorfismo, ya que presenta un alelo adicional no repetitivo denominado RO33 (11). Los rearreglos en el bloque 2 de msp1 y, por tanto, la diversidad de este gen, pueden ser generados por recombinación genética durante la reproducción sexual que se lleva a cabo en el vector (12), o por deslizamiento de cadenas ( strand-slippage) durante la reproducción asexual de los parásitos; esta última es menos frecuente (13, 14).
En la posición 108 del gen dhfr que codifica para la enzima dihidrofolato reductasa, se pueden presentar tres formas alélicas que corresponden al tipo silvestre serina (S), al tipo mutante asparagina (N) y al tipo mutante treonina (T) (15); aunque este polimorfismo está asociado con resistencia a pirimetamina y cicloguanil, en este trabajo se utilizó como marcador genético para el estudio de la diversidad de las poblaciones de parásitos.
En áreas con intensa transmisión de malaria (como algunas regiones de África) se han observado altas tasas de recombinación meiótica y un gran número de clones genéticamente distintos (16), mientras que en áreas con transmisión baja, como Brasil, o intermedia, como Vietnam, la generación de nuevos alelos es menos dinámica y está más relacionada con eventos de recombinación mitótica (13,14).
Estudios previos realizados en Colombia sobre la diversidad genética de P. falciparum mostraron que las poblaciones de parásitos eran genéticamente homogéneas al interior de cada región geográfica estudiada, aunque había algunas diferencias en la distribución entre regiones (17-20). En el presente trabajo se analizó la diversidad genética de las poblaciones de P. falciparum circulantes en cuatro zonas endémicas de malaria en Colombia y la variabilidad de la población de parásitos durante el tiempo de la infección malárica, utilizando como marcadores moleculares la región polimórfica del bloque 2 del gen msp1 y el codón 108 del gen dhfr.
Materiales y métodos
Áreas de estudio
El estudio se llevó a cabo en cuatro poblaciones colombianas endémicas para malaria, las cuales se seleccionaron con base en el índice parasitario anual (IPA). Las localidades fueron La Carpa, Casuarito, Tierralta y Puerto Libertador, ubicadas en las regiones de la Amazonia, la Orinoquia y del Urabá-bajo Cauca, respectivamente (
figura 1). La región de Urabá-bajo Cauca presentó un IPA de 42,98, la región de la Amazonia de 23,78 y la de la Orinoquia de 11,83, cifras superiores al IPA nacional de 5,5 registrado en el mismo año (datos correspondientes al año 2003). La región de Urabábajo Cauca presentó el mayor IPA por regiones, y dentro de ésta, los municipios de Puerto Libertador y Tierralta presentaron los mayores IPA, y aportaron el 38% de los casos de esa región (21).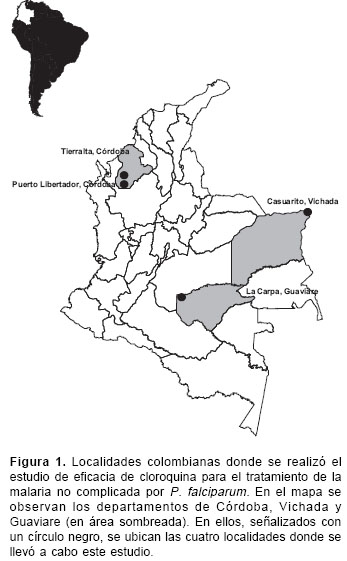
Recolección de muestras
Durante los años 2002 a 2004 se recolectaron 123 muestras de sangre de pacientes con malaria en el marco de un estudio de eficacia a la cloroquina para el tratamiento de la malaria no complicada por P. falciparum, siguiendo los protocolos estandarizados por la OMS y la OPS (22), con algunas modificaciones en los criterios de inclusión. Así, se seleccionaron pacientes de 2 o más años de edad que presentaran malaria no complicada con una parasitemia en el rango de 500 a 5.000 formas asexuales/µl de sangre.
El estudio fue aprobado por el Comité de Ética del Instituto Nacional de Salud y presentado, discutido y aprobado por parte de las autoridades departamentales de salud de los sitios en donde se realizó el trabajo de campo (23). Mediante la firma de un consentimiento escrito, los pacientes o los padres (o adultos responsables) en el caso de menores de edad, aceptaron voluntariamente participar en el estudio (24).
Antes de iniciar el tratamiento (día 0), a todos los pacientes se les tomó una muestra de sangre que se recolectó en círculos de papel filtro (Whatman 3 M) de 1 cm de diámetro. Además, al 44% (n=49) de los pacientes que presentaron falla terapéutica al tratamiento con cloroquina se les tomó una segunda muestra el día de la falla. La respuesta terapéutica de estos pacientes se clasificó en dos categorías: falla terapéutica precoz del tratamiento y falla terapéutica tardía del tratamiento (22).
Extracción de ADN del parásito
Se tomó la mitad del círculo del papel filtro impregnado con la sangre del paciente, se resuspendió en saponina al 0,5% (Sigma) y se incubó a 4°C durante toda la noche. El tubo con la muestra se centrifugó a 12.000 g durante 3 minutos, se lavó tres veces con solución salina amortiguada con fosfatos (PBS) y, luego, se resuspendió en Chelex-100 (Bio-Rad), incubando primero a 56°C durante 15 minutos y, luego, a 100°C durante 10 minutos. Se recuperó el AND presente en el sobrenadante, se distribuyó en alícuotas y se almacenó a -20°C (18,25).
Detección del alotipo en el bloque 2 del gen msp1
La mezcla de la reacción en cadena de la polimerasa (PCR universal) se ajustó a un volumen final de 25 µl y se preparó con 200 µM de dNTP, 0,5 µM de los iniciadores OK11 y OK12 (
cuadro 1), 1,5 mM de MgCl2 y 2,5 U de Taq ADN polimerasa (Promega). El programa de amplificación consistió en un ciclo de denaturación inicial a 94°C durante 2 minutos, seguido por 35 ciclos a 94°C durante 30 segundos, a 55°C durante 30 segundos y a 72°C durante 2 minutos, y una extensión final a 72°C durante 2 minutos. Para definir el alotipo presente se realizó PCR anidada utilizando los iniciadores OK1 y OK2 para el alotipo K1, OK3 y OK4 para el alotipo MAD20 y OK5 y OK6 para el alotipo RO33 (cuadro1), usando las mismas condiciones de la PCR universal. Como control positivo se utilizó ADN de las cepas de referencia Haiti135 (para el alotipo K1), FCB1 (para el alotipo MAD20) y 7G8 (para el alotipo RO33) (19).
Identificación del genotipo en el codón 108 del gen dhfr
El gen dhfr fue amplificado usando los iniciadores AMP1 y AMP2 (cuadro 1). La PCR (universal) se realizó en un volumen final de 20 µl usando 200 µM de dNTPs, 1 µM de cada iniciador, 2,5 U de Taq ADN polimerasa (Promega) y 1,5 mM de MgCl2. El programa de amplificación consistió en un ciclo inicial de denaturación a 94°C durante 3 minutos, 45 ciclos a 94°C durante 30 segundos, a 45°C durante 45 segundos y a 72°C durante 45 segundos, y una extensión final a 72°C durante 5 minutos. Para la segunda PCR (PCR anidada) se usó como iniciador sentido SP1 en todas las reacciones y los iniciadores antisentido DIA3, DIA9 y DIA12 para definir el genotipo silvestre serina-108 (S108), el genotipo mutante treonina- 108 (T108) y el genotipo mutante asparagina-108 (N108), respectivamente (cuadro 1). Las reacciones de la segunda PCR se realizaron utilizando las mismas condiciones descritas para la PCR universal. El programa de amplificación consistió en un ciclo inicial de denaturación a 94°C durante 3 minutos, 20 ciclos a 94°C durante 30 segundos, a 55°C durante 45 segundos y a 74°C durante 45 segundos, y un ciclo final de extensión a 74°C durante 5 minutos. Como control positivo se utilizó ADN de las cepas de referencia 3D7 (para el alotipo S108), FCB1 (para el alotipo T108) y T4 (para el alotipo N108) (19, 26).
Complejidad de la infección
Las infecciones se clasificaron en simples o mixtas con base en la detección de uno (infección simple) o más (infección mixta) alelos de cada gen en cada muestra evaluada (19). Una muestra se consideró como simple cuando en los ensayos de PCR se produjo señal sólo para un alelo del gen msp1 y sólo para un alelo del gen dhfr.
Análisis de los productos de PCR
Los productos de PCR para los dos marcadores genéticos probados fueron resueltos por electroforesis en gel de agarosa al 2%, teñidos en solución acuosa de 1 µg/ml de bromuro de etidio, visualizados sobre luz UV y analizados por el programa One-Dscan one-dimensional electrophoresis (Scanalytic Fairfax, VA). Se creó una base de datos en Microsoft Excel (Office 2000 para Windows) en la que se registró la información de cada paciente. Para todos los análisis se calcularon los intervalos de confianza del 95% (IC 95%).
Resultados
En el estudio se incluyeron 52 pacientes de Tierralta, 53 de Puerto Libertador, 10 de La Carpa y 8 de Casuarito, para un total de 123 pacientes. Ciento doce pacientes no respondieron al tratamiento con cloroquina, 46 con falla terapéutica tardía y 66 con falla terapéutica precoz.
Genotipificación del bloque 2 de msp1
Se detectaron las 3 formas alélicas del bloque 2 del gen msp1 (MAD20, K1 y RO33) y, en todos los casos, el tamaño del producto de PCR obtenido para el alotipo K1 fue de 210 pb, aproximadamente; el del alotipo MAD20 fue de 150 pb, aproximadamente; y el del alotipo RO33 fue de 190 pb, aproximadamente. Se encontró que en el total de la población estudiada la frecuencia de MAD20 fue de 95,9% (118/123) (IC 95%: 90,8 a 98,7), la de K1 fue de 6,5% (8/123) (IC 95%: 2,8 a 12,4) y la de RO33 fue de 2,4% (3/123) (IC 95%: 0,5 a 6,9). El alotipo MAD20 se presentó con una frecuencia de 100% en La Carpa y en Puerto Libertador; de 92,3% (48/52) (IC 95%: 81,5 a 97,9) en Tierralta, y de 87,5% (7/8) (IC 95%: 47,3 a 99,7) en Casuarito. El alotipo K1 se encontró en Tierralta y en Casuarito con frecuencias de 11,5% (6/52) (IC 95%: 4,4 a 23,4) y 25% (2/8) (IC 95%: 3,2 a 65,1), respectivamente. No se encontró el alotipo K1 en La Carpa ni en Puerto Libertador. El alelo RO33 se halló en tres de los sitios estudiados: en Tierralta, con una frecuencia de 1,9% (1/52) (IC 95%: 0,05 a 10,3); en Puerto Libertador, también con una frecuencia de 1,9% (1/53) (IC 95%: 0,05 a 10,1), y en Casuarito con una frecuencia de 12,5% (1/8) (IC 95%: 0,3 a 52,7).
Genotipificación del codón 108 de dhfr
Para el gen dhfr, en las 123 muestras analizadas se detectó el alotipo mutante N108. Se observó también una frecuencia de 34,1% (42/123) (IC 95%: 25,8 a 43,2) para el alotipo silvestre S108 y de 3,2% (4/123) (IC 95%: 0,9 a 8,1) para el alotipo mutante T108. La presencia del alotipo S108 se observó en todos los sitios de estudio a excepción de Casuarito, y la mutación T108 se observó únicamente en Tierralta y Puerto Libertador.
Complejidad de la infección
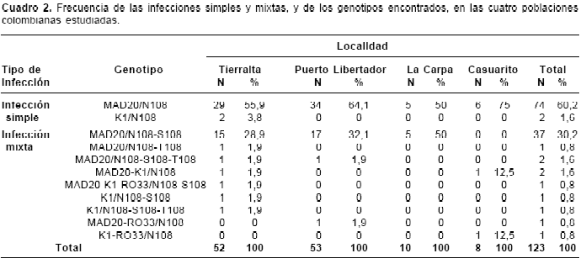
De las 123 muestras, 76 (61,8%; IC 95%: 52,6 a 70,4) correspondieron a infecciones simples y 47 (38,2%; IC 95%: 29,6 a 47,4) a infecciones mixtas. Entre las infecciones simples se encontraron 2 genotipos, MAD20/N108 con una frecuencia de 60,2% (74/123) (IC 95%: 50,9 a 68,9) y K1/N108 con una frecuencia de 1,6% (2/ 123) (IC 95%: 0,2 a 5,8), y para las infecciones mixtas hubo 9 combinaciones distintas, siendo la de mayor frecuencia la MAD20/N108-S108 con 30,2% (37/123) (IC 95%: 22,1 a 39). Las 8 combinaciones restantes fueron MAD20/N108- T108-S108 y MAD20-K1/N108, con una frecuencia de 1,6% cada una (2/123) (IC 95%: 0,2 a 5,8), y MAD20/N108-T108, MAD20-RO33/N108, MAD20- K1-RO33/N108-S108, K1/N108-S108, K1/N108- S108-T108 y K1-RO33/N108, con una frecuencia cada una de 0,8% (1/123) (IC 95%: 0,02 a 4,4).
Los resultados de la complejidad de la infección en cada una de las localidades estudiadas se presentan en el cuadro 2. Casuarito tuvo la mayor frecuencia de infecciones simples con 75% (6/8) (IC 95%: 34,9 a 96,8), seguida por Puerto Libertador con 64,1% (34/53) (IC 95%: 49,8 a 76,9), Tierralta con 59,7% (31/52) (IC 95%: 45,1 a 73) y La Carpa con 50% (5/10) (IC 95%: 18,7 a 81,3). La mayor frecuencia de infecciones mixtas se encontró en La Carpa con 50% (5/10) (IC 95%: 18,7 a 81,3) y el genotipo presente en las cinco muestras fue MAD20/N108-S108. En Tierralta, aunque la frecuencia fue menor (40,3%) (IC 95%: 27 a 54,9) que la encontrada en La Carpa, sobresale el hecho de haber detectado siete genotipos distintos (cuadro 2). En Puerto Libertador se encontraron tres combinaciones distintas de alelos y en Casuarito, dos (cuadro 2).
Variabilidad genética de la infección por P. falciparum en el tiempo
Se evaluó el 44% de las muestras de los pacientes que presentaron falla al tratamiento con cloroquina (n=112); así, se analizaron 49 muestras pareadas (tanto del día 0 como del día de la falla) con los marcadores msp1 y dhfr. Cuando se genotipificaron las muestras del día de la falla con msp1 (bloque 2) sólo se obtuvo amplificación en 42 muestras, y cuando se utilizó dhfr (codón 108) sólo hubo productos de PCR en 23 muestras; con el resto de las muestras no se logró amplificar ninguna de las formas alélicas. Teniendo en cuenta lo anterior, sólo 22 muestras pudieron analizarse en forma pareada: 8 provenían de Tierralta, 6 de Puerto Libertador, 3 de La Carpa y 5 de Casuarito.
Los resultados obtenidos se muestran en el
cuadro 3. En general no hubo variación en el genotipo de las muestras pareadas de los pacientes que se analizaron; de las 22 muestras, 15 conservaron el mismo genotipo tanto en el día 0 como en el día de la falla, y 7 presentaron cambios. Las muestras de dos pacientes de Tierralta y dos de Casuarito fueron clasificadas en el día 0 como infecciones simples y para el día de la falla se detectaron infecciones mixtas, y dos muestras de Puerto Libertador y una de la Carpa iniciaron como infecciones mixtas y para el día de la falla la infección se había transformado en simple.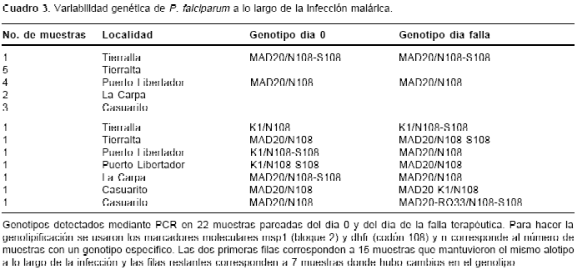
Discusión
Los resultados de las cuatro localidades indican que las poblaciones naturales de P. falciparum que circulan en estas áreas son muy homogéneas. Aunque se encontraron nueve genotipos distintos distribuidos entre los 47 pacientes con infección mixta, sólo detectamos 3 alelos para el gen msp1 (MAD20 de 150 pb, aproximadamente, RO33 de 190 pb y K1 de 210 pb) y los tres alelos posibles (S108, N108 y T108 de 339 pb) para el codón 108 del gen dhfr.
Estos resultados concuerdan con los de un estudio que se realizó en la región de Urabá-bajo Cauca con muestras de pacientes recolectadas entre los años 2000 y 2001, en el cual la cantidad de alelos diferentes para los genes msp1, msp2 y glurp fue muy baja; MAD20 de 147 pb fue el único alelo encontrado para msp1 (20). Además, otro estudio realizado en una localidad de la Costa Pacífica colombiana con muestras tomadas entre julio y septiembre de 1997, reportó 4 alelos para msp1; MAD20-1 (218 pb), MAD20-2 (238 pb), K1 (210 pb) y RO33 (190 pb) (19). En nuestros resultados, al igual que en los de Montoya y Gómez, el alelo predominante para el gen msp1 fue MAD20. Aunque a simple vista los resultados de los tres estudios muestran alelos de diferentes tamaños, situación que se podría interpretar como variación y cambio de alelos en el tiempo, debe tenerse cautela en el análisis de estas observaciones, ya que se tiene evidencia del alto grado de variabilidad entre laboratorios cuando se comparan los resultados de distintos estudios epidemiológicos (27). Resulta interesante que al comparar los resultados de los tres estudios con los deSnewin (17), se observa cómo han ido cambiando las poblaciones de parásitos a lo largo del tiempo. Así, antes de 1991 el alelo predominante en Colombia era RO33, con una frecuencia de 83,8%, seguido por MAD20 con 48,3% y K1 con 9,6%. De la misma forma, el alelo K1 ha disminuido progresivamente su frecuencia como lodemuestran nuestros hallazgos (6,5%) y los de Montoya (0%). Gómez, sin embargo, reportó una frecuencia alta para K1 (11%), pero esto pudo corresponder a una propagación de tipo epidémico teniendo en cuenta que las muestras de los pacientes se tomaron durante un brote de malaria en una población residente en un área pequeña y aislada, donde la tasa de migración era baja (14,28).
Nuestros resultados y, en general, los de otros autores colombianos contrastan fuertemente con los de países africanos, donde se pueden encontrar un sinnúmero de alelos para msp1, como fuereportado en estudios realizados en Uganda, donde se encontraron 45 alelos distintos (26 para K1, 13 para MAD20 y 6 para RO33) (10); en Senegal, donde se detectaron 36 alelos (21 para K1, 13 para MAD20 y 2 para RO33) (29), y en Nigeria, que presentó 9 alelos diferentes (4 para K1, 2 para MAD20 y 2 para RO33) (30).
El escaso polimorfismo encontrado en nuestros resultados coincide con lo reportado por Sallenave- Sales en dos localidades brasileras donde detectaron cinco alelos para el gen msp1 (2 para K1, 2 para MAD20 y 1 para RO33) (31) y por Haddad en Honduras, quien encontró 4 alelos (2 para K1 y 2 para MAD20) (32).
Ya que nuestros resultados aportan información de tres regiones geográficas distantes unas de otras y donde la población humana mantiene altas tasas de movilidad debido a las condiciones sociales y económicas, estos aspectos sugieren que el escaso polimorfismo alélico encontrado es la condición general en Colombia, a pesar de la posible migración de poblaciones humanas entre regiones endémicas de malaria.
Aunque Colombia es considerada una zona de baja transmisión comparada con otras regiones en África y Asia, la localidad de Tierralta presenta índices parasitarios anuales de más de 10, mayores que el promedio nacional, lo que la define como una región de alto riesgo de transmisión de malaria en el país (21). Esto sugiere que en Tierralta es más probable encontrar un mayor número de alelos y, por ello, a diferencia de las otras localidades, se presenta con mayor frecuencia el alelo K1. Sin embargo, existen otros factores que pueden afectar la estructura de las poblaciones de parásitos como son el esquema terapéutico empleado, la movilidad de las poblaciones humanas, las características ecoepidemiológicas de la región y la inmunidad del hospedero (8,9).
La complejidad de la infección también se determinó utilizando cada gen por separado; así, cuando sólo se usó msp1, se obtuvo un porcentaje de infecciones simples de 96%, y de mixtas de 4%. Cuando se utilizó sólo dhfr, el porcentaje de infecciones simples fue de 65% y el de mixtas de 35%. Aunque, los genes que codifican para antígenos de superficie se utilizan para llevar a cabo los estudios de diversidad genética, en este trabajo se puede observar que los resultados con el gen dhfr aportaron una mayor información para determinar la complejidad de la infección malárica. Lo anterior sugiere que, a diferencia de los estudios realizados en zonas de alta transmisión, donde algunos autores aconsejan el uso de un solo gen para genotipificar muestras del día 0 y del día de la falla al tratamiento (10,30), en zonas de baja transmisión es necesario el uso de varios marcadores moleculares para establecer realmente la diversidad genética de la población de parásitos circulantes. El uso del marcador dhfr permitió detectar 42 infecciones que contenían, además del alelo mutante N108, el alelo silvestre S108. Casuarito, localidad que tuvo el mayor porcentaje de infecciones simples, no presentó ninguna muestra con el genotipo S108. Esta situaciónpodría explicarse por el hecho de que en estalocalidad fronteriza con Venezuela posiblemente la presión ejercida con sulfadoxina-pirimetamina (S-P) ha ido eliminando gradualmente el genotipo silvestre.
Aunque la mutación puntual N108 se considera clave en el desarrollo de resistencia a los antifolatos, se necesita un efecto acumulativo de mutaciones en los genes dhfr y dhps para que ésta se desarrolle (33,34). En Suramérica se han observado niveles importantes de resistencia a S-P relacionados con ciertas mutaciones puntuales en estos genes. Así, en Venezuela, país donde se presentan altos niveles de resistenciaclínica a S-P, la frecuencia de la mutación N108 es del 96%. Estos genotipos resistentes han tenido una difusión masiva, debido a que la propagación clonal ha facilitado su dispersión, aumentando la diseminación de la multirresistencia a antimaláricos (35-37). La prevalencia y alta frecuencia de la mutación N108 en las poblaciones colombianas estudiadas (18, 19,34,38), puede ser una señal de alerta que indica la necesidad de vigilar la aparición de mutaciones en otros codones en los genes dhfr y dhps.
Al comparar los resultados del día 0 y del día de la falla, 15 de 22 muestras conservaron el mismo genotipo, lo cual demuestra la permanencia de las poblaciones de parásitos a lo largo de la infección. Esta permanencia de las poblaciones puede darse por varios factores que permiten la selección de cepas, entre ellos, la presión con medicamentos, la cual genera resistencia (8, 39). Los resultados de los estudios de eficacia in vivo de tratamiento de la malaria no complicada por P. falciparum con cloroquina, a partir de los cuales se tomaron las muestras para el presente estudio de diversidad genética, mostraron un porcentaje global de falla terapéutica de 93,4%, con la presencia del alelo mutante K76T en el gen pfcrt en todas las muestras de P. falciparum estudiadas tanto en el día 0 como en el día de la falla (datos sin publicar). Esto indica que las poblaciones de parásitos con los distintos alelos de msp1 o de dhfr son resistentes a cloroquina y predominan ampliamente en estas áreas, limitando la circulación de otras poblaciones. Otro de los mecanismos de selección es la evasión del parásito a la respuesta inmune, gracias a la capacidad que tienen ciertas poblaciones de evadir la inmunidad del hospedero activando los mecanismos de variación antigénica, lo cual determina las poblaciones circulantes en sangre periférica y puede contribuir a un aumento de la virulencia (40).
En las siete muestras restantes, a diferencia del caso anterior, se detectaron nuevas poblaciones de parásitos. Estos casos se consideran recrudescencias y pertenecen al tipo en el cual se observa, en infección simple o mixta, la presencia de bandas idénticas en las muestras pre y postratamiento, con la aparición de una banda adicional después del tratamiento (10,41). Se descartó la posibilidad de una reinfección porque todas las fallas (22) se dieron dentro de los primeros 14 días de tratamiento (datos sin publicar), y la tasa de picadura del vector no es lo suficientemente alta para que ocurra este evento (42).
En Puerto Libertador dos muestras del día 0 con el genotipo K1/N108-S108 sufrieron un cambio en el día de la falla: desapareció el alelo K1 y se detectó MAD20. Esta desaparición de alelos se puede atribuir a mecanismos que incluirían el secuestro vascular de parásitos, la competencia de poblaciones, la respuesta inmune específica del huésped o la presencia de gametocitemia, lo que puede confundir los análisis de genotipificación (10,43), ya que los resultados pueden ser clasificados erróneamente. También podría atribuirse a limitaciones en la sensibilidad de la PCR (10,44); así, cuando se tienen diferentes poblaciones y una presenta menor densidad parasitaria con respecto a las otras, la PCR no tiene la suficiente sensibilidad para detectar la cepa que está en menor cantidad (45). Las limitaciones en la sensibilidad de la PCR, la presencia de inhibidores (por ejemplo hemoglobina) de la PCR en el ADN extraído, la baja eficiencia en la extracción del ADN o el efecto del almacenamiento y la metodología de recolección de las muestras (temperatura, humedad, calidad del papel de filtro) (46,47) podrían explicar la ausencia de amplificación en las 27 muestras correspondientes al día de la falla terapéutica.
En conclusión, las poblaciones de parásitos analizadas se caracterizaron por ser homogéneas genéticamente, observándose una amplia circulación de parásitos que presentan una alta frecuencia del alelo MAD20 en msp1 y de la mutación N108 en el gen dhfr. Estos resultados contribuyen al entendimiento de la transmisión de la malaria en Colombia y podrían ser útiles para la vigilancia y el control de la resistencia de parásitos a los antimaláricos.
Conflicto de intereses
Los autores manifestamos expresamente que durante la realización del presente trabajo no existió conflicto de interés alguno que pudiera haber afectado los resultados obtenidos.
Financiación
Este estudio fue cofinanciado con recursos del Fondo de Investigaciones en Salud, Ministerio de la Protección Social-Colciencias, Proyecto Código 2104-04-11813, Contrato No. RC 134-2002, y del Instituto Nacional de Salud.
John Alexander Galindo estuvo vinculado a este proyecto gracias al Programa Jóvenes Investigadores e Innovadores de Colciencias, convenio interadministrativo especial de cooperación No. 92-2003 Colciencias-INS y el contrato de prestación de servicios profesionales No. 014-2004.
Correspondencia:
Ángela Patricia Guerra, Laboratorio de Bioquímica y Biología
Celular, Instituto Nacional de Salud, Avenida calle 26 No. 51-60, Bogotá, D.C.
Teléfono: 2 207700, ext. 425
Recibido:16/09/05; aceptado: 27/12/05
Referencias
1. Ranjit MR, Das A, Das BP, Das BN, Dash BP, Chhotray GP. Distribution of Plasmodium falciparum genotypes in clinically mild and severe malaria cases in Orissa, India. Trans R Soc Trop Med Hyg 2005;99: 389-95. [ Links ]
2. World Health Organization. Infectious Diseases. Malaria. Fact sheet No. 94. Consultado: 2005 diciembre 3. Disponible en:
http://mosquito.who.int/cmc_upload//000/015/372/RBMInfosheet_1.htm.3. World Health Organization. World Malaria Report. 2005. [ Links ] Consultado: 2005 diciembre 3. Disponible en:
http://rbm.who.int/wmr2005/pdf/adv_e.pdf.4. Guerin PJ, Olliaro P, Nosten F, Druilhe P, Laxminarayan R, Binka F et al. Malaria: current status of control, diagnosis, treatment, and a proposed agenda for research and development. Lancet Infect Dis 2002;2:564-73. [ Links ]
5. Ministerio de Salud. Situación de las enfermedades transmisibles objeto de vigilancia intensificada en salud pública, Colombia, 2002. SIVIGILA: semanas epidemiológicas 47 y 48 (18 al 9 de noviembre de 2002). Inf Quinc Epidemiol Nac 2002;7:463-78. Disponible en:
http://www.ins.gov.co/iquen/2002_iqen_24.pdf. [ Links ]6. Ministerio de la Protección Social. Malaria en Inírida- Guainía 2002-2003. SIVIGILA: semana epidemiológica 43 y 44 (20 al 31 de octubre de 2003). Inf Quinc Epidemiol Nac 2003;8:385-400. Disponible en:
http://www.ins.gov.co/iquen/2003_iqen_24.pdf. [ Links ]7. Babiker HA, Lines J, Hill WG, Walliker D. Population structure of Plasmodium falciparum in villages with different malaria endemicity in east Africa. Am J Trop Med Hyg 1997;56:141-7. [ Links ]
8. Snounou G, Zhu X, Siriporn N, Jarra W, Thaithong S, Brown KN et al. Biased distribution of msp1 and msp2 allelic variants in Plasmodium falciparum populations in Thailand. Trans R Soc Trop Med Hyg 1999;93:369-74. [ Links ]
9. Babiker HA, Ranford-Cartwright LC, Walliker D. Genetic structure and dynamics of Plasmodium falciparum infections in the Kilombero region of Tanzania. Trans R Soc Trop Med Hyg 1999;93(Suppl.1):11-4. [ Links ]
10. Cattamanchi A, Kyabayinze D, Hubbard A, Rosenthal PJ, Dorsey G. Distinguishing recrudescence from reinfection in a longitudinal antimalarial drug efficacy study: comparison of results based on genotyping of msp-1, msp-2, and glurp. Am J Trop Med Hyg 2003;68:133-9. [ Links ]
11. Tanabe K, Mackay M, Goman M, Scaife JG. Allelic dimorphism in a surface antigen gene of the malaria parasite Plasmodium falciparum. J Mol Biol 1987;195:273-87. [ Links ]
12. Kerr PJ, Ranford-Cartwright LC, Walliker D. Proof of intragenic recombination in Plasmodium falciparum. Mol Biochem Parasitol 1994;66:241-8. [ Links ]
13. Ferreira MU, Ribeiro WL, Tonon AP, Kawwamoto F, Rich SM. Sequence diversity and evolution of the malaria vaccine candidate merozoite surface protein-1 (MSP-1) of Plasmodium falciparum. Gene 2003;304:65-75. [ Links ]
14. Hoffmann EH, Ribolla PE, Ferreira MU. Genetic relatedness of Plasmodium falciparum isolates and the origin of allelic diversity at the merozoite surface protein- 1 (MSP-1) locus in Brazil and Vietnam. Malar J 2003;2:24. [ Links ]
15. Foote SJ, Galatis D, Cowman AF. Amino acids in the dihydrofolate reductase-thymidylate synthase gene of Plasmodium falciparum involved in cycloguanil resistance differ from those involved in pyrimethamine resistance. Proc Natl Acad Sci USA 1990;87:3014-7. [ Links ]
16. Conway DJ, Roper C, Odouola AM, Arnot D, Kremsner PG, Grobuschi MP et al. High recombination rate in natural populations of Plasmodium falciparum. Proc Natl Acad Sci USA 1999;96:4506-11. [ Links ]
17. Snewin VA, Herrera M, Sanchez G, Scherf A, Langsley G, Herrera S. Polymorphism of the alleles of the merozoite surface antigens MSA1 and MSA2 in Plasmodium falciparum wild isolates from Colombia. Mol Biochem Parasitol 1991;49:265-75. [ Links ]
18. Giraldo LE, Acosta MC, Labrada LA, Praba A, Montenegro-James S, Saravia NG et al. Frequency of the Asn-108 and Thr-108 point mutations in the dihydrofolate reductase gene in Plasmodium falciparum from southwest Colombia. Am J Trop Med Hyg 1998;59:124-8. [ Links ]
19. Gómez D, Chaparro J, Rubiano C, Rojas MO, Wasserman M. Genetic diversity of Plasmodium falciparum field samples from an isolated Colombian village. Am J Trop Med Hyg 2002;67:611-6. [ Links ]
20. Montoya L, Maestre A, Carmona J, Lopes D, Do Rosario V, Blair S. Plasmodium falciparum: diversity studies of isolates from two Colombian regions with different endemicity. Exp Parasitol 2003;104:14-9. [ Links ]
21. Ministerio de la Protección Social. Enfermedades transmitidas por vectores (ETV). SIVIGILA: semana Epidemiológica 45-53 (5 al 16 de enero de 2004). Inf Quinc Epidemiol Nac 2004;9:1-16. Disponible en:
http://www.ins.gov.co/iquen/2004_iqen_01.pdf [ Links ]22. Organización Panamericana de la Salud. Evaluación de la eficacia terapéutica de los medicamentos para el tratamiento del paludismo por Plasmodium falciparum sin complicaciones en las Américas. (OPS/HCP/HCT/ 113/98). Washington DC.: Panamerican Health Organization; 1998. [ Links ]
23. World Medical Association Declaration of Helsinki. Ethical principles for medical research involving human subjects. Edinburgh, Scotland. October 2000. Disponible en: http://www.wma.net/e/policy/b3.htm. [ Links ] 24. Ministerio de Salud. Código del menor. Derechos del niño. Bogotá: Ministerio de Salud; 1989. [ Links ] 25. Kain KC, Lanar DE. Determination of genetic variation within Plasmodium falciparum by using enzymatically amplified DNA from filter paper disks impregnated with whole blood. J Clin Microbiol 1991;29:1171-4. [ Links ] 26. Plowe C, Djimde A, Bouare M, Doumbo O, Wellems TE. Pyrimethamine and proguanil resistance conferring mutations in Plasmodium falciparum dihydrofolate reductase: polymerase chain reaction methods for surveillance in Africa. Am J Trop Med Hyg 1995;52:565-8. [ Links ] 27. Färnert A, Arez AP, Babiker HA, Beck HP, Benito A, Bjorkman A et al. Genotyping of Plasmodium falciparum infections by PCR: a comparative multicenter study. Trans R Soc Trop Med Hyg 2001;95:225-32. [ Links ] 28. Smith JM, Smith NH, ORourke M, Spratt BG. How clonal are bacteria? Proc Natl Acad Sci USA 1993;90:4384-8. [ Links ] 29. Konate L, Zwetyenga J, Rogier C, Bischoff E, Fontenille D, Tall A et al. Variation of Plasmodium falciparum msp1 block 2 and msp2 allele prevalence and of infection complexity in two neighbouring Senegalese villages with different transmission conditions. Trans R Soc Trop Med Hyg 1999;93(Suppl 1):21-8. [ Links ] 30. Happi CT, Gbotosho GO, Sowunmi A, Falade CO, Akinboye DO, Gerena L et al. Molecular analysis of Plasmodium falciparum recrudescent malaria infections in children treated with chloroquine in Nigeria. Am J Trop Med Hyg 2004;70:20-6. [ Links ] 31. Sallenave-Sales S, Daubersies P, Mercereau-Puijalon O, Rahimalala L, Contamin H, Druilhe P et al. Plasmodium falciparum: a comparative analysis of the genetic diversity in malaria-mesoendemic areas of Brazil and Madagascar. Parasitol Res 2000;86:692-8. [ Links ] 32. Haddad D, Snounou G, Mattei D, Enamorado IG, Figueroa J, Stahls S et al. Limited genetic diversity of Plasmodium falciparum in field isolates from Honduras. Am J Trop Med Hyg 1999;60:30-4. [ Links ] 33. Sibley CH, Hyde JE, Sims PF, Plowe CV, Kublin JG, Mberu EK et al. Pyrimethamine-sulfadoxine resistance in Plasmodium falciparum: what next? Trends Parasitol 2001;17:582-8. [ Links ] 34. Schmider N, Peyerl-Hoffmann G, Restrepo M, Jelinek T. Short communication: point mutations in the dihydrofolate reductase and dihydropteroate synthase genes of Plasmodium falciparum isolates from Colombia. Trop Med Int Health 2003;8:129-32. [ Links ] 35. Cortese JF, Caraballo A, Contreras CE, Plowe CV. Origin and dissemination of Plasmodium falciparum drugresistance mutations in South America. J Infect Dis 2002;186:999-1006. [ Links ] 36. Urdaneta L, Plowe C, Goldman I, Lal AA. Point mutation in dihydrofolate reductase and dihydropteroate synthase genes of Plasmodium falciparum isolates from Venezuela. Am J Trop Med Hyg 1999;61:457-62. [ Links ] 37. Urdaneta L, Lal A, Barnabé C, Oury B, Goldaman I, Ayala FJ et al. Evidence for clonal propagation in natural isolates of Plasmodium falciparum from Venezuela. Proc Natl Acad Sci USA 2001;98:6725-9. [ Links ] 38. Rallón NI, Osorio LE, Giraldo LE. Lack of an association between the Asn-108 mutation in the dihydrofolate reductase gene and in vivo resistance to sulfadoxine/pyrimethamine in Plasmodium falciparum. Am J Trop Med Hyg 1999;61:245-8. [ Links ] 39. Blair-Trujillo S, Lacharme-Lora L, Carmona- Fonseca J. Resistance of Plasmodium falciparum to antimalarial drugs in Zaragoza (Antioquia, Colombia), 1998. Mem Inst Oswaldo Cruz 2002;97:401-6. [ Links ] 40. Sinden R, Pilles HM. The malaria parasites. In: Warrell DA, Pilles HM, editors. Essential malariology. 4 ed. London: Arnold; 2002. p.18. [ Links ] 41. Basco LK, Ndounga M, Ngane VF, Soula G. Molecular epidemiology of malaria in Cameroon. XIV. Plasmodium falciparum chloroquine resistance transporter (pfcrt) gene sequences of isolates before and after chloroquine treatment. Am J Trop Med Hyg 2002;67:392-5. [ Links ] 42. Gonzalez JM, Olano V, Vergara J, Arevalo-Herrera M, Carrasquilla G, Herrera S et al. Unstable, lowlevel transmission of malaria on the Colombian Pacific Coast. Ann Trop Med Parasitol 1997;91:349-58. [ Links ] 43. Aubouy A, Migot-Nabias F, Deloron P. Polymorphism in two merozoite surface proteins of Plasmodium falciparum isolates from Gabon. Malar J 2003;2:12. [ Links ] 44. Ranford-Cartwright LC, Johnston KL, Abdel- Muhsin AM, Khan BK, Babiker HA. Critical comparison of molecular genotyping methods for detection of drug-resistant Plasmodium falciparum. Trans R Soc Trop Med Hyg 2002;96:568-72. [ Links ] 45. Chaparro J, Rojas MO, Wasserman M. Plasmodium falciparum: underestimation of dihydrofolate reductase and dihydropteroate synthase polymorphism in field samples: a technical shortcoming of nested PCR assays with mutation-specific primers. Exp Parasitol 2001;99:115-22. [ Links ] 46. Chaorattanakawee S, Natalang O, Hananantachai H, Nacher M, Brockman A, Krudsood S et al. Storage duration and polymerase chain reaction detection of Plasmodium falciparum from blood spots on filter paper. Am J Trop Med Hyg 2003;69:42-4. [ Links ] 47. Färnert A, Arez AP, Correia AT, Bjorkman A, Snounou G, do Rosario V. Sampling and storage of blood and the detection of malaria parasites by polymerase chain reaction. Trans R Soc Trop Med Hyg 1999;93:50-3














