Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.26 no.2 Bogotá June 2006
Caracterización molecular de aislamientos invasores colombianos de Streptococcus pneumoniae serotipo 5 recuperados entre 1994 y 2004
Carolina Firacative, Jaime Moreno, Elizabeth Castañeda
Grupo de Microbiología, Instituto Nacional de Salud, Bogotá, D.C., Colombia.
Introducción. Streptococcus pneumoniae serotipo 5 es causa importante de enfermedad invasora en Colombia, donde se ha demostrado la circulación del clon 19-Colombia5 susceptible a penicilina pero resistente a tetraciclina y a cloranfenicol.
Objetivo. Establecer las relaciones genéticas de aislamientos invasores colombianos de S. pneumoniae serotipo 5 recuperados entre 1994 y 2004 con el clon 19-Colombia5.
Materiales y métodos. Se estudiaron 83 aislamientos que tenían datos de susceptibilidad a penicilina, vancomicina, ceftriaxona, eritromicina, trimetoprim sulfametoxazol, cloranfenicol y tetraciclina, de los cuales 29 fueron obtenidos de niños menores de cinco años. El patrón de restricción del ADN se determinó por electroforesis en gel de campo pulsado, usando la enzima Sma I. La similitud genética entre los aislamientos y el clon se estableció según los criterios de Tenover y utilizando el programa Fingerprinting II.
Resultados. Todos los aislamientos se relacionaron con el clon 19-Colombia5 y se agruparon en el patrón electroforético A con 17 subtipos. El patrón A se estableció en 32 aislamientos (38,6%), el A8 en 18 (21,7%) y el A5 en 10 (12%), los 15 patrones restantes agruparon los otros 23 aislamientos. Los 34 aislamientos resistentes a tetraciclina y cloranfenicol mostraron relación con los patrones electroforéticos A (n = 32), A16 (n = 1) y A28 (n = 1), caracterizados, al igual que el clon, por presentar una banda de 340 Kb.
Conclusión. Estos resultados muestran la circulación continua en el país de aislamientos de S. pneumoniae serotipo 5 genéticamente relacionados con el clon 19-Colombia5.
Palabras clave: Streptococcus pneumoniae, meningitis, neumonía, electroforesis en gel de campo pulsado, resistencia a la tetraciclina, resistencia a cloranfenicol.
Molecular characterization of Colombian invasive serotype 5 Streptococcus pneumoniae isolates recovered between 1994 and 2004
Background. Streptococcus pneumoniae serotype 5 is an important cause of invasive disease in Colombia where the circulation of the clone Colombia5-19, penicillin susceptible but tetracycline and chloramphenicol resistant, has been demonstrated.
Objective. To establish the genetic relatedness among Colombian invasive S. pneumoniae serotype 5 isolates, recovered between 1994 and 2004, and the clone Colombia5-19.
Materials and methods. Eighty three isolates with penicillin, vancomycin, ceftriaxone, erythromycin, trimethoprim-sulfametoxazole, chloramphenicol and tetracycline susceptibility patterns were studied. Of these isolates, 29 were recovered from children less than 5 years of age. DNA restriction patterns of all isolates were determined for pulsed field gel electrophoresis, using the SmaI enzyme. Genetic similarity among isolates and the clone Colombia5-19 was established according to Tenovers criteria and the Fingerprintingä II program.
Results. All isolates were related with the Colombia5-19 clone and belonged to the electrophoretic pattern A, of which 17 subtypes were derived. Most of the isolates belonged to patterns A (38,6%), A8 (21,7%) and A5 (12%), the other 23 isolates belonged to 15 electrophoretic patterns. Thirty -four isolates resistant both to tetracycline and chloramphenicol were related with electrophoretic patterns A ( n = 32), A16 ( n = 1) and A28 ( n = 1), which have a band of 340 Kb in common with the clone.
Conclusion. These results showed the continuing circulation of S pneumoniae serotype 5 isolates in the country, and which are genetically related with the clone Colombia5-19.
Keywords: Streptococcus pneumoniae; meningitis; pneumonia; electrophoresis, pulsed-field gel; clones, tetracycline resistance, chloramphenicol resistance
En Colombia , Streptococcus pneumoniae serotipo 5 representa el 7% de los aislamientos recuperados de enfermedad invasora como meningitis y neumonía tanto en niños como en adultos (1). El total de estos aislamientos presenta susceptibilidad a penicilina y algunos presentan resistencia a trimetoprim sulfametoxazol (SXT) (85%), tetraciclina (60%) y cloranfenicol (60%) (1).
Se ha demostrado que estos aislamientos invasores serotipo 5 se relacionan genéticamente según los patrones de restricción de ADN cromosomal obtenidos por electroforesis en gel de campo pulsado ( Pulsed Field Gel Electrophoresis, PFGE), lo cual ha establecido la circulación de un clon específico, reconocido por la red de epidemiología molecular de neumococo ( Pneumococcal Molecular Epidemiology Network, PMEN) como el clon 19-Colombia5 (2-4).
La vigilancia de la prevalencia y dispersión de este clon en el país y en otros países de Latinoamérica y del mundo (3-6) resulta útil para evaluar la presión selectiva que pueda generarse por la introducción de las vacunas conjugadas que incluyan el serotipo 5 (7,8). Por tal razón, el objetivo de este estudio fue establecer, por medio de la determinación de los patrones de restricción de PFGE, las relaciones genéticas entre los aislamientos invasores de S. pneumoniae serotipo 5 recuperados entre 1994 y 2004 y el clon 19- Colombia5, con el fin de actualizar la información y contribuir con el programa de vigilancia molecular del neumococo. Este programa se desarrolla en Colombia como parte del programa de vigilancia de la distribución de los tipos capsulares y la susceptibilidad antimicrobiana de aislamientos de S. pneumoniae, y forma parte del proyecto del Sistema Regional de Vacunas (SIREVA II) de la Organización Panamericana de la Salud (OPS) (9).
Materiales y métodos
Aislamientos
Se estudiaron 83 aislamientos invasores colombianos de S. pneumoniae serotipo 5, 29 de niños menores de cinco años recuperados entre 2000 y 2004, y 54 de pacientes mayores de cinco años recuperados entre 1994 y 2004. Por procedencia, 33 aislamientos fueron remitidos de Antioquia (39,8%), 22 de Bogotá (26,5%), 11 de Valle (13,3%), ocho de Risaralda (9,6%) y los nueve restantes (10,8%) de Amazonas, Caldas, Córdoba, Magdalena y Santander.
El 62,7% de los aislamientos se recuperó de pacientes con neumonía, de los cuales el 81% provenía de pacientes mayores de cinco años, el 26,5% de pacientes con meningitis, 63% de los cuales eran niños menores de cinco años, y el 10,8 % de los aislamientos se recuperó de pacientes con otras enfermedades invasoras como sepsis, síndrome febril y bacteremia.
Identificación y susceptibilidad antimicrobiana
En el Grupo de Microbiología del Instituto Nacional de Salud los aislamientos fueron confirmados por métodos estandarizados, y serotipificados utilizando la reacción de Quellung con antisueros del Statens Seruminstitut (Copenhague, Dinamarca) (10). También se determinaron los patrones de susceptibilidad antimicrobiana a penicilina, vancomicina, ceftriaxona, eritromicina, trimetoprim sulfametoxazol (SXT), cloranfenicol y tetraciclina por el método de Kirby-Bauer, y la concentración inhibitoria mínima (CIM) por microdilución en caldo según el Comité Nacional para Estándares de Laboratorio Clínico ( National Committee for Clinical Laboratory Standards, NCCLS) (11) (ahora CLSI). El
cuadro 1 muestra los porcentajes de resistencia a antibióticos de los aislamientos estudiados por grupo de edad.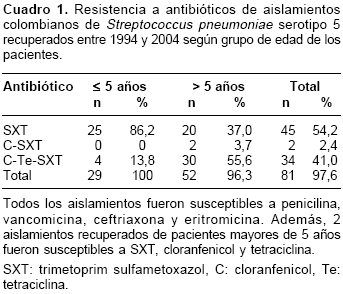
Los aislamientos se encontraban conservados a -70°C en leche descremada al 20% y fueron recuperados en agar sangre de cordero al 5%, e incubados a 37°C durante 18 horas; posteriormente fueron reconfirmados por morfología de colonia, presencia de a-hemólisis y prueba de solubilidad en bilis (10).
Electroforesis de campo pulsado (PFGE)
Se realizó según el protocolo de Vela y colaboradores (12); los aislamientos fueron inoculados en 10 ml de caldo Todd-Hewitt (Difco) al 3% suplementado con extracto de levadura al 1%, glucosa al 1% y glutamina (22 mg/ml), e incubados durante 6 horas a 37°C. Las células fueron recuperadas por centrifugación, resuspendidas en PIV (NaCl y Tris 1 M pH 8,0) y mezcladas con agarosa de bajo punto de fusión (Bio-Rad) al 1,5% para inmovilizarlas en discos.
Los discos obtenidos fueron tratados con 5 mg/ml de RNasa (Sigma), 1mg/ml de proteinasa K (Promega) y fueron posteriormente digeridos con 20 U de Sma I (Promega). El aislamiento de S. pneumoniae Spn 1439 -106 (ATCC BAA - 341) se utilizó como referencia del patrón electroforético del clon 19-Colombia5 y la cepa R6 (donada por Alexander Tomasz de la Universidad Rockefeller, Nueva York) se utilizó como control interno del procesamiento de muestras y de las condiciones de la electroforesis. Los fragmentos de restricción fueron separados por PFGE (equipo CHEF DRII, Bio-Rad) en un gel de agarosa (Bio-Rad) al 1%, a 6V/cm, con un tiempo inicial de 1 segundo, un tiempo final de 30 segundos y por un periodo de 23 horas a 11,3°C. El marcador de peso molecular utilizado fue el fago lambda (New England Biolabs) El gel fue teñido con bromuro de etidio (3 mg/ml) y lavado con agua destilada. Por último se le tomó una fotografía utilizando una cámara de sistema instantáneo (Polaroid MP4+).
Análisis de los patrones de restricción de ADN
Los patrones de bandas de PFGE, algunos identificados en estudios anteriores (2, 3), fueron clasificados de acuerdo con los criterios de Tenover (13). Los resultados fueron analizados con el programa Fingerprinting II (Bio-Rad), que compara los patrones electroforéticos de los aislamientos y calcula el coeficiente de similitud de Dice (SD), para generar dendrogramas que asocian los patrones según sus relaciones genéticas por medio del método no ponderado de agrupamiento que utiliza promedios aritméticos ( Unweighted pair group method using arithmetic averages, UPGMA).
Resultados
Los 83 aislamientos analizados por PFGE según los criterios de Tenover mostraron un patrón electroforético predominante identificado como patrón A con 17 subtipos (A1, A3, A5, A8, A16- A28), de los cuales los subtipos del A16 al A28 fueron identificados por primera vez en este estudio. Los aislamientos pertenecientes al patrón A no presentaron diferencias en el número ni en el tamaño de las bandas, y por lo tanto, fueron indistinguibles del clon 19-Colombia5 (100% de similitud genética). El patrón A se estableció como estrechamente relacionado con los subtipos A5, A8, A16, A20-A25 y A28 por diferenciarse en dos o tres bandas, con un porcentaje de similitud genética entre 95,6 y 96,8%, y posiblemente relacionado con los subtipos A1, A3, A17-A19, A26 y A27 por diferenciarse en cuatro a seis bandas, con un porcentaje de similitud genética entre 81,9 y 93,3% (
figura 1).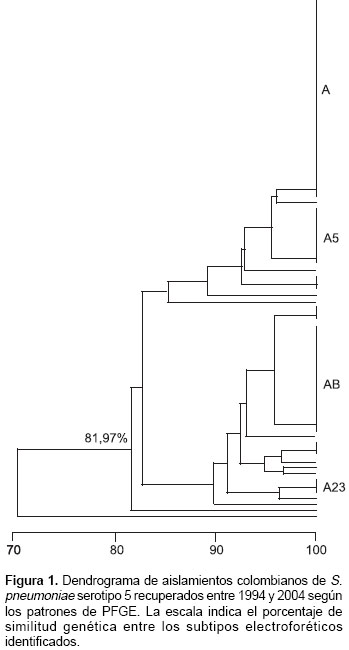
El 85% de los aislamientos recuperados de pacientes mayores de cinco años y el 62% de los de niños menores de 5 años fueron agrupados en cuatro patrones electroforéticos comunes a los dos grupos de edad, 32 aislamientos en el patrón A (38,6%), 18 en el A8 (21,7%), 10 en el A5 (12%) y tres en el A23 (3,6%). De los 14 subtipos restantes, con tres o menos aislamientos, en siete (A18 - A22, A24, A27) se agruparon los recuperados de niños menores de cinco años y en los otros siete (A1, A3, A16, A17, A25, A26, A28) los de pacientes mayores de cinco años.
De los 81 aislamientos (97,6%) resistentes a SXT, 34 (42%) fueron resistentes a cloranfenicol y tetraciclina, considerándose multirresistentes. Estos 34 aislamientos, recuperados de pacientes mayores de 5 años en su mayoría (88,2%), pertenecieron a los patrones electroforéticos A (n = 32), A16 (n = 1) y A28 (n = 1), y al igual que el clon 19-Colombia5, se caracterizaron por presentar una banda de 340 Kb. Los demás subtipos no se relacionaron con patrones de resistencia a antibióticos.
Discusión
Los patrones de restricción del ADN cromosomal obtenidos por PFGE de aislamientos invasores de S. pneumoniae serotipo 5 determinados en este estudio fueron genéticamente similares al clon 19 -Colombia5, lo que demuestra la clonalidad de los aislamientos. El hallazgo de estos aislamientos clonales en el periodo estudiado y en diferentes departamentos indica la prevalencia y circulación del clon en el país. Al igual que en estudios anteriores (2,3), el patrón electroforético A es el más frecuente, aunque su frecuencia ha disminuido, debido principalmente al aumento del número de subtipos electroforéticos identificados, de cinco descritos anteriormente a 17 reportados en este estudio.
Uno de los factores que pueden explicar la poca diversidad genética de los aislamientos de S. pneumoniae serotipo 5 es la mayor frecuencia de recuperación de aislamientos de este serotipo en pacientes con enfermedades invasivas que en portadores, a diferencia de los aislamientos de otros serotipos (14-17). La poca adaptación de los aislamientos serotipo 5 a las superficies mucosas hace que se encuentren ocasionalmente en la nasofaringe, donde se puede llevar a cabo el proceso de transformación (18), por lo que tienen poca oportunidad de intercambiar material genético con su propia especie o con especies relacionadas, y de estar expuestos a la presión selectiva que generan los antibióticos. Además, la capacidad de un serotipo de ser clonal puede representar una ventaja en cuanto a la capacidad de ser invasor (5) y se ha demostrado que los aislamientos provenientes de portadores tienen más diversidad genética que los aislamientos invasores (6,14).
Los aislamientos con los patrones A, A16 y A28 presentaron un fragmento de 340 Kb que se relaciona con la resistencia a tetraciclina y cloranfenicol, lo que coincide con lo encontrado en otro estudio (3). De igual manera, la ausencia de la banda se asoció con aislamientos susceptibles a estos dos antibióticos (5). Este fragmento de 340 Kb contiene los genes tetM y cat (2), que confieren resistencia a tetraciclina y cloranfenicol respectivamente, y que hacen parte del transposón conjugativo Tn 5253 (19). La presencia de la banda puede explicarse por la inserción de este transposón en el genoma del neumococo, evento genético que puede alterar los sitios de restricción de la enzima Sma I y generar nuevos patrones electroforéticos (13,20).
En Colombia existen pocos datos sobre el uso de cloranfenicol y tetraciclina en los hospitales o en la comunidad. Sin embargo, se ha reportado que para niños menores de cinco años no se recomienda el uso de cloranfenicol como alternativa para el tratamiento efectivo de la meningitis neumocóccica (21), mientras que para los adultos se recomienda la tetraciclina como primera elección para el tratamiento de la neumonía adquirida en la comunidad en pacientes que no presenten factores de riesgo y teniendo en cuenta los patrones locales de resistencia a antibióticos (22).
Se han recuperado aislamientos de S. pneumoniae serotipo 5 relacionados genéticamente con el clon 19-Colombia5 en Israel (5) y Portugal (23) y en países de Latinoamérica como Argentina, Uruguay, Brasil, México y Guatemala, donde se han identificado los subtipos electroforéticos A, A3 y A5, al igual que en Colombia (3,6). Sin embargo, es necesario establecer las relaciones genéticas de los aislamientos serotipo 5 que circulan en otros países como Perú (24) y España (25) con el clon 19-Colombia5, para contribuir con la epidemiología y conocimiento de su dispersión clonal.
Los clones se caracterizan por ser resistentes a antimicrobianos de uso común, por persistir en el tiempo y por estar en diversas áreas geográficas dentro de un país o internacionalmente (2,5,26), como ha sucedido con algunos clones de neumococo resistentes a drogas que han conseguido exitosamente la dispersión mundial (27). Estas características pueden hacer que la introducción de vacunas conjugadas lleve a la emergencia de nuevos clones resistentes que expresen serotipos no presentes en la vacuna (20).
El aumento de resistencia a antimicrobianos en el neumococo se debe al éxito de unos pocos clones multirresistentes, razón por la cual el uso racional de antibióticos, la vigilancia de los clones resistentes y el uso más amplio de vacunas que disminuyan el número de portadores y pacientes con enfermedades invasoras son estrategias críticas en el control de la resistencia en neumococo. Se ha reportado que la vigilancia de los clones es importante para supervisar patrones de resistencia y ayudar en la elección de agentes antimicrobianos apropiados contra enfermedades (27). Por lo tanto, la vigilancia del clon 19- Colombia5 sigue siendo importante debido a sus características de resistencia y a la capacidad de diseminación que puede permitirle una dispersión mundial parecida a la de otros clones.
Agradecimientos
Al Grupo de Microbiología del Instituto Nacional de Salud por su colaboración durante el desarrollo del trabajo. Al Instituto Nacional de Salud y a la Organización Panamericana de la Salud a través de su proyecto SIREVA II por la financiación de este estudio. A Liliana Gamboa por su aporte de resultados iniciales para la realización de este trabajo.
Conflicto de intereses
Los autores de este trabajo declaran que no tienen ningún conflicto de intereses.
Financiación
El trabajo realizado estuvo financiado por el Instituto Nacional de Salud.
Correspondencia:
Elizabeth Castañeda
Instituto Nacional de Salud.
Avenida calle 26 No. 51-60
Teléfono 57-1-220-0920
Fax 57-1-220-0901
Recibido: 16/12/05; aceptado: 06/04/06
Referencias [ Links ] Disponible en
http://www.ins.gov.co/pdf_investiga/microbiologia_spn_05.pdf.2. Tamayo M, Sá-Leâo R, Santos Sanches I, Castañeda E, De Lencastre H. Dissemination of chloramphenicol -and tetracycline-resistant but penicillin susceptible invasive clone of serotype 5 Streptococcus pneumoniae in Colombia. J Clin Microbiol 1999;37:2337-42. [ Links ]
3. Gamboa L, Camou T, Hortal M, Castañeda E, Sireva- Vigía Working Group. Dissemination of Streptococcus pneumoniae clone Colombia5-19 in Latin America. J Clin Microbiol 2002;40:3942-50. [ Links ]
4. The Pneumococcal Molecular Epidemiology Network (PMEN). Página de internet. Consultado: 6 de octubre de 2005. Disponible en: http://
www.sph.emory.edu/PMEN/index.html. [ Links ]5. Porat N, Trefler R, Dagan R. Persistence of two invasive Streptococcus pneumoniae clones of serotypes 1 and 5 in comparison to that of multiple clones of serotypes 6B and 23F among children in Southern Israel. J Clin Microbiol 2001;39:1827-32. [ Links ]
6. emlièková H, Crisóstomo MI, Brandileone MC, Camou T, Castañeda E, Corso A, et al. Serotypes and clonal types of penicillin-susceptible Streptococcus pneumoniae causing invasive disease in children in five Latin American countries. Microb Drug Resist 2005;11:195-204. [ Links ]
7. Klugman KP, Madhi SA, Huebner RE, Kohberger R, Mbelle N, Pierce N. A trial of a 9-valent pneumococcal conjugate vaccine in children with and those without HIV infection. N Engl J Med 2003;349:1341-8. [ Links ]
8. Dagan R, Käyhty H, Wuorimaa T, Yaich M, Bailleux F, Zamir O, et al. Tolerability and immunogenicity of an 11-valent mixed carrier Streptococcus pneumoniae capsular polysaccharide-diphteria toxoid or tetanus protein conjugate vaccine in Finnish and Israeli infants. Pediatr Infect Dis J 2004;23:91-8. [ Links ]
9. Di Fabio JL, Castañeda E, Agudelo CI, de la Hoz F, Hortal M, Camou T, et al. Evolution of Streptococcus pneumoniae serotypes and penicillin susceptibility in Latin America, Sireva-Vigía Group.1993-1999. PAHO Sireva-Vigía Study Group. Pan American Health Organization. Pediatr Infect Dis J 2001;20:959-67. [ Links ]
10. Organización Panamericana de la Salud e Instituto Nacional de Salud Colombia. Programa de vigilancia de los serotipos y resistencia antimicrobiana de Streptococcus pneumoniae y Haemophilus influenzae. Manual de procedimientos del proyecto SIREVA II, p. 41-99. 2004. Consultado: 18 de octubre de 2005. Disponible en:
http://www.paho.org/Spanish/AD/THS/EV/LABS-manual-vigilancia-serotipos.pdf [ Links ]11. National Committee for Clinical Laboratory Standards (NCCLS). Performance standards for antimicrobial susceptibility testing. Eighth edition informational supplement. Vol. 23. M2-A8. Wayne PA: National Committee for Clinical Laboratory Standards; 2003. [ Links ]
12. Vela MC, Fonseca N, Di Fabio JL, Castañeda E. Presence of international multiresistant clones of Streptococcus pneumoniae in Colombia. Microb Drug Resist 2001;7:153-64. [ Links ]
13. Tenover FC, Arbeit RD, Goering RV, Mickelsen PA, Murray BE, Persing DH, et al. Interpreting chromosomal DNA restriction patterns produced by pulsed-field gel electrophoresis: criteria for bacterial strain typing. J Clin Microbiol 1995;33:2233-9. [ Links ]
14. Takala AK, Vuopio-Varkila J, Tarkka E, Leinonen M, Musser JM. Subtyping of common pediatric pneumococcal serotypes from invasive disease and pharyngeal carriage in Finland. J Infect Dis 1996;173:128-35. [ Links ]
15. Saha SK, Baqui AH, Darmstadt GL, Ruhulamin N, Hanif M, Arifeen SE, et al. Comparison of antibiotic resistance and serotype composition of carriage and invasive pneumococci among Bangladeshi children: implications for treatment policy and vaccine formulation. J Clin Microbiol 2003;41:5582-7. [ Links ]
16. Hausdorff WP, Feikin DR, Klugman KP. Epidemiological differences among pneumococcal serotypes. Lancet Infect Dis 2005;5:83-93. [ Links ]
17. Brueggemann AB, Peto TE, Crook DW, Butler JC, Kristinsson KG, Spratt BG. Temporal and geographic stability of the serogroup-specific invasive disease potential of Streptococcus pneumoniae in children. J Infect Dis 2004;190:1203-11. [ Links ]
18. Yother J, McDaniel LS, Briles DE. Transformation of encapsulated Streptococcus pneumoniae. J Bacteriol 1986;168:1463-5. [ Links ]
19. Ayoubi P, Kilic AO, Vijayakumar MN. Tn 5253, the pneumococcal W ( cat tet) BM6001 element, is a composite structure of two conjugative transposons, Tn 5251 and Tn 5252. J Bacteriol 1991;173:1617-22. [ Links ]
20. Sousa NG, Sá-Leão R, Crisóstomo I, Simas C, Nuñes S, Frazão N, et al. Properties of novel international drug-resistant pneumococcal clones identified in day-care centers of Lisbon, Portugal. J Clin Microbiol 2005;43:4696-703. [ Links ]
21. Hernández M, Mejía GI, Trujillo H, Robledo J. Evidencia in vitro de la utilidad de cloranfenicol y rifampicina para el tratamiento de infecciones sistémicas y meningitis causadas por Streptococcus pneumoniae aislados de niños menores de 5 años en Colombia. Biomédica 2003;23:456-61. [ Links ]
22. Caballero AS, Martínez E, Giraldo H, Álvarez CA, Torres CA, Saavedra C, et al. Recomendaciones para el diagnóstico, tratamiento y prevención de la neumonía adquirida en la comunidad en adultos. Infectio 2004;8:8-44. [ Links ]
23. Serrano I, Melo-Cristino J, Carriço JA, Ramirez M. Characterization of the genetic lineages responsible for pneumococcal invasive disease in Portugal. J Clin Microbiol 2005;43:1706-15. [ Links ]
24. Cullotta AR, Kalter HD, Delgado J, Gilman RH, Facklam RR, Velapatino B, et al. Antimicrobial susceptibilities and serotype distribution of Streptococcus pneumoniae isolates from a low socioeconomic area in Lima, Peru. Clin Diagn Lab Inmunol 2002;9:1328-31. [ Links ]
25. Aspa J, Rajas O, Rodríguez de Castro F, Blanquer J, Zalacain R, Fenoll A, et al. Drug resistant pneumococcal pneumonia: clinical relevance and related factors. Clin Infect Dis 2004;38:787-98. [ Links ]
26. McGee L, McDougal L, Zhou J, Spratt BG, Tenover FC, George R, et al. Nomenclature of major antimicrobial- resistant clones of Streptococcus pneumoniae defined by the pneumococcal molecular epidemiology network. J Clin Microbiol 2001;39:2565-71. [ Links ]
27. Klugman KP. The successful clones: the vector of dissemination of resistance in Streptococcus pneumoniae. J Antimicrob Chemother 2002;50 (Suppl. 2):1-5. [ Links ]














