Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.26 no.3 Bogotá Sept. 2006
Caracterización por electroforesis de campo pulsado de aislamientos de Salmonella Typhimurium recuperados en el programa de vigilancia de enfermedad diarreica aguda en Colombia, 1997-2004
Nélida Muñoz, María Elena Realpe, Elizabeth Castañeda, Clara Inés Agudelo
Grupo de Microbiología, Instituto Nacional de Salud, Bogotá, D.C. Colombia.
Introducción. En Colombia, Salmonella Typhimurium es el segundo serotipo más frecuentemente aislado en muestras clínicas humanas después de S. Enteritidis, y representa el 28,2% (n = 468) de los 1.659 aislamientos recuperados en el programa de vigilancia por el laboratorio de enfermedad diarreica aguda liderado por el Grupo de Microbiología del Instituto Nacional de Salud.
Objetivo. Caracterizar molecularmente los aislamientos de S. Typhimurium que circulan en el país con el fin de establecer su perfil y determinar su relación genética.
Materiales y métodos. En 468 aislamientos remitidos por 14 laboratorios de salud pública departamentales, recuperados entre enero de 1997 y diciembre de 2004, se determinó el perfil genómico por electroforesis de campo pulsado con la enzima de restricción XbaI según el protocolo de la Red PulseNet (CDC, Atlanta); el análisis de los geles se realizó con el programa Fingerprinting II.
Resultados. Se obtuvieron 180 patrones electroforéticos. Los patrones prevalentes fueron COINJPX.X01.0062, encontrado en 101 aislamientos (21,3%), seguido por los patrones COINJPX.X01.0001 en 40 (8,4%), COINJPX.X01.0058 en 13 (3%), COINJPX.X01.0003 en 12 (2,5%), COINJPX.X01.0066 en 11 (2,3%) y COINJPX.X01.0108 en 6 (1,4%). COINJPX.X01.0001, fue el patrón predominante en Bogotá hasta 2002, reemplazado a nivel nacional por el COINJPX.X01.0062. El análisis de agrupamiento mostró tres grupos con similitudes genéticas entre 53% y 82%, lo que revela la heterogeneidad genética del serotipo.
Conclusiones. El estudio permitió identificar patrones electroforéticos prevalentes, tener una base de datos nacional disponible para la comparación de los perfiles en tiempo real, detectar brotes y relacionar casos aparentemente aislados.
Palabras clave: Salmonella Typhimurium, vigilancia epidemiológica, diarrea, electroforesis en gel de campo pulsado, Colombia.
Characterization by pulsed-field gel electrophoresis of Salmonella Typhimurium isolates recovered in the acute diarrheal disease surveillance program in Colombia, 1997-2004
Introduction. In Colombia Salmonella Typhimurium is the second most frequently isolated Salmonella serotype (after S. Enteritidis) from human clinical samples. This serotype represents 28.2% (n = 468) of the 1659 Salmonella isolates recovered by the Instituto Nacional de Salud (INS) Acute Diarrheal Diseases Surveillance Program from 1997 to 2004. Previously, little was known about the molecular attributes of these strains and commonality of genetic subtypes among S. Typhimurium isolated from humans in Colombia.
Objective. The purpose of this study was to characterize S. Typhimurium isolates circulating in Colombia using pulsed-field gel electrophoresis in order to establish genetic profiles and determine their potential relatedness.
Materials and methods. 468 S. Typhimurium isolates were submitted to INS by 14 Public Health Laboratories from January 1997 to December 2004. The pulsed-field gel electrophoresis was performed using restriction enzyme XbaI according to the PulseNet standard protocol (CDC, Atlanta). Restriction pattern analysis and cluster definition were performed using Molecular Analyst Fingerprinting II software.
Results. 180 distinct electrophoretic patterns were obtained. Pattern COINJPX.X01.0062 was the most common (21.3% of the 473 isolates, n=101), followed by COINJPX.X01.0001 (8.4%, n=40), COINJPX.X01.0058 (3%, n=13), COINJPX.X01.0003 (2.5%, n=12), COINJPX.X01.0066 (2.3%, n=11), and COINJPX.X01.0108 (1.4%, n=6). COINJPX.X01.0001, was the most prevalent pattern among S. Typhimurium isolated in Bogotá from 1997-2002, but appeared to be replaced both in Bogotá and nationally by COINJPX.X01.0062 in 2003-2004. Cluster analysis demonstrated three distinct groups with 53% and 82% similarity indicating heterogeneity within the serotype.
Conclusions. The study provides important electrophoretic pattern baseline data, which will be useful for prospective real-time comparison of profiles of future isolates. Cluster detection using this method will facilitate outbreak detection and will enhance our ability to find common sources for seemingly unrelated infections.
Keywords: Salmonella Typhimurium, epidemiologic surveillance, diarrhea, electrophoresis, gel, pulsed-field, Colombia.
Salmonella spp. son los principales patógenos causantes de las enfermedades transmitidas por alimentos en países en vías de desarrollo; al igual que, en países desarrollados donde Salmonella spp. suelen ser más importantes que Escherichia coli enterotoxigénica y Shigella sp, los cuales a menudo también son superados por otros agentes como Campylobacter spp. (1,2). Con base en la diversidad de los antígenos somáticos y flagelares, el género Salmonella se clasifica en 2.523 serotipos descritos en el esquema de Kauffmann y White (3,4). S. Typhimurium es uno de los serotipos más frecuentemente aislados en pacientes con salmonelosis en países como EEUU (5,6), Argentina y Brasil, entre otros (7). En nuestro país, la importancia de S. Typhimurium se debe a su frecuencia y perfil de multirresistencia antimicrobiana, representando el 28,2% del total de aislamientos de Salmonella spp. confirmados por el Grupo de Microbiología del Instituto Nacional de Salud en el Programa de Vigilancia de la Enfermedad Diarreica Aguda entre 1997 y 2004. Este serotipo presenta una resistencia a tetraciclina de 79,2%, a ampicilina de 69%, a trimetoprim sulfametoxazol de 54,2%, a cloranfenicol de 23,8%, y una multirresistencia de 53,1%, predominantemente a ampicilina, tetraciclina y trimetoprim sulfametoxazol (7,8).
Debido a la elevada prevalencia del serotipo Typhimurium y a la limitación de la técnica de serotipificación para diferenciar entre los aislamientos del mismo serotipo, se hace difícil el reconocimiento de brotes cuando éstos se relacionan con un número bajo de casos y se aislan con frecuencia (9,10); por consiguiente, se han empleado otras técnicas de tipificación de Salmonella spp. como la determinación de la susceptibilidad antimicrobiana (3,7,10) y la fagotipificación (11), las cuales permiten establecer relaciones epidemiológicas. Las técnicas de subtipificación molecular como la electroforesis en gel de campo pulsado (PFGE), considerada la prueba de oro para la diferenciación y el agrupamiento de aislamientos de Salmonella spp. de un serotipo específico, la cual es en la actualidad el método más ampliamente utilizado en las redes de vigilancia de enfermedades transmitidas por alimentos (12-16).
En 1996 se creó en EEUU la Red Nacional de Subtipificación Molecular de Patógenos Transmitidos por Alimentos, PulseNet, la cual emplea la técnica PFGE. PulseNet no es un sistema de vigilancia como tal, pero sí propone un método de subtipificación recomendado por el CDC de Atlanta para los programas de vigilancia de las enfermedades transmitidas por alimentos que permite establecer a través de las bases de datos de los patrones de PFGE una rápida comparación electrónica entre los perfiles de ADN de los diferentes laboratorios en cualquiera de las redes, logrando identificar los grupos clonales asociados con brotes a nivel local, nacional y regional. PulseNet cuenta con redes en EEUU, Canadá, Europa, Asia y América Latina (17).
El Grupo de Microbiología del Instituto Nacional de Salud hace parte de la red PulseNet latino-americana desde el 2004, lo que le ha permitido participar en los programas de control de calidad y comparar los resultados obtenidos con la técnica estandarizada de subtipificación molecular PFGE.
Dado que en Colombia S. Typhimurium es el segundo serotipo más frecuentemente aislado en muestras clínicas y que su virulencia está rela-cionada con la multirresistencia a los antibióticos, se consideró necesario conocer más sobre su epidemiología molecular; este estudio buscó caracterizar por PFGE los aislamientos de S. Typhimurium recuperados en el Programa de Vigilancia de la Enfermedad Diarreica Aguda entre 1997 y 2004, con el fin de establecer su perfil y determinar su relación genética. Este conoci-miento permitirá realizar la vigilancia activa de la salmonelosis mediante la comparación de los perfiles genéticos en tiempo real con el fin de detectar brotes y sus posibles fuentes de infección y establecer la relación de casos aparentemente aislados.
Materiales y métodos
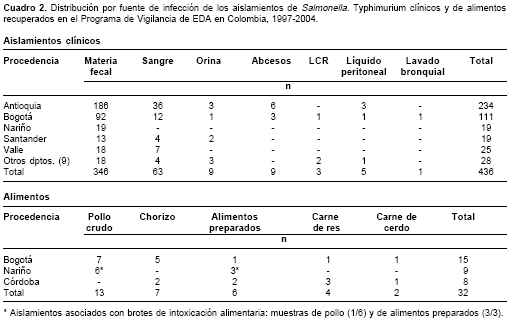
Se estudiaron un total de 468 aislamientos de S. Typhimurium de los cuales 436 eran de material clínico humano y 32 de alimentos. Nueve aislamientos se relacionaron epidemiológicamente con dos brotes de intoxicación alimentaria ocurridos en Nariño; el primer brote, en el año 2000, relacionó dos casos (uno de muestra clínica y otro de alimento), y el segundo, en el 2003, involucró 60 casos clínicos de los cuales siete fueron confirmados por el laboratorio (cuatro muestras clínicas y tres de alimentos) (18). Los aislamientos fueron remitidos por 14 laboratorios de salud pública (Antioquia, Atlántico, Caldas, Córdoba, Cundinamarca, Guainía, Huila, Nariño, Norte de Santander, Risaralda, Santander, Tolima, Vichada y el Distrito Capital). La distribución de los aislamientos de S. Typhimurium por año y departamento se presenta en el
cuadro 1 y por fuente de infección en el cuadro 2.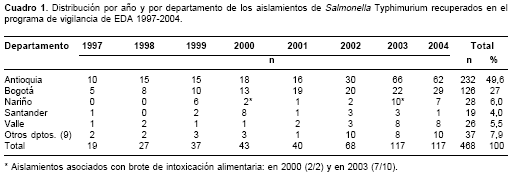
De los 436 aislamientos clínicos, el 42% se recuperó de mujeres y el 58%, de hombres. La edad estuvo en un rango entre 0 y 83 años, con una media de 15 años y una desviación de 20,8 años con respecto a la media. Los menores de 15 años constituyeron el grupo de edad con mayor número de aislamientos 268(67,5%) y en este grupo, 146 (54,5%) correspondían a menores de cinco años, 140 (34,3%) a mayores de 15 años y 28 no registraban la edad de los pacientes.
Los aislamientos se encontraban almacenados a -70 °C en leche descremada (Difco) al 20% como parte del cepario del Grupo de Microbiología. Para su recuperación, cada uno de ellos se resembró dos veces en agar infusión cerebro corazón (BHI) (Becton, Dickinson) a 37°C en aerobiosis. El 10% del total de los aislamientos se confirmó bioquímicamente de acuerdo con el esquema de identificación estandarizado en el laboratorio (19), y serológicamente siguiendo el esquema de Kauffmann-White (3) con antisueros (Difco).
PFGE
El perfil genómico se estableció mediante la técnica de PFGE. El total de aislamientos fue procesado con el protocolo estandarizado por el CDC de Atlanta para la subtipificación molecular de Salmonella, que utiliza discos de agarosa y el fago Lambda como marcador de peso molecular. Por la técnica de bloques que utiliza el protocolo de PFGE modificado por la Red PulseNet-EEUU (año 2000) y Salmonella Braenderup H9812 como marcador de peso de molecular se procesaron el 10% de los aislamientos que presentaron por la técnica de disco el mismo patrón electroforético y todos los aislamientos que presentaron un solo patrón.(16).
Para la preparación del ADN se mezclaron las suspensiones bacterianas con una absorbancia de 1,1 (a una longitud de onda de 610 nm) con 0,5 mg/ml de proteinasa K (Promega) y luego fueron embebidas en agarosa de bajo punto de fusión (Low-melting, Bio-Rad) al 2% en buffer TE para elaborar los discos. En la técnica de bloques, se mezclaron 200 ml de la suspensión con igual volumen de agarosa SeaKem Gold 1% (Cambrex, Cambridge Maryland, USA) y de dodecil sulfato de sodio (SDS) 1%. Posteriormente se sometieron a lisis celular en una solución tamponada (Tris 50mM, EDTA 50mM, pH 8,0), sarcosil 1% proteinasa K a 0,1 mg/ml) a 54 °C en baño de María con agitación constante a 200 rpm durante dos horas, y posteriormente se realizaron seis lavados a 50 °C por 15 minutos, dos con agua Milli Q y cuatro con buffer TE (Tris 10 mM, EDTA 1 mM). Todos los aislamientos se analizaron usando la enzima de restricción XbaI (Promega). Los discos y los bloques se separaron en geles de agarosa al 1% (PFGE- grade, Bio-Rad y Seakem Gold, Cambrex, respectivamente). La PFGE se realizó en el CHEF DRII-Bio-Rad, con un tiempo inicial de 2,2 segundos, tiempo final de 63,8 segundos, voltaje de 6V/cm y tiempo de corrido de 18 horas a 14°C. Después del corrido electroforético, los geles se tiñeron con bromuro de etidio (1 µg/ml) en agitación constante durante una hora y se destiñeron después con agua destilada durante el mismo tiempo. Posterior-mente, el ADN se visualizó en un transiluminador con luz UV y la imagen se registró con una cámara Polaroid para su análisis.
Análisis de los geles
Los geles de agarosa preparados con discos y con bloques fueron normalizados en el programa Fingerprinting II versión 3.0 (Bio-Rad) con el marcador de peso molecular respectivo (20). Las bandas obtenidas se definieron visualmente teniendo en cuenta su número y su posición y se compararon con la foto correspondiente.
Siguiendo las recomendaciones de la Red PulseNet, los perfiles de PFGE identificados en los aislamientos de S. Typhimurium para ingresarlos a la base de datos nacional se designaron con el código COINJPXX01.0001, que corresponde al país Colombia (CO), Instituto Nacional (IN), Typhimurium (JPX), enzima de restricción XbaI (X01) y número del patrón (0001) de S. Typhimurium. La numeración de los patrones se continuó de acuerdo a la secuencia de la base de datos descrita por Wiesner et al (21).
Los coeficientes de similitud se calcularon con el algoritmo Dice utilizado para comparar los patrones de macrorrestricción y para establecer la relación genética entre los aislamientos. Los elementos de la matriz fueron sometidos a análisis de agrupamientos empleando la técnica del algoritmo de emparejamiento de bandas con base en promedios aritméticos (UPGMA- unweighted pair-group metered using arithmetic average), método que utiliza un agrupamiento secuencial. La identificación y ordenamiento de las relaciones de manera decreciente con respecto a la similitud permitió una variación de 1,5% en la posición de la banda, de esta forma se construyeron los respectivos dendrogramas.
Análisis estadístico
Se utilizaron tablas cruzadas para describir el comportamiento de los patrones electroforéticos con relación al lugar de procedencia y la fuente del aislamiento. La asociación entre estas variables se exploró utilizando la prueba estadística de ji cuadrado, generada con un 95% de confianza. Para su procesamiento se utilizó el software estadístico MINITAB versión 14.0.
Resultados
Como resultado de la digestión enzimática del ADN cromosomal con XbaI se obtuvieron los perfiles de ADN constituidos por 11 a 15 fragmen-tos de restricción, con pesos moleculares entre 20,5 y 1.135 kb. El análisis de los 468 aislamientos generó 180 patrones de PFGE diferentes. Los patrones de PFGE prevalentes fueron COINJPX.X01.0062, encontrado en 101 (21,6%) aislamientos, seguido por COINJPX.X 01.0001 en 40 (8,5%), COINJPX.X01.0058 en 13 (2,7%), COINJPX.X01.0003 en 12 (2,6%), COINJPX.X01.0066 en 11 (2,3%) y COINJPX. X01.0108 en 6 (1,3%); los 285 (61%) aislamientos restantes se agruparon en 174 patrones diferentes; en la
figura 1 se presenta el perfil genómico de los cinco patrones más frecuentes.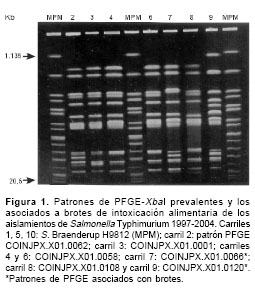
De los 101 aislamientos que presentaron el patrón COINJPX.X01.0062, 53 (52,4%) provenían de Antioquia y 24 (23,8%) de Bogotá. Este patrón también fue dominante en los 24 (23,8%) aislamientos restantes provenientes de otros departamentos. Otros patrones comunes en Antioquia fueron el COINJPX.X01.0058 y el COINJPX.X01.0108, los cuales se encontraron respectivamente en 92% (11/12) y 83,3% (5/6) del total de los aislamientos con estos genotipos. Los patrones COINJPX.X01.0001 y COINJPX. X01.0003 se encontraron respectivamente en el 48,8% (20/41) y 85,7% (12/14) del total de los aislamientos con estos genotipos provenientes de Bogotá. En Nariño, el patrón COINJPX.X01.0066 se presentó en 90% (9/10) del total de los aislamientos con este genotipo. En Santander y Valle no se encontraron patrones predominantes.
Al analizar la evolución de los patrones por año se encontró que el patrón COINJPX.X01.0001 tuvo un comportamiento parabólico desde 1998 hasta 2003, caracterizado por un crecimiento entre los años 1998 y 2000 (cuando aparece la máxima frecuencia), y un decrecimiento posterior hasta el 2003. En contraste, el patrón COINJPX.X01.0062 mostró un incremento sostenido entre el 2002 y el 2004 (
figura 2).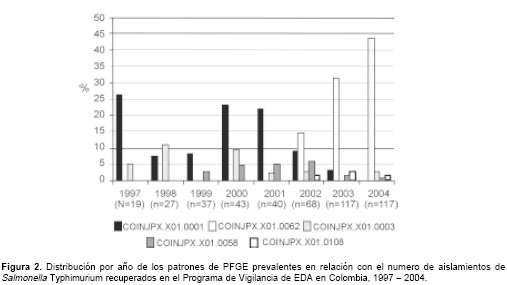
En cuanto a la distribución de los patrones por grupos de edad, en el grupo de niños entre 0 y 5 años y mayores de 15 años, el predominante fue COINJPX.X01.0062 con 25,7 y 28,5%, respec-tivamente. En el grupo de edad entre 6 y 14 años no se demostró un patrón común. En ambos géneros los patrones COINJPX.X01.0062 y COINJPX.X01.0001 se relacionaron con 21,3 y 8,3%, respectivamente.
La relación de los patrones electroforéticos por fuente de infección mostró que en las 346 mues-tras de materia fecal, los patrones más frecuentes fueron COINJPX COINJPX.X01.0058 en 12 (3,4%), COINJPX.X01.0003 en 10 (2,8%) y COINJPX.X01.0108 en 5 (1,4%). No se encontró asociación estadísticamente significativa entre el patrón electroforético y la fuente (ji cuadrado = 10,6; p > 0,05).
El análisis de los perfiles de PFGE estableció relación genética de los dos aislamientos que epidemiológicamente estuvieron involucrados en el brote de enfermedades transmitidas por alimentos en Nariño en el 2000, con cuatro aislamientos adicionales recuperados de pollo crudo en el mismo año y en diferentes localidades del departamento, ya que presentaron el mismo patrón electroforético, COINJPX.X01.0066. En el segundo brote ocurrido en el 2003, se encontró el mismo patrón de PFGE, COINJPX.X01.0120, en tres alimentos preparados y en cuatro pacientes.
Análisis de agrupamiento
El dendrograma del total de aislamientos de S. Typhimurium permitió determinar un rango de similitud genética de entre 53 y 82% con tres grupos principales.
El grupo I presentó 53% de similitud e incluyó 9 (1,9%) aislamientos; el grupo II, con 82% de similitud, incluyó 449 (96%), encontrándose en este grupo los cinco patrones PFGE prevalentes, y el grupo III, con 64% de similitud, incluyó 10 (2,1%) aislamientos (
figura 3).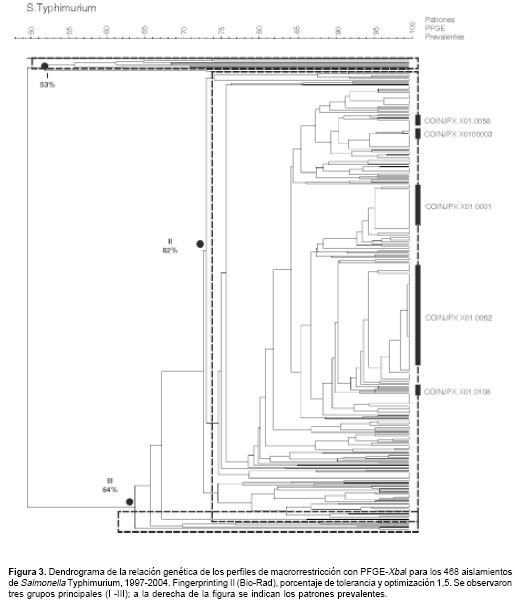
Los aislamientos del primer y tercer grupo presentaron una baja similitud genética (inferior a 80%), lo cual confirma la heterogeneidad genética de este serotipo.
El grupo II se dividió a su vez en 46 subgrupos que relacionaron aislamientos indistinguibles genéticamente (100% de similitud) y 255 (53,9%) aislamientos que presentaron 149 patrones electroforéticos diferentes con una similitud genética superior a 80%. Esto indica que a pesar de la alta diversidad genética dada por el número de patrones electroforéticos encontrados, la mayoría de los aislamientos colombianos de S. Typhimurium están relacionados genéticamente.
Análisis estadístico
Al explorar la asociación entre el lugar de procedencia y el patrón electroforético se encontró que esta fue estadísticamente significativa (ji cuadrado = 193,6; p <0,001).
Discusión
Los métodos de subtipificación molecular aplicables a un amplio rango de serovariedades de Salmonella se consideran ideales cuando más de un serotipo está implicado en un brote y cuando los brotes se relacionan con un bajo número de casos. Este estudio se orientó hacia la sero-variedad Typhimurium, asociada comúnmente con gastroenteritis en muestras clínicas humanas, y que constituye en nuestro país el segundo serotipo más frecuentemente aislado después de S. Ente-ritidis (39%), representando el 28,2% del total de aislamientos de Salmonella spp. recuperados por el Laboratorio del Grupo de Micro-biología del INS entre 1997 y 2004 en el marco del Programa de Vigilancia de la Enfermedad Diarreica Aguda (8).
Este trabajo determinó la relación genética en los 468 aislamientos colombianos de S. Typhimurium, observándose que, debido al número de patrones electroforéticos encontrados, el serotipo Typhimurium presenta una considerable diversidad genética, lo que coincide con los hallazgos de otros autores que han utilizado la PFGE y describen aislamientos muy heterogéneos recuperados en una misma región (21,22). Esta diversidad, representada por los subtipos de PFGE, probablemente muestra que las infecciones proceden de diferentes fuentes (10). Así, siempre que exista una estrecha relación epidemiológica, el hallazgo de aislamientos con perfiles de PFGE indistinguibles en este serotipo podría evidenciar la relación con un brote (14).
El análisis numérico de los perfiles de macrorrestricción es de gran utilidad para evaluar las interrelaciones entre los aislamientos de S. Typhimurium y tiene aplicaciones prácticas en los estudios de vigilancia epidemiológica a corto y largo plazo (23-25). Al explorar la relación entre el lugar de procedencia y el patrón electroforético se encontró que el patrón COINJPX.X01.0062 fue predominante en los departamentos de Antioquia (53/232 [22,8%]) y en el Distrito Capital (24/126 [19%]; p <0,001). Este patrón se relacionó, además, con una alta frecuencia en el grupo de niños menores de cinco años (25,7%) y fue común en las muestras de sangre (36,5%).
La investigación espacial y temporal de los patrones de PFGE en este estudio estableció que hubo un cambio en la circulación de los patrones, pues se encontró que el genotipo COINJPX.X01.0001 fue predominante a partir de 1998 para luego ser desplazado por el genotipo COINJPX.X01.0062 en años posteriores (2002). A pesar de observarse un incremento en el número de aislamientos provenientes de Antioquia y Bogotá a partir del año 2002, podría pensarse que tal situación es resultado del muestreo que se obtuvo, pero surge entonces la pregunta de por qué el patrón COINJPX.X01.0001 desaparece cuando aparece el COINJPX.X01.0062.
El estudio de 958 aislamientos de S. Typhimurium con la PFGE permitió a Bender y col (10) confirmar diez brotes de intoxicación alimentaria que involucraban un número bajo de casos en centros institucionales, los cuales no habrían sido reconocidos sin la subtipificación molecular. En forma similar, el análisis de los perfiles de ADN por PFGE en uno de los dos brotes estudiados en Colombia demostró la relación del alimento contaminado (pollo) con cuatro aislamientos más obtenidos de muestras de pollo crudo procedentes de cuatro localidades diferentes, pero que según los datos epidemiológicos provenían de una fuente común (un mismo distribuidor). El hecho de que las muestras clínicas y las de alimentos presentaran el mismo genotipo sugiere una posible ruta de infección y demuestra que sin la subtipificación por PFGE estos aislamientos no se habrían podido relacionar epidemiológicamente.
El alto poder discriminatorio de la PFGE aplicada a los 468 aislamientos de S. Typhimurium estudiados no constituyó una sorpresa, ya que la PFGE ha sido exitosa en la subtipificación de Salmonella spp., según se informa en varios estudios (10,13,24-26). Nuestro estudio confirma que, en las regiones donde S. Typhimurium es altamente endémico, la PFGE es de gran utilidad epidemio-lógica para la caracterización y comparación de aislamientos de este serotipo, a diferencia de los métodos de vigilancia convencionales que carecen de la sensibilidad para detectar aislamien-tos relacionados con brotes de S.Typhimurium, especialmente cuando involucran un número reducido de casos con cercanía en el tiempo.
S. Typhimurium es un organismo considerable-mente diverso en comparación con el serotipo S. Enteritidis, que es altamente clonal (21,27). Por ello, la detección de aislamientos estrechamente relacionados en diferentes áreas geográficas podría ser una señal de diseminación o aparición de grupos clonales que han incrementado su virulencia (28). Este hallazgo puede confirmarse con el uso de protocolos de PFGE estandarizados y la existencia de una base de datos nacional compartida, condiciones que fortalecen a los laboratorios que cuentan con programas de vigilancia molecular.
La identificación por medio de la PFGE de un incremento de casos en un subtipo específico puede ser la indicación del comienzo de un brote. En la labor de la Red Nacional de Subtipificación Molecular de Patógenos Transmitidos por Alimentos, Swaminathan y col (14) recomiendan que una vez detectado un grupo de aislamientos genéticamente relacionados, la investigación epidemiológica debe iniciarse para determinar si hay una fuente común, facilitando así el rápido reconocimiento de brotes y la definición de los casos aislados que se agrupan en el tiempo. Cuando los patrones no están relacionados por PFGE no es necesario realizar dichas acciones (25,29).
En este trabajo, los patrones de PFGE demostraron ser estables a través del tiempo, hallazgo que sugiere que la PFGE es un método altamente confiable no solamente para la discriminación de brotes en un tiempo corto sino también para la vigilancia de la epidemiología del serotipo Typhimurium a través de períodos prolongados (10).
La identificación y control de las infecciones transmitidas por alimentos dependen del rápido diagnóstico microbiológico y de la subtipificación molecular para identificar el agente causal. Se requiere de esfuerzos continuos para incrementar el número de aislamientos remitidos por los laboratorios de salud pública al Laboratorio Nacional de Referencia del INS con el fin de que el estudio molecular de Salmonella spp. sea de rutina y refleje la situación real de la vigilancia molecular de este importante enteropatógeno en el país.
Agradecimientos
Agradecemos a la Red Nacional de Laboratorios participantes en el Programa de Vigilancia de la Enfermedad Diarreica Aguda por el esfuerzo continuo para la recuperación de los aislamientos y reconocemos con gratitud el aporte de sus conocimientos a Jean Wichard (CDC, Atlanta) y a Elkin Hernández.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses.
Financiación
Este estudio fue financiado por el Instituto Colombiano para el Desarrollo de la Ciencia y la Tecnología, Francisco José de Caldas, Colciencias, código 2104-04-12679.
Correspondencia:
Nélida Muñoz, Grupo de Microbiología, Instituto Nacional de Salud, Avenida calle 26 # 51-60 zona 6 CAN, Bogotá, D. C., Colombia.
Teléfono: 2207700 extensión 445
Fax: (+571) 2207700-446
Recibido: 28/04/06; aceptado: 03/08/06
Referencias
1. Valenzuela C. Control de enfermedades diarreicas. sl: OPS/OMS, 2006. Consultado: 3 de marzo de 2006
2. Jaramillo E, Estrada S, Ospina S. Etiología de la enfermedad diarreica aguda (EDA) de origen bacteriano, utilizando un protocolo estandarizado de laboratorio. Infectio 1999; 3: 95-9. [ Links ]
3. Popoff MY. Antigenic formulas of the Salmonella serovars. 8th revision. Paris: Pasteur Institute, World Health Organization Collaborating Centre for Reference and Research on Salmonella; 2001. [ Links ]
4. Wang L, Andrianopoulos K, Liu D, Popoff MY, Reeves PR. Extensive variation in the O-antigen gene cluster within one Salmonella enterica serogroup reveals an unexpected complex history. J Bacteriol 2002; 184: 1669-77. [ Links ]
5. Olsen SJ, Bishop R, Brenner FW, Roels TH, Bean N, Tauxe RV, et al. The changing epidemiology of Salmonella: trends in serotypes isolated from humans in the United States, 1987-1997. J Infect Dis 2001; 183: 753-61. [ Links ]
6. CDC Salmonella surveillance. Annual summary, 2002. Atlanta, GE: US Deparment of Health and Human Services, CDC; 2003. [ Links ]
7. Organización Panamericana de la Salud. Reunión anual regional de los países participantes en la red de monitoreo de vigilancia de la resistencia a los antibióticos. Asunción, Paraguay, 31 Enero - 2 Febrero, 2001. Consultado: 3 de marzo de 2006 OPS HCP/HCT/201/02.
http://www.paho.org/Spanish/AD/DPC/CD/amr-resultados-2000.htm [ Links ]8. Grupo de Microbiología, Instituto Nacional de Salud. Distribución de los aislamientos de Salmonella spp. por procedencia y por año 1997-2003. En Vigilancia de serotipos y patrones de susceptibilidad antimicrobiana de los patógenos de importancia en salud pública. Agudelo CI. Consultado: 3 de marzo de 2006 Disponible: http://www.ins.gov.co/pdf_investiga/Microbiologia_salm_05.pdf [ Links ]
9. Guzmán-Blanco M, Casellas JM, Sader HS. Bacterial resistance to antimicrobial agents in Latin America. The giant is awakening. Infecf Dis Clin North Am 2000; 14: 67-81. [ Links ]
10. Bender JB, Hedberg CW, Boxrud DJ, Besser JM, Wicklund JH, Smith KE, et al. Use of molecular subtyping in surveillance for Salmonella enterica serotype Typhimurium. N Engl J Med 2001; 344: 189-95. [ Links ]
11. Lindstedt BA, Heir E, Gjernes E, Kapperud G. DNA fingerprinting of Salmonella enterica subsp. enterica serovar Typhimurium with emphasis on phage type DT104 based on variable number of tandem repeat loci. J Clin Microbiol 2003; 41: 1469-79. [ Links ]
12. Lailler R, Grimont F, Jones Y, Sanders P, Brisabois A. Subtyping of Salmonella Typhimurium by pulsed-field gel electrophoresis and comparisons with phage types and resistance types. Pathol Biol 2002; 50: 361-8. [ Links ]
13. Tamada Y, Nakaoka Y, Nishimori K, Doi A, Kumaki T, Uemura N, et al. Molecular typing and epidemiological study of Salmonella enterica serotype Typhimurium isolates from cattle by fluorescent amplified-fragment length polymorphism fingerprinting and pulsed-field gel electrophoresis. J Clin Microbiol 2001; 39: 1057-66. [ Links ]
14. Swaminathan B, Barret TJ, Hunter SB, Tauxe RV, CDC PulseNet Task Force. PulseNet: the molecular subtyping network for foodborne bacterial disease surveillance, United States. Emerg Infect Dis 2001; 7: 382-9. [ Links ]
15. Soto S, Guerra B, Gonzáles M, Mendoza M. Potencial of three way randomly amplified polimorphic DNA analysis as a typing method for twelve Salmonella serotypes. Appl Environ Microbiol 1999; 65: 4830-6. [ Links ]
16. CDC, APHL. PulseNet. Atlanta, GA: U.S. Department of Health and Human Services, CDC; 2004. Consultado 3 de marzo de 2006. Disponible en: www.cdc.gov/pulsenet. [ Links ] 17. Red PulseNet América Latina. Pulsenet America Latina - Plan de Acción - 2004-2005. Consultado: 3 de abril de 2006. Disponible en: 18. Hidalgo M, Gracia M, Ovalle M, Wiesner M, Chávez J, Realpe ME, et al. Caracterización de los aislamientos de Salmonella Typhimurium, asociados con un brote de intoxicación alimentaria en una localidad de Pasto, Nariño. Inf Quinc Epidemiol Nac 2004; 9: 89-92. [ Links ] 19. Bopp CA, Brenner FW, Fields PI, Wells JG, Srockbine NA. Escherichia, Shigella and Salmonella. In: Murray PR, Baron EJ, Pfaller MA, Tenover FC, Yolken RH, editors. Manual of clinical microbiology. 8th ed. Washington, DC: American Society for Microbiology Press; 2003.p. 654-83. [ Links ] 20. Hunter SB, Vauterin P, Lambert-Fair MA, Van Duyne S, Kubota K, Graves L, et al. Establishment of a universal size standard strain for use with the pulseNet standardize pulse-field gel electrophoresis protocols: converting the national data bases to the new size standard. J Clin Microbiol 2005; 43: 1045-50. [ Links ] 21. Wiesner M, Hidalgo M, Castañeda E, Agudelo CI. Molecular analysis of Salmonella Enteritidis and Typhimurium clinical and food isolates by Pulse-Field Gel Electrophoresis in Bogotá, Colombia. Microb Drug Resistant 2006; 12: 68-74. [ Links ] 22. Zhao S, Fedorka-Cray PJ, Friedman S, McDermott PF, Walter RD, Qaiyumi S, et al. Characterization of Salmonella Typhimurium origin obtained from the National Antimicrobial Resistance Monitoring System. Foodborne Pathog Dis 2005; 2: 169-81. [ Links ] 23. Guerra B, Schrors P, Mendoza MC. Application of PFGE performed with XbaI to an epidemiological and phylogenetic study of Salmonella serotype Typhimurium. Relations between genetic types and phage types. New Microbiol 2000; 23: 11-20. [ Links ] 24. Tsen HY, Lin JS, Hsih HY. Pulse field electrophoresis for animal Salmonella enterica serovar Typhimurium isolates in Taiwan. Vet Microbiol 2002; 87: 73-80. [ Links ] 25. On SL, Baggesen DL. Determination of clonal relationships of Salmonella Typhimurium by numerical analysis of macrorestriction profiles. J Appl Microbiol 1997; 83: 699-706. [ Links ] 26. Fakhr MK, Nolan LK, Logue CM. Multilocus sequence typing lacks the discriminatory ability of pulsed-field gel electrophoresis for typing Salmonella enterica serovar Typhimurium. J Clin Microbiol 2005; 43: 2215-9. [ Links ] 27. Oshibe T, Hamada K. Phylogenetic analysis of genotypic variations of Salmonella Enteritidis isolates from sporadic infections using pulsed field gel electrophoresis from December 1996 to November 2000 in Hyogo Prefecture. Jpn J Infect Dis 2001; 54: 40-3. [ Links ] 28. Sulakvelidze A, Kekelidze M, Turabelidze D, Tsanava S, Tevsadze L, Devdariani L, et al. Salmonellosis in the republic of Georgia: using molecular typing to identify the outbreak-causing strain. Emerg Infect Dis 2000; 6: 70-3. [ Links ] 29. Refsum T, Heir E, Kapperud G, Vardund T, Holstad T. Molecular epidemiology of Salmonella enterica serovar Typhimurium isolates determined by pulsed-field gel electrophoresis: comparison of isolates from avian wildlife, domestic animals and the environment in Norway. Appl Environ Microbiol 2002; 68: 5600-6. [ Links ]














