Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.26 n.3 Bogotá set. 2006
Resistencia a antimicrobianos de bacilos Gram negativos aislados en unidades de cuidado intensivo en hospitales de Colombia, WHONET 2003, 2004 y 2005
María Consuelo Miranda, Federico Pérez, Tania Zuluaga, María del Rosario Olivera, Adriana Correa, Sandra Lorena Reyes, María Virginia Villegas y Grupo de Resistencia Bacteriana Nosocomial de Colombia.
Centro Internacional de Entrenamiento e Investigaciones Médicas, CIDEIM, Cali, Colombia.
El Grupo de Resistencia Bacteriana Nosocomial de Colombia está conformado por las siguientes personas en su correspondiente ciudad e institución hospitalaria: Bogotá, Clínica San Pedro Claver: Carlos Alquichire, Aura Lucía Leal, Martha Ruiz, Pilar Hurtado, Gladys Ceballos, María Mercedes Muñoz; Hospital Central de la Policía: Henry Mendoza, Alba Lucía Sanín, Olga Pinilla, Nancy Botia, Janeth Hernández, Cristina Matiz; Instituto Nacional de Cancerología: Sonia Cuervo, Jorge Cortés, Ruth Quevedo, María Cristina Paredes, Patricia Arroyo, Diana Bermúdez, Carlos Revelo. Medellín: Hospital Pablo Tobón Uribe: Carlos Ignacio Gómez, Jaime López, Mónica Cuartas, Celina Gómez, Ana Lucía Correa; Clínica de las Américas: Julián Betancourth, Diego Lalinde, Esteban Echavarría, Juan David Villa, Luz A. Álvarez, Jorge Nagles, Ana Cristina Quiroga. Cali, Hospital Universitario del Valle: Ernesto Martínez, Lena Barrera, Luz Marina Gallardo, Alba Lucía Bohorquez, Nancy Villamarín, Hilda M. Gómez, Sandra L. Ossa. Bucaramanga, Fundación Cardiovascular: Claudia Bárcenas, Adriana Pinto; La Foscal: Luis Ángel Villar. Barranquilla, Clínica General del Norte: Rubén D. Camargo, Adriana Marín, Ángela Mendoza. Pereira, Hospital Universitario San Jorge: Juan C. Cobo, Martha L. Gómez, Carmen E. Llano, Myriam Gómez, Araceli Cano.
Introducción. Los sistemas de vigilancia son una pieza clave para la detección y control de la resistencia bacteriana. Es indispensable recolectar constantemente la información de cada institución por la variabilidad existente entre países, ciudades y hospitales frente a los mecanismos de resistencia bacteriana y así plantear intervenciones apropiadas para cada institución. De enero 2003 a diciembre de 2005, el Centro Internacional de Entrenamiento e Investigaciones Médicas (CIDEIM) desarrolló un proyecto de vigilancia en un grupo de 10 hospitales de tercer nivel, en seis ciudades de Colombia.
Objetivos. Presentar el comportamiento de Escherichia coli, Klebsiella pneumoniae, Pseudomonas aeruginosa, Acinetobacter baumannii y Enterobacter cloacae, considerados los Gram negativos patógenos más relevantes en infección nosocomial, frente a antimicrobianos seleccionados.
Materiales y métodos. Las pruebas de susceptibilidad se realizaron por métodos automatizados en 9 hospitales y por Kirby Bauer en un hospital. Se eligieron antibióticos con actividad reconocida contra Gram negativos, de acuerdo con las guías del Comité Nacional para el Control de Estándares en el Laboratorio Clínico (NCCLS). Los laboratorios realizaron control de calidad interno y externo. Mensualmente se recibió la información procedente del laboratorio de microbiología de cada institución y se centralizó en una base de datos en WHONET 5.3 en CIDEIM. Se realizó un análisis conglomerado de frecuencias y porcentajes de resistencia a antibióticos.
Resultados. Los altos porcentajes de resistencia reportados en especial para A. baumannii, corroboraron la presencia de bacterias multirresistentes en las UCI en las instituciones participantes durante el periodo de estudio.
Conclusiones. Es urgente crear una red nacional de vigilancia de la resistencia a antimicrobianos de los patógenos hospitalarios y de esta manera mejorar nuestra habilidad para detectar, supervisar y manejar la resistencia a antimicrobianos en Colombia.
Palabras clave: Vigilancia epidemiológica, resistencia antimicrobiana a drogas, antibióticos, bacilos Gram negativos, software.
Antimicrobial resistance in Gram negative bacteria isolated from intensive care units of Colombian hospitals, WHONET 2003, 2004 and 2005
Introduction. Surveillance systems play a key role in the detection and control of bacterial resistance. It is necessary to constantly collect information from all institutions because the mechanisms of bacterial resistance can operate in different ways between countries, cities and even in hospitals in the same area. Therefore local information is important in order to learn about bacterial behaviour and design appropriate interventions for each institution. Between January 2003 and December 2004, the Centro Internacional de Entrenamiento e Investigaciones Médicas (CIDEIM) developed a surveillance project in 10 tertiary hospitals in 6 cities of Colombia.
Objectives. Describe the trends of antibiotic resistance among the isolates of Escherichia coli, Klebsiella pneumoniae, Pseudomona aeruginosa, Acinetobacter baumannii and Enterobacter cloacae, five of the most prevalent nosocomial Gram negative pathogens.
Materials and Methods: The susceptibility tests were performed by automated methods in 9 hospitals and by Kirby Bauer in 1 hospital. Antibiotics with known activity against Gram negatives, according to the Clinical Laboratory Standards Institute guidelines, were selected. The laboratories performed internal and external quality controls. During the study period, the information was downloaded monthly from the databases of each microbiology laboratory and sent to CIDEIM where it was centralized in a database using the system WHONET 5.3.
Results. The high resistance rates reported especially for A. baumannii, evidenced the presence of multidrug resistant bacteria in both ICUs and wards at every studied institution.
Conclusions. The creation of a national surveillance network to improve our capabilities to detect, follow up, and control the antibiotic resistance in Colombia is urgently needed.
Key words: Epidemiologic surveillance, drug resistance, bacterial, Gram-negative bacteria, antibiotic, software.
La resistencia bacteriana a antimicrobianos es un problema emergente a nivel mundial (1,2). El incremento de la prevalencia de Streptococcus pneumonie resistente a la penicilina, Staphylococcus aureus resistente a la meticilina, Enterococcus resistente a la vancomicina y de bacilos Gram negativos productores de beta-lactamasas es apenas un ejemplo del creciente problema de la resistencia documentado recientemente por sistemas de vigilancia nacionales e interna-cionales (2). Existen diferencias geográficas, con tasas de resistencia y mecanismos subyacentes característicos de cada región y de cada institución (3). Por ello es indispensable conocer la epidemiología local para plantear estrategias encaminadas a disminuir y controlar la resistencia bacteriana en cada institución.
Los sistemas de vigilancia realizan el seguimiento de los cambios de sensibilidad a antimicrobianos. Según el Centro para el Control y Prevención de Enfermedades de Atlanta (CDC, EEUU), el funcionamiento ideal de tales drogas implica la recolección, análisis e interpretación de datos en forma permanente y sistemática con el fin de utilizar los resultados adecuadamente en las instituciones hospitalarias (4). Además, esta información debe ayudar a definir acciones de salud pública y a medir los efectos de cualquier intervención (5).
Con el propósito de permitir la comparación de la información generada en los laboratorios de microbiología, John Stelling y Thomas OBrien, bajo el auspicio de la Organización Mundial de la Salud (OMS / WHO), desarrollaron WHONET, una aplicación diseñada para la recolección y análisis de información proveniente de los laboratorios de microbiología (6). La aplicación permite que los laboratorios guarden sus datos en un lenguaje común, comparable con el de otros laboratorios alrededor del mundo, y de esta manera facilita la conformación de redes de información en resistencia bacteriana. Gracias a estos avances, en la última década han surgido múltiples sistemas de vigilancia de la resistencia bacteriana. Como ejemplo se pueden citar la Red Internacional para el Estudio y Prevención de la Resistencia Antimicrobiana Emergente (INSPEAR) (160 centros en 40 países), el Sistema Europeo de Vigilancia de la Resistencia Antimicrobiana (EARSS) (375 centros en 15 países) y el programa de vigilancia antimicrobiana SENTRY (80 centros en todo el mundo, de ellos 10 en 30 países de Latinoamérica) (7). Desde hace varios años, países latinoamericanos como Argentina, Chile, Brasil y Venezuela vienen desarrollando sus propios sistemas de vigilancia (8-11). En Colombia, WHONET se empezó a utilizar en grupos pequeños, pero sólo en el año 2002, con la automatización de los laboratorios de microbiología, aumentó su popularidad entre los grupos de microbiología y comités de infecciones del país. En ese año, el CIDEIM utilizó el WHONET 5.1 para un estudio descriptivo de la resistencia a los antimicrobianos en las bacterias Gram negativas más frecuentes a nivel hospitalario en un grupo de nueve hospitales en tres ciudades de Colombia. En este primer estudio se encontró una mayor frecuencia de fenotipos sugestivos de beta-lactamasas de espectro extendido (BLEE) que la descrita en otros países como Estados Unidos. Mientras que en Estados Unidos 3 a 5% de los aislamientos de E. coli y 10 a 12% de K. pneumoniae tienen fenotipos sugestivos de BLEE, en Colombia se encontró entre 8 y 11% en E. coli y 20 a 30% en K. pneumoniae (12). Adicionalmente, se describió la primera BLEE tipo cefotaximasa (CTX-M-12) en el país (13). La epidemiología de estos hospitales evidenció también la presencia de P. aeruginosa y A. baumannii multirresistentes a la mayoría de antibióticos, tales como cefalosporinas, quinolonas y betalactámicos, e incluso los carba-penemos. Además, este primer estudio realizado en el 2002, evidenció algunas deficiencias de los laboratorios de microbiología en el tamizaje de las BLEE y permitió dar pautas para aumentar la detección de estas enzimas con el uso mínimo de dos cefalosporinas de tercera generación en las pruebas de sensibilidad, así como la inclusión de la prueba confirmatoria para BLEE (14,15).
Este trabajo tiene como objetivo describir los hallazgos del análisis de los datos recolectados y centralizados en WHONET por el CIDEIM en los años 2003, 2004 y 2005 con el fin de mostrar los patrones de resistencia a los antibióticos de los Gram negativos más frecuentemente hallados a nivel hospitalario en un grupo de 10 hospitales, y conocer su comportamiento en Colombia.
Materiales y métodos
Este es un estudio descriptivo realizado en una muestra por conveniencia en 10 instituciones hospitalarias de seis ciudades de Colombia, que describe el comportamiento de los Gram negativos E. coli, K. pneumoniae, P. aeruginosa, A. baumannii y E. cloacae en términos de porcentajes de resistencia a los antimicrobianos en aislamientos clínicos provenientes de pacientes hospitalizados en las unidades de cuidado intensivo (UCI) durante el periodo comprendido entre el 1 de enero del 2003 y el 31 de diciembre del 2005.
Instituciones participantes y metodología utilizada
Se incluyeron diez hospitales de tercer nivel en las siguientes ciudades: Bogotá, Clínica San Pedro Claver, Instituto Nacional de Cancerología y Hospital Central de la Policía; Barranquilla, Clínica General del Norte; Bucaramanga, Clínica La Foscal y Fundación Cardiovascular; Medellín, Hospital Pablo Tobón Uribe y Clínica Las Américas; Cali, Hospital Universitario del Valle, y Pereira, Hospital Universitario San Jorge. Tres de estos hospitales son públicos, seis privados y uno de régimen especial, y tienen un promedio de 342 camas (rango 63 a 965 camas). Todos cuentan con UCI de adultos con un promedio de 14 camas. Nueve de estas instituciones tienen, además, UCI neonatales con un promedio de ocho camas. Además, en cinco de estas instituciones existe un programa de trasplantes. Nueve instituciones realizan las pruebas de sensibilidad a los antibióticos con métodos automatizados (3 MicroScan y 6 Vitek) y una las realiza por el método de difusión de disco Kirby-Bauer. Se seleccionó un grupo de antibióticos con actividad reconocida contra bacilos Gram negativos de acuerdo con las guías del NCCLS, que a partir de enero del 2005 se conoce como Instituto de Estándares Clínicos y de Laboratorio (CLSI) (15).
Control de calidad en los laboratorios participantes
Los laboratorios de microbiología de estas instituciones llevaron a cabo el control de calidad interno con cepas ATCC de referencia (E. coli ATCC 25922 y P. aeruginosa ATCC 27853) y control de calidad externo mediante programas de evaluación del desempeño con cepas de referencia y contrarreferencia en laboratorios establecidos para este fin así: ocho instituciones con el Grupo de Microbiología del Instituto Nacional de Salud, y, además, cuatro de estas instituciones con la Corporación de Investigaciones Biológicas. Seis instituciones con el Programa de Control de Eficiencia Americano, denominado Medical Laboratory Control - Randox International Quality Assessment Scheme; dos de ellas con el Laboratorio Departamental de Antioquia y una de éstas también con el Colegio Americano de Patología. Uno de los centros refirió no tener control de calidad interno, pero sí haber participado en un programa de control de calidad externo mensual durante los tres periodos.
Recolección de la información
Durante los tres años del periodo de estudio se recibieron mensualmente los resultados de todos los cultivos bacterianos obtenidos de muestras clínicas de pacientes hospitalizados en cada institución (se excluyeron urgencias y consulta externa). Esta información se obtuvo exportando a un disquete los datos de los equipos automatizados MicroScan y Vitek del laboratorio de microbiología de cada hospital. Uno de los hospitales, que realiza las pruebas de identificación y sensibilidad a antibióticos mediante Kirby-Bauer, envió los resultados ya incluidos en archivos WHONET 5.1 en el 2003 y WHONET 5.3 en el 2004 y 2005. La información procedente de los hospitales se incorporó mediante la aplicación BacLink a una base de datos administrada en el CIDEIM con el programa WHONET 5.3 (OMS, 2003).
Análisis de la información
Basados en las recomendaciones de la OMS, sólo se incluyó el primer cultivo positivo por paciente para evitar sesgos por sobreestimación de resistencia al incluir cultivos duplicados de pacientes con gérmenes resistentes (3). En este manuscrito sólo se presentan los datos de los aislamientos procedentes de las UCI.
Se realizó el análisis estadístico descriptivo de datos del conglomerado de los 10 hospitales utilizando los comandos del programa WHONET 5.3 (listado de frecuencias de microorganismos y porcentajes de resistencia a antibióticos seleccionados). Primero se obtuvo una lista de los microorganismos más frecuentes en muestras procedentes de las UCI. Luego se obtuvieron los porcentajes de resistencia a los antibióticos más usados para tratamiento de los bacilos Gram negativos y los utilizados como marcadores epidemiológicos de resistencia de acuerdo con los estándares del CLSI. Se realizó la comparación múltiple de los porcentajes de frecuencia y de resistencia en los años 2003, 2004 y 2005 por medio del estadístico ji cuadrado para probar la diferencia entre proporciones (k = 3). Se utilizó un nivel de significación de 0,05 para establecer diferencias estadísticamente significativas.
El análisis de los porcentajes de resistencia a antibióticos de los bacilos Gram negativos más frecuentes en estos hospitales se realizó bajo criterios uniformes, ya que todos los laboratorios manejan concentraciones acordes con los criterios de resistencia y sensibilidad del CLSI. Los antibióticos utilizados para el análisis fueron cefotaxima (CTX), ceftriaxona (CRO), ceftazidima (CAZ), cefepima (FEP), ciprofloxacina (CIP), amikacina(AMK), piperacilina/tazobactam (TZP), imipenem (IPM) y meropenem (MEN) para E. coli y K. pneumoniae; CAZ, FEP, CIP, AMK, aztreonam (AZT), TZP, IPM y MEN para P. aeruginosa ; CAZ, CIP, AMK, IPM y MEN para A. baumannii, y CAZ, FEP, CIP, AMK, AZT, IMP y MEN para E. cloacae.
Actividades realizadas en los hospitales
Una vez realizados los procesos de recepción y análisis de la información, el CIDEIM elaboró reportes trimestrales que se enviaron a los comités de infecciones, al laboratorio de microbiología y al infectólogo de cada institución participante. Estos reportes incluyeron un análisis descriptivo de la información microbiológica, los probables mecanismos de resistencia y las recomendaciones a seguir para la implementación de medidas de control de infecciones con énfasis en la higiene de manos, uso racional de antibióticos, y los fenotipos de resistencia que deberían vigilar y enviar al CIDEIM para estudios moleculares posteriores.
Resultados
Se registraron 4.008 aislamientos provenientes de las UCI en el 2003, 4.004 en el 2004 y 4.304 en el 2005. Los 10 gérmenes más frecuentemente aislados en las UCI constituyeron el 70% de todos los aislamientos en los tres periodos. Entre los más frecuentes estuvieron, en su orden: S. aureus, E. coli, P. aeruginosa K. pneumoniae, A. baumannii y E. cloacae. Se observó una disminución estadísticamente significativa en el número de aislamientos de A. baumannii (p < 0,001) (
cuadro 1).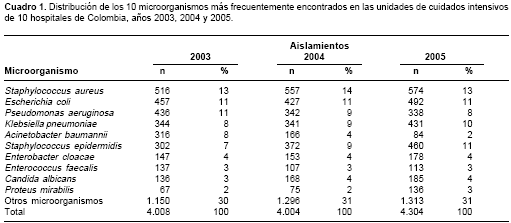
Resistencia de bacilos Gram negativos a cefalosporinas de tercera y cuarta generación
E. coli y K. pneumonie presentaron fenotipos sugestivos de BLEE durante los tres años del estudio. E. coli presentó un porcentaje de ceftazidimasas y cefotaximasas bajo (2 a 6%) en comparación con K. pneumoniae que estuvo entre 21 y 17%, al igual que la resistencia a FEP que estuvo entre 2 y 4% para E. coli y entre 10 y 13% para K. pneumoniae (cuadro 2).
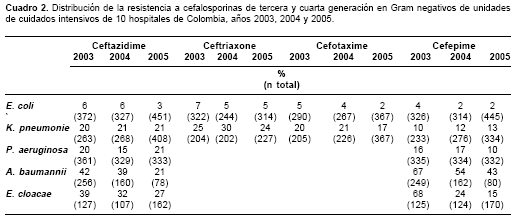
P. aeruginosa
mostró una disminución estadística-mente significativa (p < 0,021) en la resistencia a FEP, a diferencia de la resistencia a CAZ, que fue estable (cuadro 2).Aunque los porcentajes de resistencia de A. baumannii a CAZ (p< 0,003) y a FEP (p < 0,001) descendieron de forma significativa en los tres años, esta bacteria mostró un perfil de multirresistencia.
E. cloacae mantuvo porcentajes de resistencia altos frente a CAZ, mientras que la resistencia a FEP descendió en forma estadísticamente significativa (p < 0,001) (
cuadro 2).Resistencia de bacilos Gram negativos a piperacilina/tazobactam, ciprofloxacina y amikacina
E. coli mostró bajos porcentajes de resistencia a TZP y a AMK y alta frecuencia de resistencia a CIP. K. pneumonie presentó porcentajes de resistencia menores que E. coli frente a CIP y AMK durante los tres años y permaneció estable frente a TZP (
cuadro 3).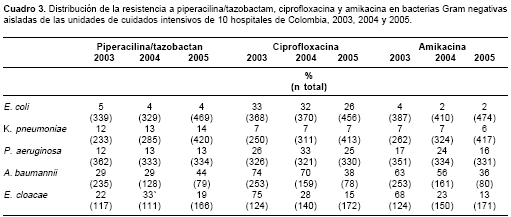
P. aeruginosa
mostró un promedio de 13% de resistencia a TZP y presentó un incremento de la resistencia a CIP y AMK del 2003 al 2004, pasan-do de 26 a 33% y de 17 a 24%, respectivamente, pero las diferencias no fueron estadísticamente significativas en los tres años (cuadro 3).A. baumannii presentó disminución significativa de la resistencia a CIP y AMK (p < 0,001) y un incremento de la resistencia a TZP (p < 0,034) (
cuadro 3).Los porcentajes de resistencia de E. cloacae descendieron significativamente frente a TZP (p < 0,024), CIP y AMK (p < 0,001) (
cuadro 3).Resistencia de bacilos Gram negativos a carbapenemos
E. coli y K. pneumonie no presentaron resistencia frente a carbapenemos (
cuadro 4).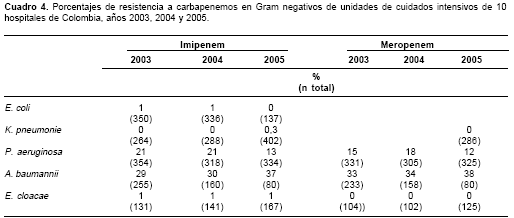
La resistencia de P. aeruginosa a IMP (p < 0.001) y MEN (p < 0.03) disminuyó en el año 2005 en forma estadísticamente significativa (
cuadro 4).Como se mencionó anteriormente, A. baumannii mostró resistencia alta y persistente a todos los antibióticos, incluso a los carbapenemos (
cuadro 4).E. cloacae mantuvo una resistencia de 1% a IMP y no se observó resistencia a MEN (
cuadro 4).Discusión
Este estudio constituye un primer paso en el desarrollo de un sistema de vigilancia para la resistencia bacteriana hospitalaria a nivel nacional, con recolección de datos microbiológicos en forma permanente y retroalimentación a las instituciones hospitalarias participantes en forma trimestral a través de reportes de los resultados y análisis de la resistencia hospitalaria, incluidas recomen-daciones para el manejo racional de antibióticos de acuerdo con los perfiles de resistencia observados. Durante este tiempo se estandarizó el tamizaje sistemático de antibióticos, lo que permitió comparar en forma más confiable y precisa el comportamiento de las bacterias hospitalarias frente a los antibióticos seleccionados y hacer un seguimiento a través del tiempo.
Los altos porcentajes de resistencia reportados coinciden con los resultados de otros informes de vigilancia de la resistencia antimicrobiana en el mundo, tales como los hallazgos de SENTRY presentados en la 37 Conferencia Internacional de Agentes Antimicrobianos y Quimioterapia (ICAAC). Este informe muestra la emergencia de la alta resistencia de K. pneumoniae y destaca que, especialmente en Latinoamérica, las cepas resistentes son tres veces más prevalentes que en los Estados Unidos y tienen un mayor porcentaje de resistencia (37%) a cefalosporinas de tercera generación como la CAZ. Un informe más reciente (16) resalta entre los resultados importantes la resistencia que presentan la mayoría de bacterias, incluidas las Gram negativas, a las fluoroquinolonas (CIP) y la resistencia de P. aeruginosa y Acinetobacter ssp a todos los antimicrobianos.
Los fenotipos encontrados en cada bacteria ayudaron a sospechar mecanismos de resistencia específicos presentes en estos tres años.
Lo más notorio en E. coli es la alta resistencia a CIP, la cual se utiliza como marcador, ya que una vez una bacteria Gram negativa es resistente a este antibiótico debe considerársele resistente a las demás quinolonas (17). El fenotipo de resistencia a las cefalosporinas de tercera generación sugiere la producción de BLEE durante estos tres años.
K. pneumonie presentó un fenotipo productor de BLEE más frecuente que E. coli, como se ha descrito en otras partes del mundo y en Colombia (12,13,18-21). La resistencia baja de Klebsiella a CIP, contraria a la alta resistencia de E. coli, es un buen ejemplo de cómo los mecanismos de resistencia operan en forma diferente en cada bacteria y de qué manera la selección de resistencia puede ser específica para cada antibiótico.
P. aeruginosa presentó perfiles de resistencia más elevados a los antibióticos tamizados y especialmente es notoria la resistencia a CIP, por lo que es importante insistir en restringir su uso empírico. Nuevamente aparecen diferencias entre bacterias de distintas especies en su comportamiento frente a los antibióticos, así, es inusual encontrar resistencia a carbapenemos en enterobacterias, mientras que P. aeruginosa puede seleccionar mayor resistencia a este grupo de antibióticos (22). El amplio espectro de un antibiótico y su potencia in vitro no necesariamente se relacionan con la capacidad de la bacteria de seleccionar ciertos mecanismos de resistencia inherentes; el caso específico es el cierre de la porina Opr D para el paso de los carbapenemos al interior de P. aeruginosa, que asociada con una producción de AmpC, puede generar resistencia a carbapenemos y no necesariamente a otros betalactámicos (22).
De todas las bacterias estudiadas, A. baumannii presentó los porcentajes más elevados de multirresistencia. Por ello, aunque hubo una disminución importante en el número de aislamientos de esta bacteria en las UCI, constituye una gran preocupación, ya que su presencia está ligada a brotes de infección hospitalarias y a una gran dificultad terapéutica debido a su elevada multirresistencia. Su presencia siempre obliga a investigar la transmisión cruzada de clones secundaria a una falla en el lavado de manos y barreras de contacto (23-25) (Hospital Universitario del Valle, CIDEIM. Poster S10, Congreso Nacional de Infectología. ACIN, Cartagena, Colombia, 2003). La multirresistencia de A. baumannii también puede deberse al uso indiscriminado de antibióticos de amplio espectro, los cuales están asociados con la selección de bacterias multirresistentes (26).
E. cloacae posee el gen capaz de producir la betalactamasa AmpC, y el uso de cefalosporinas de tercera generación está asociado con la selección de cepas de-represoras, lo que lleva a fallas terapéuticas durante el tratamiento con estos antibióticos (27). Los perfiles de resistencia a cefalosporinas de tercera generación presentes en E. cloacae sugieren una elevadasíma producción de AmpC. Entre todas las enterobacterias prevalentes en este estudio, E. cloacae podría ser la de mayor capacidad de seleccionar resistencia, incluso a los carbapenemos, al producir altísimas cantidades de AmpC y cerrar porinas (21). Los antibióticos con los que mostró una menor resistencia fueron los carbapenemos FEP y AMK.
La información de los estudios de vigilancia coordinados nos enseña cómo la resistencia antimicrobiana varía geográfica y temporalmente. La vigilancia constante de las tasas de resistencia puede ayudar a asignar eficientemente los recursos, racionalización que a su vez puede disminuir los costos de los cuidados en salud preservando el poder de los antibióticos considerados de primera línea. El desarrollo y la expansión de los sistemas de laboratorio responsables de recolectar los datos de la vigilancia es un componente esencial en la tarea de detectar la resistencia antimicrobiana. Estos laboratorios ayudan a la difusión de guías de tratamiento y estrategias de control. Por otra parte, en el marco del sistema de vigilancia de la resistencia es importante implementar un sistema unificado de control de calidad interno y externo para los laboratorios hospitalarios. Un estudio de vigilancia realizado en 90 centros de INSPEAR (28) con el objetivo de medir los métodos usados para la identificación de S. aureus y la determinación de la susceptibilidad a antibióticos reveló muchas deficiencias en los laboratorios de estos centros hospitalarios, entre otras, en el reporte y la confirmación de S. aureus resistente a vancomicina y a teicoplanina, en el uso de discos de oxacilina con concentración del antimicrobiano diferente a la recomendada por el laboratorio de referencia, y, adicionalmente, el 20% de los centros no tenía control de calidad interno y el 36% de ellos no participaba en programas de control de calidad externos. Esta información demostró claramente la necesidad de fortalecer la capacidad de los laboratorios en el área de microbiología para detectar la resistencia y apoyar la vigilancia epidemiológica en estos centros. Además, el conocimiento de la epidemiología de la resistencia hospitalaria y de los fenotipos bacterianos permite sospechar mecanismos de resistencia específicos para ciertos antibióticos. Ello permite, a su vez, seleccionar con mayor precisión el antibiótico más estable frente a este mecanismo de resistencia y lograr su uso adecuado.
De acuerdo con los hallazgos de este estudio, en Colombia es urgente crear una red nacional de vigilancia de la resistencia de los patógenos hospitalarios para mejorar nuestra habilidad para detectar, supervisar y manejar la resistencia a los antimicrobianos.
Este trabajo es el producto de un esfuerzo interinstitucional por establecer un sistema de vigilancia que suministre información útil y ayude a optimizar estrategias para contener la resistencia bacteriana a nivel hospitalario.
Agradecimientos
Agradecemos a John Stelling por su permanente asesoría en el uso de WHONET, a Nancy Gore Saravia del CIDEIM, y a Elizabeth Castañeda por la revisión de este manuscrito.
Conflicto de intereses
María Virginia Villegas ha trabajado como asesora y conferencista de AstraZéneca, Bristol Myers Squibb, Merck Sharp & Dohme, Pfizer y Wyeth-Ayerst.
Financiación
Este trabajo fue financiado por la Gobernación del Valle del Cauca, AstraZéneca, Bristol Myers Squibb, Merck Sharp & Dohme, Pfizer, Wyeth-Ayerst y CIDEIM.
Correspondencia:
María Virginia Villegas, CIDEIM, Av 1N #3-03, Cali, Colombia.
Teléfono: 668 2164, Fax: 6642989.
Recibido: 10/10/05; aceptado: 10/07/06
Referencias
1. Tenover FC, Mohammed MJ, Stelling J, O´Brien T, Williams R. Ability of laboratories to detect emerging antimicrobial resistance: proficiency testing and quality control results from the World Health Organization´s external quality assurance system for antimicrobial susceptibility testing. J Clin Microbiol 2001; 39: 241-50. [ Links ]
2. Organización Mundial de la Salud. Estrategia mundial de la OMS para contener la resistencia a los antimicrobianos. Ginebra: WHO; 2001.p. 11-7. [ Links ]
3. Winokur PL, Canton R, Casellas JM, Legakis N. Variations in the prevalence of strains expressing an extended-spectrum beta-lactamase phenotype and characterization of isolates from Europe, the Americas, and the Western Pacific region. Clin Infect Dis 2001; 32 (Suppl. 2): S94-103. [ Links ]
4. Klaucke DN, Buehler JW, Thacker SB, Parrish RG, Tiowbridge FL, Berkelman RL. Guidelines for evaluating surveillance systems. MMWR Morb Mortal Wkly Rep 1988; 37 (Suppl 5): 1-18. [ Links ]
5. World Health Organization. Surveillance standards for antimicrobial resistance. Geneva: WHO; 2002. [ Links ]
6. OBrien TF, Stelling JM. WHONET: an information system for monitoring antimicrobial resistance. Emerg Infect Dis 1995: 1; 66. [ Links ]
7. Bax R, Bywater R, Cornaglia G, Goossens H, Hunter P, Isham V, et al. Surveillance of antimicrobial resistance: what, how and whither? Clin Microbiol Infect 2001; 7: 316-25. [ Links ]
8. Rossi A, Tokumoto M, Galas M, Soloaga R, Corso A. Monitoring antibiotic resistance in Argentina. The WHONET program 1995-1996. Rev Panam Salud Publica 1999; 6: 234-41. [ Links ]
9. Sader HS, Gales AC, Pfaller MA, Mendes RE, Zoccoli C, Barth A, et al. Pathogen frequency and resistance patterns in Brazilian hospitals: summary of results from three years of the SENTRY Antimicrobial Surveillance Program. Braz J Infect Dis 2001; 5: 200-14. [ Links ]
10. Valdivieso F, Trucco O, Prado V, Diaz MC, Ojeda A. Antimicrobial resistance of agents causing urinary tract infections in 11 Chilean hospitals. PRONARES project. Rev Med Chil 1999; 127: 1033-40. [ Links ]
11. Comegna M, Guzmán BM, Carmona O, Molina M, Grupo Colaborativo Venezolano de Resistencia Bacteriana. Resistencia bacteriana a los antimicrobianos en Venezuela-Nuevos hallazgos. Bol Soc Ven Microb 2000; 20: 58-63 [ Links ]
12. Villegas MV, Correa A, Perez F, Miranda MC, Zuluaga T, Quinn JP, et al. Prevalence and characterization of extended-spectrum beta-lactamases in Klebsiella pneumoniae and Escherichia coli isolates from Colombian hospitals. Diagn Microbiol Infect Dis 2004; 49: 217-22. [ Links ]
13. Villegas MV, Correa A, Perez F, Zuluaga T, Radice M, Gutkind G, et al. CTX-M-12 beta-lactamase in a Klebsiella pneumoniae clinical isolate in Colombia. Antimicrob Agents Chemother 2004; 48: 629-31. [ Links ]
14. Centers for Disease Control and Prevention (CDC). Laboratory capacity to detect antimicrobial resistance 1998. MMWR Morb Mortal Wkly Rep 2000; 48: 1167-71. [ Links ]
15. NCCLS. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard M7-A6. 6th edition. Vol. 23. Wayne PA: National Committee for Clinical Laboratory Standards; 2003. [ Links ]
16. Sader HS, Jones RN, Gales AC, Silva JB, Pignatari AC, SENTRY Participants Group (Latin America). SENTRY antimicrobial surveinllance program report: Latin American and Brazilian results for 1997 through 2001. Braz J Infect Dis 2004; 8: 25-79. [ Links ]
17. Mandel GL, Bennett JE, Dolin R. Principles and practices of infectious diseases. 6th edition. New York: Churchill Livingston Inc; 2005. [ Links ]
18. Naumovski L, Quinn JP, Miyashiro D, Patel M, Bush K, Singer SB, et al. Outbreak of ceftazidime resistance due to a novel extended-spectrum b-lactamase in isolates from cancer patients. Antimicrob Agents Chemother 1992; 36: 1991-6. [ Links ]
19. Schiappa DA, Hayden MK, Matushek MG, Hashemi FN, Sullivan J, Smith KY, et al. Ceftazidime-resistant Klebsiella pneumoniae and Escherichia coli bloodstream infection: a case-control and molecular epidemiologic investigation. J Infect Dis 1996; 174: 529-36. [ Links ]
20. Wong-Beringer A, Hindler J, Loeloff M, Queenan AM, Lee N, Peggues DA, et al. Molecular correlation for the treatment outcomes in bloodstream infections caused by Escherichia coli and Klebsiella pneumoniae with reduced susceptibility to ceftazidime. Clin Infect Dis 2002; 34: 135-46. [ Links ]
21. Livermore DM. Beta-lactamases in laboratory and clinical resistance. Clin Microbiol 1995; 8: 557-84. [ Links ]
22. Livermore DM. Multiple mechanism of antimicrobial resistance in Pseudomonas aeruginosa: Our worst nightmare? Clin Infect Dis 2002; 34: 634-40. [ Links ]
23. Higgins PG, Wisplinghoff H, Stefanik D, Seifert H. In vitro activities of beta-lactamase inhibitors clavulanic acid sulbactam, and tazobactam alone or in combination with beta-lactams against epidemiologically characterized multidrug-resistant Acinetobacter baumannii strains. Antimmicrob Agents Chemother 2004; 48: 1586-92. [ Links ]
24. Gales AC, Jones RN, Forward KR, Liñares J, Sader HS, Verhoef J. Emerging importance of multidrug-resistant Acinetobacter species and Stenotrophomonas maltophilia as pathogens in seriously ill patients: geographic patterns, epidemiological features and trends in the SENTRY Antimicrobial Surveillance Program (1997-1999). Clin Infect Dis 2001; 32 (Suppl 2): S104-13. [ Links ]
25. Villegas MV, Hartstein AI. Acinetobacter outbreaks, 1977-2000. Infect Control Hosp Epidemiol 2003; 24: 284-95. [ Links ]
26. Scerpella EG, Wanger AR, Armitige L, Anderlini P, Ericsson CD. Nosocomial outbreak caused by a multiresistant clone of Acinetobacter baumannii: results of the case-control and molecular epidemiologic investigations. Infect Control Hosp Epidemiol 1995; 2: 92-7. [ Links ]
27. Chow JW, Fine MJ, Shlaes DM, Quinn JP, Hooper DC, Johnson MP, et al. Enterobacter bacteremia: Clinical features and emergence of antibiotic resistance during therapy. Ann Intern Med 1991; 115: 585-90. [ Links ]
28. Richet HM, Mohammed J, McDonald C, Jarvis WR, INSPEAR. Building communication networks: international network for the study and prevention of emerging antimicrobial resistance. Emerg Infect Dis 2001; 7: 319-22. [ Links ]














