Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.26 n.3 Bogotá set. 2006
Tipificación molecular de Listeria monocytogenes aisladas de muestras clínicas y alimentos
Mayra Viviana Medrano 1, Silvia Restrepo 2, María Consuelo Vanegas 1,
1 Laboratorio de Ecología Microbiana de Alimentos, Departamento de Ciencias Biológicas, Universidad de Los Andes, Bogotá, Colombia.
2 Laboratorio de Micología y Fitopatología, Departamento de Ciencias Biológicas, Universidad de Los Andes, Bogotá, Colombia.
Introducción. Listeria monocytogenes es un patógeno emergente adquirido por el consumo de alimentos contaminados. Causa una enfermedad llamada listeriosis, cuya tasa de mortalidad a nivel mundial varía entre 20% y 30%, alcanzando hasta un 80% en casos de infecciones neonatales. La técnica del ADN polimorfo amplificado aleatorio permite distinguir entre diferentes aislamientos y caracterizarlos molecularmente, lo cual aporta información útil acerca de la diversidad de este patógeno en Colombia.
Objetivo. Caracterizar molecularmente diferentes aislamientos de L. monocytogenes aisladas de muestras clínicas y alimentos utilizando ésta técnica para determinar posibles relaciones entre estos dos orígenes.
Materiales y métodos. Se analizaron 38 aislamientos de L. monocytogenes; 22 de muestras clínicas y 16 de alimentos y plantas procesadoras de alimentos utilizando dos oligonucléotidos de 10pb (HLWL-74 y Arbitrario). Los datos se analizaron utilizando los programas Quantity One y SYN-TAX.
Resultados. Se detectó un alto porcentaje de polimorfismo mediante los oligonucleótidos HLWL-74 (81,81%) y Arbitrario (85,71%). Se pudieron describir dos linajes superiores luego del análisis, los cuales se dividieron a su vez en cuatro grupos mayores (A, B C y D) donde se observó una gran diversidad genética. La mayoría de aislamientos clínicos se agruparon bajo el mismo grupo y se encontraron alejados de los aislamientos de alimentos.
Conclusión. Los resultados de este estudio demuestran que existe una gran diversidad de polimorfismos de ADN entre los aislamientos de L. monocytogenes que circulan en Colombia, lo que podría reflejar diferencias a nivel fenotípico y patogénico en estos aislamientos.
Palabras claves: Listeria monocytogenes, listeriosis, muestras de alimentos, reacción en cadena de la polimerasa, técnica del ADN polimorfo amplificado aleatorio, polimorfismo genético.
Molecular typing of Listeria monocytogenes isolated from clinical and food samples
Introduction. Listeria monocytogenes is an emergent foodborne pathogen acquired by the ingestion of contaminated food. This bacterium causes a disease called listeriosis, whose mortality rate world wide is around 20% to 30%, reaching up to 80% in cases of neonatal infections. The random amplified polymorphic DNA technique allows different isolates to be distinguished and characterized at the molecular level, which can provide useful information about the diversity of this pathogen in Colombia.
Objective. To molecularly characterize different L. monocytogenes isolates from food and clinical samples using this technique to determine possible relationships among these two origins.
Materials and methods. Thirty eight L. monocytogenes isolates were analyzed; 22 from human clinical samples and 16 from food processing plants and food using two 10bp primers (HLW74, Arbitrary). The data were analyzed using Quantity One and SYN-TAX software.
Results. A high percentage of polymorphism was detected with both primers (HLWL-74, 81,81%; Arbitrary, 85,71%). Two major lineages were found, which were divided into four major clusters (A, B C y D) and great genetic diversity was observed. Most of the clinical isolates were grouped within the same cluster, and were more distantly related to the food isolates.
Conclusion. The results of this study demonstrate a high degree of genetic diversity of DNA polymorphisms among the L. monocytogenes isolates circulating in Colombia, which could reflect phenotypic and pathogenic differences in these isolates.
Key words: Listeria monocytogenes, Listeria infections, food samples, polymerase chain reaction, random amplified polymorphic DNA technique, polymorphism, genetic.
L. monocytogenes es un patógeno emergente adquirido por el consumo de alimentos contaminados y que causa listeriosis, una enfermedad invasiva que involucra complicaciones muy serias como meningitis, septicemia e infecciones perinatales (1,2), y con una mortalidad a nivel mundial que varía entre 20 y 30%, alcanzando hasta un 70% en casos de meningitis, 50% en casos de septicemia y hasta un 80% en casos de infecciones perinatales o neonatales (3). Entre las infecciones perinatales, la infección por L. monocytogenes es de baja frecuencia y con características clínicas poco específicas que la diferencien de otras infecciones perinatales (4), lo que dificulta su diagnóstico. La listeriosis es más frecuente en grupos considerados de alto riesgo como niños, ancianos y mujeres embarazadas, ya que puede causar parto prematuro, aborto, muerte del feto o enfermedad grave en el recién nacido e incluso su muerte; también es de alto riesgo en personas inmunosuprimidas. Las personas que no pertenecen a estos grupos se pueden enfermar, pero la probabilidad es más baja (5-7).
La listeriosis ha surgido como una enfermedad muy importante de origen alimentario en las últimas dos décadas; el primer brote estudiado ocurrió en 1981 en Nueva Escocia, Canadá, por ensalada de repollo contaminada (1). Debido a la habilidad de sobrevivir y crecer bajo condiciones adversas y en refrigeración puede transmitirse por consumo de una amplia variedad de alimentos (8), particularmente en alimentos listos para el consumo, tales como jamones, salchichas, etc. (9-11). Diversos estudios han reportado los alimentos RTE (del inglés, Ready To Eat) como un vehículo frecuente en la transmisión de L. monocytogenes; específicamente en Estados Unidos hay informes de brotes en varios estados relacionados con el consumo de salchichas tipo "frankfurters" y jamones, entre otros (9). En otros países como Portugal se ha encontrado que cerca del 15% de los alimentos listos para el consumo están contaminados con L. monocytogenes (6,12,13). En Colombia se ha determinado que la incidencia de este microorganismo en quesos y leche no pasteurizada distribuida en Boyacá es muy alta (29,6 y 16%, respectivamente) (14,15); en derivados cárnicos listos para el consumo también se ha encontrado una alta incidencia (24%) (Vanegas MC, Vega C, Martínez AJ, Forero P, Casas C. Aislamiento de Listeria monocytogenes a partir de cárnicos embutidos en supermercados y plazas de mercado de Bogotá. Memorias VIII Congreso Latinoamericano de Microbiología e Higiene de Alimentos. Bogotá, Mayo 18-21 de 2005, p 27).
En Colombia existen pocos datos sobre la epidemiología de la listeriosis debido a que no se diagnostica con frecuencia y, por lo tanto, presenta subregistro epidemiológico; uno de los estudios epidemiológicos más conocidos fue el realizado en 1994 en un hospital de tercer nivel (Cali, Valle), en el cual se informó sobre 19 casos clínicos de listeriosis: 10 en adultos inmunosuprimidos, dos en mujeres embarazadas, seis en neonatos y un caso en una adolescente de 12 años (16). En la actualidad es posible que los casos de listeriosis en el territorio nacional hayan aumentado, especialmente debido a la creciente población inmunosuprimida por factores como el cáncer, los trasplantes y las terapias inmunosupresoras. En general, la listeriosis se considera una enfermedad inusual en Colombia, ya que se presentan casos esporádicos de los cuales se tiene poca documentación; esto, sumado a su difícil diagnóstico, dificulta la obtención de datos epidemiológicamente representativos. Este patógeno cobra cada vez mayor importancia en salud pública a nivel mundial, y se conocen datos epidemiológicos de algunos países desarrollados; en Estados Unidos la bacteria es responsable de aproximadamente 2.500 casos y 500 muertes al año (5,17,18).
Debido a la importancia de este patógeno, se han desarrollado diversos estudios sobre los factores de virulencia y los mecanismos de patogenicidad de L. monocytogenes, y cada vez se utilizan más las técnicas moleculares para conocerlo mejor. Recientemente se han realizado análisis genéticos de L. monocytogenes para determinar marcadores moleculares que permitan entender la circulación de genotipos (19). Con la técnica del ADN polimorfo amplificado aleatorio (RAPD por su sigla en inglés) se pueden establecer patrones genéticos de bandeo en condiciones de amplificación de baja astringencia, lo cual permite caracterizar los aislamientos de acuerdo a productos específicos de amplificación según la naturaleza del genoma (20); debido a su acentuada habilidad discriminatoria (21) ya se ha empleado como método de diferenciación de L. monocytogenes (22).
El Instituto Nacional de Medicamentos y Alimentos de Colombia (Invima) realiza la serotipificación de L. monocytogenes aisladas de muestras de alimentos, pero no hay publicaciones al respecto (23). Los reportes de la literatura indican que el 95% de los aislamientos humanos corresponde a los serotipos 1/2 a, 1/2b y 4b, especialmente este último, mientras que el serotipo 1/2 se ha aislado principalmente de alimentos y del ambiente de producción; sin embargo, en el país no se conoce información al respecto (23). La serotipificación es una herramienta útil para tipificar los diferentes aislamientos de L. monocytogenes, pero es difícil conseguir los antisueros. Adicionalmente, no aporta información directa acerca de la diversidad molecular de los aislamientos circulantes de L. monocytogenes en nuestro medio, por lo que en este estudio no se utilizó. La caracterización molecular genera información epidemiológicamente útil, pues al establecer la relación entre los aislamientos de origen alimentario y clínico, se pueden desarrollar estudios de trazabilidad de casos y brotes, así como determinar si existen tipos particulares que se presenten con mayor frecuencia en alimentos específicos, y determinar cuáles de son los más comunes en pacientes con listeriosis o son más resistentes a antibióticos, desinfectantes o condiciones ambientales. Es por esto que se hace necesario realizar investiga-ciones que aporten datos sobre el tipo de cepas que están circulando en los alimentos y en muestras clínicas, e implementar técnicas que permitan discriminar entre las cepas circulantes tanto en alimentos como en muestras clínicas humanas o de animales.
El objetivo de este estudio fue caracterizar molecularmente diferentes aislamientos de L. monocytogenes provenientes de muestras clínicas y de alimentos utilizando el RAPD para determinar relaciones filogenéticas entre los aislamientos de estos dos orígenes.
Materiales y métodos
Aislamientos bacterianos
Como control se empleó la cepa L. monocytogenes ATCC 7644. Se emplearon 38 aislamientos de L. monocytogenes no relaciona-dos epidemiológica, geográfica ni temporalmente: 16 de alimentos y plantas procesadoras, corres-pondientes a cinco aislamientos de lácteos (LMO6, LMO7, LMO76, LMO77, LMO79); siete de jamones (LMO48, LMO49, LMO50, LMO52, LMO53, LMO54, LMO55); tres de ambientes de plantas procesadoras de alimentos (LMO94 - peto de operaria, LMO95 - báscula, LMO111 - pared cuarto frío); uno de pollo (LMO174), y 22 aislamientos clínicos: uno de tejido de pie infectado (LMO172), siete de hemocultivo (LMO175, LMO186, LMO187, LMO191-LMO194), uno de materia fecal (LMO195), uno de líquido pleural (LMO190) y 12 de líquido cefalorraquídeo (LMO176-LMO185, LMO188-LMO189). Los aislamientos de alimentos y plantas procesadoras se obtuvieron e identificaron en el Laboratorio de Ecología Microbiana y Alimentos (LEMA) mediante la metodología tradicional (BAM-FDA) (24) y reacción en cadena de la polimerasa (PCR) por medio de la detección del gen hly que codifica para la proteína listeriolisina O específica de L. monocytogenes. Los aislamientos de origen clínico fueron donados por el Grupo de Micro-biología del Instituto Nacional de Salud y por la Fundación Santafé de Bogotá, y se confirmaron por PCR en el LEMA. Todos los aislamientos se mantuvieron a -70°C en caldo infusión cerebro corazón (BHI, del inglés Brain Heart Infusion) con glicerol y en agar sangre a 4°C.
Extracción, cuantificación y preservación de ADN
Se realizaron cultivos de 18 horas a 37°C en caldo BHI; la extracción se hizo a partir de 1 ml del caldo utilizando el estuche para aislamiento de ADN procariótico ProDNA 2003 (Corporación Corpogen, Bogotá, Colombia) y siguiendo las instrucciones del fabricante; se resuspendió el ADN en 50 µl de la solución de resuspensión del estuche. Luego se cuantificó resuspendiendo 15 µl de la solución de ADN en 285 µl de solución de resuspensión (buffer TE, factor de dilución 20) en una celda de cuarzo, y se realizaron lecturas a 260, 280 y 320 nm para determinar la pureza y cantidad de ADN en el espectrofotómetro (Biomate 3, Thermos-pectronic, Rochester NY, USA). La pureza se estimó mediante la razón A260/A280, la cual osciló entre 1,7 y 1,8, lo que refleja un bajo contenido proteico. El ADN se ajustó a una concentración de 25 ng/µl, se distribuyó en alícuotas de trabajo de 60 µl y se conservó a -20°C hasta su uso.
PCR del gen hly
Se emplearon los oligonucleótidos complemen-tarios a un fragmento del gen de la listeriolisina O (hly) reportado como específico para L. monocytogenes, LM1: 5-CGGAGGTTCCGC AAAAGATG-3 y LM2: 5-CCTCCAGAGTGATCG ATGTT-3, que amplifican un producto de 234 pb (25,26) de acuerdo a las condiciones previamente reportadas (27). Para cada reacción se empleó L. monocytogenes ATCC 7644 como control positivo y como control negativo, agua libre de nucleasa (Promega Corporation, Madison USA).
Condiciones de RAPD
Se aplicó la técnica a los 38 aislamientos de L. monocytogenes mencionados. Se utilizaron dos oligonucleótidos de 10 pares de bases sintetizados por IDT (Integrated ADN Technologies, Inc. 1710 Commercial Park. Coralville, IA 5224. United States of America): HLWL-74 (5'-ACG TAT CTG C-3'), reportado previamente (1,28) como eficaz para caracterizar aislamientos de L. monocytogenes mediante la técnica de RAPD, y un oligonucleótido arbitrario de 10 pb (5' -GAG CTC GTG T-3') reportado por la Unidad de Servicio de Ácidos Nucléicos y Proteína de la Universidad British Columbia como estándar para las reacciones del RAPD (29). El ciclo general utilizado se estableció en el laboratorio con base en varios protocolos reportados en la literatura (2,19,20,28) y modificados utilizando un termociclador Gene CyclerTM (Bio-Rad Laboratories, Inc. USA): denaturación inicial a 94°C por 5 minutos seguida de 30 ciclos a 94°C por 30 segundos, a 37°C por 45 segundos, a 72 °C por un minuto y un ciclo final a 72°C por 5 minutos.
El volumen final de reacción fue de 25 µl que contenían 0,1 U/µl Taq polimerasa, 3 mM MgCl2, 1X buffer PCR (reactivos del estuche TucanTaq; Corporación Corpogen, Bogotá, Colombia), 0,2 mM de cada dNTP (dNTP Kit, Promega Corporation, Madison USA), 1,5 µM de oligonucleótido y 50 ng de ADN. Cada reacción se realizó dos veces para cada aislamiento y cada oligonucleótido. Los productos de amplificación se separaron por electroforesis tradicional en geles de agarosa al 1% en buffer TBE 1X, seguida por una tinción en piscina de bromuro de etidio (0,5 mg/ml) durante 45 minutos. En todas las reacciones se incluyó un control negativo para cada oligonuecleótido (agua libre de nucleasa en vez de ADN) y se utilizó el ADN de fago l digerido con EcoRI+HindIII (Promega Corporation) como marcador de peso molecular. Los productos se visualizaron con el transiluminador ChemiDoc XRS system (Bio-Rad Laboratories). Todos los aislamientos se analiza-ron dos veces en reacciones independientes por cada oligonucleótido para asegurar la reprodu-cibilidad en los patrones de bandeo; en ambas reacciones se obtuvieron patrones idénticos con pequeñas variaciones en la resolución de algunas bandas de muy baja intensidad que no se tuvieron en cuenta para la construcción de las matrices, lo cual asegura una mayor precisión en los resultados obtenidos.
Se analizaron los datos de manera independiente para cada oligonucleótido mediante el programa Quantity One de Bio-rad y SYN-TAX (2000). Se construyó una matriz binaria (1 = banda presente, 0 = banda ausente) para construir un dendrograma empleando el programa SYN-TAX (2000) con la herramienta de agrupación jerárquica y el coeficiente de correlación de Jaccard independientemente para cada oligonucleótido. Los porcentajes de similitud se obtuvieron de acuerdo a los resultados del dendrograma, el cual refleja el valor de disimilitud entre los aislamientos. Se consideró que había linaje cuando la disimilitud fue superior a 0,75 (75%). Esta técnica fue estandarizada previamente en el laboratorio para asegurar resultados reproducibles.
Resultados
Extracción de ADN
El índice de pureza de los ADN utilizados en el estudio osciló entre 1,7 y 1,8. Cuando se obtuvo un índice inferior, se extrajo nuevamente para obtener una óptima calidad en los resultados (datos no mostrados).
Confirmación de aislamientos por PCR
Todos los aislamientos presentaron una banda de 234 pb correspondiente a un fragmento del gen hly de L. monocytogenes. En todos los casos se obtuvo amplificación del control positivo y no se detectó contaminación en el control negativo.
Caracterización molecular de L. monocytogenes
Se obtuvieron bandas claras y reproducibles en los 38 aislamientos analizados por medio de los dos oligonucleótidos empleados. La reprodu-cibilidad de las reacciones de RAPD se evaluó con dos ensayos independientes para cada aislamiento y cada oligonucleótido, y solamente se tuvieron en cuenta aquellas bandas que se presentaron en las dos reacciones independientes con intensidad clara. No se detectaron productos de amplificación en los controles negativos, lo que confirma la ausencia de contaminaciones.
Con el oligonucleótido HLWL-74 se obtuvieron 11 bandas claras y reproducibles, de las cuales nueve (81,81%) eran altamente polimórficas entre los aislamientos analizados. Con este oligonucleótido se observaron dos linajes superiores (I y II) y los 38 aislamientos formaron un total de 20 grupos menores. El linaje I agrupó 35 aislamientos y se subdividió en dos grupos: A, el cual a su vez se dividió en dos grupos menores (1 y 2), y B, el cual también se dividió en dos grupos menores (3 y 4) (
figura 1). Todos los aislamientos clínicos, exceptuando el LMO 172, se agruparon bajo este linaje. Los aislamientos de origen alimentario se encontraron en su mayoría en el subgrupo 1, exceptuando las cepas LMO95, LMO174, LMO6 y LMO7; el subgrupo 2 quedó formado exclusivamente por cepas de origen clínico, agrupando 14 de los 22 (63,64%) aislamientos clínicos analizados, y el subgrupo 3 quedó conformado por un aislamiento de origen ambiental (LMO95) y uno de pollo (LMO174).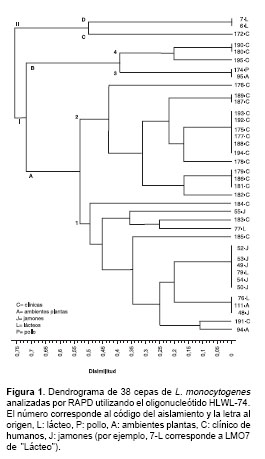
El linaje II agrupó tres aislamientos y se subdividió en dos grupos, C (LMO6 y LMO7, aislados de lácteos), y D (aislamiento LMO 172 de tejido de pie infectado), los cuales presentaron un porcentaje de disimilitud del 75% con respecto a los otros aislamientos.
Con el oligonucleótido arbitrario se obtuvieron 14 bandas, de las cuales 12 (85,71%) fueron altamente polimórficas. Con este oligonucleótido se observaron dos linajes superiores (I y II) y los 38 aislamientos formaron un total de 19 grupos menores. El linaje I agrupó 32 aislamientos y se subdividió en dos grupos: A, el cual a su vez se dividió en dos grupos menores (1 y 2), y B, el cual también se dividió en dos grupos menores (3 y 4) (
figura 2). La mayoría de los aislamientos clínicos se agruparon bajo este linaje, pero con un menor porcentaje (77,27%) al observado con el oligonucleótido HLWL-74, encontrándose 15 de los 22 bajo el subgrupo 1 (68,18%), así como la mayoría de los aislamientos de origen alimentario. El subgrupo 2 está formado exclusivamente por aislamientos de lácteos (LMO6 y LMO7); el aislamiento LMO174 es el único miembro del subgrupo 4 y se encontró más cercanamente relacionado con los clínicos.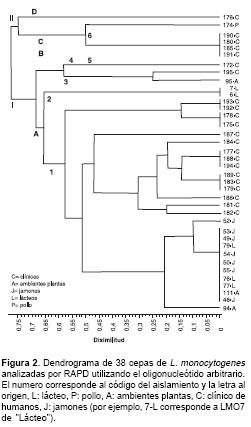
El linaje II agrupó seis aislamientos y se subdividió en dos grupos denominados C y D. El grupo C se dividió a su vez en los subgrupos 5 y 6, de los cuales el subgrupo 5 incluyó solamente aislamientos clínicos (LMO191, LMO180, LMO185 y LMO190). El subgrupo 6 incluyó un aislamiento obtenido de pollo (LMO174) y en el grupo D sólo quedó incluido un aislamiento (LMO176) de origen clínico. Los seis aislamientos presentaron un 75% de disimilitud con respecto a los otros analizados.
Discusión
En los último años se ha intensificado el estudio de L. monocytogenes como patógeno transmitido por alimentos, y aunque da origen a una enfermedad poco documentada en nuestro país, es una amenaza para la salud pública debido a su alta tasa de mortalidad (30). Varios factores influyen en la obtención de datos específicos sobre la listeriosis en Colombia: no es una enfermedad de notificación obligatoria; es una enfermedad con un largo periodo de incubación, por lo que establecer una relación directa entre el alimento contaminado y el desarrollo de la enfermedad es muy difícil, y las manifestaciones clínicas de la listeriosis aguda pueden confundirse con otras afecciones.
La listeriosis es una enfermedad muy importante que se presenta de forma aguda durante el primer trimestre del embarazo y puede causar aborto ocasional o recurrente en pacientes portadoras (31). La alta mortalidad de la listerosis, sumada a la creciente población inmunosuprimida y al incremento a nivel mundial de infecciones alimentarias, hace necesario estudiar este tema en nuestro país, conocer las cepas de L. monocytogenes que circulan en nuestro medio, así como implementar técnicas que permitan identificarlas y realizar su seguimiento epidemiológico. Por lo anterior, se analizaron 38 aislamientos de L. monocytogenes provenientes de diferentes orígenes para determinar las posibles relaciones entre los diferentes aislamientos mediante la técnica de RAPD y con dos oligonucleótidos diferentes.
La técnica de RAPD se utiliza universalmente en estudios epidemiológicos de L. monocytogenes (31,32) además de otras técnicas como la electroforésis de campo pulsado (PFGE por su sigla en inglés) y los polimorfismos en la longitud de fragmentos de restricción (RFLP por su sigla en inglés), entre otras (33). Algunos autores reportan que la técnica de RAPD es de baja reproducibilidad, y que por ende no debería utilizarse en estudios de caracterización molecular (34,35); sin embargo, al igual que lo reportado por otros autores, en este estudio se obtuvo una buena reproducibilidad (1,2,19-21,36). Se encontró que la calidad y la cantidad de ADN es fundamental para obtener patrones de bandeo reproducibles, ya que la presencia de altas cantidades de proteína o cantidades inferiores a 25ng de ADN en las reacciones puede alterar drásticamente la resolución de las bandas obtenidas. Por esta razón, es recomendable estandarizar la técnica con una cantidad y calidad específica de ADN para asegurar resultados más acertados (34).
Se observó una gran diversidad de patrones de bandeo en los aislamientos analizados, lo cual puede reflejar variaciones a nivel genético y, por consiguiente, representar cambios a nivel fenotípico, por ejemplo, la morfología de colonia, la producción de biopelículas y el tipo de hemólisis. Debido a la alta diversidad de los aislamientos analizados no es posible determinar una relación directa entre las cepas circulantes en alimentos y las cepas clínicas, a diferencia de lo logrado en otros estudios (1,2,18).
Los resultados del agrupamiento de los aislamien-tos clínicos obtenido en los dendrogramas sugieren que éstos son genéticamente distintos a los de origen alimentario, y por esta razón se agrupan más cercanamente entre sí.
Este trabajo muestra que existe diversidad molecular entre los aislamientos de L. monocytgenes presentes en plantas procesadoras de alimentos y en los alimentos, así como una relación más lejana de las cepas clínicas. Estas diferencias a nivel molecular pueden reflejar diferencias adaptativas entre las distintas cepas, y explicar la resistencia a condiciones adversas y a diferentes antibióticos, así como características propias de cada cepa, tales como la velocidad de crecimiento, la degradación de azúcares y la capacidad de colonización e invasión en el hospedero (2).
Se recomienda el uso de esta técnica para futuros estudios de caracterización molecular de aislamientos de L. monocytogenes circulantes tanto en alimentos como en infecciones humanas para generar datos epidemiológicos que puedan utilizarse en la profilaxis, tratamiento y control de la listeriosis (2,19).
La importación de nuevos productos alimenticios a Colombia podría convertirse en una fuente importante de variantes no nativas de L. monocytogenes, lo cual favorecería el flujo genético entre cepas, originando nuevas capacidades de virulencia y supervivencia (6,21,22).
Así mismo, la técnica de RAPD puede utilizarse en estudios de seguimiento y trazabilidad desde la materia prima hasta el producto terminado en la industria de alimentos, como herramienta de aseguramiento de la calidad y de protección indirecta de la salud pública, así como en medicina veterinaria para detectar una gran variedad de enfermedades causadas por este micro-organismo en animales importantes para el sector productivo del país (37).
Agradecimientos
Los autores agradecen al Grupo de Microbiología del Instituto Nacional de Salud de Colombia por la amable donación de cepas de origen clínico, sin las que no hubiera sido posible la realización de este estudio. Así mismo, agradecen a la profesora Gloria Uribe de la Universidad de los Andes y al Grupo de Microbiología de la Fundación Santa Fe de Bogotá por su apoyo en la consecución y donación de cepas clínicas.
Conflicto de intereses
Declaramos que la investigación a partir de la cual se originó el presente manuscrito no presenta conflicto de intereses.
Financiación
Este trabajo fue financiado por la Facultad de Ciencias y por el Laboratorio de Ecología Microbiana y Alimentos (LEMA) de la Universidad de los Andes.
Correspondencia:
Maria Consuelo Vanegas, Laboratorio de Ecología Microbiana y Alimentos-LEMA, Universidad de los Andes, Cra 1a No 18A-70 J209, Bogotá, Colombia.
Teléfono: 3394949 ext. 2784, fax extensión: 3339.
Recibido: 10/04/06; aceptado: 14/08/06
Referencias
1. Lawrence LM, Gilmour A. Characterization of Listeria monocytogenes isolated from poultry products and from the poultry-processing environment by random amplification of polymorphic DNA and multilocus enzyme electrophoresis. Appl Environ Microbiol 1995; 61: 2139-44. [ Links ]
2. Inoue S, Katagiri K, Terao M, Maruyama T. RAPD and actA gene-typing of Listeria monocytogenes isolates from human listeriosis, the intestinal content of cows and beef. Microbiol Immunol 2001; 45: 127-33 [ Links ]
3. US Food and Drug Administration. Bad Bug Book. Foodborne pathogenic microorganisms and natural toxins handbook. Listeria monocytogenes. Consultado: 21 de noviembre de 2005 Disponible en: http://www.cfsan.fda.gov/~mow/chap6.html [ Links ]
4. Jesam G, Cádiz F, Céspedes P, Ramírez C. Infección perinatal por Listeria monocytogenes: presentación de casos clínicos, ¿transmisión en la sala de atención inmediata? Boletín Hospital San Juan de Dios 2005; 52: 116-23. [ Links ]
5. Asociación de Enfermeras de Salud de la Mujer, de Obstetricia y del Recién Nacido (AWHONN, siglas en inglés); Fundación Internacional del Concejo de Información Alimentaría (IFIC, siglas en inglés); Departamento de Agricultura de los Estados Unidos (USDA, siglas en inglés); Departamento de Salud y Servicios Humanos de los Estados Unidos (DHHS, siglas en inglés). La listeriosis y el embarazo:¿Cuál es su riesgo? La manipulación adecuada de los alimentos le permitirá tener un embarazo sano. Boletín Informativo. Washington: Asociación de Enfermeras de Salud de la Mujer, de Obstetricia y del Recién Nacido (AWHONN); 2002. Consultado: 21 de noviembre de 2005. Disponible en: http://adams.unl.edu/foodsafety/nfsem_htm/resources/downloads/Listeriosis_sp.pdf [ Links ]
6. Escartin FE. Microbiología e inocuidad de los alimentos. Queretaro, México: Universidad Autónoma de Queretaro; 2000. [ Links ]
7. DAgostino MD, Wagner M, Vazquez-Boland JA, Kuchta T, Karpiskova R, Hoorfar J, et al. A validated PCR-based method to detect Listeria monocytogenes using raw milk as a food model- towards an international standard. J Food Prot 2004; 67: 1646-55. [ Links ]
8. Guerra MM, Fernando de Almeida B. Fontes de contaminacäo dos alimentos por Listeria monocytogenes. Hig Aliment 2004; 18: 12-8. [ Links ]
9. Murphy RY, Berrang ME. Thermal lethality of Salmonella Senftenberg and Listeria innocua on fully cooked and vacuum packaged chicken breast strips during hot water pasteurization. J Food Prot 2002; 65: 1561-4. [ Links ]
10. Norton DM, McCamey MA, Gall KL, Scarlett JM, Boor KJ, Wiedmann M. Molecular studies on the ecology of Listeria monocytogenes in the smoked fish processing industry. Appl Environ Microbiol 2001; 67: 198-205. [ Links ]
11. Barmpalia IM, Geornaras I, Belk KE, Scanga JA, Kendall PA, Smith GC, et al. Control of Listeria monocytogenes on frankfurters with antimicrobials in the formulation and by dipping in organic acid solutions. J Food Prot 2004; 67: 2456-64. [ Links ]
12. Al-Holy M, Lin M, Rasco B. Destruction of Listeria monocytogenes in sturgeon (Acipenser transmontanus) caviar by a combination of nisin with chemical antimicrobials or moderate heat. J Food Prot 2005; 68: 512-20. [ Links ]
13. Wallace MF, Call JE, Porto CS, Cocoma GJ, ERRC Special Projects Team, Luchansky B. Recovery rate of Listeria monocytogenes from commercially prepared frankfurters during extended refrigeration storage. J Food Prot 2003; 66: 584-91. [ Links ]
14. Vergara J. Prevalencia de Listeria monocytogenes en quesos frescos distribuidos en Bogotá D.C. (Monografía Microbiología de Alimentos e Industria). Bogotá: Universidad de los Andes; 2004. [ Links ]
15. Rueda A. Utilización de la reacción en cadena de la polimerasa (PCR) en tiempo real para determinar la incidencia de Listeria monocytogenes en leches crudas en el departamento de Boyacá. (Tesis de Maestría en Microbiología). Bogotá: Universidad de los Andes, 2005. [ Links ]
16. Crespo M, Velez J, Castañeda C, Hoyos F, López M, Salazar J. Aislamiento de Listeria monocytogenes en un hospital de tercer nivel. Colombia Médica 1999; 30: 89-90 [ Links ]
17. Murphy RY, Hanson RE, Feze N, Johnson NR, Scott LL, Duncan LK. Eradicating Listeria monocytogenes from fully cooked franks by using and integrated pasteurization-packaging system. J Food Prot 2005; 68: 507-11. [ Links ]
18. Van Coillie E, Werbrouck H, Heyndrickx M, Herman L, Rijpens N. Prevalence and typing of Listeria monocytogenes in ready-to eat food products on the Belgian market. J Food Prot 2004; 67: 2480-7. [ Links ]
19. Yoshida T, Takeuchi M, Sato M, Hirai K. Typing Listeria monocytogenes by random amplified polymorphic DNA (RAPD) fingerprinting. J Vet Med Sci 1999; 61: 857-60. [ Links ]
20. McPherson MJ, Moller SG. PCR The Basics. Oxford, U.K: BIOS Scientific Publishers Limited: 2000. p. 257-528. [ Links ]
21.; Dhanashree B, Otta SK, Karunasagar I, Karunasagar I. Typing of Listeria monocytogenes isolates by random amplification of polymorphic DNA. Indian J Med Res 2003; 117: 19-24. [ Links ]
22. Farber JM, Addison CJ. RAPD typing for distinguishing species and strains in the genus Listeria. J Appl Bacteriol 1994; 77: 242-50. [ Links ]
23. Muñoz AI, Díaz G. Listeriosis. (Monografía). Bogotá: Instituto Nacional de Vigilancia de Medicamentos y Alimentos INVIMA; 1998. [ Links ]
24. US Food and Drug Administration. FDA Bacteriological analytical manual. 1998. Consultado: 25 de febrero de 2006;. Disponible en: http://www.foodinfonet.com/publication/fdaBAM.htm [ Links ]
25. Lampel KA, Orlandi PA, Kornegay L. Improved template preparation for PCR-based assays for detection of food-borne bacterial pathogens. Appl Environ Microbiol 2000; 66: 4539-42. [ Links ]
26. Rodríguez-Lázaro D, Hernández M, Pla M. Simultaneous quantitative detection of Listeria spp. and Listeria monocytogenes using a duplex real time PCR-based assay. FEMS Microbiol Lett 2004; 233: 257-67. [ Links ]
27. Vanegas MC, Rojas J, Vergara J. Detección de Listeria monocytogenes de diferentes orígenes. Mundo Microbiológico 2003; 2: 17-20. [ Links ]
28. Lawrence LM, Harvey J, Gilmour A. Development of a random polymorphic DNA typing method for Listeria monocytogenes. Appl Environ Microbiol 1993; 59: 3117-9. [ Links ]
29. The University of British Columbia. Primer sets. Consultado: 22 de junio de 2005Disponible en: http://www.michaelsmith.ubc.ca/services/NAPS/Primer_Sets/ [ Links ]
30. Leite P, Rodrigues R, Ferreira M, Ribeiro G, Jacquet C, Martin P, et al. Comparative characterization of Listeria monocytogenes isolated from Portuguese farmhouse ewes cheese and from humans. Int J Food Microbiol 2006; 106: 111-21. [ Links ]
31. Sánchez E, Castillo A, Ortega C. Infección perinatal por Listeria. reporte de un caso. Rev Colomb Obste Ginecol 2001; 52: 199-202. [ Links ]
32. Fleming DW, Cochi SL, MacDonald KL, Brondum J, Hayes PS, Plikaytis BD, et al. Pasteurized milk as a vehicle of infection in an outbreak of listeriosis. N Engl J Med 1985; 312: 404-7. [ Links ]
33. Zhou X, Jiao X, Wiedmann M. Listeria monocytogenes in the chinese food system: strain characterization through partial actA sequencing and tissue-culture pathogenicity assays. J Med Microbiol 2005; 54: 217-24. [ Links ]
34. Penner GA, Bush A, Wise R, Kim W, Domier L, Kasha K, et al. Reproducibility of random amplified polymorphic DNA (RAPD) analysis among laboratories. PCR Methods Appl 1993; 2: 341-5. [ Links ]
. 35 Ellsworth DL, Rittenhouse KD, Honeycutt RL. Artifactual variation in randomly amplified polymorphic DNA banding patterns. Biotechniques 1993; 14: 214-7. [ Links ]
36. Soares-Ramos J, Ramos H, Cruz L, Chubatsu L, Pedrosa F, Rigo L, et al. Comparative molecular analysis of Herbaspirillum strains by RAPD, RFLP, and 16S rDNA sequencing. Genet Mol Biol 2003; 26: 537-43. [ Links ]
37. Schweizer G, Ehrensperger F, Torgerson PR, Braun U. Clinical findings and treatment of 94 cattle presumptively diagnosed with listeriosis. Vet Rec 2006; 158: 588-92. [ Links ]














