Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.26 no.4 Bogotá Dec. 2006
Premios nobel en Fisiología o Medicina y Química, año 2006.
Una nueva dimensión del ARN en la regulación de la expresión genética y como herramienta experimental y terapéutica
Nobel prizes in Physiology of Medicine and Chemistry, year 2006.
A new RNA dimension in regulation of gene expression and as experimental and therapeutic tool
Luis Alberto Gómez
Grupo de Fisiología Molecular, Instituto Nacional de Salud
El 2 de octubre de 2006 se anunció el nombre de los investigadores laureados con el premio nobel en Fisiología o Medicina en este año. Los galardonados fueron los investigadores estado-unidenses Andrew Z. Fire, profesor de la Escuela de Medicina de la Universidad de Stanford (
fotografía 1 y derecha, fotografía 2) y Craig C. Mello (fotografía 3), profesor de la Escuela de Medicina de la Universidad de Massachussets. Se les otorgó el premio por sus investigaciones en la interferencia de la expresión de genes mediada por moléculas de ARN de doble cadena, un proceso fisiológico celular endógeno de regulación genética que está altamente conservado en los organismos multicelulares. Este proceso es llamado interferencia de ARN (ARNi) y, también, se conoce en plantas como el silencio genético después de la transcripción o abatimiento de la expresión de un gen después de que ha sido transcrito "post-transcriptional gene silencing"


El 4 de octubre también se anunció el nombre del investigador laureado con el premio nobel en Química, Roger D. Kornberg
(fotografía 4 e izquierda, (fotografía 2), profesor de la Universidad de Stanford, por sus estudios sobre las bases moleculares de la transcripción del ADN en células eucarióticas, un tema que se relaciona con los trabajos de Fire y Mello como lo mencionaré a continuación.
En el dogma central de la biología, descrito desde hace varias décadas, la información contenida en el ADN se transfiere al mARN y de éste a una proteína; sin embargo, en esta descripción no se explica cómo ocurre ni cómo se controla. R. Kornberg reveló a escala atómica el primer paso en este proceso, cómo el ADN se convierte en mARN en las células, un proceso conocido como transcripción que, en otras palabras, consiste en la síntesis de ARN por la enzima ARN polimerasa II a partir de la información contenida en el ADN.
La contribución de los trabajos de R. Konberg fue la descripción estructural de la maquinaria molecular implicada en el proceso de trascripción, lo cual es uno de los avances más importantes en la bioquímica contemporánea. El grupo de Konberg purificó un conjunto de proteínas de levaduras conocido como complejo de transcripción, constituido por varias proteínas. Este complejo incluía la enzima ARN polimerasa II (Pol II) conformada por doce subunidades proteicas acopladas y varias proteínas asociadas, llamadas factores de transcripción generales, que transcriben la información genética contenida en el ADN (1-2). La construcción de las primeras imágenes de la Pol II en acción derivada de los estudios de cristalografía de rayos X dirigidos por R. Kornberg, complementa el conocimiento de la fisiología molecular del ADN (3) y expande el conocimiento de cómo son sintetizados los ácido ribonucleicos, ARN y ADN, y como éste es transferido de la célula madre a las células hijas, en lo que contribuyó Arthur Konberg
(fotografía 2, centro), el padre de Roger Konberg, quien compartió con Severo Ochoa el premio nobel en Fisiología o Medicina de 1959.La maquinaria de transcripción revelada por el grupo de R. Kornberg, junto con la participación de otro complejo de proteínas conocidas como proteínas mediadoras o coactivadoras, determinan la activación de la expresión de genes específicos (2-4). La activación génica en eucariotes superiores requiere la acción concertada de factores de transcripción y proteínas coactivadoras, que existen como complejos multiproteicos, interaccionan y modifican la cromatina y permiten la acción de la ARN Pol II para que la trascripción ocurra (4-5). La síntesis del ARN de los genes específicos y sus productos son los que finalmente determinan procesos biológicos complejos como el desarrollo, la diferenciación y el metabolismo celular, entre otros (4). El control de la expresión genética se focaliza, en gran parte, en el control de la transcripción que, a su vez, está regulada por los niveles, la interacción y la cinética de la actividad de los factores y coactivadores de transcripción que, en conjunto, inician, aceleran y determinan la expresión de genes específicos (6).
Por otra parte, los trabajos originales de Mello y Fire en interferencia genética inducida por el ARN en el nemátodo Caenorhabditis elegans abrieron el camino a nuevas investigaciones orientadas a identificar y dilucidar al ARNi como un mecanismo conservado que regula la expresión genética durante el desarrollo y la fisiología normal de los organismos, así como algunas enfermedades.
Desde los primeros años de la década de 1980 se empezó a utilizar la introducción experimental de ARN exógeno antisentido (un ARN de cadena sencilla con secuencia complementaria a un ARN mensajero endógeno, llamado ARN sentido) para inhibir la expresión de un gen específico. En el mecanismo de inhibición, se planteó la hipótesis que el ARN antisentido se unía al ARN sentido por el proceso de hibridación o asociación de dos moléculas por el grado de complementariedad, y esta hibridación interrumpía la expresión del ARN mensajero endógeno (7-9).
En estos experimentos el ARN sentido se usa frecuentemente como control y en 1995 Guo y Kemphues reportaron que la introducción del ARN sentido (control) inesperadamente provocaba una interferencia genética específica de secuencia en C. elegans (10). Este sorpresivo hallazgo condujo al grupo de investigación dirigido por Mello a investigar el mecanismo molecular que subyacía a este proceso de interferencia genética inducida por ARN (ARNi) en C. elegans. Los resultados de sus primeras investigaciones en este campo condujeron a proponer un modelo en el cual el ARN sentido o antisentido exógenos serían convertidos a una molécula de doble cadena por una polimerasa hipotética de C. elegans, y que esta molécula de doble cadena desencadenaba un proceso fisiológico inhibitorio con alto grado de fidelidad y que, además, era heredable. La mayor parte de estos resultados se presentaron y discutieron en dos reuniones científicas sobre nemátodos: 1) Driver S, Ali M, Mello C. Formation of heritable anti-transgenes after RNA microinjection in C. elegans. East Coast Meeting, abstract 33, Rutgers University, New Jersey, 1996, y 2) Driver S, Ali M, Mello C. Antisense in C. elegans: Heritable gene silencing induced by RNA microinjection. International Worm Meeting, abstract 136, Wisconsin, 1997.
No obstante, varias observaciones previas condujeron al concepto de la interferencia genética del ARN por ARN de doble cadena. El silencio genético posterior a la transcripción y los cambios epigenéticos inducidos por el ARN exógeno ya habían sido descritos previamente en las plantas. Por ejemplo, antes de los trabajos de Mello y Fire se había descrito el fenómeno de interferencia del ARN en petunias, se había descrito por degradación de un ARN específico mediada por otro ARN de secuencia complementaria, así como la supresión de la expresión de genes por secuencias homólogas asociado con la metilación en plantas de tabaco (11-15). Estas observaciones y las evidencias de que el proceso de ARNi podía ser heredable, se constituyeron en factores de confusión y contribuyeron a sesgar inicialmente la hipótesis de que el agente que interfería podía ser una molécula de ADN y no un ARN de doble cadena. Sin embargo, lo que condujo a identificar definitivamente que la molécula capaz de interferir en la expresión era una molécula de ARN de doble cadena (dsRNA, del inglés double strand) fue el trabajo de Fire, Mello et al. en 1998, quienes integraron las evidencias existentes y demostraron que la molécula que estaba mediando la interferencia era un ARN de doble cadena de 21 a 25 nucleótidos, (16).
Fire y Mello observaron que para diferentes concentraciones de ARN, tanto la cadena sentido como la antisentido inducían efectos inhibitorios semejantes, produciendo como resultado la desaparición de la proteína endógena. Este efecto era más potente cuando se introducía una molécula de ARN de doble cadena, que las cadenas sencillas purificadas y que el silencio o la interferencia se lograba si las secuencias de ARN eran dirigidas a exones (secuencias de nucleótidos que se traducen), pero que si el ARN se dirigía contra secuencias del promotor o de los intrones (secuencias de nucleótidos entre exones que no se traducen), éstas no eran efectivas (16,17). Estos datos sugirieron que la molécula que ejercía la interferencia era un intermediario estable de doble cadena y que el mecanismo de ARNi era a nivel pos-transcripción, respectivamente.
Mello y Fire también confirmaron que la molécula blanco del ARNi era el ARN mensajero y en un esfuerzo por identificar el número más pequeño de bases requeridas para el ARNi en C. elegans, el grupo de investigadores llevó a cabo un experimento detallado en el que variaban la longitud del ARNi, usando 24, 50 y 100 nucleótidos y lograron demostrar que el tamaño del ARN capaz de inducir la interferencia era de 25 a 50 nucleótidos (16-20). Además, demostraron que dos genes relacionados, con 80% de homología entre ellos, presentaban una pérdida de la función diferente, que no se superponía, lo cual demostraba que el requisito de la homología de secuencias entre el ARN blanco y el ARN pequeño que producía interferencia era indispensable para promover interferencia específica. Finalmente, fue necesario descartar que el ARNi provocaba una metilación de novo en C. elegans (18), como se había reportado en plantas (13).
Andrew Fire, Craig Mello et al. describieron que sólo unas pocas moléculas de ARN de doble cadena (dsARN) se requerían para inducir el abatimiento del mARN endógeno y sugirieron un mecanismo catalítico en el proceso de reconocimiento y degradación del mARN (16). Los resultados obtenidos en conjunto mostraban que la introducción de un dsARN exógeno causaba la pérdida de la expresión del mARN endógeno de una manera específica de la secuencia y que este proceso genético inducido por ARN pequeños que producían interferencia (siARN, de las siglas en inglés, small interfering RNA) estaba conservado en vertebrados e invertebrados (16-20).
Las evidencias bioquímicas y genéticas obtenidas con posterioridad al trabajo seminal de los laureados, están permitiendo entender el mecanismo del abatimiento de genes por ARNi. Hoy sabemos que el ARNi es un proceso de descenso de la expresión genética posterior a la transcripción que opera induciendo la degradación del ARN de manera específica, cuyo mecanismo bioquímico es similar en la mayoría de organismos estudiados (21). Como se esquematiza en el modelo simplificado en la
figura 1, el ARNi es inducido por varios tipos de moléculas de ARN que, por el número de nucleótidos y su función, se han llamado ARN pequeños que causan interferencia, siARN. Estos siARN se pueden generar, por lo menos, de tres tipos de ARN que no codifican (ncARN, del inglés non coding RNA). Se denominan ARN que no codifican porque la información contenida en su secuencia no se traduce en proteínas. Los ncARN con capacidad de generar el proceso de ARNi pueden ser (figura 1): 1) ARN de doble cadena (dsARN) de origen exógeno y que se pueden sintetizar químicamente; 2) ARN cortos que forman una estructura secundaria (plegamiento) en forma parecida a un tallo y una asa (shARN, de las siglas en inglés short hairpin RNA) de origen exógeno que, también, pueden ser introducidos por vectores virales y plasmídicos; 3) microARN con plegamiento en forma parecida a un tallo y una asa, de origen endógeno (miARN, del inglés micro hairpin RNA) que son moléculas de ARN de 21 a 25 nucleótidos, inicialmente descubiertos en el seguimiento de los genes requeridos para el desarrollo del estadio de larva de C. elegans (22). Estos miARN son codificados por genes específicos y transcritos por la ARN Pol II. Los siARN son producto del procesamiento de dsARN por la actividad ribonucleasa de la enzima Dicer, conservada evolutivamente (21-25). Una vez se generan los siARN, se hibridan con el mARN específico y son incorporados en el complejo proteico conocido como RISC (RNA-induced silencing complex), que funciona como una ARN-helicasa dependiente de ATP y como una endorribonucleasa que degrada al mARN (25,26).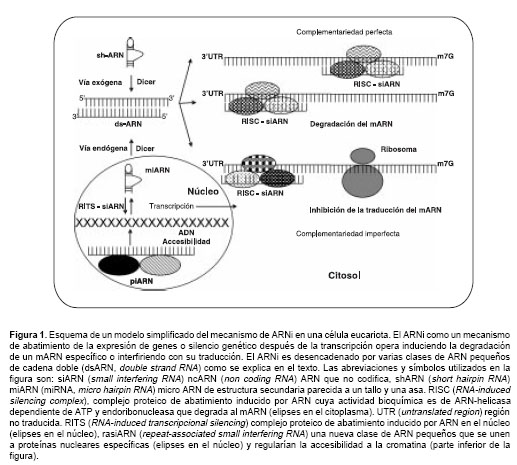
Figura 1. Esquema de un modelo simplificado del mecanismo de ARNi en una célula eucariota. El ARNi como un mecanismo de abatimiento de la expresión de genes o silencio genético después de la transcripción opera induciendo la degradación de un mARN específico o interfiriendo con su traducción. El ARNi es desencadenado por varias clases de ARN pequeños de cadena doble (dsARN, double strand RNA) como se explica en el texto. Las abreviaciones y símbolos utilizados en la figura son: siARN ( small interfering RNA) ncARN ( non coding RNA) ARN que no codifica, shARN ( short hairpin RNA) miARN (miRNA, micro hairpin RNA) micro ARN de estructura secundaria parecida a un tallo y una asa. RISC ( RNA-induced silencing complex), complejo proteico de abatimiento inducido por ARN cuya actividad bioquímica es de ARN-helicasa dependiente de ATP y endoribonucleasa que degrada al mARN (elipses en el citoplasma). UTR ( untranslated region) región no traducida. RITS ( RNA-induced transcripcional silencing) complejo proteico de abatimiento inducido por ARN en el núcleo (elipses en el núcleo), rasiARN (repeat- associated small interfering RNA) una nueva clase de ARN pequeños que se unen a proteínas nucleares específicas (elipses en el núcleo) y regularían la accesibilidad a la cromatina (parte inferior de la figura).
La interferencia del mARN desencadenada por diferentes tipos de ARN pequeños ocurre a través de vías bioquímicas comunes. La escisión del mARN blanco sólo ocurre en las regiones homólogas a los siARN, con perfecta complemen-tariedad en la secuencia que codifica o en la región que no codifica (UTR, untranslated region) del extremo 3 del mARN. En Drosophila se ha observado que, si la complementariedad no es perfecta, esta hibridación parcial también puede interferir en la síntesis de proteínas induciendo la inhibición de la traducción del mARN (síntesis de la proteína) (25). Además, en algunas plantas existe la polimerasa de ARN dependiente de ARN (RdRP, RNA dependent RNA polymerase), que está involucrada en la propagación de la señal de ARNi, por lo que puede generar una amplificación de la interferencia por la síntesis adicional de dsARN a través de esta enzima RdRP (24,27).
La conservación evolutiva del proceso biológico de ARNi se refleja en la importancia de nuevas clases de ARN pequeños que no codifican (ncARN) que también participan en el mecanismo de ARNi. Esta nueva clase de ncARN son los llamados piARN que controlan la expresión genética en células germinales; son diferentes a los miARN, en su origen, tamaño y función. Los piARN tienen una longitud de 25 a 31 nucleótidos en Drosophila y sus homólogos en mamíferos, se llaman rasiARN (repeat-associated small interfering RNA) que aunque son ARN pequeños que causan interferencia y están asociados a repeticiones, se originan de la hibridación de dos transcritos complementarios en el núcleo, se asocian a proteínas diferentes y participan del mecanismo de silencio genético acoplado a la trascripción, como se describe anteriormente y en la
figura 1 (28). Aunque ha habido un progreso significativo en el conocimiento de los mecanismos moleculares que regulan la expresión genética por ARNi, todavía quedan muchas preguntas sin respuesta; por ejemplo, ¿todos los ARN pequeños, tales como siARN, micro-ARN y piARN/rasiARN, son funcionales?, ¿cuáles serían sus blancos? ¿Estas moléculas funcionan en más de un mecanismo en la red de regulación de la expresión de la información contenida en el genoma? (29).Los temas de los trabajos científicos de los galardonados en Química y en Fisiología o Medicina se entrelazan en la compleja regulación de la expresión génica. En primer lugar, los microARN son sintetizados por la ARNpol II; en segundo lugar, se propone que los siARN pueden ser generados durante la transcripción de genes que codifican proteínas, una posibilidad sugerida recientemente (30), y en tercer lugar, hay evidencias de la regulación del genoma por ARN pequeños, lo que implica que la interferencia de ARN podría estar acoplada a la trascripción. Uno de los mecanismos sería a través de siARN generados en el núcleo, a partir de RdRP, que en conjunto con el complejo llamado RITS (RNA-induced transcripcional silencing) degradarían ARN primarios sintetizados por el complejo de iniciación de la trascripción (31). Otro mecanismo de acople entre transcripción y ARNi sería a través de la asociación de rasiARN, la otra clase de ARN pequeños que se describió recientemente, con proteínas nucleares específicas con las que forman un complejo rasiARN-proteína que podría regular la accesibilidad a la cromatina a nivel del ADN o de las histonas, en un nivel posterior a la transcripción (30,31) (
figura 1).El descubrimiento de las bases moleculares de la transcripción en células eucarióticas y del ARNi ha tenido un gran impacto sobre todos los aspectos de la regulación de la expresión genética y la fisiología celular. En especial, ha modificado las nociones que teníamos sobre cómo se controla la expresión de los genes y sus implicaciones en procesos complejos como la viabilidad, la división, la diferenciación, el desarrollo, la cinética y el equilibrio celular. El uso de este conocimiento se ha convertido en muy pocos años no sólo en una verdadera revolución en investigación biomédica al facilitar el estudio de la expresión y función de genes específicos en contextos celulares normales y patológicos, sino que, también, promete obtener nuevo conocimiento en los mecanismos moleculares de la regulación del genoma y augura un gran potencial terapéutico para el manejo de enfermedades comunes (32-34). Por lo tanto, el aprender dónde, cuándo, cómo y por qué se "prenden" o activan algunos genes y alternativamente se "apagan" o inactivan otros genes, lo que conduce generalmente al aumento o disminución de la expresión de genes, respectivamente, son algunas de las tareas centrales derivadas del trabajo de Konberg, Mello y Fire.
Los trabajos de estos investigadores han abierto campos de investigación en bioquímica y biología molecular en busca de una mejor comprensión de los procesos básicos que ocurren durante la regulación de la expresión de genes, del papel del ARNi y de los factores de transcripción durante procesos celulares complejos en el desarrollo y la fisiología celular normal, incluidos la viabilidad, la proliferación, la fertilidad, el metabolismo, la estabilidad genómica, la función mitocondrial, la senescencia, la morfología celular, la diferen-ciación celular, la señalización celular, la respuesta inmune y la apoptosis (muerte celular programada), y en enfermedades cuya patogenia está mediada por la alteración en la expresión de genes específicos (35).
En poco tiempo ya se han generado nuevas investigaciones en busca del conocimiento de la fisiopatología molecular de enfermedades con alteraciones en la regulación de la expresión de genes y su intrincada relación con esos procesos celulares y como una aproximación para conocer el posible papel del ARNi en los mecanismos patógenos de varias enfermedades comunes, crónicas e infecciosas que incluyen enfermedades virales, influenza, hepatitis, cáncer, malformacio-nes congénitas, hipercolesterolemia, algunas enfermedades cardiacas, neurodegenerativas y cerebrovasculares, entre otras (36-43). Por ejemplo, en el cáncer, las células escapan de los mecanismos normales que regulan la expresión de genes específicos cuyos productos controlan la proliferación, la diferenciación y la muerte celular. Las alteraciones de la expresión de genes que codifican proteínas específicas y que no codifican proteínas como los miARN, han sido reportadas en cáncer lo cual sugiere que en la regulación de la expresión genética, el ARNi podría contribuir en el proceso de su desarrollo (39,40). En otras entidades como en los trastornos inflamatorios, cardiovasculares y neurodegenera-tivos, el uso de ds-ARN cortos sintetizados químicamente y a través del desarrollo de sistemas de expresión que producen de manera estable siARN han permitido confirmar que estas patologías también afrontan los mecanismos de regulación del ARNi, alteraciones y cambios en expresión de genes específicos (41-44).
Los trabajos de los investigadores distinguidos con el premio nobel también han generado una explosión de investigaciones en biotecnología, genética molecular y farmacología molecular, particularmente en la caracterización de complejos moleculares que funcionan como máquinas de transcripción y en el diseño y construcción de ARN pequeños (micro-ARN, siARN, piARN) y de nuevos mediadores implicados en reducir la expresión de genes a nivel de la transcripción y después de ésta (45). Además, paralelamente se ha intensificado la búsqueda de funciones biológicas del ARNi, de genes maestros del desarrollo y la regulación del genoma. Por ejemplo, dos de las áreas de intensa investigación actual son la identificación de genes, factores de transcripción y ARN pequeños que comprometen a una célula madre a un linaje celular específico (46) y el silencio genético inducido por ds-ARN y su función en resistencia a la infección por virus en plantas y animales (47).
La profundización en la fisiología molecular de los ARN pequeños, la ARN polimerasa II, los factores de transcripción y en el papel de la cromatina contribuirá a entender por qué una célula es diferente a otra a pesar de tener el mismo contenido de información genética, que es una de las preguntas básicas de la biología (48). Además, el conocimiento de la regulación de la expresión genética en células eucariotas, incluidas la activación y la supresión de la expresión, generará nuevas alternativas para el estudio de la función de genes desconocidos y nuevas posibilidades de desarrollo de moléculas basadas en ARN para el tratamiento de enfermedades, en cuyo mecanismo patógeno está implicada una alteración de la expresión genética. Además, el uso del ARNi, como herramienta para manipular experimentalmente la expresión genética, facilitará el estudio de la función de genes individuales y sus efectos en una escala genómica, lo cual no sólo proporcionará nuevo conocimiento de los procesos biológicos básicos, sino también un medio para identificar más rápidamente blancos terapéuticos y marcadores moleculares de diagnóstico, progresión y pronós-tico de numerosas enfermedades humanas (49).
En este orden de ideas, el silencio genético ya se ha logrado in vivo con la inyección de siARN empaquetados en liposomas por vía intravenosa y se ha observado principalmente en órganos altamente perfundidos, como el pulmón, el hígado y el bazo (50). La liberación local de siARN ha sido exitosa en el sistema nervioso central (51) y por electroporación, también se ha logrado inducir ARNi en músculo, retina y cerebro (52). Varias líneas de investigación actuales están implementando con éxito el uso de vectores virales, basados en retrovirus (53), lentivirus (54) y adenovirus (55) para inducir ARNi a nivel experimental. Aunque ha sido exitoso se requieren más investigaciones para incrementar la estabilidad de moléculas siARN in vivo y la eficiencia con la que las células y tejidos captan estas moléculas.
La potencia, el alto grado de especificidad y la versatilidad del ARNi en sistemas experimentales han estimulado el esfuerzo para usar esta tecnología como herramienta para silenciar genes asociados con algunas enfermedades humanas. Hasta la fecha, algunos siARN han mostrado ser efectivos como agentes terapéuticos potenciales en cultivos celulares y modelos animales de patologías, incluidas cáncer (56), enfermedades infecciosas, principalmente virales (57), enfermedades inflamatorias y autoinmunes (58) y trastornos neurológicos (59). Las investigaciones adicionales relacionadas con la seguridad y la eficacia de usar la tecnología del ARNi como estrategia terapéutica, plantean en el futuro cercano los primeros estudios clínicos fase I. Sin embargo, para esto se requieren varios avances que incluyen, entre otros, refinamientos en los sistemas de entrega y liberación de moléculas para inducir ARNi de una manera regulada y específica de tejidos. Además, se requiere profundizar en los mecanismos moleculares del ARNi para desarrollar estrategias que disminuyan la producción de interferón inducido por dsARN, evitar el silencio o abatimiento genético inespecífico y muerte celular, como respuesta celular a un ARN exógeno (60).
En resumen, gracias a los descubrimientos de la maquinaria molecular de la transcripción, del proceso de interferencia del ARN y al avance en el conocimiento de sus mecanismos y regulación derivados de las investigaciones señaladas, hoy nos enfrentamos a una nueva dimensión del ARN en la regulación de la expresión genética y disponemos de herramientas experimentales en investigación biomédica para la lucha contra enfermedades comunes, que van desde la introducción de ácidos nucleicos exógenos, hasta la inducción de cambios en la expresión de genes específicos, en enfermedades infecciosas y en enfermedades crónicas. Igualmente, el ARNi se constituye en una herramienta para estudios preclínicos en modelos animales, de los que se espera permitan optimizar el potencial terapéutico del control de la expresión de genes y el ARNi para aplicaciones seguras y eficaces en humanos, en el menor tiempo posible.
Agradecimientos
El autor agradece a Colciencias y al INS por su apoyo para el desarrollo del proyecto de investigación "Uso de la tecnología de ARN de interferencia, ARNi, como potencial herramienta antitumoral" (código 2104-12-16780) cuya ejecución facilitó la escritura de este manuscrito.
Conflicto de intereses
El autor declara no tener conflicto de intereses, ni vinculación con algún laboratorio farmacéutico o empresa interesada en obtener resultados positivos o negativos de la información presentada.
Correspondencia:
Luis Alberto Gómez
Instituto Nacional de Salud, apartado aéreo 80080, Avenida Calle 26 Nº 51-60, Bogotá, D. C., Colombia;
teléfono (57-1) 220 7700, extensión 414; fax: (571) 220 0901;
Recibido: 23/11/06; aceptado: 30/11/06
Referencias
1. Cramer P, Bushnell DA, Fu J, Gnatt AL, Maier-Davis B, Thompson NE, et al. Architecture of RNA Polymerase II and implications for the transcription mechanism. Science 2000;288:640-9.
2. Gnatt AL, Cramer P, Fu J, Bushnell DA, Kornberg RD. Structural basis of transcription: An RNA Polymerase II elongation complex at 3.3 A resolution. Science 2001;292:1876-82.
3.Westover KD, Bushnell DA, Kornberg RD. Structural basis of transcription: separation of RNA from DNA by RNA polymerase II. Science 2004;303:1014-6.
4. Spiegelman BM, Heinrich R. Biological control through regulated transcriptional coactivators. Cell 2004;119: 157-67.
5. Annunziato AT, Hansen JC. Role of histone acetylation in the assembly and modulation of chromatin structures. Gene Expr 2000;9:37-61.
6. Maniatis T, Reed R. An extensive network of coupling among gene expression machines. Nature 2002;416:499-506.
7. Izant J, Weintraub H. Inhibition of Thymidine Kinase Gene expression by antisense RNA: A molecular approach to genetic analysis. Cell 1984;36:1007-15.
8. Harland R, Weintraub H. Translation of mRNA Injected into Xenopus oocytes is specifically inhibited by antisense RNA. Journal of Cell Biology 1985;10:1094-9.
9. Rosenberg U, Preiss A, Seifert E, Jackle H, Knipple DC. Production of phenocopies by Kruppel antisense RNA injection into Drosophila embryos. Nature 1985;313:703-6.
10. Guo S, Kemphues K. Par-1, a gene required for establishing polarity in C. elegans embryos, encodes a putative Ser/Thr kinase that is asymmetrically distributed. 1995;Cell 81:611-20.
11. van der Krol AR, Mur LA, Beld M, Mol JN, Stuitje AR. Flavonoid genes in petunia: addition of a limited number of gene copies may lead to a suppression of gene expression. Plant Cell 1990;2:291-9.
12. Napoli C, Lemieux C, Jorgensen R. Introduction of a chimeric Chalcone Synthase gene into Petunia results in reversible co-suppression of homologous genes in trans. Plant Cell 1990;2:279-89.
13. Ingelbrecht I, Van Houdt H, Van Montagu M, Depicker A. Posttranscriptional silencing of reporter transgenes in tobacco correlates with DNA methylation. Proc Natl Acad Sci U S A. 1994;91:10502-6.
14. Baulcombe DC. RNA as a target and an initiator of post-transcriptional gene silencing in transgenic plants. Plant Mol Biol 1996; 32:79-88.
15. Metzlaff M, ODell M, Cluster PD, Flavell RB. RNA-mediated RNA degradation and chalcone synthase A silencing in petunia. Cell 1997;88:845-54.
16. Fire A, Xu S, Montgomery MK, Kostas SA, Driver SE, Mello CC. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature 1998;391:806-11.
17. Montgomery MK, Xu S, Fire A. RNA as a target of double-stranded RNA-mediated genetic interference in Caenorhabditis elegans. Proc Natl Acad Sci U S A 1998;95:15502-7.
18. Tabara H, Grishok A, Mello CC. RNAi in C. elegans: soaking in the genome sequence. Science 1998;282:430-1.
19. Caplen NJ, Parrish S, Imani F, Fire A, Morgan RA. Specific inhibition of gene expression by small double-stranded RNAs in invertebrate and vertebrate systems. Proc Natl Acad Sci U S A 2001;98:9742-7.
20. Mello CC, Conte DJr. Revealing the world of RNA interference. Nature 2004;431:338-42.
21. Elbashir SM, Lendeckel W, Tuschl T. RNA interference is mediated by 21 and 22 nucleotide RNAs. Genes Dev 2001;15:188-200.
22. Lee RC, Ambros V. An extensive class of smallRNAs in Caenorhabditis elegans. Science 2001;294:862-64.
23. Hannon, GJ. RNA interference. Nature 2002;418:244-251
24. Zeng Y, Yi R, Cullen BR. MicroRNAs and and small interfering RNAs can inhibit mRNA expression by similar mechanisms, Proc Natl Acad Sci U S A 2003;100,9779-84.
25. Valencia-Sanchez MA, Liu J, Hannon GJ, Parker R. Control of translation and mRNA degradation by miRNAs and siRNAs Genes Dev 2006;20:515-24
26. Bernstein E, Caudy AA, Hammond SM, Hannon GJ. Role for a bidentate ribonuclease in the initiation step of RNA interference. Nature 2001;409:363-6.
27. Vanitharani R, Chellappan P, Fauquet CM. Short interfering RNA-mediated interference of gene expression and viral DNA accumulation in cultured plant cells. Proc Natl Acad Sci U S A. 2003;100:9632-6.
28. Lau NC, Seto AG, Kim J, Kuramochi-Miyagawa S, Nakano T, Bartel DP, et al. Characterization of the piRNA complex from rat testes. Science 2006;313:363-7.
29. Storz G, Altuvia S, Wassarman KM. An abundance of RNA regulators. Annu Rev Biochem. 2005;74:199-217.
30. Buratowski S, Moazed D. Gene regulation: expression and silencing coupled. Nature 2005;435:1174-5.
31. Buhler M, Verdel A, Moazed D. Tethering RITS to a nascent transcript initiates RNAi- and heterochromatin-dependent gene silencing. Cell 2006;125:873-86.
32. Soutchek J, Akink A, Bramlage B, Charisse K, Constien R, Donoghue M, et al. Therapeutic silencing of an endogenoose gene by systemic administration of modified siRNAs. Nature 432;2004:173-8
33. Inoue A, Sawata SY, Taira K. Molecular design and delivery of siRNA. J Drug Target 2006;14:448-55.
34. Pushparaj PN, Melendez AJ. Short interfering RNA (siRNA) as a novel therapeutic. Clin Exp Pharmacol Physiol. 2006;33:504-10.
35. Novina C, Sharp P. The iRNA revolution. Nature 430, 161-164.
36. Ge Q, McManus MT, Nguyen T, Shen CH, Sharp PA, Eisen HN, et al. RNA interference of influenza virus production by directly targeting mRNA for degradation and indirectly inhibiting all viral RNA transcription. Proc Natl Acad Sci U S A 2003;100:2718-23.
37. Lee NS, Dohjima T, Bauer G, Li H, Li MJ, Ehsani A, et al. Expression of small interfering RNAs targeted against HIV-1 rev transcripts in human cells. Nat. Biotechnol 2002;20:500-5.
38. Ying C, De Clercq E, Neyts J. Selective inhibition of hepatitis B virus replication by RNA interference. Biochem Biophys Res Comun 2003;309:482-84
39. Calin GA, Dumitru CD, Shimizu M, Bichi R, Zupo S, Notch E, et al. Frequent deletions and down-regulation of micro-RNA genes miR15 and miR16 at 13q14 in chronic lymphocytic leukemia. Proc Natl Acad Sc. U S A 2002;99:15524-29.
40. Calin GA, Sevignani C, Dumitru CD, Hyslop T, Noch E, Yendamuri S, et al. Human microRNA genes are frequently located at fragile sites and genomic regions involved in cancers. Proc Nat Acad Sci U S A 2004;101:2999-3004.
41. Kim YO, Park SJ, Balaban RS, Nirenberg M, Kim Y. A functional genomic screen for cardiogenic genes using RNA interference in developing Drosophila embryos. Proc Natl Acad Sci U S A 2004;101:159-64.
42. Miller VM, Gouvion CM, Davidson BL, Paulson HL. Targeting Alzheimers disease genes with RNA interference: an efficient strategy for silencing mutant alleles. Nucleic Acids Res. 2004;32:661-68.
43. Kao SC, Krichevsky AM, Kosik KS, Tsai LH. BACE1 suppression by RNA interference in primary cortical neurons. J Biol Chem 2004;279:1942-49.
44. Song E, Lee SK, Wang J, Ince N, Ouyang N, Min J, et al. Chen J. RNA interference targeting Fas protects mice from fulminant hepatitis. Nat Med 2003;9:347-51.
45. Reynolds A, Leake D, Boese Q, Scaringe S, Marshall WS, Khvorova A. Rational siRNA design for RNA interference. Nat Biotechnol 2004;22:326-30.
46. Hay DC, Sutherland L, Clark J, Burdon T. Oct-4 knockdown induces similar patterns of endoderm and trophoblast differentiation markers in human and mouse embryonic stem cells. Stem Cells 2004;22:225-35.
47. Wang XH, Aliyari R, Li WX, Li HW, Kim K, Carthew R, et al. RNA interference directs innate immunity against viruses in adult Drosophila. Science 2006;312:452-4.
48. Levin SA. Fundamental questions in biology. PLoS Biol 2006;4: e300. DOI: 10.1371/journal.pbio.0040300.
49. Hannon GJ, Rossi JJ. Unlocking the potential of the human genome with RNA interference. Nature 2004;431:371-8.
50. Sorensen DR, Leirdal M, Sioud M. Gene silencing by systemic delivery of synthetic siRNAs in adult mice. J Mol Biol 2003;327:761-66.
51. Baker-Herman TL, Fuller DD, Bavis RW, Zabka AG, Golder FJ, Doperalski NJ, et al. BDNF is necessary and sufficient for spinal respiratory plasticity following intermittent hypoxia. Nat Neurosci 2004,7:48-55.
52. Konishi Y, Stegmuller J, Matsuda T, Bonni S, Bonni A. Cdh1-APC controls axonal growth and patterning in the mammalian brain. Science 2004;303:1026-30
53. Lois C, Refaeli Y, Qin XF, Van Parijs L. Retroviruses as tools to study the immune system. Curr Opin Immunol 2001;13:496-504.
54. Abbas-Terki T, Blanco-Bose W, Deglon N, Pralong W, Aebischer P. Lentiviral-mediated RNA interference. Hum Gene Ther 2002;13:2197-201.
55. Zhao LJ, Jian H, Zhu H. Specific gene inhibition by adenovirus-mediated expression of small interfering RNA. Gene 2003, 316:137-41.
56. Wohlbold L, van der Kuip H, Miething C, Vornlocher HP, Knabbe C, Duister J, et al. Inhibition of bcr-abl gene expression by small interfering RNA sensitizes for imatinib mesylate (STI571). Blood 2003;102:2236-39.
57. Kitabwalla M, Ruprecht RM. RNA interference: a new weapon against HIV and beyond. N Engl J Med 2002;347:1364-67.
58. Zender L, Hutker S, Liedtke C, Tillmann HL, Zender S, Mundt B et al. Caspase 8 small interfering RNA prevents acute liver failure in mice. Proc Natl Acad Sci U S A 2003;100:7797-802.
59. Ding H, Schwarz DS, Keene A, Affar el B, Fenton L, Xia X. Selective silencing by RNAi of a dominant allele that causes amyotrophic lateral sclerosis. Aging Cell 2003;2:209-17.
60. Hutvagner G, Zamore PD. RNAi: nature abhors a double-strand. Curr Opin Genet Dev 2002;12:225-32.














