Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.26 no.4 Bogotá Dec. 2006
Epidemiología genética de la artritis reumatoide: ¿qué esperar de América Latina?
Angélica María Delgado-Vega 1, Javier Martín 2, Julio Granados 3, Juan Manuel Anaya 1
1 Unidad de Biología Celular e Inmunogenética, Corporación para Investigaciones Biológicas (CIB), Universidad del Rosario, Medellín, Colombia.
2
Instituto de Biomedicina López-Neyra, Consejo Superior de Investigación Científica (CSIC), Granada, España.3
Departamento de Inmunología y Reumatología, Instituto Nacional de Ciencias Médicas y Nutrición Salvador Zubirán, México D.F., México.Trabajo parcialmente presentado en forma oral durante el Simposio de Inmunogenética; 23 de febrero de 2006, Medellín, Colombia.
La artritis reumatoide es una enfermedad sistémica autoinmune, crónica, que afecta principalmente las articulaciones que tienen movimiento. La enfermedad es mucho más frecuente en mujeres y su prevalencia en la población latinoamericana es cercana al 0,5%. La existencia de agregación familiar (
ls= 2-17) indica su carácter hereditario. Sin embargo, la herencia de la artritis reumatoide es poligénica y no sigue un patrón mendeliano. La importancia de encontrar factores genéticos asociados con la artritis reumatoide radica en la contribución a la comprensión de los mecanismos patogénicos de la enfermedad, su posible aplicación clínica como marcadores de riesgo, diagnóstico, pronóstico, e incluso, blanco terapéutico. Mapeos genéticos llevados a cabo en diversas poblaciones en busca de loci y genes candidatos han identificado la región HLA como aquella con mayor evidencia de ligamento. Sin embargo, su fracción etiológica corresponde sólo a un tercio de la susceptibilidad genética de la enfermedad. Esto indica que genes diferentes al HLA también están implicados en la susceptibilidad a desarrollar artritis reumatoide. En Latinoamérica, los alelos HLA-DRB1*0404 y TNF -308A han sido asociados de manera uniforme con la artritis reumatoide. En el presente artículo se revisan los factores genéticos de la artritis reumatoide en el marco de una aproximación lógica y ordenada establecida por la epidemiología genética, y se ofrecen algunas recomendaciones para futuros estudios en poblaciones latinoamericanas.Palabras clave: artritis reumatoide, epidemiología, enfermedades autoinmunes, complejo mayor de histocompatibilidad, antígenos HLA, América Latina.
Genetic epidemiology of rheumatoid arthritis: What to expect from Latin America?
Rheumatoid arthritis is a chronic and systemic autoimmune disease characterized by inflammation and destruction of the synovial joints. It affects approximately 0.5% of the Latin-American population and is three times more common in women than in men. Evidence of familial aggregation
(ls=2-17) was the first indication of a genetic susceptibility to disease. As in other autoimmune diseases, it has a complex genetic basis. Results from whole-genome scans indicate that the HLA region contains a significant and consistent set of linked loci. However, HLA accounts for only one-third of the genetic susceptibility of disease, indicating that non-HLA genes are also involved in the disease susceptibility. In Latin-America, association with HLA-DRB1*0404 and TNF -308A alleles has been uniformly established; however, many other candidate genes remain to be studied. The identification of genetic factors conferring susceptibility to rheumatoid arthritis will contribute to the knowledge of the pathogenic mechanisms, ability to predict its occurrence, the development of diagnostic tools, prognosis, and treatment. The genetic epidemiology of rheumatoid arthritis is herein reviewed; a set of recommendations is provided for the design, analysis and interpretation of genetic association studies in the context of Latin-American populations.Key words: rheumatoid arthritis, epidemiology, autoimmune diseases, major histocompatibility complex, HLA antigens.
La artritis reumatoide es una enfermedad articular autoinmune, inflamatoria y crónica, que afecta principalmente a las mujeres. Con frecuencia compromete otros órganos distintos a las articulaciones y tiene un impacto adverso en la esfera biopsicosocial. Aunque su causa es desconocida, se han identificado factores endocrinos, ambientales y genéticos involucrados en su desarrollo, los cuales pueden variar de una población a otra (1). Al igual que otras enfermedades autoinmunes, desde el punto de vista genético, ésta es una enfermedad compleja, es decir, su herencia no obedece las leyes mendelianas y es poligénica (2). La complejidad se extiende a factores no genéticos, pues más allá de las exposiciones ambientales, como el cigarrillo, éstos incluyen eventos estocásticos, evolutivos e históricos que hacen de la enfermedad un fenómeno poblacional más que individual (3).
La comprensión de la relación entre genotipo y fenotipo es posible gracias a la epidemiología genética, disciplina que estudia el papel de los factores genéticos y su interacción con factores no genéticos (ej. ambientales) en la etiología de una enfermedad (4). La epidemiología genética considera también el efecto de la acción de los genes y los mecanismos de herencia de las enfermedades (4). En el presente artículo se revisan los factores genéticos de la artritis reumatoide en el marco de una aproximación lógica y ordenada, establecida por la epidemiología genética (1).
¿Qué tan fuerte es el componente genético de la artritis reumatoide?
Agrupamiento familiar
El primer paso para establecer si una enfermedad tiene un componente genético consiste en evaluar si existe agregación familiar y, posteriormente, examinar la segregación (4). La observación más simple que implica un factor genético en una enfermedad es la presencia de múltiples casos de la misma dentro de familias extendidas, fenómeno conocido como agrupamiento familiar (5). Sin embargo, este fenómeno debe cuantificarse mediante la comparación de la prevalencia de la enfermedad en grupos de individuos con diferente grado de relación genética. Entre gemelos monocigotos, genéticamente idénticos, existe una tasa de concordancia elevada de artritis reumatoide (12% a 15%) comparada con la observada en gemelos dicigotos y hermanos de individuos afectados (2% a 4%) con quienes comparten el 50% del material genético (3). A su vez, la frecuencia en los familiares cercanos de los pacientes es mayor que en la población general, en la que la prevalencia oscila entre 0,24% y 1%, siendo de aproximadamente 0,4% en la población latinoamericana (6).
Agregación familiar
A diferencia del sentido clínico propio del agrupamiento familiar, la agregación familiar es una medida epidemiológica estimada por el riesgo recurrente
lR (donde R representa el tipo de familiares de los casos afectados en los cuales se calcula la prevalencia de la enfermedad y luego se divide por la prevalencia en los familiares de los individuos sin el rasgo). Para la artritis reumatoide, la agregación familiar en hermanos de los afectados (lS hermanos) varía entre 2 y 17%, dependiendo de la prevalencia de la enfermedad en la población con la que se compare, lo que subraya la importancia de realizar este tipo de estudios en grandes cohortes compuestas por familias y pares de hermanos afectados (3). Por otro lado, también se ha demostrado aumento de la frecuencia de otras enfermedades autoinmunes en familiares de pacientes con artritis reumatoide (7), así como agregación familiar de artritis reumatoide en familiares de pacientes con otras enfermedades autoinmunes tales como lupus eritematoso sistémico (8,9) y síndrome de Sjögren primario (10), lo que indica que existe agregación familiar de autoinmunidad en general, más que de una enfermedad autoinmune en particular. Este concepto se conoce también como autoinmunidad familiar. Es importante distinguir este término del concepto de autoinmunidad compartida, el cual hace referencia a aquellos pacientes que poseen manifestaciones serológicas o clínicas de dos o más enfermedades autoinmunes (11). El punto común de estas observaciones es que sugieren determinantes genéticos compartidos para diferentes enfermedades autoinmunes (12).Heredabilidad
Para estimar de forma más precisa hasta dónde la agregación familiar es causada por factores genéticos, se requiere un modelo biológico racional que especifique cómo el fenotipo de interés puede ser modulado por el efecto de uno o más genes. Un modelo de componentes de varianza permite estimar la fracción de la variabilidad total (
s2T) atribuible a los efectos genéticos (s2G), tanto aditivos (s2A) como no aditivos (s2E), denominándola heredabilidad (13). A partir de estudios en gemelos, se estima que la heredabilidad de la artritis reumatoide es aproximadamente del 60%, independientemente de la prevalencia, sexo, edad de inicio o gravedad de la enfermedad (14). El poder estadístico de los estudios de identificación de genes es directamente proporcional a la heredabilidad del rasgo de interés.¿Existe un gen con un efecto suficiente que justifique los estudios para identificarlo?
Análisis de segregación
A partir de la evidencia de agregación familiar, el análisis de segregación investiga si la enfermedad puede explicarse por el efecto de uno o varios genes principales y busca un modelo probable de transmisión dentro de las familias. Se han postulado varios modelos de herencia para las enfermedades autoinmunes como la artritis reumatoide que incluyen un patrón monogénico autosómico recesivo (15), autosómico dominante (16, 17), de dos loci (18) y poligénico (19). El modelo más aceptado propone que la artritis reumatoide y las enfermedades autoinmunes, tomadas en conjunto, son el resultado del pleiotropismo de un gen mayor (probablemente en la región del HLA) sumado a múltiples efectos poligénicos menores (7) que podrían dirigir el blanco de la respuesta autoinmune.
¿En qué lugar del genoma puede estar el gen causante?
Análisis de ligamento
La incorporación de la biología de la gametogénesis y la recombinación meiótica a un modelo matemático permite mapear el genoma en busca de loci de susceptibilidad para una enfermedad partiendo de la tipificación de marcadores genéticos y el análisis de su cosegregación con el rasgo de interés en individuos relacionados (20). En una enfermedad compleja como la artritis reumatoide se utilizan métodos no paramétricos, es decir, que no requieren la especificación de un modelo de herencia, y en los cuales se asume que entre pares de hermanos afectados (ASP) existe un exceso de haplotipos idénticos por descendencia (IBD) en las regiones donde se ubican los genes causantes de la enfermedad (21).
A la fecha, se han realizado varios mapeos genéticos para artritis reumatoide en población japonesa (22,23), inglesa (24,25), europea (26,27) y norteamericana (28-30). Todos estos estudios, menos uno (22), identifican a la región HLA como la única que tiene evidencia significativa de ligamiento (LOD score > 3,6 o p < 2 x 10-5)(21) (cuadro 1). El locus HLA de clase II se reconoce de forma consistente como el principal factor de riesgo genético para la artritis reumatoide en diversas poblaciones, así su fracción etiológica corresponda sólo a un tercio de la susceptibilidad genética de la enfermedad (31). Esto indica que genes diferentes al HLA también están implicados. Sin embargo, la mayoría de los resultados de los estudios que han investigado loci no-HLA en artritis reumatoide no han sido replicados.
La inconsistencia de los resultados es explicable en gran parte por el bajo poder estadístico que poseen los estudios de ligamiento para detectar genes con efectos individuales moderados o bajos, poder que además se ve afectado por el tamaño limitado de la muestra, el tipo de marcadores utilizados, los errores de tipificación y la heterogeneidad tanto clínica como genética propia de la artritis reumatoide (20).
Existen varias estrategias que permiten confirmar si la ausencia de replicación se debe al poder estadístico o si los loci ligados corresponden a falsos positivos (error tipo I). La principal consiste en repetir el análisis de ligamento en otro grupo de familias provenientes de la misma población, y analizar el segundo grupo de pacientes como una muestra independiente o por medio de un análisis combinado. La
figura 1 ilustra los resultados obtenidos mediante esta aproximación por el grupo norteamericano NARAC (North American Rheumatoid Artritis Consortium) (28,29). Las regiones 1p13, 6p21 y 18q21, identificadas en el primer mapeo genético, fueron replicadas en un segundo grupo de 216 familias (332 ASP) con nueva evidencia de ligamento en 9q22 y 10q21. Es importante notar que la región no HLA con mayor ligamiento en el primer mapeo (4q22) no alcanzó evidencia nominal (p < 0,05) en el análisis combinado de las 512 familias (cuadro 1 ) (28,29).
Menos consistentes fueron los resultados en la cohorte inglesa de la ARC (Arthritis Research Campaign) (figura 1). Ninguno de los marcadores para los cuales hubo exceso de alelos IBD en el estudio de MacKay et al. (24) mostró evidencia de ligamento en un posterior análisis realizado por Eyre et al. (32). Aparentemente, el segundo grupo de familias difería significativamente del primero en términos de edad de inicio de la enfermedad y número de individuos portadores de alelos codificantes para el epítope compartido (SE), el cual corresponde a una secuencia similar en las posiciones 70-74 de la cadena
b del HLA-DRB1 que ha sido asociada a la artritis reumatoide (ver más adelante complejo mayor de histocompatibilidad).Considerando que parte de la heterogeneidad clínica (fenotipo) puede estar influida por factores genéticos (genotipo), es válido dividir o estratificar la población de estudio en subgrupos más homogéneos. El análisis estratificado de la cohorte del Reino Unido evidenció ligamento significativo en 6q cuando ambos hermanos de un ASP eran mujeres o portadores de dos copias del SE, y en 16p para individuos con artritis reumatoide de inicio temprano (< 40 años)(32). Sin embargo, al reducir aún más el tamaño de la muestra se afecta negativamente el poder estadístico. Otra alternativa para corregir la heterogeneidad sin afectar el poder consiste en realizar un análisis multivariado que considere los diferentes fenotipos clínicos como covariables, tales como las erosiones óseas, el SE, la edad de inicio, etc. Aplicando esta estrategia en una población caucásica independiente, Osorio et al. (27) obser-varon que 2p14 y 12q23 están asociados con erosiones óseas, 2q14 con AR de inicio temprano, y 12q23 y 20p12 con la presencia del SE.
En 1999, Wise et al (33) establecieron un método de clasificación no paramétrico para el metanálisis de mapeos genéticos. Este método combina los resultados de diferentes estudios con el fin de corroborar las regiones genómicas que demuestran evidencia de ligamento repetidamente, e identifica regiones nuevas que no alcanzan el nivel de significancia en estudios individuales (33). A la fecha se han llevado a cabo dos metanálisis de mapeos genéticos para artritis reumatoide (34,35). Además de confirmar al HLA como el locus más significativo (p < 2x10-5), Fisher et al. (34) identificaron otras regiones con evidencia significativa de ligamento, principalmente 16cen, 6q, 12p y, luego de considerar el peso relativo de cada estudio según el número de ASP, 1q, 3q, 4q, 8p, 9q y 14q. Choi et al. (35) encontraron evidencia consistente de ligamento en 6p22.3-p21.1 (Psumrnk= 8x10-7); 6q, 8pter y 18q (Psumrnk< 0,01); así como en 12p, 1p y 16p (Psumrnk< 0,05). En resumen, estos dos metanálisis de mapeos genéticos para artritis reumatoide confirman al HLA como el mayor factor de susceptibilidad para la enfermedad y revelan evidencia de ligamento en regiones no HLA en los cromosomas 1p, 6, 8p, 12, 16 y 18q en todos los estudios (34,35). Etzel et al. (36) publicaron recientemente un metanálisis basado en el método Loesgen, el cual promedia el Non-parametric linkage (NPL) score obtenido en los estudios de ligamento analizados, otorgando un peso a cada uno en función del contenido de información y el tamaño de la muestra. Siete de las regiones reportadas (1q, 6p, 6q, 8p, 12p, 16p y 18q) coinciden con las identificadas previamente mediante el método de metanálisis de mapeos genéticos (34,35), más dos nuevos loci ligados en 2q y 5p. Al comparar ambos métodos, vale la pena destacar que el metanálisis posee mayor poder para identificar genes que contribuyen a la susceptibilidad de la enfermedad en todas las poblaciones involucradas, mientras que el poder para analizar poblaciones diversas no fue examinado en el metanálisis de Etzel et al (36), pues sólo incluyó cuatro mapeos genómicos realizados en caucásicos (24,26,28,29). Sin embargo, el método Loesgen ha demostrado ser más robusto y consistente porque no combina resultados de pruebas de significancia (LOD score o valor p) como sí lo hace el metanálisis de mapeos genéticos y, por lo tanto, es menos susceptible a incrementos en la tasa de falsos positivos (37)
¿Pueden identificarse las regiones de susceptibilidad con mayor precisión?
Cuando no se parte de ninguna hipótesis previa sobre la localización de los genes involucrados en la enfermedad de interés, se examina el genoma con marcadores genéticos regularmente distribuidos y, una vez se identifican las regiones relevantes, se realiza un mapeo genético más fino al interior de ellas en pasos sucesivos hasta identificar el locus y su gen o el polimorfismo incriminado en la susceptibilidad.
La mayoría de los estudios de ligamento para artritis reumatoide se basan en mapas de microsatélites o secuencias cortas de ADN repetidas en tándem (STR, short tandem repeats), examinados de forma regular a lo largo de todo el genoma cada 10 a 12 centimorgans (cM) (22,26,28,29). Debido a que la densidad del mapa de marcadores está relacionada directamente con la posibilidad de encontrar un locus de susceptibilidad para la enfermedad, Osorio et al. realizaron un estudio de ligamento con microsatélites espaciados cada 3,3 cM, reduciendo a cerca de 0,8 cM (3,3/4) la distancia promedio entre cualquier posible gen de susceptibilidad y el marcador más cercano a éste (27). Sus resultados discriminaron dentro del HLA el locus HLA-DRB1 como la región con mayor evidencia de ligamento (p = 6,25 x 10-5) e identificaron 19 regiones por fuera del HLA (19). Adicionalmente, luego de realizar 10.000 simulaciones computarizadas de genotipos, se estimó que 8±4 regiones génicas eran verdaderos positivos (27) (
figura 1). La simulación de genotipos es otra herramienta que disminuye la tasa de falsos positivos al obtener un nivel de significación más astringente.En la actualidad, gracias al Proyecto Genoma Humano y al Proyecto Internacional HapMap, se cuenta con un catálogo de más de 3 millones de polimorfismos de un sólo nucleótido (SNP, single nucleotide polimorphism) y haplotipos comunes de SNP que posibilitan mapeos genéticos más densos y de mayor poder y precisión (38). Dado que el desequilibrio de ligamiento es un parámetro poblacional, se ha generado controversia sobre la aplicabilidad de los patrones descritos sobre poblaciones diferentes a las incluidas en el HapMap. Estudios recientes respaldan que los tag SNP (tSNP) capturan la mayor parte de la variabilidad genética humana [entre 90 y 95% de los SNP con una frecuencia alélica menor (MAF)
³ 5% y coeficiente de correlación r2 ³ 0,8] (39). El poder para detectar un locus de susceptibilidad en un estudio de ligamento o asociación basado en tSNP depende principalmente del tamaño de la muestra, la MAF de las variantes seleccionadas y la densidad con la cual se distribuyen sobre la región de interés, y no de si los bloques de haplo-tipos se conservan en la población de estudio (40).Es importante recordar que tanto los SNP como los microsatélites tienen ventajas y desventajas que deben considerarse en cada estudio (
cuadro 2). No obstante, dado que los SNP son los marcadores genéticos más abundantes a lo largo del genoma y los métodos para su tipificación son simples y adaptables a sistemas de alto rendimiento, se han convertido en la mejor opción para el mapeo de genes asociados a enferme-dades complejas (41). John et al. (25) confirmaron la utilidad de esta tecnología mediante la tipificación de más de 11.000 SNP en 157 familias del Reino Unido, demostrando mayor contenido de información frente a los microsatélites, así como mayor uniformidad y mejor definición de la región de ligamento. Respecto a los loci para artritis reumatoide, aunque los resultados fueron más o menos similares, con SNP se identificaron cuatro loci adicionales en 13q, 14q, 21q y Xp (25).
El estudio de ligamiento más grande basado en SNP, de los reportados hasta el momento de la elaboración de este manuscrito, fue realizado por Amos et al. (30) en 642 familias que incluían 1.371 ASP pertenecientes a la cohorte norteamericana (NARAC). Mediante la tipificación de 5.850 SNP a lo largo del genoma, identificaron dos nuevas regiones de ligamento con LOD > 3,0 en los cromosomas 2q33 y 11p12 (
cuadro 1). Nueva-mente, la evidencia más fuerte de ligamiento se encontró en la vecindad del complejo mayor de histocompatibilidad (MHC) con un LOD = 16, evidencia fuertemente influenciada por la presencia de anticuerpos anti-CCP. Sin embargo, la región de ligamiento en 6p21 es más amplia que en estudios previos, sugiriendo la influencia de múltiples loci de susceptibilidad dentro de ésta. Además, este estudio cuantificó un aumento de 44% en el contenido de información obtenido con un mapa denso de SNP comparado con el mapa de microsatélites usado previamente en 512 familias de la misma cohorte, aspecto que adquie-re especial importancia en enfermedades de inicio tardío como la artritis reumatoide, en las que la disponibilidad de ADN parental está limitada (30).¿Existe un polimorfismo genético causal o se puede identificar un bloque haplotípico de susceptibilidad?
Análisis de asociación
El objetivo general de los estudios de asociación genética es detectar si existe relación epidemiológica entre una o más variantes genéticas y un rasgo a partir de cómo difieran las frecuencias de estas variantes entre un grupo de casos y otro de controles (
cuadro 3). Sin embargo, desde el punto de vista biológico, la asociación tiene tres interpretaciones diferentes: (i) el polimorfismo tiene un papel causal (asociación directa); (ii) el polimorfismo no es causal, pero está en el desequilibrio de ligamento con la variante causal (asociación indirecta), o (iii) la asociación es producto de la estratificación o mezcla de la población (asociación espuria)(13).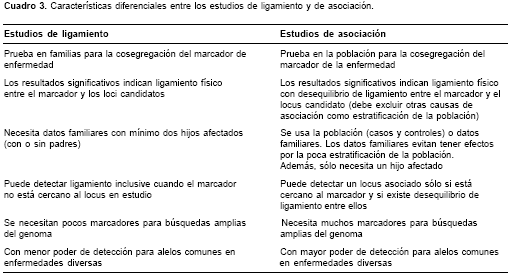
Asociación indirecta
Existen al menos dos maneras de cubrir el genoma entero con SNP; una de ellas está basada en la asociación indirecta de los genes de suscepti-bilidad con SNP altamente informativos (tSNP) distribuidos regularmente a lo largo del genoma (42). El número de tSNP necesario para representar las variaciones genéticas más comunes está entre 200.000 y 1´000.000 (43), lo cual resulta demasiado costoso para la mayoría de los grupos de investigación en enfermedades complejas, tales como la artritis reumatoide. Alternativa-mente, los investigadores del NARAC, así como Begovich et al., han identificado y desarrollado la tipificación de una serie de 12.000 SNP potencialmente funcionales dada su localización en regiones génicas codificantes o reguladoras (44). La dificultad de esta aproximación reside en el mayor tamaño de la muestra requerido para detectar ligamento con polimorfismos codificantes, ya que además de tener un efecto genético moderado, son menos frecuentes que los no codificantes (45).
Se han propuesto estrategias más costo-efectivas como la tipificación por etapas, en la que no es necesario tipificar todos los marcadores genéticos en el número total de individuos (45). Reciente-mente, Tamiya et al. (23) publicaron el primer mapeo genético de asociación realizada para artritis reumatoide en cuatro etapas: 1) identificación de los STR necesarios para escanear el genoma con una densidad de 100 kb; 2) tipificación de los 27.037 STR identificados en un pool de ADN de 375 casos y controles divididos en tres fases (sólo los STR con evidencia de ligamiento en un grupo fueron replicados en el siguiente, reduciendo el error tipo I); 3) de los 133 STR asociados se confirmaron 47 mediante tipificación individual del genotipo (no pool) en 940 casos e igual número de controles y, por último, 4) disección fina de las regiones génicas de susceptibilidad identificadas (6p21.3, 11q13.4, 10q13 y 14q23.1) usando tSNP y análisis de haplotipos (figura 1) (23). Esta aproximación en etapas conservó la mayoría del poder estadístico y disminuyó los costos derivados de la tipificación (45).
Genes candidatos
La importancia de encontrar genes asociados con la artritis reumatoide es su contribución a la comprensión de los mecanismos patogénicos de la enfermedad, su posible aplicación clínica como marcadores de riesgo, diagnóstico, pronóstico e, incluso, blanco terapéutico (43).
La selección de los genes candidatos puede tener una justificación posicional (generador de hipótesis), así como funcional (probador de hipótesis). La primera asume que los genes de susceptibilidad con mayor efecto están ubicados en las regiones de ligamento; por lo tanto, es acertado examinarlas en detalle en busca de genes con plausibilidad biológica. La segunda parte de la alteración en la función o niveles de expresión de la proteína codificada y su papel en la fisiopatología de la enfermedad. En la artritis reumatoide, virtualmente cualquier gen que codifique para una molécula involucrada en proliferación o adhesión celular, inflamación, señalización intracelular, angiogénesis o degradación de matriz extracelular puede ser razonablemente postulado como gen candidato.
Más de 180 genes han sido estudiados en la artritis reumatoide, la mayoría de ellos con funciones en el reconocimiento inmunológico (HLA, TLRll), quimiotaxis, citocinas (TNF, IL6, MIF), factores de crecimiento, ligandos celulares, moléculas de adhesión (ICAM1), señalización intracelular (PTPN22, NFKBIL1) y factores de transcripción (RUNX1), entre otros (46). Sin embargo, con excepción del HLA, la mayoría de asociaciones publicadas no han sido reproducidas en poblaciones diferentes a las que se reportan, lo que subraya la necesidad de revisar cuidadosamente el diseño experimental de los estudios de asociación, con especial atención en la selección de las variantes genéticas, la población de estudio, la selección de controles, el análisis estadístico y la replicación de los resultados (43) (cuadro 3). Al analizar polimor-fismos de genes candidatos, el tamaño de la muestra es otro de los factores importantes a considerar, ya que la magnitud de la mayoría de asociaciones dada por la razón de disparidad (odds ratio [OR]) es baja. Los principales estudios de asociación para genes no-MHC en AR están resumidos en el cuadro 4 (Disponible en: http://www.ins.gov.co/pdf/Biomedica/artritis_reumatoide_cuadro_4.pdf).
No obstante, vale la pena recordar que las discrepancias observadas en los genes asociados entre diferentes poblaciones pueden explicarse, al menos en parte, por la heterogeneidad genética de la enfermedad. Diferentes alelos, incluso diferentes genes, pueden responder por la susceptibilidad a la enfermedad en diferentes grupos étnicos. Tal es el caso de los estudios de asociación para artritis reumatoide llevados a cabo en población japonesa, los cuales han señalado evidencia significativa para alelos de los genes PADI4 y SLC22A4 (53,75); tales hallazgos no han sido reproducidos en población caucásica (96). La asociación más robusta descrita para un gen no-MHC ha sido la variante 620W del gen PTPN22, replicada en un amplio número de poblaciones de origen caucásico (97). Sin embargo, este gen prácticamente no es polimórfico en población asiática (98). En población colombiana (Medellín y Bucaramanga) no observamos que este polimorfismo confiriera riesgo de susceptibilidad para la artritis reumatoide (51). Estos últimos resultados deben interpretarse como ausencia de asociación (heterogeneidad genética) o bien como riesgo de susceptibilidad muy bajo, que implica que el estudio no alcanzó suficiente poder estadístico.
Complejo mayor de histocompatibilidad
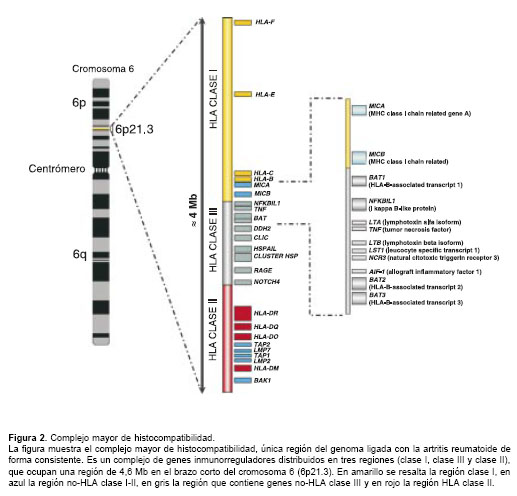
Han pasado 30 años desde que el investigador de origen peruano Peter Stastny reportó por primera vez la asociación del hoy conocido HLA-DRB1*04 con la artritis reumatoide (99); sin embargo, aún se desconoce el mecanismo exacto que determina esta predisposición. Hasta el momento, el complejo mayor de histocompati-bilidad es la única región del genoma que ha demostrado estar asociada con artritis reumatoide de manera consistente, contribuyendo con aproximadamente el 30% de la susceptibilidad genética a la enfermedad (31). El complejo mayor de histocompatibilidad es un complejo de genes, la mayoría con funciones inmunorreguladoras, cuyo haplotipo extendido cubre 7,6 Mb sobre el brazo corto del cromosoma 6 (6p21.3) (figura 2). Está subdivido en tres regiones desde el telómero hasta el centrómero así: clase I, clase III y clase II. En ésta última se encuentran los loci HLA-DR, -DP y -DQ, que codifican las cadenas a y b de las moléculas HLA de clase II (100).
Desde el informe de Stastny, diferentes alelos HLA-DRB1 se han reportado en poblaciones étnicamente diversas; tales hallazgos fueron unificados en 1987 por Peter Gregersen bajo la hipótesis del "epítope compartido" (SE) (101). El SE es una secuencia de aminoácidos común para los diferentes alelos asociados (70QRRAA74, 70RRRAA74 ó 70QKRAA74) que comprende los residuos 70 a 74 de la tercera región hipervariable de la cadena
b de la molécula HLA-DR (cuadro 5).La localización del SE dentro de la fosa de anclaje al péptido lo involucra directamente en la presentación antigénica, lo cual sugiere varios mecanismos funcionales para explicar la susceptibilidad a la enfermedad
(cuadro 6). Los aminoácidos de carga negativa (ácido aspártico, ácido glutámico) encajan perfectamente cuando esta secuencia de aminoácidos forma parte del bolsillo 4, como es el caso del péptido (261-273) del colágeno tipo II involucrado en modelos experimentales de artritis (103). Por lo tanto, aunque los alelos HLA-DR comparten ciertas características comunes, cada alelo posee una especificidad particular determinada por los aminoácidos que conforman las regiones que interactúan con los péptidos antigénicos, es decir, los bolsillos de unión.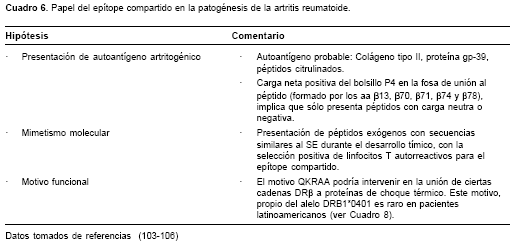
Cárdenas et al. (107) propusieron que las interacciones moleculares que se dan entre los aminoácidos de los péptidos antigénicos y los que forman las moléculas HLA de clase II pueden describirse y predecirse mediante simulaciones teóricas computacionales basadas en los tres principales momentos multipolares (monopolar, bipolar, cuadripolar) de la distribución continua de cargas electrostáticas. Estos cálculos se realizan para cada bolsillo de unión, primero vacío y luego ocupado por cada uno de los 20 posibles aminoácidos naturales, con el fin de obtener una variable que mide la magnitud de la perturbación del potencial electrostático del bolsillo producida por el efecto del aminoácido ocupante sobre los residuos con los que interactúa (108). Comparando el comportamiento de los cinco bolsillos de unión al antígeno (P1, P4, P6, P7 y P9) en los complejos HLA-DR
b1*0401-Colágeno tipo II(1168-1169) y HLA-DRb1*0101-hemaglutinina del virus de la influenza, Cárdenas et al. (108) encontraron que el bolsillo P4 tiene una ligera predominancia en cuanto a la carga general de la molécula, explicada por la presencia de un residuo b13H (histidina) más el residuo b71K (lisina) en el alelo HLA-DRB1*0401 que corresponde a b 71R (arginina) en HLA-DRB1*0101 (cuadro 5). Aunque los aminoácidos K y R poseen cadenas laterales formadas respectivamente por grupos de butilamonio y guanidina que les otorgan una carga positiva, desde el punto de vista electrostático, el efecto de la carga de la lisina resultó más evidente que el de la arginina, la cual presentó un comportamiento similar al de aminoácidos aromáticos (carga planar simétrica) (108). Los aminoácidos principales donde se encontraron las mayores diferencias están indicados en el cuadro 5, resaltando los que presentaron más perturbación. Adicionalmente, estos estudios han revelado que cada bolsillo tiene su propio tipo de comportamiento: el bolsillo P1 es predominante-mente un bolsillo de anclaje, la especificidad del alelo está modulada especialmente por los bolsillos P6 y P7, mientras que los bolsillos P4 y P9 cumplen un doble papel (109). Los resultados de esta aproximación teórica concuerdan con estudios experimentales y se convierte en una herramienta prometedora para la comprensión de la restricción genética del reconocimiento inmunológico.Aunque es razonable, la hipótesis del SE no explica toda la susceptibilidad genética conferida por el HLA, pues a pesar de la robustez de la asociación con el locus DRB1, el riesgo absoluto de desarrollar artritis reumatoide en individuos portadores de alelos codificantes para el SE es relativamente baja. Para ejemplificar la situación, se ha reportado que hasta el 30% de la población normal caucásica es positiva para HLA-DRB1*04, lo que indica que la presencia del SE no es necesaria ni suficiente para desarrollar la enfermedad (110).
Varios estudios han sugerido que el papel principal del SE consistiría en modular la gravedad de la enfermedad. Las características que han sido asociadas con la presencia de uno o más alelos SE-positivos son, principalmente, la seropositi-vidad del factor reumatoide y la enfermedad erosiva (111). Sin embargo, el poder estadístico de este tipo de estudios ha sido limitado y por lo tanto no permite establecer un consenso definitivo.
Además del fundamento epidemiológico de la hipótesis del SE, Ou et al. (112) introdujeron una clasificación funcional de los alelos HLA-DR sustentada por la evidencia experimental de la restricción-DR del receptor de células T (TCR). Dicha categorización establece siete patrones de supertipo DR-restrictivo (RSP) según los residuos polimórficos
b70, b71 y b74 del HLA-DR, aminoácidos que se unen al péptido durante la presentación antigénica y entran en contacto con el TCR, por lo cual le confieren la especificidad a la molécula, modelan el repertorio de linfocitos T, y determinan la asociación con susceptibilidad o resistencia a enfermedades (cuadro 7).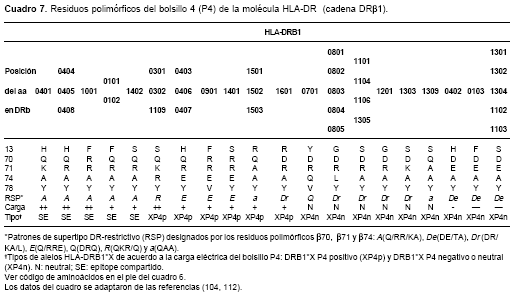
Los alelos de susceptibilidad asociados con artritis reumatoide codifican la secuencia del RSP "A" (70Q/R71R74K/A); en contraste, los RSP "De" (70D71E/T74A) y "E" (70Q/R71R74E), codificados por los alelos DRB1*0402 (De), *0403 y *0407 (E), están reportados como factores protectores para la enfermedad (112). Esta observación es coherente con el efecto protector atribuido a varios alelos HLA-DRB1 (*0103, *0402, *1102, *1103, *1301, *1302 y *1304), los cuales comparten la secuencia 70DERAA74 en vez del SE (113,114). No obstante, existe controversia sobre si este efecto es producto de la "ausencia de alelos de predisposición". Recientemente, van der Helm-van Mil et al. (115) demostraron en población caucásica (440 casos y 423 controles) que los alelos codificantes para 70DERAA74 protegen frente al desarrollo de la AR tanto en individuos SE- como en SE+. Adicionalmente, los pacientes portadores de la secuencia 70DERAA74 presentaron menor destrucción articular luego de cuatro años de seguimiento aun en presencia de otros factores asociados con la gravedad de la enfermedad, tales como el factor reumatoide positivo, los anticuerpos antipéptidos cíclicos citrulinados (CCP) y el tabaquismo (115). Así pues, la protección asociada a DERAA sería independiente del SE.
En Latinoamérica, en la población mexicana de origen mestizo se han identificado otros alelos protectores para artritis reumatoide que incluyen los DRB1*0701, *1101, *1503, *0801 y *1401 (116, 117). Estos alelos no codifican para 70DERAA74, pero comparten el ácido aspártico (D) en la posición 70, lo que sugiere que la carga positiva (D+) o negativa (Q/R) del residuo
b70 es determinante en el reconocimiento del complejo péptido-HLA-DR por parte del TCR. Reviron et al. (104) clasificaron los alelos HLA-DR de acuerdo a la carga electrostática neta del cuarto bolsillo de unión al antígeno (P4) y demostraron que los alelos con una carga neutra o negativa (DRB1*XP4n) eran factores de protección frente al desarrollo de artritis reumatoide, aunque aquellos con un P4 cargado positivamente (DRB1*XP4p) no tuvieron influencia significativa sobre la predisposición genética a desarrollar la enfermedad (cuadro 7).En Colombia, algunos estudios han examinado la relación entre HLA y la AR (118-122) (
cuadro 8). Clavijo et al. (118) mostraron que la frecuencia tanto del HLA-DR4 como del DR1 fue de 23% en 69 pacientes; este estudio no incluyó población control. Yunis et al. (119) estudiaron ocho pacientes de la tribu guambiana y 24 controles apareados por raza. La presencia del HLA-DR4 se observó en siete pacientes (87,5%) y en 15 controles (62,5%). Seis pacientes (75%) fueron HLA-DRB1*0404 mientras que este alelo fue observado en ocho de 15 controles (53,3%). Aunque la diferencia entre pacientes y controles no fue significativa, el hallazgo de genes caucásicos, tales como el DRB1*0404, en la población guambiana sugirió un efecto de fusión, que apoya la hipótesis que considera el origen de la artritis reumatoide en el antiguo continente, dada la asociación entre este alelo con la enfermedad. Ávila et al. (120), en un análisis de 60 pacientes y 60 controles pareados por sexo, reportaron un aumento de la frecuencia del HLA-DR4 en los pacientes (42 vs. 7%, riesgo relativo: 7,7, p < 0,001) (48). Ninguno de los anteriores estudios (118-120) examinó otros genes que pueden codificar para el SE, tales como el DR1, DR10 y DR14. En un pequeño grupo de pacientes afrocolombianos de Quibdó, pero que incluyó a toda la población afectada, no observamos asociación de alelos del HLA-DRB1 con la AR (121). Posteriormente, reportamos en población antioqueña la asociación del SE 70QRRAA74 en el HLA-DRB1*04, en particular el DRB1*0404, con la enfermedad (122). Este alelo corresponde, en efecto, el factor de riesgo más importante y uniforme para desarrollar artritis reumatoide en latinoamericanos (cuadro 8).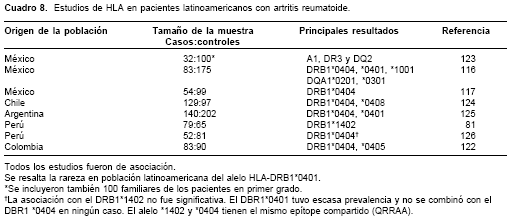
Una de las características principales del complejo mayor de histocompatibilidad es el alto desequilibrio de ligamento que existe entre los diferentes loci, lo cual dificulta la definición precisa de los genes responsables de la susceptibilidad. Algunos estudios han sugerido que el HLA-DQ es el principal locus del complejo mayor de histocompatibilidad asociado con artritis reumatoide y que los alelos HLA-DRB1 modulan la predisposición siendo permisivos o protectores (114). Sin embargo, otros estudios no respaldan esta hipótesis, indicando que la asociación DQ puede ser producto del desequilibrio de ligamento con el locus DRB1 (127).
La aplicación del análisis de ligamento de alta resolución sobre el complejo mayor de histocompatibilidad mediante STR o SNP ha revelado la existencia de un segundo locus de susceptibilidad para artritis reumatoide, que al parecer es independiente de DRB1 y está ubicado entre la porción telomérica de la región de clase III y la fracción más centromérica de la región de clase I (87,128-131). Esta región, de aproximadamente 600 kb, contiene genes candidatos para la artritis reumatoide dadas sus funciones inmunorreguladoras (
figura 2) (132). Sin embargo, hasta la fecha no se ha identificado el gen o los genes que contribuyen a la suscepti-bilidad genética de la artritis reumatoide de forma independiente a los genes HLA.Al examinar los genes con plausibilidad biológica se debe destacar el TNF (que codifica para el factor de necrosis tumoral alfa [TNF
a]) por dos razones principales. En primer lugar, por la evidencia de que el TNFa es una citocina pro-inflamatoria crítica en la fisiopatología de la artritis reumatoide: se produce en el tejido sinovial de pacientes con artritis reumatoide por macrófagos y células T, existen niveles elevados de la citocina en el suero y líquido sinovial, y los anticuerpos anti-TNFa tienen efecto terapéutico benéfico sobre la enfermedad (1). Segundo, el TNF es un gen altamente polimórfico, con cinco micro-satélites y numerosos SNP en su promotor, algunos de los cuales podrían regular la expresión génica (133).En población peruana, el alelo correspondiente al microsatélite TNF
a6 se asoció con la enfermedad independiente del HLA-DRB1 (81). Similares resultados han sido observados en población antioqueña (Gómez LM et al., sometido a publicación). En cuanto a los SNP del TNF, existen dos transiciones A>G en las posiciones –238 y –308. Ambas variantes fueron asociadas con artritis reumatoide en población mexicana, y para los alelos TNF -238GG y TNF -308A en enfermedad grave (82). En población colombiana observamos una asociación opuesta del TNF en autoinmunidad y tuberculosis (TB) (79). El estudio incluyó pacientes con artritis reumatoide (n = 165), lupus eritematoso (n = 100), síndrome de Sjögren primario (n = 67), tuberculosis (n = 135) y controles pareados étnicamente (n = 430), y demostró la asociación del alelo TNF –308A con artritis reumatoide (OR 1,8, p = 0,002), lupus eritematoso (OR 2,6, p < 0,0001), y síndrome de Sjögren primario (OR 2,9, p < 0,0001). En contraste, el alelo TNF –308G se asoció con tuberculosis (OR 1,8, p = 0,02). Igualmente, el alelo TNF –238A resultó ser un factor protector para autoinmunidad, pero de susceptibilidad para tuberculosis (OR 2,2, p < 0,0001) (79). En población chilena de Santiago se observó la presencia de TNF-308A en 23% de 92 pacientes con artritis reumatoide y en 42 controles (OR 2,8, p = 0,05)(83).La variabilidad en los polimorfismos del TNF asociados en diferentes poblaciones y la imposibilidad de afirmar que no son producto del desequilibrio de ligamento con loci diferentes del HLA-DRB1 reflejan la complejidad del estudio de genes dentro del complejo mayor de histocom-patibilidad, como también la importancia del tamaño muestral y la composición étnica de la población de estudio.
¿Qué esperar de América Latina?
Los principales retos enfrentados en la búsqueda de genes de susceptibilidad para la artritis reumatoide en América Latina se centran en garantizar la reproducibilidad de los resultados y encontrar variantes genéticas causales, es decir, demostrar que afectan la función o la cantidad de la proteína expresada. La probabilidad de que una asociación reportada sea verdadera depende del poder estadístico del estudio, el control del sesgo y el número de estudios que hayan probado la misma hipótesis. Es más factible que la asociación no sea real cuando los estudios se conducen en muestras pequeñas, la magnitud del riesgo atribuible al gen es baja, o se conducen estudios generadores de hipótesis con múltiples comparaciones, diseño experimental flexible con nuevas variables, y definiciones de fenotipos y métodos de análisis diversos (134).
En enfermedades autoinmunes, como la artritis reumatoide, debe prestarse especial atención al cálculo del tamaño de la muestra para alcanzar un poder estadístico >80%, teniendo en cuenta que la mayoría de genes involucrados en la enfermedad tiene un efecto genético moderado (OR = 1,5-2,5) o bajo (OR< 1,5) (135). Es importante recolectar un buen número de controles, de tal manera que la relación control:caso sea igual o superior a 1:1, cuidar que estén pareados de forma adecuada para evitar la estratificación de la población e incluir en el análisis de los datos la verificación de la misma mediante pruebas estadísticas como el Fst (136), así como realizar los ajustes por edad, sexo y demás covariables que puedan ser factores de confusión (137). Igualmente, se sugiere corregir la estratificación y la mezcla mediante la tipificación de marcadores informativos de ancestría (138,139).
Otra aproximación, que no se ve afectada por la estratificación de la población, es el estudio de familias basado en pruebas de desequilibrio de transmisión (TDT, Transmission Disequilibrium Test). El TDT observa las diferencias entre los alelos parentales que son transmitidos y no transmitidos a los hijos afectados para comparar la frecuencia de transmisión con lo esperado bajo un modelo de segregación independiente, en el que los alelos no transmitidos se usan como controles (3). En estudios poblacionales multicéntricos, que involucran individuos de diverso origen, esta herramienta resulta muy útil sumada al estudio tradicional de casos y controles no relacionados. En el cuadro 9 se esquematizan algunas recomendaciones que podrían contribuir en el diseño, implementación e interpretación de los estudios genéticos de asociación en América Latina.
Conclusiones
Dada la importancia de la artritis reumatoide en la población latinoamericana, tanto por su frecuencia (6) como por su impacto (140,141), y el actual interés en desarrollar estudios multicéntricos tal como el planteado por el Grupo Latinoamericano de Estudio de la Artritis Reumatoide (GLADAR), consideramos que la revisión y análisis aquí realizados podrán ser útiles para futuros estudios en nuestra población, que permitan mejorar la comprensión de los factores de riesgo genéticos de la enfermedad y su eventual implementación en la práctica clínica como factores de predicción.
Es imperiosa la necesidad de gestionar, innovar y compartir los beneficios del conocimiento entre comunidades locales, poniendo fin a la utilización comercial de los recursos genéticos por parte de grandes institutos de investigación en los países que disponen de la tecnología para su desarrollo y consiguen los derechos de propiedad intelectual. El Acuerdo sobre Aspectos Relacionados con el Comercio de los Derechos de Propiedad Intelectual (TRIPS) de la Organización Mundial del Comercio (OMC) no reconoce la necesidad de compartir beneficios, y es por eso que existe un creciente interés internacional por que se formulen políticas que reconozcan los derechos comunitarios a los recursos genéticos, protocolos y códigos de conducta para la recolección y manipulación de las muestras, medios de divulgación oportuna del conocimiento generado tanto para los sujetos de investigación como para los miembros de los grupos colaborativos (142). Estos acuerdos necesitan, por un lado, el respaldo de una legislación nacional e internacional sólida que reconozca la investigación en salud como una prioridad para el desarrollo de las naciones y le dedique la mejor inversión económica posible. De otro lado, la comunidad científica y académica debe hacer un esfuerzo concertado, en cooperación con el gobierno nacional, para diseñar un marco ético en el que se compartan los beneficios de la investigación genética basado en los principios de equidad y justicia y en el espíritu de convención (142).
Agradecimientos
Los autores expresan su gratitud a María Carolina Páez, John Castiblanco y Luis Miguel Gómez por sus comentarios y criticas constructivas al presente manuscrito.
Conflicto de intereses
Los autores del artículo hacen constar que no existe, de manera directa o indirecta, ningún tipo de conflicto de intereses financieros, académicos o personales que puedan poner en peligro la validez de lo comunicado.
Financiación
Este trabajo fue financiado por la Corporación para Investigaciones Biológicas y la Universidad del Rosario.
Correspondencia:
- Juan-Manuel Anaya, Corporación para Investigaciones
Biológicas, Carrera 72A No 78B-141, Medellín, Colombia.
Teléfono (+57-4) 4410855, fax (+57-4) 4415514
.Recibido: 28/05/06; aceptado: 22/09/06
Referencias
1. Anaya JM, Pineda-Tamayo R, Gómez LM, Galarza Maldonado C, Rojas-Villaraga A, Martin J. Artritis reumatoide: bases moleculares, clínicas y terapéuticas. Medellín: CIB, Universidad del Rosario, FUNPAR; 2006. [ Links ]
2. Worthington J. Investigating the genetic basis of susceptibility to rheumatoid arthritis. J Autoimmun 2005;25 (Suppl):16-20. [ Links ]
3. Seldin MF, Amos CI, Ward R, Gregersen PK. The genetics revolution and the assault on rheumatoid arthritis. Arthritis Rheum 1999;42:1071-9. [ Links ]
4. Burton PR, Tobin MD, Hopper JL. Key concepts in genetic epidemiology. Lancet 2005;366:941-51. [ Links ]
5. Lawrence JS, Ball J. Genetic studies on rheumatoid arthritis. Ann Rheum Dis 1958;17:160-8. [ Links ]
6. Senna ER, De Barros AL, Silva EO, Costa IF, Pereira LV, Ciconelli RM, et al. Prevalence of rheumatic diseases in Brazil: a study using the COPCORD approach. J Rheumatol 2004;31:594-7. [ Links ]
7. Lin JP, Cash JM, Doyle SZ, Peden S, Kanik K, Amos CI, et al. Familial clustering of rheumatoid arthritis with other autoimmune diseases. Hum Genet 1998;103:475-82. [ Links ]
8. Priori R, Medda E, Conti F, Cassara EA, Danieli MG, Gerli R, et al. Familial autoimmunity as a risk factor for systemic lupus erythematosus and vice versa: a case-control study. Lupus 2003;12:735-40. [ Links ]
9. Alarcon-Segovia D, Alarcon-Riquelme ME, Cardiel MH, Caeiro F, Massardo L, Villa AR, et al. Familial aggregation of systemic lupus erythematosus, rheumatoid arthritis, and other autoimmune diseases in 1,177 lupus patients from the GLADEL cohort. Arthritis Rheum 2005;52:1138-47. [ Links ]
10. Reveille JD, Wilson RW, Provost TT, Bias WB, Arnett FC. Primary Sjogrens syndrome and other autoimmune diseases in families. Prevalence and immunogenetic studies in six kindreds. Ann Intern Med 1984;101:748-56. [ Links ]
11. Sloka S. Observations on recent studies showing increased co-occurrence of autoimmune diseases. J Autoimmun 2002;18:251-7. [ Links ]
12. Alarcon-Riquelme ME. The genetics of shared autoimmunity. Autoimmunity 2005;38:205-8. [ Links ]
13. Cordell HJ, Clayton DG. Genetic association studies. Lancet 2005;366:1121-31. [ Links ]
14. MacGregor AJ, Snieder H, Rigby AS, Koskenvuo M, Kaprio J, Aho K, et al. Characterizing the quantitative genetic contribution to rheumatoid arthritis using data from twins. Arthritis Rheum 2000;43:30-7. [ Links ]
15. Lynn AH, Kwoh CK, Venglish CM, Aston CE, Chakravarti A. Genetic epidemiology of rheumatoid arthritis. Am J Hum Genet 1995;57:150-9. [ Links ]
16. Bias WB, Reveille JD, Beaty TH, Meyers DA, Arnett FC. Evidence that autoimmunity in man is a Mendelian dominant trait. Am J Hum Genet 1986;39:584-602. [ Links ]
17. Pritchard MH. Evidence for a hypothetical non-HLA susceptibility gene in rheumatoid arthritis. Br J Rheumatol 1994;33:475-9. [ Links ]
18. Rigby AS, Voelm L, Silman AJ. Epistatic modeling in rheumatoid arthritis: an application of the Risch theory. Genet Epidemiol 1993;10:311-20. [ Links ]
19. Yamashita TS, Khan MA, Kushner I. Genetic analysis of families with multiple cases of rheumatoid arthritis. Dis Markers 1986;4:113-9. [ Links ]
20. Dawn Teare M, Barrett JH. Genetic linkage studies. Lancet 2005;366:1036-44. [ Links ]
21. Lander E, Kruglyak L. Genetic dissection of complex traits: guidelines for interpreting and reporting linkage results. Nat Genet 1995;11:241-7. [ Links ]
22. Shiozawa S, Hayashi S, Tsukamoto Y, Goko H, Kawasaki H, Wada T, et al. Identification of the gene loci that predispose to rheumatoid arthritis. Int Immunol 1998;10:1891-5. [ Links ]
23. Tamiya G, Shinya M, Imanishi T, Ikuta T, Makino S, Okamoto K, et al. Whole genome association study of rheumatoid arthritis using 27 039 microsatellites. Hum Mol Genet 2005;14:2305-21. [ Links ]
24. MacKay K, Eyre S, Myerscough A, Milicic A, Barton A, Laval S, et al. Whole-genome linkage analysis of rheumatoid arthritis susceptibility loci in 252 affected sibling pairs in the United Kingdom. Arthritis Rheum 2002;46:632-9. [ Links ]
25. John S, Shephard N, Liu G, Zeggini E, Cao M, Chen W, et al. Whole-genome scan, in a complex disease, using 11,245 single-nucleotide polymorphisms: comparison with microsatellites. Am J Hum Genet 2004;75:54-64. [ Links ]
26. Cornelis F, Faure S, Martinez M, Prudhomme JF, Fritz P, Dib C, et al. New susceptibility locus for rheumatoid arthritis suggested by a genome-wide linkage study. Proc Natl Acad Sci U S A 1998;95:10746-50. [ Links ]
27. Osorio YF, Bukulmez H, Petit-Teixeira E, Michou L, Pierlot C, Cailleau-Moindrault S, et al. Dense genome-wide linkage analysis of rheumatoid arthritis, including covariates. Arthritis Rheum 2004;50:2757-65. [ Links ]
28. Jawaheer D, Seldin MF, Amos CI, Chen WV, Shigeta R, Monteiro J, et al. A genomewide screen in multiplex rheumatoid arthritis families suggests genetic overlap with other autoimmune diseases. Am J Hum Genet 2001;68:927-36. [ Links ]
29. Jawaheer D, Seldin MF, Amos CI, Chen WV, Shigeta R, Etzel C, et al. Screening the genome for rheumatoid arthritis susceptibility genes: a replication study and combined analysis of 512 multicase families. Arthritis Rheum 2003;48:906-16. [ Links ]
30. Amos CI, Chen WV, Lee A, Li W, Kern M, Lundsten R, et al. High-density SNP analysis of 642 Caucasian families with rheumatoid arthritis identifies two new linkage regions on 11p12 and 2q33. Genes Immun 2006;7:277-86. [ Links ]
31. Deighton CM, Walker DJ, Griffiths ID, Roberts DF. The contribution of HLA to rheumatoid arthritis. Clin Genet 1989;36:178-82. [ Links ]
32. Eyre S, Barton A, Shephard N, Hinks A, Brintnell W, MacKay K, et al. Investigation of susceptibility loci identified in the UK rheumatoid arthritis whole-genome scan in a further series of 217 UK affected sibling pairs. Arthritis Rheum 2004;50:729-35. [ Links ]
33. Wise LH, Lanchbury JS, Lewis CM. Meta-analysis of genome searches. Ann Hum Genet 1999;63:263-72. [ Links ]
34. Fisher SA, Lanchbury JS, Lewis CM. Meta-analysis of four rheumatoid arthritis genome-wide linkage studies: confirmation of a susceptibility locus on chromosome 16. Arthritis Rheum 2003;48:1200-6. [ Links ]
35. Choi SJ, Rho YH, Ji JD, Song GG, Lee YH. Genome scan meta-analysis of rheumatoid arthritis. Rheumatology (Oxford) 2006;45:166-70. [ Links ]
36. Etzel CJ, Chen WV, Shepard N, Jawaheer D, Cornelis F, Seldin MF, et al. Genome-wide meta-analysis for rheumatoid arthritis. Hum Genet 2006;119:634-41. [ Links ]
37. Dempfle A, Loesgen S. Meta-analysis of linkage studies for complex diseases: an overview of methods and a simulation study. Ann Hum Genet 2004; 68:69-83. [ Links ]
38. The International HapMap Consortium. The International HapMap Project. Nature 2003;426:789-96. [ Links ]
39. Montpetit A, Nelis M, Laflamme P, Magi R, Ke X, Remm M, et al. An evaluation of the performance of tag SNPs derived from HapMap in a Caucasian population. PLoS Genet 2006;2:e27. [ Links ]
40. Tenesa A, Dunlop MG. Validity of tagging SNPs across populations for association studies. Eur J Hum Genet 2006;14:357-63. [ Links ]
41. Thomas DC, Haile RW, Duggan D. Recent developments in genomewide association scans: a workshop summary and review. Am J Hum Genet 2005;77:337-45. [ Links ]
42. Yamamoto K, Yamada R. Genome-wide single nucleotide polymorphism analyses of rheumatoid arthritis. J Autoimmun 2005;25(Suppl):12-5. [ Links ]
43. Huizinga TW, Pisetsky DS, Kimberly RP. Associations, populations, and the truth: recommendations for genetic association studies in Arthritis & Rheumatism. Arthritis Rheum 2004;50:2066-71. [ Links ]
44. Criswell LA, Gregersen PK. Current understanding of the genetic aetiology of rheumatoid arthritis and likely future developments. Rheumatology (Oxford) 2005;44 (Suppl. 4):iv9-iv13. [ Links ]
45. Palmer LJ, Cardon LR. Shaking the tree: mapping complex disease genes with linkage disequilibrium. Lancet 2005;366:1223-34. [ Links ]
46. Becker KG, Barnes KC, Bright TJ, Wang SA. The genetic association database. Nat Genet 2004;36:431-2. [ Links ]
47. Carlton VE, Hu X, Chokkalingam AP, Schrodi SJ, Brandon R, Alexander HC, et al. PTPN22 genetic variation: evidence for multiple variants associated with rheumatoid arthritis. Am J Hum Genet 2005;77:567-81. [ Links ]
48. Begovich AB, Carlton VE, Honigberg LA, Schrodi SJ, Chokkalingam AP, Alexander HC, et al. A missense single-nucleotide polymorphism in a gene encoding a protein tyrosine phosphatase (PTPN22) is associated with rheumatoid arthritis. Am J Hum Genet 2004;75:330-7. [ Links ]
49. Hinks A, Barton A, John S, Bruce I, Hawkins C, Griffiths CE, et al. Association between the PTPN22 gene and rheumatoid arthritis and juvenile idiopathic arthritis in a UK population: further support that PTPN22 is an autoimmunity gene. Arthritis Rheum 2005;52:1694-9. [ Links ]
50. Lee AT, Li W, Liew A, Bombardier C, Weisman M, Massarotti EM, et al. The PTPN22 R620W polymorphism associates with RF positive rheumatoid arthritis in a dose-dependent manner but not with HLA-SE status. Genes Immun 2005;6:129-33. [ Links ]
51. Gómez LM, Anaya JM, González CI, Pineda-Tamayo R, Otero W, Arango A, et al. PTPN22 C1858T polymorphism in Colombian patients with autoimmune diseases. Genes Immun 2005;6:628-31. [ Links ]
52. Orozco G, Sanchez E, Gonzalez-Gay MA, Lopez-Nevot MA, Torres B, Caliz R, et al. Association of a functional single-nucleotide polymorphism of PTPN22, encoding lymphoid protein phosphatase, with rheumatoid arthritis and systemic lupus erythematosus. Arthritis Rheum 2005;52:219-24. [ Links ]
53. Suzuki A, Yamada R, Chang X, Tokuhiro S, Sawada T, Suzuki M, et al. Functional haplotypes of PADI4, encoding citrullinating enzyme peptidylarginine deiminase 4, are associated with rheumatoid arthritis. Nat Genet 2003;34:395-402. [ Links ]
54. Plenge RM, Padyukov L, Remmers EF, Purcell S, Lee AT, Karlson EW, et al. Replication of putative candidate-gene associations with rheumatoid arthritis in >4,000 samples from North America and Sweden: association of susceptibility with PTPN22, CTLA4, and PADI4. Am J Hum Genet 2005;77:1044-60. [ Links ]
55. Caponi L, Petit-Teixeira E, Sebbag M, Bongiorni F, Moscato S, Pratesi F, et al. A family based study shows no association between rheumatoid arthritis and the PADI4 gene in a white French population. Ann Rheum Dis 2005;64:587-93. [ Links ]
56. Barton A, Bowes J, Eyre S, Spreckley K, Hinks A, John S, et al. A functional haplotype of the PADI4 gene associated with rheumatoid arthritis in a Japanese population is not associated in a United Kingdom population. Arthritis Rheum 2004;50:1117-21. [ Links ]
57. Dieude P, Petit E, Cailleau-Moindrault S, Osorio J, Pierlot C, Martinez M, et al. Association between tumor necrosis factor receptor II and familial, but not sporadic, rheumatoid arthritis: evidence for genetic heterogeneity. Arthritis Rheum 2002;46:2039-44. [ Links ]
58. Kyogoku C, Tsuchiya N, Shibue T, Tokunaga K, Matsuta K. TNFR2 position 196 polymorphism in Japanese patients with rheumatoid arthritis: comment on the article by Dieude et al. Arthritis Rheum 2003;48:273-4. [ Links ]
59. Glossop JR, Nixon NB, Dawes PT, Hassell AB, Mattey DL. No association of polymorphisms in the tumor necrosis factor receptor I and receptor II genes with disease severity in rheumatoid arthritis. J Rheumatol 2003;30:1406-9. [ Links ]
60. Constantin A, Dieude P, Lauwers-Cances V, Jamard B, Mazieres B, Cambon-Thomsen A, et al. Tumor necrosis factor receptor II gene polymorphism and severity of rheumatoid arthritis. Arthritis Rheum 2004;50:742-7. [ Links ]
61. Kyogoku C, Tsuchiya N, Matsuta K, Tokunaga K. Studies on the association of Fc gamma receptor IIA, IIB, IIIA and IIIB polymorphisms with rheumatoid arthritis in the Japanese: evidence for a genetic interaction between HLA-DRB1 and FCGR3A. Genes Immun 2002;3:488-93. [ Links ]
62. Nieto A, Caliz R, Pascual M, Mataran L, Garcia S, Martin J. Involvement of Fcgamma receptor IIIA genotypes in susceptibility to rheumatoid arthritis. Arthritis Rheum 2000;43:735-9. [ Links ]
63. Huizinga TW, Keijsers V, Yanni G, Hall M, Ramage W, Lanchbury J, et al. Are differences in interleukin 10 production associated with joint damage? Rheumatology (Oxford) 2000;39:1180-8. [ Links ]
64. Lard LR, van Gaalen FA, Schonkeren JJ, Pieterman EJ, Stoeken G, Vos K, et al. Association of the -2849 interleukin-10 promoter polymorphism with autoantibody production and joint destruction in rheumatoid arthritis. Arthritis Rheum 2003;48:1841-8. [ Links ]
65. Pascual M, Lopez-Nevot MA, Caliz R, Ferrer MA, Balsa A, Pascual-Salcedo D, et al. A poly(ADP-ribose) polymerase haplotype spanning the promoter region confers susceptibility to rheumatoid arthritis. Arthritis Rheum 2003;48:638-41. [ Links ]
66. Gonzalez-Escribano MF, Rodriguez R, Valenzuela A, Garcia A, Garcia-Lozano JR, Nunez-Roldan A. CTLA4 polymorphisms in Spanish patients with rheumatoid arthritis. Tissue Antigens 1999;53:296-300. [ Links ]
67. Matsushita M, Tsuchiya N, Shiota M, Komata T, Matsuta K, Zama K, et al. Lack of a strong association of CTLA-4 exon 1 polymorphism with the susceptibility to rheumatoid arthritis and systemic lupus erythematosus in Japanese: an association study using a novel variation screening method. Tissue Antigens 1999;54:578-84. [ Links ]
68. Barton A, Myerscough A, John S, Gonzalez-Gay M, Ollier W, Worthington J. A single nucleotide polymorphism in exon 1 of cytotoxic T-lymphocyte-associated-4 (CTLA-4) is not associated with rheumatoid arthritis. Rheumatology (Oxford) 2000;39:63-6. [ Links ]
69. Milicic A, Brown MA, Wordsworth BP. Polymorphism in codon 17 of the CTLA-4 gene (+49 A/G) is not associated with susceptibility to rheumatoid arthritis in British Caucasians. Tissue Antigens 2001;58:50-4. [ Links ]
70. Vaidya B, Pearce SH, Charlton S, Marshall N, Rowan AD, Griffiths ID, et al. An association between the CTLA4 exon 1 polymorphism and early rheumatoid arthritis with autoimmune endocrinopathies. Rheumatology (Oxford) 2002;41:180-3. [ Links ]
71. Orozco G, Torres B, Nunez-Roldan A, Gonzalez-Escribano MF, Martin J. Cytotoxic T-lymphocyte antigen-4-CT60 polymorphism in rheumatoid arthritis. Tissue Antigens 2004;64:667-70. [ Links ]
72. Barton A, Jury F, Eyre S, Bowes J, Hinks A, Ward D, et al. Haplotype analysis in simplex families and novel analytic approaches in a case-control cohort reveal no evidence of association of the CTLA-4 gene with rheumatoid arthritis. Arthritis Rheum 2004;50:748-52. [ Links ]
73. Lei C, Dongqing Z, Yeqing S, Oaks MK, Lishan C, Jianzhong J, et al. Association of the CTLA-4 gene with rheumatoid arthritis in Chinese Han population. Eur J Hum Genet 2005;13:823-8. [ Links ]
74. Lin SC, Yen JH, Tsai JJ, Tsai WC, Ou TT, Liu HW, et al. Association of a programmed death 1 gene polymorphism with the development of rheumatoid arthritis, but not systemic lupus erythematosus. Arthritis Rheum 2004;50:770-5. [ Links ]
75. Tokuhiro S, Yamada R, Chang X, Suzuki A, Kochi Y, Sawada T, et al. An intronic SNP in a RUNX1 binding site of SLC22A4, encoding an organic cation transporter, is associated with rheumatoid arthritis. Nat Genet 2003;35:341-8. [ Links ]
76. Martinez A, Fernandez-Arquero M, Balsa A, Rubio A, Alves H, Pascual-Salcedo D, et al. Primary association of a MICA allele with protection against rheumatoid arthritis. Arthritis Rheum 2001;44:1261-5. [ Links ]
77. Singal DP, Li J, Zhang G. Microsatellite polymorphism of the MICA gene and susceptibility to rheumatoid arthritis. Clin Exp Rheumatol 2001;19:451-2. [ Links ]
78. Shibue T, Tsuchiya N, Komata T, Matsushita M, Shiota M, Ohashi J, et al. Tumor necrosis factor alpha 5'-flanking region, tumor necrosis factor receptor II, and HLA-DRB1 polymorphisms in Japanese patients with rheumatoid arthritis. Arthritis Rheum 2000;43:753-7. [ Links ]
79. Correa PA, Gomez LM, Cadena J, Anaya JM. Autoimmunity and tuberculosis. Opposite association with TNF polymorphism. J Rheumatol 2005;32:219-24. [ Links ]
80. Hajeer AH, Dababneh A, Makki RF, Thomson W, Poulton K, Gonzalez-Gay MA, et al. Different gene loci within the HLA-DR and TNF regions are independently associated with susceptibility and severity in Spanish rheumatoid arthritis patients. Tissue Antigens 2000;55:319-25. [ Links ]
81. Castro F, Acevedo E, Ciusani E, Angulo JA, Wollheim FA, Sandberg-Wollheim M. Tumour necrosis factor microsatellites and HLA-DRB1*, HLA-DQA1*, and HLA-DQB1* alleles in Peruvian patients with rheumatoid arthritis. Ann Rheum Dis 2001;60:791-5. [ Links ]
82. Rodriguez-Carreon AA, Zuniga J, Hernandez-Pacheco G, Rodriguez-Perez JM, Perez-Hernandez N, Montes de Oca JV, et al. Tumor necrosis factor-alpha -308 promoter polymorphism contributes independently to HLA alleles in the severity of rheumatoid arthritis in Mexicans. J Autoimmun 2005;24:63-8. [ Links ]
83. Cuenca J, Cuchacovich M, Perez C, Ferreira L, Aguirre A, Schiattino I, et al. The -308 polymorphism in the tumour necrosis factor (TNF) gene promoter region and ex vivo lipopolysaccharide-induced TNF expression and cytotoxic activity in Chilean patients with rheumatoid arthritis. Rheumatology (Oxford) 2003;42:308-13. [ Links ]
84. Singal DP, Ye M, Qiu X, DSouza M. Polymorphisms in the TAP2 gene and their association with rheumatoid arthritis. Clin Exp Rheumatol 1994;12:29-33. [ Links ]
85. Yu MC, Huang CM, Wu MC, Wu JY, Tsai FJ. Association of TAP2 gene polymorphisms in Chinese patients with rheumatoid arthritis. Clin Rheumatol 2004;23:35-9. [ Links ]
86. Collado L, Rueda B, Caliz R, Torres B, Garcia A, Nunez-Roldan A, et al. Lack of association between the I kappa BL promoter polymorphism and rheumatoid arthritis. Arthritis Rheum 2004;50:2032-3. [ Links ]
87. Okamoto K, Makino S, Yoshikawa Y, Takaki A, Nagatsuka Y, Ota M, et al. Identification of I kappa BL as the second major histocompatibility complex-linked susceptibility locus for rheumatoid arthritis. Am J Hum Genet 2003;72:303-12. [ Links ]
88. Dieude P, Osorio J, Petit-Teixeira E, Moreno S, Garnier S, Cailleau-Moindrault S, et al. A TNFR1 genotype with a protective role in familial rheumatoid arthritis. Arthritis Rheum 2004;50:413-9. [ Links ]
89. Zapico I, Coto E, Rodriguez A, Alvarez C, Torre JC, Alvarez V. A DNA polymorphism at the alpha2-macroglobulin gene is associated with the severity of rheumatoid arthritis. J Rheumatol 2000;27:2308-11. [ Links ]
90. Swanberg M, Lidman O, Padyukov L, Eriksson P, Akesson E, Jagodic M, et al. MHC2TA is associated with differential MHC molecule expression and susceptibility to rheumatoid arthritis, multiple sclerosis and myocardial infarction. Nat Genet 2005;37:486-94. [ Links ]
91. Gonzalez-Gay MA, Llorca J, Sanchez E, Lopez-Nevot MA, Amoli MM, Garcia-Porrua C, et al. Inducible but not endothelial nitric oxide synthase polymorphism is associated with susceptibility to rheumatoid arthritis in northwest Spain. Rheumatology (Oxford) 2004;43:1182-5. [ Links ]
92. Frances R, Munoz C, Zapater P, Uceda F, Gascon I, Pascual S, et al. Bacterial DNA activates cell mediated immune response and nitric oxide overproduction in peritoneal macrophages from patients with cirrhosis and ascites. Gut 2004;53:860-4. [ Links ]
93. Baugh JA, Chitnis S, Donnelly SC, Monteiro J, Lin X, Plant BJ, et al. A functional promoter polymorphism in the macrophage migration inhibitory factor (MIF) gene associated with disease severity in rheumatoid arthritis. Genes Immun 2002;3:170-6. [ Links ]
94. Barton A, Lamb R, Symmons D, Silman A, Thomson W, Worthington J, et al. Macrophage migration inhibitory factor (MIF) gene polymorphism is associated with susceptibility to but not severity of inflammatory polyarthritis. Genes Immun 2003;4:487-91. [ Links ]
95. Donn RP, Shelley E, Ollier WE, Thomson W. A novel 5'-flanking region polymorphism of macrophage migration inhibitory factor is associated with systemic-onset juvenile idiopathic arthritis. Arthritis Rheum 2001;44:1782-5. [ Links ]
96. Martinez A, Valdivia A, Pascual-Salcedo D, Lamas JR, Fernandez-Arquero M, Balsa A, et al. PADI4 polymorphisms are not associated with rheumatoid arthritis in the Spanish population. Rheumatology (Oxford) 2005;44:1263-6. [ Links ]
97. Gregersen PK, Batliwalla F. PTPN22 and rheumatoid arthritis: gratifying replication. Arthritis Rheum 2005;52:1952-5. [ Links ]
98. Mori M, Yamada R, Kobayashi K, Kawaida R, Yamamoto K. Ethnic differences in allele frequency of autoimmune-disease-associated SNPs. J Hum Genet 2005;50:264-6. [ Links ]
99. Stastny P. Association of the B-cell alloantigen DRw4 with rheumatoid arthritis. N Engl J Med 1978;298:869-71. [ Links ]
100. Horton R, Wilming L, Rand V, Lovering RC, Bruford EA, Khodiyar VK, et al. Gene map of the extended human MHC. Nat Rev Genet 2004;5:889-99. [ Links ]
101. Gregersen PK, Silver J, Winchester RJ. The shared epitope hypothesis. An approach to understanding the molecular genetics of susceptibility to rheumatoid arthritis. Arthritis Rheum 1987;30:1205-13. [ Links ]
102. Robinson J, Waller MJ, Parham P, de Groot N, Bontrop R, Kennedy LJ, et al. IMGT/HLA and IMGT/MHC: sequence databases for the study of the major histocompatibility complex. Nucleic Acids Res 2003;31:311-4. [ Links ]
103. Dessen A, Lawrence CM, Cupo S, Zaller DM, Wiley DC. X-ray crystal structure of HLA-DR4 (DRA*0101, DRB1*0401) complexed with a peptide from human collagen II. Immunity 1997;7:473-81. [ Links ]
104. Reviron D, Perdriger A, Toussirot E, Wendling D, Balandraud N, Guis S, et al. Influence of shared epitope-negative HLA-DRB1 alleles on genetic susceptibility to rheumatoid arthritis. Arthritis Rheum 2001;44:535-40. [ Links ]
105. Roudier J. Association of MHC and rheumatoid arthritis. Association of RA with HLA-DR4: the role of repertoire selection. Arthritis Res 2000;2:217-20. [ Links ]
106. Anaya JM. Genes y artritis reumatoidea. Rev Colomb Reumatol 1999;6:240-50. [ Links ]
107. Cardenas C, Villaveces JL, Bohorquez H, Llanos E, Suarez C, Obregon M, et al. Quantum chemical analysis explains hemagglutinin peptide-MHC Class II molecule HLA-DRbeta1*0101 interactions. Biochem Biophys Res Commun 2004;323:1265-77. [ Links ]
108. Cardenas C, Villaveces JL, Suarez C, Obregon M, Ortiz M, Patarroyo ME. A comparative study of MHC Class-II HLA-DRbeta1*0401-Col II and HLA-DRbeta1*0101-HA complexes: a theoretical point of view. J Struct Biol 2005;149:38-52. [ Links ]
109. Cardenas C, Ortiz M, Balbin A, Villaveces JL, Patarroyo ME. Allele effects in MHC-peptide interactions: a theoretical analysis of HLA-DRbeta1*0101-HA and HLA-DRbeta1*0401-HA complexes. Biochem Biophys Res Commun 2005;330:1162-7. [ Links ]
110. Newton JL, Harney SM, Wordsworth BP, Brown MA. A review of the MHC genetics of rheumatoid arthritis. Genes Immun 2004;5:151-7. [ Links ]
111. Gorman JD, Criswell LA. The shared epitope and severity of rheumatoid arthritis. Rheum Dis Clin North Am 2002;28:59-78. [ Links ]
112. Ou D, Mitchell LA, Tingle AJ. A new categorization of HLA DR alleles on a functional basis. Hum Immunol 1998;59:665-76. [ Links ]
113. Vos K, van der Horst-Bruinsma IE, Hazes JM, Breedveld FC, le Cessie S, Schreuder GM, et al. Evidence for a protective role of the human leukocyte antigen class II region in early rheumatoid arthritis. Rheumatology (Oxford) 2001;40:133-9. [ Links ]
114. Zanelli E, Gonzalez-Gay MA, David CS. Could HLA-DRB1 be the protective locus in rheumatoid arthritis? Immunol Today 1995;16:274-8. [ Links ]
115. van der Helm-van Mil AH, Huizinga TW, Schreuder GM, Breedveld FC, de Vries RR, Toes RE. An independent role of protective HLA class II alleles in rheumatoid arthritis severity and susceptibility. Arthritis Rheum 2005;52:2637-44. [ Links ]
116. Debaz H, Olivo A, Vazquez Garcia MN, de la Rosa G, Hernandez A, Lino L, et al. Relevant residues of DRbeta1 third hypervariable region contributing to the expression and to severity of rheumatoid arthritis (RA) in Mexicans. Hum Immunol 1998;59:287-94. [ Links ]
117. Ruiz-Morales JA, Vargas-Alarcon G, Flores-Villanueva PO, Villarreal-Garza C, Hernandez-Pacheco G, Yamamoto-Furusho JK, et al. HLA-DRB1 alleles encoding the "shared epitope" are associated with susceptibility to developing rheumatoid arthritis whereas HLA-DRB1 alleles encoding an aspartic acid at position 70 of the beta-chain are protective in Mexican Mestizos. Hum Immunol 2004;65:262-9. [ Links ]
118. Clavijo Acero E, Molina Ramos T, Bernal Gómez A, Pinilla Roa A. Incidencia del HLA en artritis reumatoidea (resumen). Acta Med Colomb 1990;15(suppl):267. [ Links ]
119. Yunis JJ, Salazar M, Deulofeut R. DRB1*0404 allele and rheumatoid arthritis in the Guambiano amerindian tribe of Colombia. Arthritis Rheum 1994;38(suppl):4. [ Links ]
120. Avila LM, Garzón AM, Talero OH, Reyes E, Valle RR. Estudio inmunogenético en pacientes colombianos con artritis reumatoide. Rev Colomb Reumatol 1995;2:90. [ Links ]
121. Anaya JM, Correa P, Mantilla RD, Jiménez F, Kuffner T, McNicholl JM. Prevalencia y severidad de la artritis reumatoidea en población afrocolombiana de Quibdó. Acta Med Colomb 1998;23:322-33. [ Links ]
122. Anaya JM, Correa PA, Mantilla RD, Arcos-Burgos M. Rheumatoid arthritis association in Colombian population is restricted to HLA-DRB1*04 QRRAA alleles. Genes Immun 2002;3:56-8. [ Links ]
123. Avila-Portillo LM, Vargas-Alarcon G, Andrade F, Alarcon-Segovia D, Granados J. Linkage disequilibrium of HLA-DR3 and HLA-DR4 with HLA-B alleles in Mexican patients with rheumatoid arthritis. Clin Exp Rheumatol 1994;12:497-502. [ Links ]
124. Gonzalez A, Nicovani S, Massardo L, Aguirre V, Cervilla V, Lanchbury JS, et al. Influence of the HLA-DR beta shared epitope on susceptibility to and clinical expression of rheumatoid arthritis in Chilean patients. Ann Rheum Dis 1997;56:191-3. [ Links ]
125. Citera G, Padulo LA, Fernandez G, Lazaro MA, Rosemffet MG, Maldonado Cocco JA. Influence of HLA-DR alleles on rheumatoid arthritis: susceptibility and severity in Argentine patients. J Rheumatol 2001;28:1486-91. [ Links ]
126. Angulo J, Miraval T, Ponce de León H, Reveille J. Epítope reumatoide y alelo HLA-DRB1*0404 se asocian con susceptibilidad para artritis reumatoide en mestizos peruanos. Alelo HLA-DRB1*1402 en duda y la combinación maligna *0401/0404 ausente. Rev Peru Reumatol 2003;9:26-32. [ Links ]
127. de Vries N, van Elderen C, Tijssen H, van Riel PL, van de Putte LB. No support for HLA-DQ encoded susceptibility in rheumatoid arthritis. Arthritis Rheum 1999;42:1621-7. [ Links ]
128. Ota M, Katsuyama Y, Kimura A, Tsuchiya K, Kondo M, Naruse T, et al. A second susceptibility gene for developing rheumatoid arthritis in the human MHC is localized within a 70-kb interval telomeric of the TNF genes in the HLA class III region. Genomics 2001;71:263-70. [ Links ]
129. Jawaheer D, Li W, Graham RR, Chen W, Damle A, Xiao X, et al. Dissecting the genetic complexity of the association between human leukocyte antigens and rheumatoid arthritis. Am J Hum Genet 2002;71:585-94. [ Links ]
130. Zanelli E, Jones G, Pascual M, Eerligh P, van der Slik AR, Zwinderman AH, et al. The telomeric part of the HLA region predisposes to rheumatoid arthritis independently of the class II loci. Hum Immunol 2001;62:75-84. [ Links ]
131. Kilding R, Iles MM, Timms JM, Worthington J, Wilson AG. Additional genetic susceptibility for rheumatoid arthritis telomeric of the DRB1 locus. Arthritis Rheum 2004;50:763-9. [ Links ]
132. Kilding R, Wilson AG. Mapping of a novel susceptibility gene for rheumatoid arthritis in the telomeric MHC region. Cytokine 2005;32:71-5. [ Links ]
133. Hajeer AH, Hutchinson IV. Influence of TNFalpha gene polymorphisms on TNFalpha production and disease. Hum Immunol 2001;62:1191-9. [ Links ]
134. Ioannidis JP. Why most published research findings are false. PLoS Med 2005;2:e124. [ Links ]
135. Gregersen PK. Pathways to gene identification in rheumatoid arthritis: PTPN22 and beyond. Immunol Rev 2005;204:74-86. [ Links ]
136. Weir BS, Hill WG. Estimating F-statistics. Annu Rev Genet 2002;36:721-50. [ Links ]
137. Hattersley AT, McCarthy MI. What makes a good genetic association study? Lancet 2005;366:1315-23. [ Links ]
138. Rosenberg NA, Li LM, Ward R, Pritchard JK. Informativeness of genetic markers for inference of ancestry. Am J Hum Genet 2003;73:1402-22. [ Links ]
139. Choudhry S, Coyle NE, Tang H, Salari K, Lind D, Clark SL, et al. Population stratification confounds genetic association studies among Latinos. Hum Genet 2006;118:652-64. [ Links ]
140. Cadena J, Vinaccia S, Pérez A, Rico MI, Hinojosa R, Anaya JM. The impact of disease activity on the quality of life, mental health status, and family dysfunction in colombian patients with rheumatoid arthritis. J Clin Rheumatol 2003;9:142-50. [ Links ]
141. Pineda-Tamayo R, Arcila G, Restrepo P, Anaya JM. Impact of cardiovascular illness on hospitalization costs in patients with rheumatoid arthritis. Biomedica 2004;24:366-74. [ Links ]
142. Schroeder D, Ladikas M, Schuklenk U, Lasen Diaz CL, Kleinsmidt A, Alvarez-Castillo F, et al. Sharing the benefits of genetic research. BMJ 2005;331: 1351-2. [ Links ]














