Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.27 no.2 Bogotá Apr./June 2007
Polimorfismos del gen pfmdr1 en muestras clínicas de Plasmodium falciparum y su relación con la respuesta terapéutica a antipalúdicos y paludismo grave en Colombia
Paula Montoya, Alberto Tobón, Silvia Blair, Jaime Carmona, Amanda Maestre
Grupo de Malaria, Sede de Investigación Universitaria, Universidad de Antioquia, Medellín, Colombia
Recibido: 04/07/06; aceptado: 27/02/07
Introducción.
El gen pfmdr1 de Plasmodium falciparum se describió como un gen de resistencia a diversos antipalúdicos. Sin embargo, no se han estudiado el papel de su polimorfismo en la respuesta terapéutica al tratamiento con antipalúdicos en Colombia, ni su relación con paludismo grave.Objetivos. Este trabajo determinó la asociación entre los polimorfismos Asn86Tir y Asp1246Tir del gen pfmdr1 con la respuesta terapéutica a cloroquina, amodiaquina y mefloquina, en tres municipios antioqueños, y la asociación de estos polimorfismos con paludismo grave en muestras de pacientes del municipio de Tumaco.
Materiales y métodos. Los polimorfismos del gen pfmdr1 se determinaron mediante amplificación de ácidos nucleicos y análisis con enzimas de restricción.
Resultados. El alelo silvestre Asn86 se encontró en 97% (137/141) de las muestras en el estudio de respuesta terapéutica a cloroquina, amodiaquina y mefloquina; no se observó ninguna asociación entre su presencia y la falla terapéutica como sí lo reportan otros autores. El alelo 1246Tir se encontró en una alta proporción en el estudio de respuesta terapéutica, tanto en las muestras del día cero como en los del día de la falla después del tratamiento con los antipalúdicos.
Conclusiones. Los polimorfismos 86Tir y 1246Tir en el gen pfmdr1 no son útiles como factores de predicción de falla terapéutica o paludismo grave en los municipios estudiados. Este estudio describe por primera vez la presencia del alelo 86Tir en cuatro muestras clínicas de Suramérica.
Palabras clave: Plasmodium falciparum/efectos de drogas, antipalúdicos/uso terapéutico, malaria, cloroquina, amodiaquina, mefloquina.
Polymorphisms of the pfmdr1 gene in field samples of Plasmodium falciparum and their association with therapeutic response to antimalarial drugs and severe malaria in Colombia
Introduction. The pfmdr1 gene of Plasmodium falciparum has been described as a gene conferring resistance to several antimalarial drugs. In particular, polymorphisms on specific codons have been associated with resistance and treatment failure with cloroquine, amodiaquine and mefloquine. However, the role of these polymorphisms in treatment response to antimalarials remains unexplored in Colombia. Furthermore, the relationship of these polymorphisms to severe malaria is unknown.
Objective. This work studied the association of the Asn86Tyr and Asp1246Tyr pfmdr1 polymorphisms with response to cloroquine, amodiaquine and mefloquine treatment in three municipalities of Antioquia, and severe malaria cases from the municipality Tumaco.
Materials and methods.The polymorphisms were assessed by nucleic acid amplification followed by restriction length polymorphism analysis.
Results. The wild-type codon Asn86 was detected in 97% of the clinical samples from the treatment response study. No association was detected between this polymorphism and treatment failure to the three antimalarials administered. The 1246Tyr polymorphism was detected with a higher frequency in the samples from Antioquia 92% (130/141) than in those from Tumaco 22% (20/89). However, again, no association was found between the presence of a specific polymorphism and the presence of severe malaria in the municipality of Tumaco.
Conclusions. The 86Tyr and 1246Tyr polymorphisms of the pfmdr1 gene are not useful as predictors of treatment failure or severe malaria in the municipalities studied. In addition, we report for the first time, the presence of the mutant codon 86Tyr in field samples in South America.
Key words: Plasmodium falciparum/drug effects, antimalarial/therapeutic use, malaria, chloroquine, amodiaquine, mefloquine.
Entre las especies de Plasmodium, Plasmodium falciparum produce la enfermedad más seria y la tasa de mortalidad por paludismo más alta en el mundo. Esta especie ha desarrollado resistencia a la mayoría de los medicamentos disponibles contra el paludismo, lo que resulta en una gran frecuencia de fallas terapéuticas. Este fenómeno contribuye en gran medida a que el paludismo sea uno de los principales problemas de salud pública en las regiones tropicales de todo el mundo (1). En el 2005 se registraron en Colombia 107.866 casos de paludismo, de los cuales, 36.190 fueron producidos por P. falciparum (2).
Al estudiar la resistencia a los antipalúdicos en P. falciparum, se describieron algunos polimor-fismos en diversos genes del parásito; por ejemplo, en los genes dhfr (dihydrofolate reductase) y dhps (dihydropteroate synthase), que codifican las enzimas blanco de la pirimetamina y la sulfadoxina, respectivamente, y así confieren resistencia a estos medicamentos (3,4). Igualmente, los genes pfcrt (P. falciparum chloroquine resistance transporter) y pfmdr1 (P. falciparum multidrug resistance 1), que codifican proteínas trans-membrana de transporte en la vacuola digestiva del parásito, se relacionan con resistencia y falla terapéutica a la cloroquina, en el caso de pfcrt (5-7), y con resistencia a cloroquina y a diversos medicamentos, en el caso de pfmdr1 (8,9).
No obstante, algunos autores reportan falta de asociación entre la falla terapéutica o la resistencia y la presencia de mutaciones en el gen pfmdr1 (10-14). La mutación 86Tir en pfmdr1 asociada con resistencia a la cloroquina, también puede incrementar la sensibilidad a mefloquina y halofantrina (15), mientras que la presencia del alelo silvestre Asn86 se ha correlacionado con resistencia a la mefloquina (16,17).
Las mutaciones en los codones 1034, 1042 y, principalmente, en el 1246, parecen incrementar la sensibilidad a mefloquina y halofantrina (9). Esta última mutación también se describió en muestras resistentes a la cloroquina en Brasil (18), pero no se encontró en muestras resistentes a la cloroquina de Asia y África (16,19).
Los hallazgos descritos anteriormente sugieren que los mecanismos de resistencia a la cloroquina y mefloquina son inversos y que los polimorfismos en pfmdr1 parecen modular la respuesta a medicamentos como cloroquina, mefloquina y halofantrina. Lo anterior confirma la necesidad de realizar estudios enfocados a aclarar el papel de los polimorfismos en la resistencia y la falla terapéutica a compuestos antipalúdicos sin relación estructural, como lo son cloroquina y mefloquina.
En Colombia, Blair y colaboradores, en 1998 y 2001 (20-22), reportaron altos y crecientes índices de falla terapéutica a los antipalúdicos de primera línea en Antioquia, alcanzando entre 77% y 87% con cloroquina, 22% y 26% con sulfadoxina/pirimetamina y 12% y 22% con la combinación cloroquina-sulfadoxina/pirimetamina; mientras que en la Costa Pacífica, González y colaboradores reportaron el problema con la utilización de amodiaquina y sulfadoxina/pirimetamina (23).
Los municipios de Turbo, El Bagre, Zaragoza y Tumaco son zonas de alta endemicidad de paludismo por P. falciparum, pero con diferente índice parasitario anual, así como diferentes condiciones socioeconómicas y epidemiológicas (24). En estas regiones no se conocen las características genotípicas de los parásitos con respecto a genes de resistencia como el pfmdr1, ni su posible relación con la respuesta terapéutica a los antipalúdicos.
Teniendo en cuenta los datos anteriores, planteamos el presente trabajo con el fin de conocer las frecuencias de alelos mutantes y silvestres de los codones 86 y 1246 de pfmdr1 en muestras de Plasmodium falciparum, su relación con la respuesta terapéutica a medicamentos como cloroquina, amodiaquina y mefloquina en tres municipios de Antioquia, y la relación de su presencia con el desarrollo de paludismo grave en el municipio de Tumaco, Nariño.
Para el desarrollo del presente estudio se usaron muestras de P. falciparum de tres municipios antioqueños provenientes de pacientes con evaluación de respuesta terapéutica a cloroquina, amodiaquina o mefloquina, y muestras de pacientes con paludismo complicado y no complicado del municipio de Tumaco. Estas muestras se emplearon para realizar el primer estudio en Colombia de caracterización de los polimorfismos Asn86Tir y Asp1246Tir en el gen pfmdr1 de muestras clínicas de estas regiones.
Materiales y métodos
Poblaciones de estudio
El trabajo correspondió a un estudio de tipo descriptivo, transversal y prospectivo. Se llevó a cabo con dos poblaciones de estudio, una para evaluar los polimorfismos frente a la respuesta terapéutica y la otra para evaluar su relación con paludismo grave.
La población número uno la conformaron las muestras de pacientes con paludismo no compli-cado que recibieron cloroquina, amodiaquina o mefloquina en monoterapia, obtenidos de estudios de respuesta terapéutica realizados por el Grupo Malaria de la Universidad de Antioquia empleando el protocolo de la Organización Mundial de la Salud de 1998 (25), en los municipios de Zaragoza, Turbo y El Bagre.
La población número dos la conformaron las muestras de pacientes con malaria complicada y no complicada de un estudio de casos y controles en el municipio de Tumaco.
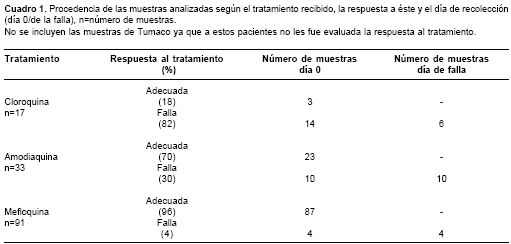
El estudio de respuesta terapéutica a la cloroquina comprendió la evaluación de 17 pacientes en los municipios de Turbo y Zaragoza (8 en Turbo y 9 en Zaragoza). El diseño de la muestra del estudio se realizó como se describe en el trabajo de eficacia terapéutica realizado por Blair y colaboradores en el 2003 (22). Las muestras se recolectaron entre los meses de febrero y abril de 2002.
Los estudios de respuesta terapéutica a amodiaquina y mefloquina evaluaron 33 y 91 pacientes, respectivamente, en los municipios de Turbo y El Bagre. El tamaño de la muestra se calculó por el sistema de calidad de lotes según el protocolo de la Organización Mundial de la Salud de 1998 (25). El número de pacientes evaluados con amodiaquina fue de 33 (21 en Turbo y 12 en El Bagre). Las muestras se recolectaron entre los meses de octubre de 2003 y marzo de 2004. El estudio de respuesta terapéutica a mefloquina evaluó 91 pacientes (44 en Turbo y 47 en El Bagre); las muestras se recolectaron entre octubre de 2002 y junio de 2003.
La investigación sobre paludismo complicado por P. falciparum en el municipio de Tumaco se trató de un estudio de casos (pacientes con malaria grave o complicada) y controles (pacientes con malaria no complicada) en el que se captaron 89 pacientes (30 casos y 59 controles); las muestras se recolectaron entre noviembre de 2002 y julio de 2003.
Se analizaron 250 muestras, de las cuales, 230 provenían del día 0.
Diagnóstico parasitológico
El diagnóstico de paludismo se realizó por medio de gota gruesa y extendido de sangre periférica, los cuales se colorearon con las coloraciones de Field y Giemsa, respectivamente. Un microsco-pista experto en malaria hizo el recuento de parásitos, número de trofozoítos jóvenes (anillos) presentes en 200 leucocitos, y la parasitemia se calculó teniendo en cuenta una constante de 8.000 leucocitos/mm3 y se expresó en parásitos/mm3 de sangre (26).
Clasificación de la respuesta terapéutica
Se clasificó en respuesta clínica adecuada o falla terapéutica (tardía o temprana) con base en el protocolo estándar de la Organización Mundial de la Salud, 1998 (25). Se hizo un seguimiento clínico y parasitológico en los días 1, 2, 3, 7, 14, 21 y 28 después de haber iniciado el tratamiento y, si era necesario, los pacientes podían consultar cualquier día fuera de los mencionados. Los medicamentos se administraron de acuerdo con el peso, por vía oral, según lo establecido por el Ministerio de Salud de Colombia (26).
Criterios de clasificación para paludismo grave
Según la Organización Mundial de la Salud, en un paciente con parasitemia asexual por P. falciparum, que no tenga otra causa para sus síntomas y presente uno o más de los siguientes síntomas o signos, se configura el diagnóstico de paludismo grave (25): paludismo cerebral (coma profundo), anemia grave (Hb<5 g/dl), insuficiencia renal (creatinina sérica>3,0 mg/dl), colapso circulatorio (presión arterial sistólica en adultos <70 mm Hg, <50 mm Hg en niños menores de cinco años de edad), edema pulmonar, hemorragia espontánea y coagulación intravascular diseminada, dificultad respiratoria, acidemia o acidosis, hiperparasitemia (>5% de los eritrocitos infectados), ictericia y alteración hepática (bilirrubina sérica total>3mg/dl). Dos de los pacientes de la población fueron incluidos en el estudio luego de verificar que cumplían alguno de estos requisitos.
Obtención y procesamiento de la muestra
Las muestras de sangre se obtuvieron mediante punción digital y se depositaron en láminas portaobjeto para la gota gruesa y extendido de sangre periférica, y en círculos de papel de filtro Whatman #3 para los análisis moleculares. A los pacientes que presentaron falla terapéutica durante los 28 días de seguimiento al tratamiento, se les recolectó una segunda muestra de sangre el día de la falla. El papel de filtro se dejó secar al aire libre y se almacenó a 4°C en bolsa plástica; posteriormente, las muestras se transportaron al laboratorio de biología molecular del Grupo Malaria y allí se almacenaron a -20°C, hasta su análisis.
Detección de polimorfismos en el gen pfmdr1 mediante reacción en cadena de la polimerasa anidada- análisis con enzimas de restricción
La extracción de ácidos nucleicos a partir de muestras de sangre en papel filtro se hizo empleando el método de Chelex (27). El ADN se repartió en microtubos y se almacenó a -20°C hasta su posterior utilización.
Para determinar la presencia de los polimorfismos en los codones 86 y 1246, se amplificaron las regiones del gen pfmdr1 que los contienen según el protocolo de Djimdé et al. 2001 (6). Posterior-mente, los fragmentos amplificados fueron digeridos con enzimas de restricción. Por esta tecnología se identificaron los polimorfismos Asn86Tir que se presentan por el cambio de una adenina (A) por una timina (T) en el nucleótido 754 y Asp1246Tir que se da por el cambio de una guanina (G) por una tirosina (T) en el nucleótido 4234. Para el codón 86 se utilizaron las enzimas de restricción Afl-III (New England Biolabs, Beverly, MA) y XapI (Fermentas, USA). La enzima Bgl-II (Fermentas, USA) se empleó para el codón 1246.
El análisis se realizó según el patrón de bandas obtenido así:
Corte con la enzima Afl-III: codón silvestre Asn86, se observa una banda de 291 pb; codón mutado 86Tir, se observan dos fragmentos, uno de 165 y otro de 126 pb.
Corte con la enzima Xapl: codón silvestre Asn86, se observan dos fragmentos, uno de 165 y otro de 126 pb. Codón mutado 86Tir, se observa una banda de 291 pb.
Corte con la enzima Bgl-II: codón silvestre Asp1246, se observan dos fragmentos, uno de 113 pb y otro de 90 pb. Codón mutado 1246Tir, se observa una banda de 203 pb.
En todos los casos, la observación de tres bandas corresponde con la presencia de los dos alelos simultáneamente.
Como controles se emplearon las cepas K1 y 3D7. La cepa K1 es 86Tir y Asp1246, mientras que la 3D7 es Asn86. Para el control de la 1246Tir, se utilizó un aislamiento de un paciente de Tumaco cuya secuencia se había obtenido previamente. Los fragmentos obtenidos se visualizaron en geles de agarosa (SeaKem) al 2%-2,5% coloreados con bromuro de etidio a una concentración de 0,5 mg/ml y las bandas se visualizaron con luz ultravioleta y se analizaron empleando el programa Quantity One (Bio-Rad).
Análisis de resultados
La información obtenida se procesó con el programa estadístico SPSS 10.0 (28). Se determinaron las frecuencias absolutas y relativas de cada uno de los polimorfismos estudiados por tratamiento y por municipio. Para determinar la asociación entre la presencia de los polimorfismos y la respuesta terapéutica (falla o respuesta clínica adecuada) o la presencia de paludismo complicado, se emplearon tablas de dos por dos y se excluyeron las muestras de pacientes que presentaron más de un alelo. Se utilizó la prueba de ji al cuadrado de Pearson y, cuando hubo valores esperados menores de cinco en las celdas, se utilizó la prueba exacta de Fisher (29). Se consideró una diferencia estadísticamente significativa cuando el valor de p fue <0,05.
Consideraciones éticas
Para la inclusión en los estudios de investigación de eficacia terapeútica y paludismo complicado iniciales, se obtuvo el consentimiento informado de los pacientes o sus parientes en caso de tratarse de menores de edad. En el consentimiento se especificaba la utilización del material donado por ellos. Todos los estudios recibieron el aval del Comité de Ética del Centro de Investigaciones Médicas de la Facultad de Medicina de la Universidad de Antioquia. Los procedimientos se clasificaron como de riesgo mínimo.
Resultados
Descripción de las poblaciones
La población uno estaba constituida por 141 muestras del estudio de respuesta terapéutica, de las cuales, 59 provenían de El Bagre, 9 de Zaragoza y 73 de Turbo. Los 89 pacientes de la población dos provenían exclusivamente de Tumaco.
La edad en años de las poblaciones evaluadas fluctuó entre 2 y 82; la mayoría residía en zona rural. El rango de parasitemia en la población uno se encontró entre 320 y 41.300 parásitos por mm3, mientras que en la población dos este rango fue de 240 a 298.000 parásitos por mm3.
Respuesta terapéutica a cloroquina, amodiaquina y mefloquina
En los trabajos de evaluación de la respuesta terapéutica que suministraron las muestras para este estudio, se encontró falla terapéutica de 82% (14/17) a cloroquina y de 30% (10/33) a amodiaquina, mientras que la falla a mefloquina fue de 4% (4/91). Los detalles acerca de la respuesta al tratamiento y el número de muestras analizadas se observan en el cuadro 1.
Polimorfismo Asn86Tir en pfmdr1
La evaluación de la mutación Asn86Tir y su relación con la respuesta terapéutica se analizaron en 161 muestras, 141 del día cero y 20 del día de la falla. Para la evaluación de la mutación Asn86Tir y su asociación con paludismo grave, se evaluaron 89 muestras del municipio de Tumaco.
El alelo Asn86 estuvo presente en 97,2% de las muestras del día cero (137/141) en los estudios de respuesta terapéutica a cloroquina, amodiaquina y mefloquina, y la mutación 86Tir se encontró sólo en una muestra (0,7%) o, en combinación con el alelo silvestre (86Asn/Tir), en el 2,1% de ellas (3/141). Las cuatro muestras que tenían el alelo mutante corresponden a pacientes que presentaron una respuesta adecuada al tratamiento con mefloquina en el municipio de Turbo. Las 20 muestras del día de la falla presentaron Asn86 (codón silvestre). En el trabajo de paludismo complicado el alelo Asn86 se encontró en el 100% de las muestras. Globalmente, el alelo Asn86 se observó en el 98,4% de las muestras, 86Tyr estaba en el 0,4%, mientras que un 1,2% de las muestras tenían una combinación de estos dos polimorfismos.
Debido a la alta proporción del alelo silvestre en las muestras del estudio, no se pudo establecer ninguna asociación entre la presencia de alelos (mutado o silvestre) y la respuesta terapéutica a cada uno de los esquemas de tratamiento con cloroquina, amodiaquina y mefloquina. Tampoco se encontró ninguna asociación entre paludismo complicado y la presencia de polimorfismos en el codón 86.
Al analizar la presencia del polimorfismo Asn86Tir y la procedencia de la muestra, se encontró en el municipio de Turbo el alelo silvestre Asn86 en el 94,5% de ellas (69/73), mientras que el alelo mutado 86Tir se encontró, solo o en combinación con el silvestre (86 Asn/Tir), en el 5,5% (4/73). En el Bajo Cauca y en Tumaco el alelo silvestre Asn86 estuvo presente en el 100% de las muestras.
Polimorfismo Asp1246Tir en pfmdr1
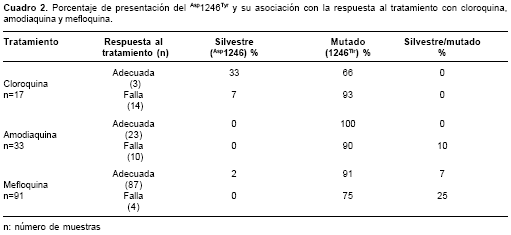
El día cero, el 66,7% (2/3) de las muestras obtenidas de pacientes que recibieron cloroquina y tuvieron respuesta clínica adecuada, presentaron el alelo 1246Tir. Entretanto, el 92,8% (13/14) de las muestras de pacientes con falla terapéutica precoz o tardía presentaron el alelo 1246Tir.
En las 33 muestras del día 0 del grupo que recibió amodiaquina, se encontró el alelo 1246Tir indepen-dientemente de la respuesta al tratamiento. Sin embargo, un paciente con falla precoz portaba tanto el alelo silvestre como el mutado. Es importante anotar que en la muestra obtenida de este paciente el día de la falla se observó exclusivamente la presencia del alelo 1246Tir. Las nueve muestras restantes del día de la falla portaban 1246Tir.
En el grupo de mefloquina, la distribución fue más heterogénea. En las muestras de pacientes que tuvieron respuesta clínica adecuada, se encontró 1246Tir en el 90,8% (79/87), Asp1246 en el 2,3% (2 /87) y ambos alelos en el 6,9% (6/87). Un paciente con falla precoz a mefloquina portaba los dos alelos (mutado y silvestre) del codón 1246, tanto el día cero como el día de la falla. La falla tardía a mefloquina sólo se presentó en tres pacientes y todas las muestras tenían 1246Tir tanto el día cero como el día de la falla.
En este estudio no se encontró una asociación significativa entre la presencia del alelo 1246Tir y la falla terapéutica al tratamiento con cloroquina p=0,33, amodiaquina p=0,30 o mefloquina p=0,92. Tampoco se encontró asociación entre la presencia del codón silvestre Asp1246 y una respuesta clínica adecuada a cualquiera de los medicamentos empleados (cuadro 2).
Polimorfismo Asp1246Tir en el gen pfmdr1 y paludismo grave
El alelo silvestre Asp1246 estuvo presente en gran proporción, tanto en las muestras de pacientes con paludismo complicado, 76,7% (23/30), como en los no complicados, 69,5% (41/59). El alelo mutado 1246Tir se encontró en 23,3% (7/30) de los pacientes con paludismo complicado y en 22% (13/59) de los no complicados. Ambos alelos se encontraron en 8,5% (5/59) de los pacientes no complicados. No se encontró ninguna asociación (p=0,94) entre 1246Tir y la presencia de paludismo complicado o entre Asp1246 y el éxito del tratamiento con los antimaláricos utilizados en Tumaco.
Distribución del polimorfismo Asp1246Tir del gen pfmdr1 según la procedencia de la muestra
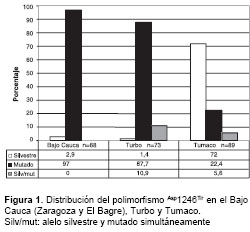
El análisis del codón 1246 en los municipios se hizo a partir de las 230 muestras obtenidas el día cero (141 del estudio de respuesta terapéutica y 89 del estudio de paludismo grave) en las dos poblaciones estudiadas. El alelo 1246Tir predominó en el municipio de Turbo en 87,7% (64/73) de las muestras y, en el bajo Cauca, en 97% (66/68) de las muestras; el porcentaje más bajo se presentó en el municipio de Tumaco: 22,4% (20/89) de las muestras. Por consiguiente, el codón silvestre Asp1246 estuvo presente en una mayor proporción en este municipio; se encontró en 72% (64/89) de las muestras y, en proporción más baja, en los municipios de Turbo, 1,3%, (1/73), y el bajo Cauca, 2,9% (2/68).
La presencia de ambos alelos 1246Asp/Tir se observó en mayor proporción en las muestras de P. falciparum de Turbo, 10,9% (8/73), que en el municipio de Tumaco, 5,6% (5/89), y no se presentó en el bajo Cauca (figura 1).
Discusión
La caracterización de la susceptibilidad in vivo de P. falciparum a los antipalúdicos es de gran importancia, debido a que aporta conocimiento sobre la epidemiología del paludismo, evalúa la eficacia terapéutica de los tratamientos y permite vigilar el desarrollo de la falla al tratamiento. Desde hace 15 años se empezó a utilizar la caracterización molecular de genes de resistencia como un método que, potencialmente, permite vigilar la falla terapéutica y la resistencia de forma más rápida y sencilla que los métodos convencionales, como el ensayo in vitro y la susceptibilidad in vivo.
Algunos de los genes candidatos más frecuentemente utilizados como marcadores de resistencia son dhps y dhfr para sulfadoxina y pirimetamina, respectivamente (4,30); el gen pfcrt, para cloroquina y el gen pfmdr1, para varios medicamentos, entre ellos cloroquina, mefloquina, artemisinina, halofantrina y quinina (9); sin embargo, el papel que juega este último gen en el desarrollo de resistencia o falla terapéutica aun no está definido.
Este es el primer estudio realizado en Colombia para determinar la frecuencia y la relación entre la presencia de los polimorfismos Asn86Tir y Asp1246Tir del gen pfmdr1 en muestras de P. falciparum y la respuesta terapéutica al tratamiento con cloroquina, amodiaquina y mefloquina; igualmente, también es el primer trabajo en valorar estos polimorfismos y su relación con paludismo grave.
En este trabajo no se encontró asociación entre el alelo 86Tir y la falla terapéutica a cloroquina o amodiaquina, lo que confirma lo descrito por Ochong et al., en 2003 (31); sin embargo, otros autores como Duraisingh et al. 1997 encontraron asociación entre la falla a estos medicamentos y el alelo 86Tir (32). Nuestros resultados concuerdan mejor con los obtenidos por Zalis et al., 1998 (18), y Povoa et al., 1998 (33), en estudios in vitro con muestras de Brasil de P. falciparum resistentes a cloroquina y con los realizados por Huaman, en 2004 (34), y Pillai, en 2003 (35), con muestras del Perú, en los que no se encontró este alelo. En conjunto, nuestros resultados y los de otros autores sugieren que 86Tir no se correlaciona en Suramérica con resistencia a la cloroquina o amodiaquina in vitro, ni in vivo.
Con respecto a la frecuencia del alelo silvestre Asn86, mediante este estudio se confirma su presencia en el 100% de las muestras de los municipios de Tumaco, Zaragoza y El Bagre. La alta frecuencia de este alelo silvestre coincide con los estudios realizados en la Amazonia peruana (34) y la brasilera (18,33), muestras resistentes in vitro a cloroquina y sensibles a mefloquina.
Con respecto al municipio de Turbo, también se observó predominio del alelo Asn86, que se encontró en 94,5% (69/73) de las muestras, y la presencia de 86Tir se confirmó en 5,5% (4/73) de las muestras en este municipio.
Esta es la primera vez que se reporta la presencia del alelo mutante para este gen en Suramérica. Esta mutación se encontró en pacientes con respuesta clínica adecuada al tratamiento con mefloquina y, además, no se encontró ninguna asociación entre la presencia del alelo 86Tir y la falla terapéutica a mefloquina o a otro de los esquemas de tratamiento empleados en este estudio.
En conclusión, la gran prevalencia en nuestro estudio del alelo Asn86, tanto en casos de respuesta adecuada como de falla al tratamiento, no nos permite afirmar que la presencia del alelo se relacione con uno u otro evento, como lo informan diversos autores (6,32,36,37). Con respecto a la distribución geográfica de alguno de los polimorfismos, podemos afirmar que, a diferencia de lo reportado en África y Asia, el alelo silvestre es mucho más frecuente en nuestras poblaciones de P. falciparum.
Algunos autores han postulado que la capacidad de eliminar la infección en presencia de alelos mutantes del codón 86 aumenta con la edad en regiones geográficas con alta endemicidad y que, por lo tanto, la inmunidad adaptativa podría jugar un papel central en la respuesta al tratamiento (38). Por lo tanto, nuestros hallazgos podrían explicarse, en parte, por los menores índices de infección por P. falciparum de nuestras poblaciones (en promedio 17,06 x 1.000 habitantes, con base en los datos reportados por el Sivigila para el 2003), cuando se comparan con los de África.
Además, en Antioquia, la recomendación de uso de los antipalúdicos ha sido diferente de la del resto del país. Por ejemplo, la amodiaquina se empleó muchos años antes en Antioquia que a nivel nacional y se han administrado drogas de segunda línea, como la mefloquina, durante periodos de falta de disponibilidad de ciertos medicamentos. El empleo de una amplia variedad de antipalúdicos podría tener un papel en seleccionar la forma mutante del gen, pero la significancia de los fenómenos de endemicidad y de utilización previa de antipalúdicos aun está por ser explorada.
Algunos estudios in vitro sugieren que el alelo mutado 86TIr incrementa la sensibilidad y no la resistencia a la mefloquina y a otros medica-mentos como la halofantrina y la artemisinina (36,39); sin embargo, esta afirmación es inconsistente con el resultado de nuestro estudio, en el cual observamos una respuesta clínica adecuada al tratamiento con mefloquina de 96% y el alelo 86TIr se encontró solo o en combinación con el silvestre en 4,6% de las muestras. La obser-vación de este alelo mutante se realizó en muestras de pacientes con respuestas clínicas adecuadas y representaron 2,8% del total de los pacientes con respuesta clinica adecuada. Nuestros resultados tampoco son coherentes con los de Mawili-Mboumba et al., donde el alelo 86TIr estuvo presente en 100% de las muestras de pacientes gaboneses sensibles a mefloquina (40). En conjunto, estos hallazgos podrían sugerir que el polimorfismo descrito en el codón 86 del gen pfmdr1 no es determinante de la respuesta a la mefloquina.
En Gambia, África, se describió una alta prevalencia del alelo 86TIr el día de la falla después del tratamiento con cloroquina y amodiaquina, presentándose con más alta frecuencia en el grupo de amodiaquina (32). Los autores sugieren que un mecanismo genético común podría mediar la resistencia para estos dos compuestos como consecuencia de su semejanza estructural.
En nuestro estudio no se confirmó esta asociación al observar el alelo silvestre Asn86 tanto en el día cero como en el día de la falla en todas las muestras de los pacientes que recibieron cloroquina y amodiaquina. Esto nos permite sugerir que el mecanismo de resistencia a cloroquina y amodiaquina en los parásitos que circulan en Suramérica puede ser diferente, ya sea al involucrar mutaciones en otros codones o en otros genes (como sería el caso del gen Pfcrt).
En 1998, Povoa y colaboradores (33) sugirieron que el alelo 86TIr podría emplearse como marcador para la detección de cepas importadas de Asia o de África hacia Brasil. Con base en esto se podría pensar que las cuatro muestras que portan el alelo 86TIr en Turbo corresponden a cepas importadas de Asia o África. Alternativamente, su existencia podría explicarse simplemente por un cambio natural inducido por diversos factores de la región (endemicidad y uso de antipalúdicos) que pueden ser explorados más a fondo con microsatélites u otras metodologías.
Todos las muestras del grupo de pacientes que recibieron amodiaquina presentaron el alelo 1246Tir tanto el día cero como el día de la falla, con excepción de un paciente que presentó una infección mixta con ambos polimorfismos (mutado y silvestre) el día cero, pero no el día de la falla, en el cual sólo presentó el alelo mutado 1246Tir. A pesar de no ser significativo, este hallazgo parece confirmar lo reportado por otros autores (32) con respecto a que, bajo la presión de amodiaquina, más que de cloroquina, se pueden seleccionar poblaciones de parásitos que portan alelos mutados, lo que explicaría la mayor frecuencia de 1246Tir en Antioquia que en la Costa Pacífica nariñense. En Antioquia, el uso de amodiaquina se ha recomendado y administrado desde 1985, a diferencia del resto del país, en donde su utilización se inició en 1998. Además, la observación de que el alelo mutado 1246Tir es más frecuente en Antioquia que en Tumaco coincide con la afirmación hecha por Winstanley y col. en 2003 (41), en la que sugieren que en regiones de menor endemicidad de P. falciparum, como en Antioquia, es más probable que se seleccionen y establezcan mutaciones, ya que todos los individuos infectados son sintomáticos y reciben tratamiento, mientras que, en zonas de mayor endemicidad, muchos individuos permanecen asintomáticos debido al desarrollo de inmunidad y una proporción menor podrían recibir tratamiento.
El hallazgo de doble alelo en alguno de los codones evaluados, permite concluir la existencia de infección policlonal en el paciente, es decir, presencia de infección por más de un clon de P. falciparum.
Este fenómeno se observó en tres muestras del día cero, tanto para el codón 86 como para el codón 1246, lo que confirma la existencia de infecciones policlonales o mixtas que no pudieron ser confirmadas por la amplificación de los genes convencionalmente utilizados (MSP1, MSP2 y GLURP), ni se pudo confirmar su posible proceden-cia de otra región, ya que las bandas obtenidas para estos genes fueron iguales a las reportadas en otros estudios realizados por Montoya y col. en Turbo (42). Estos resultados sugieren que la caracterización del gen pfmdr1 podría proporcionar información adicional sobre infecciones mixtas que no se pueden detectar con el empleo de marcadores como MSP1, MSP2 y GLURP.
Algunos autores han reportado aumento en el número de copias del gen pfmdr1 como factor determinante crucial de la resistencia in vitro y de la falla al tratamiento a mefloquina (15,43,44) y su disminución, como determinante en el aumento de la sensibilidad de P. falciparum a múltiples antipalúdicos (45). El estudio de este fenómeno no era el objeto de este estudio, pero, a la luz de nuestros resultados y los de otros autores (46), parece lógico explorar tanto el número de copias como los niveles de expresión del gen en algunas de las muestras.
Sólo se ha realizado un estudio que relaciona el desarrollo de paludismo complicado y polimorfismos en los codones 86 y 1246 de pfmdr1, en el cual se encontraron mutantes en los dos codones en 82% (18/24) de las muestras provenientes de pacientes con paludismo complicado en Kenia (47). En nuestro estudio, el alelo 86Tir no se encontró en ninguno de las 89 muestras de Tumaco (30 de pacientes con paludismo complicado y 59 no complicados), mientras que 1246Tir sólo se encontró en 23% (7/30) de las muestras de pacientes con paludismo complicado.
El análisis de un mayor número de muestras provenientes de pacientes complicados, así como de un grupo de controles en la misma región, nos permitiría confirmar que estos polimorfismos no se relacionan con paludismo grave o complicado en la región estudiada.
Los resultados de este estudio nos permiten concluir que los alelos 86Tir 1246Tir en el gen pfmdr1 no son útiles en estas regiones como marcadores moleculares de falla terapéutica o sensibilidad a medicamentos como cloroquina, amodiaquina, o mefloquina, con lo cual queda claro que en nuestras poblaciones parasitarias de P. falciparum, diversos factores genéticos del parásito, diferentes de los polimorfismos aquí estudiados, además de otros factores atribuibles al hospedero, están involucrados en la respuesta terapéutica o en el desarrollo de paludismo grave.
Conflictos de interés
Los autores declaran que no existen conflictos de interés.
Financiación
Esta investigación fue financiada por la Universidad de Antioquia, Colciencias, contrato RC 156-2002, código 1115-04-11948, y la Dirección Seccional de Salud de Antioquia.
Correspondencia:
Amanda Maestre, Grupo Malaria, Sede de Investigación Universitaria, Universidad de Antioquia, Medellín, Colombia.
Teléfono: 210 6490, fax: 210 6487
aemaestre@quimbaya.udea.edu.co
Referencias
1. Wongsrichanalai C, Pickard AL, Wernsdorfer WH, Meshnick SR. Epidemiology of drug-resistant malaria. Lancet Infect Dis. 2002; 2:209-18. [ Links ]
2. Zambrano P. Informe final de malaria, semanas 1 a 52, Colombia, 2005. Inf Quinc Epidemiol Nac. 2006; 11:49-64. [ Links ]
3. Basco LK, Tahar R, Ringwald P. Molecular basis of in vivo resistance to sulfadoxine-pyrimethamine in African adult patients infected with Plasmodium falciparum malaria parasites. Antimicrob Agents Chemother. 1998;42:1811-4. [ Links ]
4. Nzila AM, Mberu EK, Sulo J, Dayo H, Winstanley PA, Sibley CH, et al. Towards an understanding of the mechanism of pyrimethamine-sulfadoxine resistance in Plasmodium falciparum: genotyping of dihydrofolate reductase and dihydropteroate synthase of Kenyan parasites. Antimicrob Agents Chemother. 2000;44:991-6. [ Links ]
5. Fidock DA, Nomura T, Talley AK, Cooper RA, Dzekunov SM, Ferdig MT, et al. Mutations in the Plasmodium falciparum digestive vacuole transmembrane protein PfCRT and evidence for their role in chloroquine resistance. Mol Cell. 2000;6:861-71. [ Links ]
6. Djimde A, Doumbo OK, Cortese JF, Kayentao K, Doumbo S, Diourte Y, et al. A molecular marker for chloroquine-resistant falciparum malaria. N Engl J Med. 2001;344:257-63. [ Links ]
7. Dorsey G, Kamya MR, Singh A, Rosenthal PJ. Polymorphisms in the Plasmodium falciparum pfcrt and pfmdr-1 genes and clinical response to chloroquine in Kampala, Uganda. J Infect Dis. 2001;183:1417-20. [ Links ]
8. Peel SA. The ABC transporter genes of Plasmodium falciparum and drug resistance. Drug Resist Updat. 2001;4:66-74. [ Links ]
9. Reed MB, Saliba KJ, Caruana SR, Kirk K, Cowman AF. Pgh1 modulates sensitivity and resistance to multiple antimalarials in Plasmodium falciparum. Nature. 2000;403:906-9. [ Links ]
10. Basco LK, de Pecoulas PE, Le Bras J, Wilson CM. Plasmodium falciparum: molecular characterization of multidrug-resistant Cambodian isolates. Exp Parasitol. 1996;82:97-103. [ Links ]
11. Basco LK, Ringwald P. Molecular epidemiology of malaria in Cameroon. X. Evaluation of PFMDR1 mutations as genetic markers for resistance to amino alcohols and artemisinin derivatives. Am J Trop Med Hyg. 2002;66:667-71. [ Links ]
12. Awad-el-Kariem FM, Miles MA, Warhurst DC. Chloroquine-resistant Plasmodium falciparum isolates from the Sudan lack two mutations in the pfmdr1 gene thought to be associated with chloroquine resistance. Trans R Soc Trop Med Hyg. 1992;86:587-9. [ Links ]
13. Bhattacharya PR, Biswas S, Kabilan L. Alleles of the Plasmodium falciparum Pfmdr1 gene appear not to be associated with chloroquine resistance in India. Trans R Soc Trop Med Hyg 1997;91:454-5. [ Links ]
14. Bhattacharya PR, Pillai CR. Strong association, but incomplete correlation, between chloroquine resistance and allelic variation in the pfmdr-1 gene of Plasmodium falciparum isolates from India. Ann Trop Med Parasitol. 1999;93:679-84. [ Links ]
15. Peel SA, Bright P, Yount B, Handy J, Baric RS. A strong association between mefloquine and halofantrine resistance and amplification, overexpression, and mutation in the P-glycoprotein gene homolog (pfmdr) of Plasmodium falciparum in vitro. Am J Trop Med Hyg. 1994;51:648-58. [ Links ]
16. Pickard AL, Wongsrichanalai C, Purfield A, Kamwendo D, Emery K, Zalewski C et al. Resistance to antimalarials in Southeast Asia and genetic polymorphisms in pfmdr1. Antimicrob Agents Chemother. 2003;47:2418-23. [ Links ]
17. Lopes D, Rungsihirunrat K, Nogueira F, Seugorn A, Gil JP, do Rosario VE et al. Molecular characterisation of drug-resistant Plasmodium falciparum from Thailand. Malar J. 2002;1:12. [ Links ]
18. Zalis MG, Pang L, Silveira MS, Milhous WK, Wirth DF. Characterization of Plasmodium falciparum isolated from the Amazon region of Brazil: evidence for quinine resistance. Am J Trop Med Hyg. 1998;58:630-7. [ Links ]
19. Cox-Singh J, Singh B, Alias A, Abdullah MS. Assessment of the association between three pfmdr1 point mutations and chloroquine resistance in vitro of Malaysian Plasmodium falciparum isolates. Trans R Soc Trop Med Hyg. 1995;89:436-7. [ Links ]
20. Blair S, Lacharme LL, Fonseca JC, Tobon A. Resistance of Plasmodium falciparum to 3 antimalarials in Turbo (Antioquia, Colombia), 1998. Rev Panam Salud Pública. 2001;9:23-9. [ Links ]
21. Blair S, Lacharme LL, Carmona J, Tobon A. Resistencia de Plasmodium falciparum a los antimaláricos en Urabá y Bajo Cauca antioqueño. 1998. Revista Epidemiológica de Antioquia. 1999; 24:207-15. [ Links ]
22. Blair S, López ML, Pineros JG, Álvarez T, Tobón A, Carmona J. Eficacia terapéutica de tres esquemas de tratamiento de malaria no complicada por Plasmodium falciparum, Antioquia, Colombia, 2002. Biomédica. 2003;23:318-27. [ Links ]
23. González IJ, Padilla JC, Giraldo LE, Saravia NG. Eficacia de amodiaquina y sulfadoxina/pirimetamina en el tratamiento de malaria no complicada por Plasmodium falciparum en Nariño, Colombia, 1999-2002. Biomédica. 2003;23:38-46. [ Links ]
24. Carmona-Fonseca J. La malaria en Colombia, Antioquia y las zonas de Urabá Bajo Cauca: panorama para interpretar la falla terapeútica antimalárica. Parte 1. Iatreia. 2003;16:299-318. [ Links ]
25. OMS/OPS. Evaluación de la eficacia terapéutica de los medicamentos para el tratamiento del paludismo por Plasmodium falciparum sin complicaciones en las Américas. Washington D.C.: OPS/HCP/HCT/113/98; 1998. [ Links ]
26. Ministerio de Salud. Guía de atención de la malaria. En: Diario Oficial. 43956 ed. Bogotá: Ministerio de Salud; 2000. [ Links ]
27. Snounou G, Viriyakosol S, Zhu XP, Jarra W, Pinheiro L, do Rosario VE et al. High sensitivity of detection of human malaria parasites by the use of nested polymerase chain reaction. Mol Biochem Parasitol. 1993;61:315-20. [ Links ]
28. SPSS. Standard version 7.5. Chicago: SPSS for Windows; 1996. [ Links ]
29. CDC, WHO. Epiinfo 6.04. Geneve: WHO; 1996. [ Links ]
30. Basco LK, Ringwald P. Molecular epidemiology of malaria in Yaounde, Cameroon. III. Analysis of chloroquine resistance and point mutations in the multidrug resistance 1 (pfmdr1) gene of Plasmodium falciparum. Am J Trop Med Hyg. 1998;59:577-81. [ Links ]
31. Ochong EO, van den Broek IV, Keus K, Nzila A. Short report: association between chloroquine and amodiaquine resistance and allelic variation in the Plasmodium falciparum multiple drug resistance 1 gene and the chloroquine resistance transporter gene in isolates from the upper Nile in southern Sudan. Am J Trop Med Hyg. 2003;69:184-7. [ Links ]
32. Duraisingh MT, Drakeley CJ, Muller O, Bailey R, Snounou G, Targett GA, et al. Evidence for selection for the tyrosine-86 allele of the pfmdr 1 gene of Plasmodium falciparum by chloroquine and amodiaquine. Parasitology. 1997;114:205-11. [ Links ]
33. Povoa MM, Adagu IS, Oliveira SG, Machado RL, Miles MA, Warhurst DC. Pfmdr1 Asn1042Asp and Asp1246Tyr polymorphisms, thought to be associated with chloroquine resistance, are present in chloroquine-resistant and -sensitive Brazilian field isolates of Plasmodium falciparum. Exp Parasitol. 1998;88:64-8. [ Links ]
34. Huaman MC, Roncal N, Nakazawa S, Long TT, Gerena L, Garcia C, et al. Polymorphism of the Plasmodium falciparum multidrug resistance and chloroquine resistance transporter genes and in vitro susceptibility to aminoquinolines in isolates from the Peruvian Amazon. Am J Trop Med Hyg. 2004;70:461-6. [ Links ]
35. Pillai DR, Hijar G, Montoya Y, Marouino W, Ruebush TK 2nd, Wongsrichanalai C, et al. Lack of prediction of mefloquine and mefloquine-artesunate treatment outcome by mutations in the Plasmodium falciparum multidrug resistance 1 (pfmdr1) gene for Plasmodium falciparum malaria in Peru. Am J Trop Med Hyg. 2003; 68:107-10. [ Links ]
36. Duraisingh MT, Jones P, Sambou I, von Seidlein L, Pinder M, Warhurst DC. The tyrosine-86 allele of the pfmdr1 gene of Plasmodium falciparum is associated with increased sensitivity to the anti-malarials mefloquine and artemisinin. Mol Biochem Parasitol. 2000;108:13-23. [ Links ]
37. Holmgren G, Gil JP, Ferreira PM, Veiga MI, Obonyo CO, Bjorkman A. Amodiaquine resistant Plasmodium falciparum malaria in vivo is associated with selection of pfcrt 76T and pfmdr1 86Y. Infect Genet Evol. 2006;6:309-14. [ Links ]
38. Happi CT, Gbotosho GO, Folarin OA, Bolaji OM, Sowunmi A, Kyle DE et al. Association between mutations in Plasmodium falciparum chloroquine resistance transporter and P. falciparum multidrug resistance 1 genes and in vivo amodiaquine resistance in P. falciparum malaria-infected children in Nigeria. Am J Trop Med Hyg. 2006;75:155-61. [ Links ]
39. Duraisingh MT, Roper C, Walliker D, Warhurst DC. Increased sensitivity to the antimalarials mefloquine and artemisinin is conferred by mutations in the pfmdr1 gene of Plasmodium falciparum. Mol Microbiol. 2000; 36: 955-61. [ Links ]
40. Mawili-Mboumba DP, Kun JF, Lell B, Kremsner PG, Ntoumi F. Pfmdr1 alleles and response to ultralow-dose mefloquine treatment in Gabonese patients. Antimicrob Agents Chemother. 2002;46:166-70. [ Links ]
41. Winstanley PA, Ward SA, Snow RW. Clinical status and implications of antimalarial drug resistance. Microbes Infect. 2002;4:157-64. [ Links ]
42. Montoya L, Maestre A, Carmona J, Lopes D, Do Rosario V, Blair S. Plasmodium falciparum: diversity studies of isolates from two Colombian regions with different endemicity. Exp Parasitol. 2003;104:14-9. [ Links ]
43. Price RN, Uhlemann AC, Brockman A, McGready R, Ashley E, Phaipun L, et al. Mefloquine resistance in Plasmodium falciparum and increased pfmdr1 gene copy number. Lancet 2004;364:438-47. [ Links ]
44. Nelson AL, Purfield A, McDaniel P, Uthaimongkol N, Buathong N, Sriwichai S, et al. Pfmdr1 genotyping and in vivo mefloquine resistance on the Thai-Myanmar border. Am J Trop Med Hyg. 2005;72:586-92. [ Links ]
45. Sidhu AB, Uhlemann AC, Valderramos SG, Valderramos JC, Krishna S, Fidock DA. Decreasing pfmdr1 copy number in Plasmodium falciparum malaria heightens susceptibility to mefloquine, lumefantrine, halofantrine, quinine, and artemisinin. J Infect Dis. 2006;194:528-35. [ Links ]
46. Anderson TJ, Nair S, Qin H, Singlam S, Brockman A, Paiphun L et al. Are transporter genes other than the chloroquine resistance locus (pfcrt) and multidrug resistance gene (pfmdr) associated with antimalarial drug resistance? Antimicrob Agents Chemother. 2005;49:2180-8. [ Links ]
47. Omar SA, Adagu IS, Gump DW, Ndaru NP, Warhurst DC. Plasmodium falciparum in Kenya: high prevalence of drug-resistance-associated polymorphisms in hospital admissions with severe malaria in an epidemic area. Ann Trop Med Parasitol. 2001;95:661-9. [ Links ]














