Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.27 suppl.1 Bogotá Jan. 2007
Estructura poblacional y variabilidad genética de Rhodnius prolixus (Hemiptera: Reduviidae) procedente de diferentes áreas geográficas de Colombia
Diana Carolina López, Carlos Jaramillo, Felipe Guhl
Centro de Investigaciones en Microbiología y Parasitología Tropical (CIMPAT), Facultad de Ciencias, Universidad de los Andes, Bogotá D.C., Colombia.
Recibido: 16/12/05; aceptado: 14/03/06
Introducción. Rhodnius prolixus es el vector más importante de la enfermedad de Chagas en Colombia. La caracterización genética de esta especie resulta útil para comprender su potencial de dispersión. La distribución del vector y la estructura genética de sus poblaciones, son factores importantes para implementar de manera adecuada las estrategias de control y vigilancia epidemiológica de la enfermedad de Chagas.
Objetivo. Establecer relaciones genéticas entre poblaciones de R. prolixus capturadas en diferentes hábitat y áreas geográficas de Colombia, para dilucidar la estructura genética y dispersión del vector en el territorio colombiano.
Materiales y métodos. Se analizaron tres poblaciones domiciliadas de R. prolixus provenientes de Tolima, Cundinamarca y la Sierra Nevada de Santa Marta; y una población silvestre procedente de Casanare. Se emplearon dos técnicas moleculares para evaluar la estructura genética de las poblaciones: análisis del ITS-2 del ADN ribosomal por PCR/RFLP y RAPDs.
Resultados. R. prolixus presenta variabilidad genética moderada (Fst 0,057-0,15), entre las poblaciones domiciliadas se encontraron tasas de migración (Nm>1) que revelan flujo genético. Se encontró diferenciación genética de moderada-alta entre la población silvestre de Casanare y las poblaciones domésticas del centro del país (Tolima y Cundinamarca).
Conclusión. Las poblaciones domiciliadas de R. prolixus son homogéneas debido a que existe flujo genético entre éstas; lo cual es favorable para el control químico, mientras que la población silvestre agrupa aparte de las domiciliadas. Se evidencia la necesidad de estudiar la estructura genética de los focos silvestres, sus posibles rutas de dispersión y el riesgo epidemiológico que representan.
Palabras clave: enfermedad de Chagas, vigilancia epidemiológica, control vectorial, Rhodnius, ADN espaciador ribosomal, reacción en cadena de la polimerasa, polimorfismo de longitud del fragmento de restricción, técnica del ADN polimorfo amplificado aleatorio.
Population structure and genetic variability of Rhodnius prolixus (Hemiptera: Reduviidae) from different geographic areas of Colombia.
Introduction. Rhodnius prolixus is the most important vector of Chagas disease in Colombia. Genetic characterization of this species is useful to understand its potential of dispersion. The distribution of the vector and the genetic population structure are important factors for the adequate implementation of control programs and epidemiological surveillance of Chagas disease.
Objective. Genetic relationships were established for populations of R. prolixus collected from several habitat types and representative geographic areas of Colombia. A second aim was to assess its population genetic structure and dispersion across Colombia.
Materials and methods. Genetic comparisons were made from three domestic populations of R. prolixus from (1) Tolima Province, (2) Cundinamarca Province and (3) the Sierra Nevada of Santa Marta in northern Colombia, and (4) one sylvatic population from Casanare. Two molecular techniques were used to evaluate the genetic structure of these populationsanalysis of the ITS-2 of ribosomal DNA by PCR/RFLP and RAPDs.
Results. Rhodnius prolixus shows a moderate genetic variability (Fst 0.06–0.15). Among domestic populations, the migration rates found were adequate (Nm>1) to maintain gene flow. A moderate to large degree of genetic differentiation was observed between the sylvatic population from Casanare and the domestic populations from the centre of the country (Tolima and Cundinamarca).
Conclusion. The domestic populations of R. prolixus are homogeneous because genetic flow exists between them, and this is favourable to chemical control, while the sylvatic population clusters apart from the domestic populations. Hence the need to study the genetic structure of the sylvatic foci, their possible dispersion routes and the epidemiological risk that they represents.
Key words. Chagas disease, epidemiologic surveillance, vector control, Rhodnius, DNA, ribosomal spacer, polymerase chain reaction, polymorphism, restriction fragment length, random amplified polymorphic DNA technique.
La enfermedad de Chagas, exclusiva del continente americano, se extiende desde México hasta Argentina y es considerada una de las enfermedades parasitarias de mayor impacto social y económico (1). En Colombia constituye un problema de salud pública importante; se estima que la prevalencia de la infección en la población es del 5% y que existen alrededor de 3,5 millones de personas en riesgo de adquirir la enfermedad, dependiendo de la distribución geográfica de los insectos vectores (Hemiptera: Reduviidae) capaces de transmitir el parásito Trypanosoma cruzi (2).
El vector más importante de la enfermedad de Chagas en Colombia, al igual que en Venezuela y Centro América, es Rhodnius prolixus, debido a su amplia distribución geográfica, sus hábitos domiciliarios, su alta frecuencia de dispersión y buena capacidad para infectarse y transmitir el parásito.
Estudios recientes reportan la presencia de R. prolixus silvestre en el departamento de Casanare en plantaciones agroindustriales de palma africana (Elaeis guineensis) y palma nativa (Attalea butyracea), con índices de infestación del 46,6 y 100% e índices de infección de 41,17 y 67,18%, respectivamente (Guhl et al., Primer reporte de Rhodnius prolixus Stal, en Elaeis guineensis I, variedad Papúa, en plantaciones agroindustriales de Villanueva, Casanare (Pinto et al., Comprobación del ciclo silvestre de Rhodnius prolixus Stal en reductos de Attalea butyracea, en el departamento del Casanare. En: Memorias del XII Congreso Colombiano de Parasitología y Medicina Tropical. Biomédica 2005;25(Suppl I):158-9). Reportes anteriores describen poblaciones silvestres de R. prolixus en 13 especies de palmas en Venezuela (3).
La caracterización genética del vector con base en el análisis de especímenes domiciliados y silvestres provenientes de diferentes áreas del territorio colombiano resulta importante para comprender su distribución geográfica, potencial de dispersión y flujo genético, aspectos importantes en la endemicidad de la enfermedad de Chagas en las zonas donde se puede llevar a cabo la transmisión vectorial de T. cruzi.
Diversas técnicas moleculares como la amplificación aleatoria de ADN polimorfo (RAPD, por sus siglas en inglés), la secuenciación de genes mitocondriales, microsatélites, y el análisis de genes y espaciadores ribosomales se han utilizado ampliamente en el estudio de la estructura genética de varias especies de triatominos para resolver relaciones filogenéticas y evolutivas entre especies, complejos de especies e incluso para la caracterización de poblaciones de una misma especie (4-7).
Algunas regiones del ADN ribosomal (rADN) han demostrado ser importantes marcadores moleculares en estudios sistemáticos y filogenéticos de triatominos, debido a que presentan secuencias microsatélites útiles para la caracterización de poblaciones (8-10). El análisis de estas regiones mediante la reacción en cadena de la polimerasa (PCR) en búsqueda de polimor-fismos de longitud y polimorfismos de fragmentos de restricción (RFLP) ha probado que son eficientes en la diferenciación de especies de Anopheles sp y cepas de Angiostrongylus sp (11).
Los RAPDs se basan en la amplificación al azar de segmentos de ADN en búsqueda de poli-morfismos, para así determinar relaciones genéticas entre individuos de una población o entre diferentes poblaciones. Esta técnica se ha utilizado para análisis de la estructura poblacional de varias especies de triatominos como Triatoma dimidiata, T. brasiliensis, R. prolixus, entre otras, permitiendo estimar el flujo genético entre poblaciones silvestres y domésticas de estos vectores (12-14).
En este estudio se empleó la técnica RAPD y se analizó el espaciador interno transcrito2 (ITS-2) del rADN por PCR/RFLP con el propósito de evaluar la variabilidad intra-específica de R. prolixus a partir de cuatro poblaciones colombianas y determinar posibles relaciones genéticas entre ellas.
Materiales y métodos
Insectos
Se analizaron en total 48 insectos adultos R. prolixus, 12 por población (seis hembras y seis machos). Las poblaciones domiciliadas fueron capturadas en tres áreas geográficas endémicas para la enfermedad de Chagas en Colombia: en los municipios de Fomeque y Ubaque en Cundinamarca, en el municipio de Coyaima, Tolima, en el asentamiento indígena Kasakumake en la Sierra Nevada de Santa Marta, y en Dibuya, La Guajira; la población silvestre fue capturada en dos lotes de cultivos agroindustriales de palma de aceite (Elaeis guineensis) en el municipio de Villanueva, Casanare (
figura 1).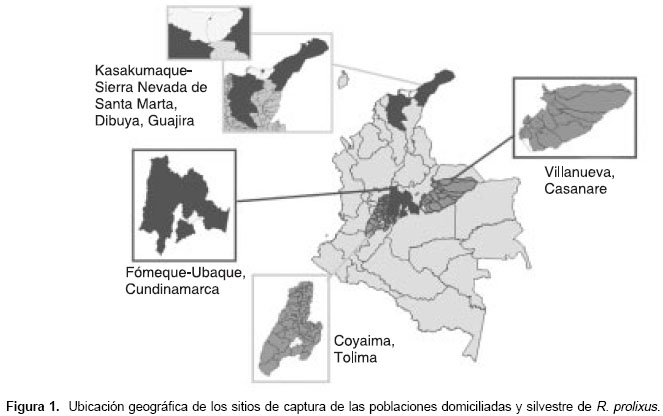
Adicionalmente, se incluyó un espécimen de R. prolixus domiciliado de Maracaibo, Venezuela, para compararlo con las poblaciones colombianas. Como grupos de exclusión se usaron un ejemplar de R. pallescens procedente de Vegachí, Antioquia, uno de R. colombiensis procedente de Totarco, Tolima, y un individuo T. dimidiata de Soatá, Boyacá. Todos los insectos utilizados en el estudio fueron clasificados siguiendo las claves de Lent & Wygodzinsky (15).
Extracción del ADN
El ADN se obtuvo a partir de las seis patas de cada insecto, que fueron retiradas y conservadas en alcohol en un tubo plástico de 1,5 ml. Las patas se lavaron con hipoclorito 1:50 y etanol al 70% para evitar la contaminación con ADN foráneo. Se empleó el estuche de extracción AquaPure Genomic de BIORAD®. El ADN fue almacenado a -20°C.
Cuantificación del ADN
La concentración de ADN se determinó por espectrofotometría (barrido de 234 a 320 nm). Se cuantificó el ADN extraído (15 µg/ml en promedio) y el ADN de los productos de PCR (329 µg/ml en promedio) para los protocolos de restricción enzimática.
Amplificación del ITS-2 por PCR
Se emplearon los iniciadores 5,8T (5-CTA AGC GGT GGA TCA CTC GG-3) y 28 T (5-GCA CTA TCA AGC AAC ACG ACT C-3) (10), que amplifican un fragmento de 127 pb del gen 5,8S, el ITS-2 completo y un fragmento de 350 pb del gen 28S. Se usó el estuche PCR Ready to go de Pharmacia®; cada perla fue hidratada con 15,25 µl de agua destilada estéril, se adicionó 1 pmol de cada iniciador y 3,75 µl de MgCl2 (10 mM), así como 5 µl del ADN molde (15 ng/µl), para un volumen final de 25 µl. En todas las reacciones se incluyó un blanco de reacción sin ADN. El perfil térmico usado fue el descrito por Jaramillo et al. (13). La electroforesis se realizó en geles de agarosa al 1,5%; en cada pozo se sembraron 6 ml de muestra previamente mezclada con 2 ml de buffer de carga. Los geles se corrieron 100 minutos a 70 voltios y luego fueron digitalizados en un documentador de geles CHEMI-DOC SYSTEM y analizados con el software Quantity One 1D de BIORAD®.
Análisis de restricción por PCR/RFLP
Para la elección de las enzimas se elaboró un mapa de restricción del ITS-2 de R. prolixus con el programa NEBcutter V2.0 de New England Biolabs disponible en Internet a partir de secuencias publicadas en Genbank (AJ286888, AY345868). Se buscaron enzimas que tuvieran uno o dos cortes sobre la secuencia y un sitio de reconocimiento de 4 a 6 nucleótidos. La digestión de los productos de PCR se realizó por separado con las enzimas Rsa I, Nhe I, Dde I y Hpy 188 I. La enzima Rsa I tiene un sitio de corte sobre el ITS-2 de R. prolixus en la secuencia de reconocimiento GTnAC; la enzima Nhe I tiene un sitio de corte sobre la secuencia G nCTAG ? C del ITS-2 de R. prolixus; Dde I realiza dos cortes en el fragmento del rADN amplificado, uno sobre el gen 5,8S y el otro sobre el gen 28S de R. prolixus, en la secuencia de reconocimiento C?TNA?G; la enzima Hpy 188 I presenta dos sitios de corte sobre el fragmento del rADN de R. prolixus en la secuencia TC?N?GA. En las restricciones se siguió el protocolo sugerido por Promega®: en un tubo plástico estéril de 1,5 ml se mezcló agua destilada estéril, 2 ml del buffer de la enzima (10X) y 0,4 ml de BSA acetilada (5 mg/µl) (únicamente para las digestiones con Rsa I y Nhe I); se adicionaron 3,5 µl ADN (0,3 µg/µl) y cinco unidades de enzima (10 U/µl) para un volumen final de 20 µl. La digestión se realizó a 37°C durante 3 a 4 horas. Los patrones de restricción se visualizaron en geles de agarosa al 1,5 o 2%, dependiendo del tamaño de los fragmentos obtenidos. El producto se mezcló con 4ml de buffer de carga 6X y se sembraron 10 µl en cada pozo. La electroforesis, la digitalización de los geles y el cálculo del peso de los fragmentos se realizaron tal como se describió para los productos de PCR. Como controles experimentales, paralelos a los ensayos de restricción, se usaron el ADN no digerido y la reacción de restricción sin enzima.
RAPD
Se usó el estuche Ready To Go RAPD y los iniciadores RTG 2 (5 -GTTTCGCTCC-3) ,3 (5 -GTAGACCCGT-3) y 4 (5-AAGAGCCCGT-3) de Pharmacia®; cada perla se hidrató con 19 µl de agua destilada y se agregaron 5 µl de iniciador (25 pmol) y posteriormente se adicionaron 4 µl del ADN molde (15 ng/µl) para un volumen final de reacción de 28 µl. Como control de amplificación se utilizó ADN de E. coli BL21 y como control negativo se realizó un blanco de reacción sin ADN. El perfil térmico consistió en un paso inicial de denaturación a 95°C por cinco minutos, seguido de 44 ciclos: a 95°C por 1 minuto, a 36°C por 1 minuto y a 72°C por 2 minutos. Las reacciones se visualizaron en geles de poliacrilamida al 6% con un tiempo de corrido electroforético de 3 horas a 80 V. En cada pozo se sembraron 7 ml del producto previamente mezclado con 2 ml de buffer de carga. Los geles fueron teñidos usando el estuche Silver Stain Plus de Pharmacia®.
Análisis de datos RAPD
El análisis se realiza bajo tres supuestos: a) las poblaciones están en equilibrio de Hardy - Weinberg, o sea que se asume que no hay presiones de selección que favorezcan a alguna población; b) los marcadores RAPD segregan en forma mendeliana con tasas constantes de evolución y sustitución, y c) los alelos recesivos son idénticos entre los individuos.
Se elaboró una matriz binaria con la información genética obtenida de cada individuo bajo el criterio de presencia [1] y ausencia [0] de bandas de acuerdo con Welsh et al. (16). Teniendo en cuenta que en los marcadores RAPDs el fenotipo dominante de un locus se considera como la presencia de una banda, y el fenotipo recesivo es la ausencia de esa banda, los individuos se comparan fenotípicamente en cada locus.
La matriz binaria se analizó con los programas RAPDPLOT (17) y SYNTAX 2000 (18). En RAPDPLOT se generó una matriz de distancias a partir de la matriz binaria usando el índice de similitud de Nei y Li (19), y en SYNTAX se empleó el índice de disimilitud de Jaccard para datos binarios; en los dos programas se generaron dendrogramas con el algoritmo de agrupamiento UPGMA (unweighted pair-group method using an arithmetic average).
Se usó el programa RAPDDIST para calcular las distancias genéticas de Nei (20) entre las poblaciones y se realizó un análisis bootstrap de 1000 réplicas, método estadístico que se usa para evaluar la confiabilidad de un árbol y permite examinar qué tan frecuentemente se forma un conglomerado particular; el árbol consenso resultante se da por el número de veces que una ramificación o nodo se presenta y esto es producto de la proporción bootstrap. Los árboles fueron generados con el programa NEIGHBOR de PHYLIP 3.5C.
El estadístico F (Fst) y la tasa efectiva de migración (Nm) entre poblaciones se estimaron con RAPDFST mediante tres metodologías: Wright (21), Weir & Cockerham (22) y Lynch & Milligan (23). El Fst es la razón de la varianza observada en la frecuencia de un alelo en un locus RAPD entre subpoblaciones y su máxima varianza en la población total; el programa computa los valores Fst para cada locus RAPD asumiendo que las subpoblaciones están en equilibrio de Hardy-Weinberg y la dominancia de cada loci.
Los valores Fst y Nm son estimaciones indirectas del flujo genético y la migración entre poblaciones, en las cuales se asume que no hay selección, ya que ésta puede incrementar o disminuir el Fst, y que no hay mutaciones si la tasa de mutación es alta en relación a la tasa de migración, como sucede con microsatélites o en ADN mitocondrial; puede haber desviaciones en la estimación.
Resultados
Amplificación del ITS-2
Todos los R. prolixus presentaron una única banda de 1.180 pb (
figura 2A). Se analizaron también ejemplares de R. colombiensis, R. pallescens y T. dimidiata, usados como grupos de exclusión en la técnica RAPD. R. colombiensis presentó una banda de 1.175 pb, R. pallescens una de 709 pb y T. dimidiata una de 964 pb (figura 2B).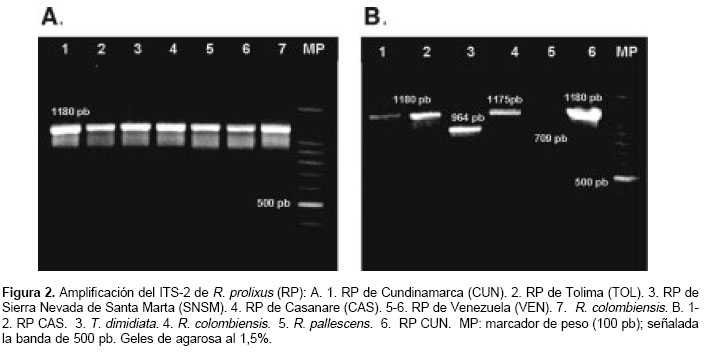
Restricción con Rsa I
Se encontró el mismo patrón de restricción en todos los individuos de las poblaciones estudiadas, y dos fragmentos, uno de 833 y otro de 343 pb. El espécimen R. prolixus de Venezuela mostró un patrón de restricción idéntico al de las poblaciones colombianas (figura 3A). T. dimidiata mostró un sitio de corte y generó dos fragmentos, uno de 738 y otro de 239 pb; R. pallescens no fue cortado por la enzima y R. colombiensis mostró un patrón de restricción con un fragmento de 804 pb y otro de 362 pb (figura 3B).
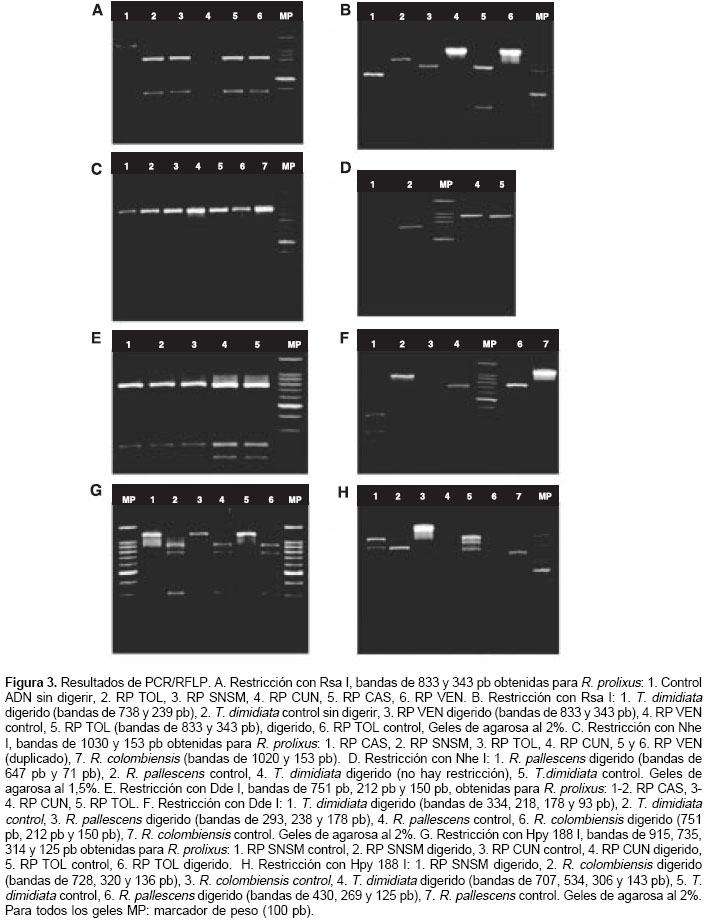
Restricción con Nhe I
Todos los individuos R. prolixus analizados mostraron el mismo patrón de restricción conformado por dos bandas de 1.030 y 153 pb (figura 3C). También hay restricción en R. colombiensis, generando dos fragmentos de 1.020 y 153 pb; R. pallescens presenta un sitio de restricción para esta enzima generando fragmentos de 647 pb y 71 pb. En T. dimidiata no hay restricción (figura 3D).
Restricción con Dde I
Todos los R. prolixus evaluados presentaron dos sitios de corte y el mismo patrón de restricción, una banda de 751 pb, una de 212 pb y una de 150 pb (figura 3E). R. colombiensis presentó un patrón igual al de R. prolixus con dos sitios de corte; T. dimidiata presentó tres sitios de corte, originando cuatro fragmentos de 334, 218, 178 y 93 pb; R. pallescens mostró dos sitios de corte generando tres fragmentos de 293, 238 y 178pb (figura 3F).
Restricción con Hpy 188 I
Esta enzima teóricamente origina un patrón de restricción con tres fragmentos de aproximada-mente 737, 323 y 120 pb; después de la digestión se obtuvo un patrón homogéneo de cuatro bandas en todas las poblaciones: 915, 735, 314 y 125 pb (figura 3G); estos fragmentos podrían sugerir la existencia de un sitio de corte adicional; sin embargo, la suma de los tamaños de las bandas sobrepasa el tamaño del producto sin digerir. R. colombiensis no presentó la banda de 915 pb, pero sí tres bandas de 728, 320 y 136 pb. En T. dimidiata se encontraron cuatro bandas producto de la restricción: de 707, 534, 306 y 143 pb. R. pallescens presentó tres bandas: de 430, 269 y 125 pb (figura 3H).
RAPD
Con los iniciadores RTG 2,3 y 4 se visualizaron un total de 71 bandas o marcadores que representan cada uno un locus RAPD (figura 4). Los dendrogramas generados con los programas SYNTAX y RAPDPLOT muestran una distribución heterogénea de los individuos; no hay agrupamientos definidos por lugar de origen, aunque se observan conglomerados discretos entre individuos de Cundinamarca y Tolima. Todos los individuos R. prolixus del estudio se encuentran por debajo de un valor de disimilitud de Jaccard de 0,475 (en una escala de 0 a 1). R. colombiensis es el grupo de exclusión mas cercano a R. prolixus, seguido por R. pallescens y T. dimidiata (figura 5).
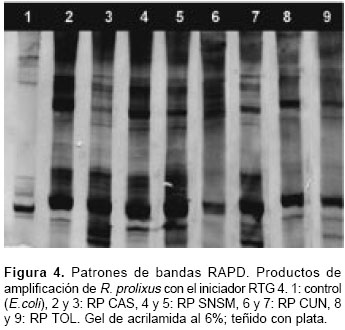
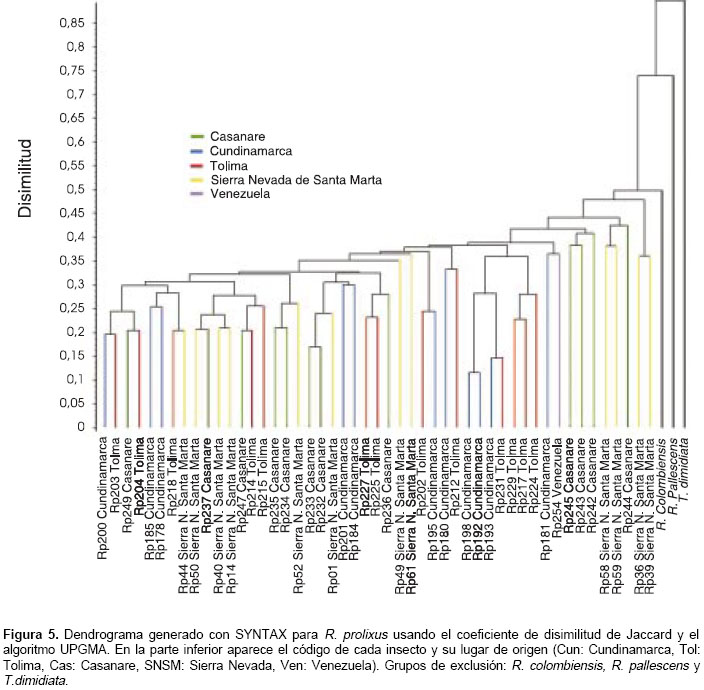
En el análisis con RAPDDIST se generaron árboles con diferentes grupos de exclusión que muestran las relaciones genéticas entre las poblaciones. Se realizó un análisis bootstrap"de 1.000 repeticiones para comprobar la consistencia de los datos RAPD que soportan las relaciones representadas por las ramas del árbol.
Las poblaciones de Tolima y Cundinamarca se agrupan en un conglomerado con una distancia genética de 0,03175, mientras que las poblaciones de la Sierra Nevada y Casanare se encuentran aparte, un poco más alejadas genéticamente de las dos primeras, con distancias de 0,03605 y 0,03733, respectivamente, siendo la población de Casanare la más alejada (
figura 6).
R. prolixus
de Venezuela se encontró separado de las poblaciones de Colombia, con una distancia genética de 0,12521. Para R. colombiensis, usado como grupo de exclusión, se obtuvo una distancia de 0,19167; R. pallescens mostró una distancia de 0,22180 y T. dimidiata enraizó el árbol a una distancia de 0,29351.Se estimó el índice de fijación o estadístico F (Fst), que mide el grado de diferenciación genética entre poblaciones y la tasa efectiva de migración (Nm) entre todas las poblaciones en conjunto y por pares. Los valores Fst obtenidos en todos los casos indican una diferenciación genética moderada (Fst 0,05 a 0,15) y una tasa de migración (Nm>1), suficiente para mantener homogeneidad genética entre las poblaciones (cuadro 1).
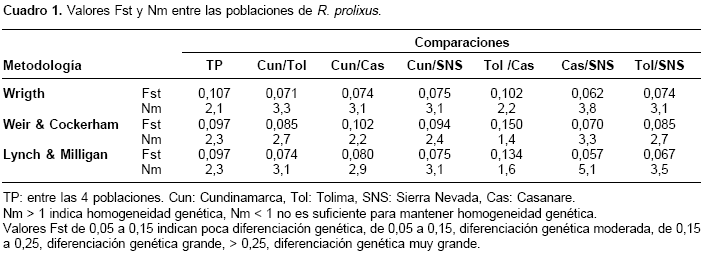
El valor Fst más alto se encontró entre las poblaciones de Tolima y Casanare (Fst 0,150) siguiendo la metodología de Weir & Cockerham, que realiza una corrección en muestras de tamaño pequeño, e indicando una diferenciación genética grande; el siguiente valor Fst más alto se obtuvo entre Cundinamarca y Casanare (Fst 0,102). Las estimaciones del Fst y Nm concuerdan con lo observado en el dendrograma de RAPDDIST, en el que la población de Casanare proveniente de un hábitat silvestre se diferencia de las otras poblaciones.
Discusión
No se detectaron polimorfismos de longitud en la región del ITS-2; la digestión del fragmento amplificado con las enzimas Nhe I, Rsa I, Dde I y Hpy I mostró patrones de restricción homogéneos, indicando que los individuos evaluados no difieren en ninguno de los sitios de clivaje de las enzimas, y por lo tanto no se presentaron polimorfismos de fragmentos de restricción; por ello no es posible, usando estas enzimas, diferenciar a nivel molecular las poblaciones de R. prolixus con estos marcadores.
El patrón de restricción obtenido con la enzima Hpy I presentó una banda adicional que no desapareció ni modificando el tiempo de incubación ni la cantidad de enzima, por lo que puede tratarse de un genotipo heterocigoto, en el que se presenta el sitio de restricción en algunas de las cadenas y en otras no. El hecho de que el patrón se haya presentado en todos los individuos indicaría un alto porcentaje de heterocigocidad en la población. Sería conveniente corroborar estos resultados mediante la secuenciación de dichos fragmentos.
El marcador molecular ITS-2 presentó alta homogeneidad intra-específica, lo que concuerda con lo encontrado en otro estudio, en el cual secuencias del ITS-2 de dos individuos R. prolixus de Colombia fueron idénticas (Vergel C et al. Marcadores especie-específicos para la diferenciación entre Rhodnius prolixus y Rhodnius colombiensis. En: Memorias Curso Taller Internacional 2002: sistemas de información geográfica, sensores remotos y genética poblacional de vectores y parásitos aplicados al control de la enfermedad de Chagas; 2002 Dic 2-6; Universidad de los Andes, Bogotá. Colombia. p. 45-64).
Algo similar fue reportado entre dos variedades cromáticas de R. stali (8), por lo cual se sugiere usar otros marcadores moleculares de evolución más rápida como lo son algunos genes mitocondriales.
Los dendrogramas obtenidos con RAPDDIST indican que las poblaciones de Tolima y Cundinamarca son las más cercanas genéticamente, probablemente debido a la cercanía geográfica, y un Fst de 0,071 muestra un flujo genético de moderado a alto entre estas dos poblaciones.
El conglomerado de Tolima y Cundinamarca se agrupa con la población de la Sierra Nevada, probablemente debido a un proceso de migración pasiva entre las cordilleras central y oriental a lo largo de todo el valle del Magdalena.
La región del Casanare se encuentra separada por la Cordillera Oriental de las otras áreas de estudio; esta barrera geográfica al parecer ha conllevado a un aislamiento geográfico entre las poblaciones del occidente y oriente del país, lo cual concuerda con lo reportado en otro estudio realizado con patrones de sensilla entre poblaciones de R. prolixus de la zona andina y los llanos orientales (Esteban L et al. Variación fenotípica antenal de poblaciones domésticas de Rhodnius prolixus (Hemiptera:Reduviidae) de Colombia. En: Resúmenes del XXXII Congreso Sociedad Colombiana de Entomología; 2005 Jul 27-29; Ibagué. Colombia. p.110).
Las estimaciones del índice de fijación (Fst) y la tasa efectiva de migración (Nm) indican que entre las poblaciones Tolima-Casanare y Cundinamarca-Casanare existe una diferenciación genética moderada a grande. Sin embargo, a nivel general se mantiene entre todas las poblaciones una diferenciación genética moderada, debida a tasas de migración de por lo menos un individuo por generación. Este movimiento de insectos entre poblaciones separadas por grandes distancias y barreras geográficas puede ser resultado del transporte pasivo asociado a migraciones rural-urbanas (24), por lo tanto, las migraciones humanas y barreras geográficas parecen haber influido en la dinámica de dispersión de las poblaciones de R. prolixus, creando una diferenciación entre las poblaciones del oriente y el occidente del país.
Los resultados obtenidos por la técnica de RAPD para las poblaciones domiciliadas concuerdan con lo reportado en estudios anteriores realizados por morfometría y patrones isoenzimáticos, en los cuales se demuestra que aunque R. prolixus presenta baja variabilidad genética, se observan algunas diferencias significativas entre poblaciones locales y otras variantes geográficas (25-28); otros estudios realizados por medio de análisis de secuencias de ADN mitocondrial y ADN nuclear reportan resultados similares a los obtenidos por medio de las otras técnicas, mostrando que R. prolixus es una especie muy homogénea genéticamente (29-32); sin embargo en ninguno de estos estudios se incluyen las poblaciones silvestres colombianas recientemente reportadas e incluidas en el presente estudio, razón por la cual encontramos una variabilidad genética moderada entre las poblaciones silvestres y las domiciliadas.
Por otro lado, la población silvestre de Casanare parecer ser la menos relacionada con las otras poblaciones evaluadas y puede representar un riesgo epidemiológico para esa área. Estas poblaciones silvestres han mostrado ser un riesgo inminente en la transmisión de la enfermedad. Se ha reportado que el 3% de los niños infectados se debe a los insectos que están llegando de las palmas a las viviendas (Informe de la Secretaria de Salud del Casanare. En: Memorias del Primer Taller Internacional sobre Control de la Enfermedad de Chagas; 2005 Mayo 2-6; Universidad de los Andes. Bogotá, Colombia, p. 245-9).
Los hallazgos de R. prolixus en hábitats silvestres en el departamento de Casanare sugieren que en la región oriental del país el vector podría ser autóctono, y que en las otras regiones fue introducido, encontrándose estrictamente domiciliado.
Es indispensable determinar la distribución y variabilidad genética de las poblaciones que se encuentran asociadas al domicilio y su relación con las poblaciones silvestres, para establecer el riesgo que éstas representan y poder así realizar una adecuada vigilancia epidemiológica.
Los resultados presentados en este estudio constituyen una primera aproximación a la estructura genética poblacional de R. prolixus en Colombia y muestra datos importantes acerca de la dispersión en el país.
La variabilidad genética moderada detectada entre las poblaciones domiciliadas es favorable para las intervenciones de control químico y plantea que la eliminación del vector domiciliado es viable en Colombia a pesar de la existencia de focos silvestres, como se demostró en Venezuela (32), y como se ha observado en Centro América con el vector doméstico (33).
Se puede concluir que las poblaciones domicilia-das de R. prolixus son homogéneas debido a que existe flujo genético entre ellas, mientras que la población silvestre se encuentra separada de las domiciliadas. Se evidencia la necesidad de estudiar la estructura genética de los focos silvestres, sus posibles rutas de dispersión y el riesgo epidemiológico que representan.
Conflicto de intereses
Los autores declaran no tener conflicto de intereses con respecto al estudio.
Financiación
Este estudio se realizó con el apoyo financiero del Fondo de Investigaciones y Posgrados de la Facultad de Ciencias de la Universidad de los Andes y el CDIA-Chagas Disease Intervention Activities-EC Contract N° ICA4CT-2003-10049.
Correspondencia:
Felipe Guhl, CIMPAT, Departamento de Ciencias Biológicas, Universidad de los Andes, bloque A, oficina 201. A.A. 4976.
Carrera 1ª N° 18A -10, Bogotá, Colombia. Tel/Fax: +57 1 3324540.
Referencias
1.Moncayo A. Chagas disease: current epidemiological trends after the interruption of vectorial and transfusional transmission in the Southern cone countries. Mem Inst Oswaldo Cruz 2003;98:577-91. [ Links ]
2.Guhl F, Restrepo M, Angulo VM, Antunes CM, Campbell-Lendrum D, Davies C. Lessons from a national survey of Chagas disease transmission in Colombia. Trends Parasitol 2005;21:259-62. [ Links ]
3.Gamboa CJ. Dispersión de Rhodnius prolixus en Venezuela. Bol Dir Malariol Saneam Ambient 1962;3:262-72. [ Links ]
4.Garcia AL, Carrasco HJ, Shofield CJ, Stothard JR, Frame IA, Valente SA, et al. Random amplification of polymorphism DNA as a tool for taxonomics studies of triatomine bugs (Hemiptera: Reduviidae). J Med Entomol 1998;35:38-45. [ Links ]
5.Monteiro FA, Escalante AA, Beard CB. Molecular tools and triatomine systematics: a public health perspective. Trends Parasitol 2001;17:344-7. [ Links ]
6.Bargues MD, Marcilla A, Ramsey JM, Dujardin JP, Schofield CJ, Mas-Coma S. Nuclear rDNA-based molecular clock of the evolution of triatominae (Hemiptera: Reduviidae) vectors of Chagas disease. Mem Inst Oswaldo Cruz 2000;95:567-73. [ Links ]
7.Pacheco RS, Almeida CE, Costa J, Klisiowicz DR, Mas-Coma S, Bargues MD. RAPD analyses and rDNA intergenic–spacer sequences discriminate Brazilian populations of Triatoma rubrovaria. Ann Trop Med Parasitol 2003;97:757-68. [ Links ]
8.Bargues MD, Marcilla A, Dujardin JP, Mas-Coma S. Triatominae vectors of Chagas disease: a molecular perspective based on nuclear ribosomal DNA markers. Trans R Soc Trop Med Hyg 2002;96(Suppl 1):159-64. [ Links ]
9.García BA, Manfredi C, Fichera L, Segura EL. Variation in mitochondrial 12S and 16S ribosomal DNA sequences in natural populations of Triatoma infestans (Hemiptera, Reduviidae). Am J Trop Med Hyg 2003;68:692-4. [ Links ]
10.Marcilla A, Bargues MD, Ramsey MJ, Magallon-Gastelum E, Salazar-Schettino PM, Abad-Franch F, et al. The ITS-2 of the nuclear rDNA as a molecular marker for populations, species, and phylogenetic relationships in Triatominae (Hemiptera: Reduviidae) vectors of Chagas disease. Mol Phylogenet Evol 2001;18:136-42. [ Links ]
11.Caldeira RL, Carvalho OS, Mendonça CL, Graeff-Teixeira C, Silva MC, Ben R, et al. Molecular differentiation of Angiostrongylus costaricensis, A. Cantonensis, and A. Vasorum by polymerase chain reaction-restriction fragment length polymorphism. Mem Inst Oswaldo Cruz 2003;98:1039-43. [ Links ]
12.Jaramillo C, Montaña MF, Castro LR, Vallejo GA, Guhl F. Differentiation and genetic analysis of Rhodnius prolixus and Rhodnius colombiensis by rDNA and RAPD amplification. Mem Inst Oswaldo Cruz 2001;96:1043-8. [ Links ]
13.Ramírez CJ, Jaramillo CA, Delgado M, Pinto NA, Aguilera G, Guhl F. Genetic structure of sylvatic, peridomestic and domestic populations of Triatoma dimidiata (Hemiptera: Reduviidae) from an endemic zone of Boyaca, Colombia. Acta Trop 2005;93:23-9. [ Links ]
14.Borges EC, Romanha AJ, Diotaiuti L. Uso do random amplified polymorphic DNA (RAPD) no estudo populacional do Triatoma brasiliensis Neiva, 1911. Cad Saúde Pública 2000;16:97-100. [ Links ]
15.Lent H, Wygodzinsky P. Revision of the Triatominae (Hemiptera, Reduviidae) and their significance as vectors of Chagas disease. Bull Am Mus Nat Hist 1979;163:175-499. [ Links ]
16.Welsh J, Pretzrnan C, Postic D, Girons IS, Baranton G, McClelland M. Genomic fingerprinting by arbitrary primer polymerase chain reaction resolves Borrelia burgeri into three distinct phylogenetic groups. Int J Syst Bacteriol 1992; 42:370-7. [ Links ]
17.Kambhampati S, Black WC 4th, Rai KS. Random amplified polymorphic DNA of mosquito species and populations (Diptera: Culicidae): techniques, statistical analysis, and applications. J Med Entomol 1992;29: 939-45. [ Links ]
18.Podani J. SYNTAX. Computer programs for multivariate data analysis in ecology and systematics. Budapest: Scienta Publishing; 1993. [ Links ]
19.Nei M, Li WH. Mathematical model for studying genetic variation in terms of restriction endonucleases. Proc Natl Acad Sci USA 1979;76:5269-73. [ Links ]
20.Nei M. Genetic distance between populations. Am Nat 1972;106:283-92. [ Links ]
21.Wright S. Evolution in Mendelian populations. Genetics 1931;16:97-159. [ Links ]
22.Weir BS, Cockerham CC. Estimating F-statistics for the analysis of populations structure. Evolution 1984;38:1358-70. [ Links ]
23.Lynch M, Milligan BG. Analysis of population genetic structure with RAPD markers. Mol Ecol 1994;3:91-9. [ Links ]
24.Dias JC. Rural resource development and its potential to introduce domestic vectors into a new epidemiological situation. Rev Argent Microbiol 1988;20(Suppl. 1):81-5. [ Links ]
25.Harry M, Galindez I, Cariou ML. Isozyme variability and differentiation between Rhodnius prolixus, R. robustus and R. pictipes, vectors of Chagas disease in Venezuela. Med Vet Entomol 1992;6:37-3. [ Links ]
26.Harry M. Isozymic data question the specific status of some blood-sucking bugs of the genus Rhodnius, vectors of Chagas disease. Trans R Soc Trop Med Hyg 1993;87:492. [ Links ]
27.Harry M. Morphometric variability in the Chagas´disease vector Rhodnius prolixus. Jpn J Genet 1994;69:233-50. [ Links ]
28.Monteiro FA, Lazoski C, Noireau F, Sole-Cava AM. Allozyme relationships among ten species of Rhodniini, showing paraphyly of Rhodnius including Psammolestes. Med Vet Entomol 2002;16:83-90. [ Links ]
29.Monteiro FA, Wesson DM, Dotson EM, Schofield CJ, Beard CB. Phylogeny and molecular taxonomy of the Rhodniini derived from mitochondrial and nuclear DNA sequences. Am J Trop Med Hyg 2000;62:460-5. [ Links ]
30.Monteiro FA, Barrett TV, Fitzpatrick S, Cordon-Rosales C, Feliciangeli D, Beard CB. Molecular phylogeography of the Amazonian Chagas disease vectors Rhodnius prolixus and R. robustus. Mol Ecol 2003;12:997-1006. [ Links ]
31.Abad-Franch F, Monteiro FA. Molecular research and the control of Chagas disease vectors. An Acad Bras Cienc 2005;77:437-54 [ Links ]
32.Feliciangeli MD, Campbell-Lendrum D, Martinez C, Gonzales D, Coleman P, Davies C. Chagas disease control in Venezuela: lessons for the Andean region and beyond. Trends Parasitol 2003;19:44-9. [ Links ]
33.Zeledón R. Some historical facts and recent issues related to the presence of Rhodnius prolixus (Stal, 1859) (Hemiptera:Reduviidae) in Central America. Entomología y vectores 2004;11:233-46. [ Links ]














