Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.27 suppl.1 Bogotá Jan. 2007
Comparación de una prueba de PCR basada en los genes codificantes para la histona H2A/SIRE con pruebas serológicas convencionales para el diagnóstico de la enfermedad de Chagas crónica en pacientes colombianos
Juliana Gil 1, Paula Pavía 1, Marleny Montilla 2, Astrid C. Florez 2, laudia Quintero 3, Marcela Mercado 1, Miguel Vacca 4, Santiago Nicholls 2, Concepción Puerta 1
1 Laboratorio de Parasitología Molecular, Departamento de Microbiología, Facultad de Ciencias, Pontificia Universidad Javeriana, Bogotá D.C., Colombia.
2
Laboratorio de Parasitología, Instituto Nacional de Salud, Bogotá D.C., Colombia.3
Unidad de Epidemiología, Departamento de Microbiología, Pontificia Universidad Javeriana, Bogotá D.C., Colombia.4 Unidad de Cardiología, Hospital Universitario San Ignacio, Pontificia Universidad Javeriana, Bogotá D.C., Colombia.
Recibido: 15/02/06; aceptado: 09/06/06
Introducción. El diagnóstico de la enfermedad de Chagas en sus fases latente y crónica se dificulta por la baja parasitemia, razón por la cual se recurre a métodos serológicos y moleculares.
Objetivo. Comparar la prueba de reacción en cadena de la polimerasa basada en la amplificación del elemento SIRE insertado en el gen que codifica para la histona H2A con las pruebas serológicas convencionales para el diagnóstico de la enfermedad de Chagas en pacientes colombianos.
Materiales y métodos. Se realizó un estudio de concordancia comparando la PCR TcH2AF/R con las pruebas de inmunoensayo enzimático e inmunofluorescencia indirecta, determinándose además la sensibilidad y especificidad de la prueba. Se clasificaron y examinaron 156 individuos según los hallazgos clínicos y epidemiológicos y los resultados de las pruebas serológicas. Adicionalmente, 97 de las 156 muestras fueron comparadas con la PCR S35/S36.
Resultados. De 156 muestras, 89 (57%) fueron positivas por IFI y ELISA, y 84 (53,8%) presentaron el perfil de amplificación correspondiente a la banda esperada de 234 pb, obteniéndose una sensibilidad de 88% (I.C. 95%: 75% - 95%) y especificidad de 92,5% (I.C. 95%: 87,7% - 97,2%). El índice kappa, indicador de concordancia entre las pruebas serológicas y TcH2AF/R fue de 0,8 (I.C. 95%: 73% - 86%), en tanto entre las PCR TcH2AF/R y S35/S36 fue de 0,9 (I.C. 95%: 84% - 96%), interpretados como una concordancia buena y casi perfecta, respectivamente.
Conclusiones. La PCR TcH2AF/R es una prueba diagnóstica promisoria complementaria a las pruebas serológicas, para la detección de Trypanosoma cruzi.
Palabras clave: reacción en cadena de la polimerasa, histonas, miocardiopatía chagásica, Trypanosoma cruzi.
Comparison of a PCR test based on the histone H2A/SIRE genes with classical serological tests for the diagnosis of chronic Chagas disease in Colombian patients
Introduction. Diagnosis of Chagas disease in its latent and chronic phase is difficult because of the low parasitemia levels. Therefore, serological and molecular techniques are necessary to achieve an appropriate diagnosis.
Objective. The polymerase chain reaction based on the amplification of the SIRE element inserted into H2A encoding genes was compared with classical serological tests for the diagnosis of Chagas disease in Colombian patients.
Materials and methods. An agreement study was carried out by comparing the PCR with ELISA (enzyme linked immuno sorbent assay) and IFAT (indirect immunofluorescence) tests. In addition, the PCR sensitivity and specificity were determined. A sample of 156 individuals was tested with the H2A PCR primers after a Chagas disease classification based on clinical, epidemiological and serological data associated with each patient. In addition, 97 out of 156 samples were also compared with the S35/S36 PCR primers.
Results. Eighty-nine of 156 samples (57%) were positive by both IFAT and ELISA and 84 (53.8%) presented the expected 234 bp amplification fragment. The sensitivity of the TcH2AF/R PCR was 88% (95% C.I.: 75%--95%) and its specificity 92.5% (95% C.I.: 87.7%--97.2%). The kappa index for concordance between serological tests and TcH2AF/R PCR was 0.8 (95% C.I.: 73%--86%), and between the TcH2AF/R and S35/S36 PCR primers was 0.9 (95% C.I.: 84%--96%). These indices indicated a "good" and "almost perfect" agreement, respectively.
Conclusions. The TcH2AF/R PCR is a promising diagnostic tool for the detection of T. cruzi and is suggested as a tool complementary to the classical serological tests.
Key words: polymerase chain reaction, histones, Chagas myocardiopathy, Trypanosoma cruzi
Trypanosoma cruzi es un parásito protozoo hemoflagelado, agente causal de la enfermedad de Chagas, entidad que afecta a 18 millones de personas en 15 países endémicos, con una incidencia anual de 200.000 nuevos casos (1). En Colombia se estima que hay cerca de 700.000 personas infectadas con un 23% de la población en riesgo de contraer la enfermedad y entre 30 y 40 mil nuevos casos anuales (2).
T. cruzi se distribuye ampliamente en Sur América, presentando un amplio rango de vectores, hospederos y reservorios, así como un amplio pleomorfismo genético y biológico (3-6). En Colombia se ha visto que aun cuando la mayoría de las cepas del parásito pertenecen al grupo T. cruzi I o zimodema Z1 (7-13, Dib J, Restrepo M, Parra G, Tibayrenc M, Barnabe C, Triana O. Definición de dos poblaciones de Trypanosoma cruzi I en el norte de Colombia mediante análisis de RAPD y el gen de la proteína flagelar. Memorias XII Congreso Colombiano de Parasitología y Medicina Tropical. Biomédica 2005;25:207-8.), éstas varían en sus caracterís-ticas genéticas dependiendo especialmente de su ciclo de transmisión (7, Dib J, Restrepo M, Parra G, Tibayrenc M, Barnabe C, Triana O. Definición de dos poblaciones de Trypanosoma cruzi I en el norte de Colombia mediante análisis de RAPD y el gen de la proteína flagelar. Memorias XII Congreso Colombiano de Parasitología y Medicina Tropical. Biomédica 2005;25:207-8.). Por otra parte, T. cruzi presenta morfología similar y reacción inmunológica cruzada con Trypanosoma rangeli, parásito con el cual también comparte zonas endémicas, vectores y reservorios (14,15). Debido a lo anterior, se hace necesario el desarrollo de pruebas específicas que disminuyan o eliminen el riesgo de falsos negativos por variabilidad intra-cepa y falsos positivos por la presencia de T. rangeli.
Algunos estudios han demostrado la utilidad y el alto potencial de la prueba de reacción en cadena de la polimerasa (PCR) para el diagnóstico de la enfermedad de Chagas, tanto en su etapa aguda como en la crónica (16,17). Una de las pruebas más usadas es la PCR basada en los iniciadores S35/S36, los cuales amplifican los minicírculos del ADN del cinetoplasto (kADN) del parásito (18). Sin embargo, estos iniciadores también amplifican el kADN de T. rangeli, presentándose perfiles que sólo pueden ser distinguidos en geles de poliacrilamida y que, en caso de infecciones mixtas por ambos tripanosomas, pueden enmascarar la presencia de T. rangeli haciéndolo pasar por T. cruzi (19). Además, recientemente se ha visto que el kADN es capaz de integrarse al ADN de la célula hospedera, de manera que existe la posibilidad de amplificación de las bandas esperadas en ausencia de infección (20). Por estas razones se requiere explorar el uso de otras pruebas para la identificación específica de T. cruzi.
Teniendo en cuenta lo anterior, en este trabajo se propuso la evaluación de la prueba de PCR basada en la amplificación del gen que codifica para la histona H2A en pacientes colombianos para la detección de T. cruzi y su comparación con los métodos serológicos convencionales de inmunofluorescencia indirecta (IFI) e inmuno-ensayo enzimático (Elisa) (21).
Materiales y métodos
Pacientes y grupos de estudio
En este estudio participaron 156 individuos entre los 18 y 65 años, de quienes se obtuvo consentimiento informado. Dichos individuos fueron clasificados de acuerdo con el cuadro clínico, las variables epidemiológicas y el resultado de las pruebas serológicas IFI y Elisa (22) en 4 grupos: grupo control (A), conformado por 55 (35,3%) individuos sin riesgo epidemiológico, examen físico y electrocardiograma normal y resultados de IFI y Elisa negativos; grupo de pacientes infectados sin cardiopatía (B), conformado por 16 (10,2%) individuos asintomáticos, con riesgo epidemiológico, examen físico y electrocardiograma normales y pruebas serológicas positivas; grupo de pacientes infectados con cardiopatía (C), constituído por 73 (46,8%) individuos sintomáticos con riesgo epidemiológico, examen físico y electrocardiograma anormales con síntomas cardiovasculares clínicamente significativos y pruebas serológicas positivas, y grupo control de individuos no infectados con cardiopatía (D), conformado por 12 (7,7%) individuos con examen físico y electrocardiograma anormales, con cardiopatía dilatada de origen no chagásico y pruebas serológicas negativas.
Este estudio corresponde a una investigación con riesgo mínimo, de acuerdo con la resolución No. 08430 del Ministerio de Salud de Colombia, y fue aprobado por el Comité de Investigación y Ética de la Facultad de Ciencias y del Hospital Universitario San Ignacio de la Pontificia Universidad Javeriana y del Instituto Nacional de Salud.
Extracción de ADN a partir de sangre
A partir de una muestra de 5 ml de de sangre anticoagulada con EDTA se realizó la extracción de ADN utilizando una alícuota de 0,5 ml mediante el estuche comercial "GFX™ Genomic Blood DNA Purification Kit" (Amersham Pharmacia) y el método fenol-cloroformo-alcohol isoamílico según el protocolo descrito por Vallejo y colaboradores (18), realizando el paso de extracción con fenol-cloroformo-alcohol isoamílico tres veces. El ADN obtenido fue resuspendido en 10 mM de Tris y 1 mM de EDTA, pH 8,0 (TE) estéril.
Extracción de ADN del parásito
Se trabajó con formas epimastigotas de las cepas de T. cruzi, D.A. (MHOM/CO/01/DA) y Munantá (IRHO/CO/98/MTA). El cultivo en masa de los parásitos en fase logarítmica se centrifugó a 4.000 r.p.m. durante una hora. El precipitado obtenido se lavó tres veces con 1 ml de solución salina amortiguada con fosfatos (PBS); se centrifugó nuevamente a 6.000 r.p.m. durante 10 min a 4°C y se resuspendió en 1 ml de PBS y 100 µl de NP40 al 10% en agua; se mezcló, centrifugó a 10.000 r.p.m. por 5 min y se resuspendió en 2 ml de PBS y SDS al 10%. El ADN se extrajo según el método de fenol-cloroformo-alcohol isoamílico, seguido de precipitación con etanol. Finalmente, se midió la concentración del ADN por espectrofotometría a 260/280 nm y se evaluó en geles de agarosa al 1% coloreados con bromuro de etidio (23).
Pruebas de PCR
Para la reacción de PCR TcH2AF/R se utilizaron los oligonucleótidos TcH2AF (5´- AGT GGC AGA CTT TGG GGT C -3´) y TcH2AR (5´- GAG AGT GAT GGG AGA GC -3´), los cuales anillan en el elemento SIRE insertado en la región no codificante de la unidad de 1,2 kb del gen de la histona H2A de T. cruzi (24). La reacción de PCR se llevó a cabo de acuerdo con lo descrito por Pavia y colaboradores (21), utilizando el siguiente programa en un termociclador PTC -100 MJ Research®: denaturación inicial a 95°C por 5 min, 15 ciclos con el siguiente perfil: denaturación a 95°C por 30 segundos, anillaje y extensión a 72°C por 30 segundos cada uno. Luego, se realizaron 30 ciclos con denaturación a 95°C por 30 segundos, anillaje a 65°C por 30 segundos y extensión a 72°C por 30 segundos, seguidos de la extensión final a 72°C por 5 min. Como blanco de reacción se reemplazó el ADN de la muestra por agua o por TE; como control negativo se utilizó ADN humano y como positivo ADN de la cepa Munantá (IRHO/CO/98/MTA) del parásito. Los productos de amplificación fueron analizados mediante electroforesis en gel de agarosa al 1,5% teñido con bromuro de etidio (23). La prueba de PCR S35/S36 se realizó de acuerdo a lo descrito por Vallejo y colaboradores (18). Con el fin de eliminar la posibilidad de inhibición de la reacción de PCR en las muestras negativas, éstas fueron amplificadas nuevamente tras la adición de 50 ng ADN de la cepa Munantá (IRHO/CO/98/MTA) del parásito.
Determinación del poder de detección de la PCR
Se determinó la cantidad mínima de parásitos a detectar por ambas reacciones de PCR mezclando cantidades en base 10 de formas epimastigotas del parásito con sangre humana libre de infección, seguida de la extracción de ADN y ensayos de PCR. Ambos ensayos se realizaron tres veces para asegurar la reproducibilidad de los resultados.
Análisis estadístico
Los datos fueron consignados en una base de datos elaborada con el paquete estadístico EPI-INFO 6.0. El análisis estadístico descriptivo de las pruebas diagnósticas se realizó a través del paquete estadístico Stata 6.0. Se analizaron las características operativas de la prueba (sensibilidad y especificidad) y se realizó el cálculo del índice de concordancia kappa con sus respectivos intervalos de confianza (25).
Resultados
Determinación de la capacidad de detección de la prueba de PCR TcH2AF/R
Se observó el producto de amplificación esperado de 234 pb con la PCR TcH2AF/R hasta con una forma del parásito en ambas cepas a partir del ADN extraído por el método de triple extracción con fenol-cloroformo-alcohol isoamílico en tres réplicas del experimento (
figura 1). Igualmente, con la PCR S35/S36 se pudo detectar hasta un parásito utilizando el método de extracción de triple fenol-cloroformo-alcohol isoamílico, independientemente de la cepa empleada. No obstante, con el estuche comercial disminuyó el poder de detección de ambas pruebas de PCR de 1 a 100 parásitos en el caso de la cepa D.A. y de 1 a 10 parásitos en el caso de la PCR S35/S36 usando el ADN de la cepa Munantá como blanco de amplificación.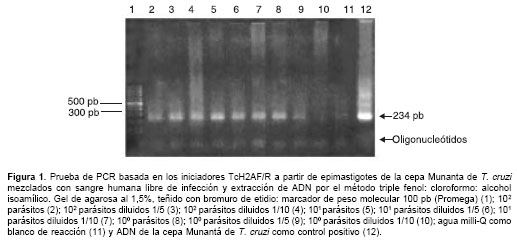
Con el fin de corroborar los resultados anteriores, se realizó una prueba piloto con 31 muestras de pacientes chagásicos y 26 de individuos sanos, en la cual se observó que de 57 muestras a las cuales se les realizó la prueba de PCR TcH2AF/R a partir del ADN extraído con ambos métodos de extracción, 18 (31,5%) fueron positivas para ambos métodos, dos (3,5%) fueron positivas únicamente para el estuche comercial y 13 (22,8%) para el de triple extracción con fenol-cloroformo-alcohol isoamílico. Se obtuvo un índice de concordancia kappa de 0,49 (IC 95%: 36 - 62%), interpretado como una concordancia moderada entre estos dos métodos de extracción. Con base en estos resultados, se seleccionó el método de triple extracción con fenol-cloroformo-alcohol isoamílico para la extracción del ADN de las muestras a analizar.
Comparación de la prueba de PCR TcH2AF/R con las pruebas serológicas y la PCR S35/S36
Se realizó un estudio de concordancia en donde se comparó la PCR TcH2AF/R con las pruebas serológicas de IFI y Elisa, técnicas utilizadas en el Instituto Nacional de Salud como referencia para el diagnóstico y confirmación de la enfermedad de Chagas, utilizando cepas nativas colombianas (22). Ambas pruebas serológicas en este estudio arrojaron resultados idénticos.
Del total de 156 muestras, 89 (57%) fueron positivas por IFI y Elisa y 84 (53,8%) presentaron el perfil de amplificación correspondiente a la banda esperada de 234 pb, presentándose 79 muestras positivas por ambos métodos (
figura 2). Por su parte, de las 67 muestras negativas por ambas pruebas serológicas, 62 de ellas también fueron negativas por la prueba de PCR TcH2AF/R (figura 2).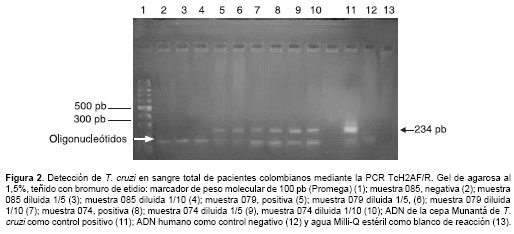
Se obtuvo una sensibilidad de 88% (IC 95%: 75 - 95%) y especificidad de 92,5% (IC 95%: 87,7 - 97,2%) al comparar la prueba de PCR TcH2AF/R con las pruebas serológicas.
El índice de concordancia kappa entre la prueba de PCR TcH2AF/R y ambas pruebas serológicas fue de 0,8 (IC 95%: 73 - 86%), interpretado como una buena concordancia.
El análisis del desempeño de la PCR TcH2AF/R en cada grupo de estudio fue el siguiente: en el grupo control de pacientes no infectados (A), de un total de 55 muestras, cuatro (7,3%) fueron positivas por PCR y 51 (92,7%) fueron negativas. En el grupo de pacientes infectados asintomáticos (B), de un total de 16 muestras, 15 (93,7%) fueron positivas por la PCR TcH2AF/R. En el grupo de pacientes sintomáticos (C), de un total de 73 muestras, 64 (87,6%) fueron positivas por PCR y 9 (12,3%) fueron negativas. En el grupo de individuos control con cardiopatía (D), de un total de 12 muestras, 11 (91,7%) fueron negativas por la PCR TcH2AF/R y una fue positiva.
Considerando los individuos seropositivos (grupos B y C) o seronegativos (grupos A y D), se tiene que en los primeros la PCR TcH2AF/R detectó como positivos al 88,7% (79 de 89), en tanto que en los segundos, de 67 muestras, 5 (7,5%) de ellas fueron positivas por la prueba de PCR.
Por otra parte, de las 156 muestras, 96 de ellas también fueron analizadas mediante la PCR S35/S36, encontrando un índice kappa de 0,9 (IC 95%: 84 -96%), interpretado como una concordancia casi perfecta.
Discusión
El diagnóstico de la enfermedad de Chagas depende de la etapa de la enfermedad en la que se encuentre el paciente y se debe apoyar en la información tanto epidemiológica como clínica del paciente, así como en las pruebas de laboratorio.
Mientras en la etapa aguda de la enfermedad el diagnóstico no presenta problemas debido a la elevada parasitemia, en las etapas indeterminada y crónica, el diagnóstico es muy complejo y requiere de varias pruebas de laboratorio para su confirmación (1,17,22). Entre las pruebas de laboratorio disponibles para el diagnóstico en las etapas indeterminada y crónica de la enfermedad, actualmente se encuentran las pruebas serológicas y el diagnóstico molecular directo por PCR. Mientras las primeras son pruebas indirectas que se basan en la detección de anticuerpos contra el parásito y dependen del estado inmunológico del paciente, de los antígenos, de la fase de desarrollo del parásito empleado en la prueba y del tiempo transcurrido desde la infección, los segundos se basan en la detección directa del ADN del parásito y no dependen del estado inmunológico del paciente ni del tiempo de adquisición de la infección (16,26).
En este trabajo se comparó el desempeño de la prueba de PCR TcH2AF/R para el diagnóstico de pacientes chagásicos indeterminados y crónicos con miras a su utilización como una prueba de apoyo en el diagnóstico de esta enfermedad.
El poder de detección de la prueba TcH2AF/R, evidenciada por la detección de hasta una forma parasitaria en los ensayos de infección artificial, se explica por la abundancia de los genes H2A (24,27) y por que el elemento SIRE short interspersed repetitive element, con 2.000 a 3.000 copias por genoma, se encuentra integrado en el extremo 3 no traducido (UTR) de los genes codificantes para la histona H2A del parásito y sus nucleótidos 16 a 248 (no. de acceso al GenBank, AF098063) corresponden al fragmento amplificado con los cebadores TcH2AF/R (27,28).
Los resultados obtenidos en este trabajo se apoyan en la especificidad de la prueba TcH2AF/R reportada por Pavía y colaboradores, según la cual los iniciadores TcH2AF/R amplifican exclusivamente el ADN de cepas de T. cruzi pertenecientes a los grupos I y II hasta ahora descritos del parásito (29), en tanto que no amplifican el ADN de otros tripanosomátidos que circulan en Colombia, como T. rangeli tanto de cepas KP 1(+) como KP 1(-), representantes de los linajes de este parásito (30) ni de Leishmania spp. y Crithidia spp., entre otros (21).
Es así como la PCR TcH2AF/R fue capaz de detectar el 88% de los individuos serológicamente positivos, porcentaje similar al 84,8% descrito por Gutiérrez y colaboradores en un estudio llevado a cabo en 79 individuos seropositivos en Santander (Colombia) utilizando como blanco el ADN del cinetoplasto del parásito (31). Resultados similares de 83,5 y 90% de positividad fueron reportados por Gomes y colaboradores en 79 pacientes seropositivos y por Britto y colaboradores en 61 en Brasil (32,33). Porcentajes superiores de positividad por PCR en pacientes crónicos han sido descritos con un número menor de pacientes (34) o sometiendo los productos de amplificación a un paso posterior de hibridación con un cebador interno dentro de la secuencia amplificada (35).
En este estudio, el 12% de los individuos seropositivos no fue detectado a pesar de que se descartó la presencia de inhibición de la PCR. Estos resultados pueden deberse a alguna de las siguientes razones o a una combinación de las mismas: (i) niveles escasos de parasitemia, de forma que en los 500 µl de la muestra no estuviera presente ningún parásito (36); (ii) intermitencia en la parasitemia (37), o (iii) presencia de falsos positivos en las pruebas serológicas posiblemente por reacción cruzada con otros tripanosomátidos como Leishmania y T. rangeli (38).
En el caso de los cinco pacientes positivos por PCR y negativos por serología, en primer lugar llama la atención cómo estos pacientes fueron positivos tanto por la prueba TcH2AF/R, que amplifica blancos nucleares (21), como por la prueba S35/S36, que amplifica blancos mitocondriales (18), lo cual apoya la presencia de infección. Se ha reportado que pacientes chagásicos pueden presentar pruebas de xenodiagnóstico positivas y serologías negativas en algunos momentos de la enfermedad, especialmente en individuos recién infectados o con estado inmunológico comprometido (39), situación que puede explicar la presencia de PCR positiva en ausencia de anticuerpos IgG anti-T.cruzi. Este hallazgo también fue observado en el estudio reportado por Gutiérrez y colaboradores, en el que uno de los 25 individuos serológicamente negativos (4%) fue positivo por PCR (31); por Salomone y colaboradores, quienes encontraron que de 80 individuos seronegativos, 12 (15%) tenían PCR positiva y tres de éstos presentaban síntomas clínicos compatibles con la enfermedad de Chagas (40); por Gomes y colaboradores, quienes reportaron que 10 de 21 (47,6%) pacientes con serología negativa presentaron PCR y ensayos de lisis mediada por el complemento positivas (32), y por Avila y colaboradores, quienes encontraron PCR positiva en tres de cuatro pacientes seronegativos (35).
Adicionalmente, analizando los posibles factores de riesgo de contaminación durante el proceso de manipulación, extracción y amplificación del ADN, se tiene que cada uno de los pasos de las pruebas de PCR (extracción del ADN, reacción de pre-PCR, amplificación y electroforesis) se realizó en áreas diferentes y separadas, utilizando material anti-aerosol, blancos de reacción y controles negativos.
Es de especial interés el buen desempeño presen-tado por la prueba de PCR TcH2AF/R en el grupo de pacientes crónicos asintomáticos, en los cuales no se dispone de la ayuda que brinda la sintomato-logía para el diagnóstico basado en la tríada pruebas de laboratorio, sintomatología y epidemiología. Así mismo, en pacientes cardiópatas con síntomas compatibles con la enfermedad de Chagas, la PCR TcH2AF/R puede constituir una herramienta valiosa para descartar la infección por T. cruzi.
Teniendo en cuenta los resultados de este trabajo y las deficiencias de las pruebas serológicas convencionales en cuanto a la posibilidad de reacción cruzada con otros tripanosomátidos, lo que da lugar a falsos positivos y también a falsos negativos en casos de infección reciente o estado inmunológico comprometido, la PCR TcH2AF/R se perfila como una prueba diagnóstica promisoria de apoyo para la detección de T. cruzi en pacientes crónicos.
Con el fin de aumentar el poder de detección de la prueba de PCR se proponen las siguientes estrate-gias: (i) aumentar el volumen de sangre a analizar a una alícuota de 0,7-1 ml; (ii) preservar la muestra de sangre en clorhidrato de guanidina; (iii) tomar muestras seriadas con intervalos mínimos de dos a tres meses, e (iv) hibridar el producto de amplificación obtenido con un iniciador interno.
A pesar de los resultados prometedores de las pruebas de PCR, se requiere de un mayor número de estudios sobre consistencia y conformidad con la PCR TcH2AF/R y otras pruebas de PCR que utilizan blancos diferentes de amplificación, para postular su uso como patrón de oro en el diagnóstico de la enfermedad de Chagas. Adicionalmente, el requerimiento de equipo especializado y elevado costo de las pruebas de PCR hace difícil su uso común y de rutina, característica primordial de todo patrón de oro. No obstante, estudios paralelos del grupo de investigación proponen el uso de un "constructo" como patrón de oro, utilizando la PCR como prueba diagnóstica, además de los hallazgos clínicos y epidemiológicos (41).
Agradecimientos
Los autores expresan su agradecimiento a la Unidad de Electrofisiología y Cardiología del Hospital Universitario San Ignacio (HUSI), a la Clínica Shaio y al Centro de Salud del municipio de Santana, departamento de Boyacá.
Conflicto de intereses
Los autores manifestamos expresamente que durante la realización del presente trabajo no existió conflicto de intereses que pudiera afectar los resultados obtenidos.
Financiación
Este estudio fue financiado por la Vicerrectoría Académica de la Pontificia Universidad Javeriana. Registro de proyecto 1702.
Correspondencia:
Concepción Puerta, Laboratorio de Parasitología Molecular, Departamento de Microbiología, Facultad de Ciencias, Pontificia Universidad Javeriana, Bogotá D.C., Colombia.
Teléfono:(+571) 3208320 ext. 4024, fax (571) 3208320 ext. 4021
cpuerta@javeriana.edu.co
Referencias
1. World Health Organization. Control of Chagas Disease. Second report of the WHO Expert Committee. Technical Report 2002. Series 905. Geneva: WHO;2002. p.39-40. [ Links ]
2. Moncayo A. Chagas disease: current epidemiological trends after the interruption of vectorial and transfusional transmission in the Southern Cone countries. Mem Inst Oswaldo Cruz 2003;98:577-91. [ Links ]
3. Macedo AM, Machado CR, Oliveira RP, Pena SD. Trypanosoma cruzi: genetic structure of populations and relevance of genetic variability to the pathogenesis of Chagas disease. Mem Inst Oswaldo Cruz 2004;99:1-12. [ Links ]
4. Huete-Perez JA, Flores-Obando RE, Ghedin E, Caffrey CR. Genomic and proteomic approaches for Chagas disease: critical analysis of diagnostic methods. Expert Rev Mol Diagn 2005;5:521-30. [ Links ]
5. DAvila DA, Gontijo ED, Lages-Silva E, Meira WS, Chiari E, Galvao LM. Random amplified polymorphic DNA profiles of Trypanosoma cruzi isolates from chagasic patients with different clinical forms. Parasitol Res 2006;98:455-61. [ Links ]
6. Machado CR, Augusto-Pinto L, McCulloch R, Teixeira SM. DNA metabolism and genetic diversity in Trypanosomes. Mutat Res 2006;612:40-57. [ Links ]
7. Saravia NG, Holguin AF, Cibulskis RE, DAlessandro A. Divergent isoenzyme profiles of sylvatic and domiciliary Trypanosoma cruzi in the eastern plains, piedmont, and highlands of Colombia. Am J Trop Med Hyg 1987;36:59-69. [ Links ]
8. Rodríguez P, Montilla M, Nicholls RS, Zarante I, Puerta, C. Isoenzymatic characterization of Colombian strains of Trypanosoma cruzi. Mem Inst Oswaldo Cruz 1998;93:739-40. [ Links ]
9. Marquez E, Arcos-Burgos M, Triana O, Moreno J, Jaramillo N. Clonal population structure of Colombian sylvatic Trypanosoma cruzi. J Parasitol 1998;84: 1143-9. [ Links ]
10. Triana O, Jaramillo N, Moreno J. Genetic variability of Colombian populations of Trypanosoma cruzi and Trypanosoma rangeli. Biol Res 1999;32:1-10. [ Links ]
11. Montilla MM, Guhl F, Jaramillo C, Nicholls S, Barnabe C, Bosseno MF, et al. Isoenzyme clustering of Trypanosomatidae Colombian populations. Am J Trop Med Hyg 2002;66:394-400. [ Links ]
12. Rodríguez P, Escalante M, Díez H, Cuervo C, Montilla M, Nicholls RS, et al. Estudio de la variabilidad de seis cepas colombianas de Trypanosoma cruzi mediante polimorfismos de longitud de fragmentos de restricción (RFLP) y amplificación aleatoria de ADN polimórfico (RAPD). Biomédica 2002;22:263-71. [ Links ]
13. Mejia AM, Triana O. Análisis por LSSP-PCR de la variabilidad genética de Trypanosoma cruzi en sangre y órganos de ratones infectados. Biomédica 2005;25:76-86. [ Links ]
14. DAlessandro A, Saravia N. Trypanosoma rangeli. En: Gilles HM, editor. Protozoal diseases. London: Arnold Press;1999. p.398-412. [ Links ]
15. Guhl F, Vallejo GA. Trypanosoma (Herpetosoma) rangeli Tejera, 1920: an updated review. Mem Inst Oswaldo Cruz 2003;98:435-42. [ Links ]
16. Vallejo GA. Estudios comparativos entre las secuencias de kDNA de Trypanosoma cruzi y Trypanosoma rangeli y su aplicación en el diagnóstico molecular de la tripanosomiasis americana. Actual Biol 1998;20:43-56. [ Links ]
17. Guhl F, Jaramillo C, Carranza JC, Vallejo GA. Molecular characterization and diagnosis of Trypanosoma cruzi and Trypanosoma rangeli. Arch Med Res 2002;33:362-70. [ Links ]
18. Vallejo GA, Gulh F, Chiari E, Macedo AM. Species specific detection of Trypanosoma cruzi and Trypanosoma rangeli in vector and mammalian hosts by polymerase chain reaction amplification of kinetoplast minicircle DNA. Acta Trop 1999;72:203-12. [ Links ]
19. Vargas N, Souto RP, Carranza JC, Vallejo GA, Zingales B. Amplification of a specific repetitive DNA sequence for Trypanosoma rangeli identification and its potential application in epidemiological investigations. Exp Parasitol 2000;96:147-59. [ Links ]
20. Nitz N, Gomes C, de Cassia Rosa A, DSouza-Ault MR, Moreno F, Lauria-Pires L, et al. Heritable integration of kDNA minicircle sequences from Trypanosoma cruzi into the avian genome: insights into human Chagas disease. Cell 2004;118:175-86. [ Links ]
21. Pavía P, Cuervo C, Montilla M, Nicholls S, Puerta C. Diseño y estandarización de una prueba de PCR para la detección específica de Trypanosoma cruzi. Infectio 2003;7:129-36. [ Links ]
22. Beltrán M, Duque S, Guhl F, Herrera CP, López MC, Moreno AL, et al. Prueba de ELISA y prueba de inmunofluorescencia indirecta (IFI). En: Guhl F, Nicholls RS, editores. Manual de procedimientos para el diagnóstico de la enfermedad de Chagas. Bogotá: Ministerio de Salud; 2001. p.32-48. [ Links ]
23. Sambrook JF, Russell DW. Gel electrophoresis of DNA. En: Molecular cloning A laboratory manual. Tercera edición. Cold Spring Harbor, New York: Cold Spring Harbor Laboratory Press; 2001.p.5.1-17. [ Links ]
24. Puerta C, Martin J, Alonso C, Lopez MC. Isolation and characterization of the gene encoding histone H2A from Trypanosoma cruzi. Mol Biochem Parasitol 1994;64:1-10. [ Links ]
25. Sackett D, Haynes R, Guyatt G, Tugwell P. Selección de pruebas diagnosticas. En: Epidemiologia Clínica. Segunda Edición. México, D.F.: Editorial Médica Panamericana; 1998. p.64-7. [ Links ]
26. Wincker P, Britto C, Pereira JB, Cardoso MA, Oelemann W, Morel CM. Use of a simplified polymerase chain reaction procedure to detect Trypanosoma cruzi in blood samples from chronic chagasic patients in a rural endemic area. Am J Trop Med Hyg 1994;51:771-7. [ Links ]
27. Thomas MC, Olivares M, Escalante M, Maranon C, Montilla M, Nicholls S, et al. Plasticity of the histone H2A genes in a Brazilian and six Colombian strains of Trypanosoma cruzi. Acta Trop 2000;75:203-10. [ Links ]
28. Vázquez MP, Schijman AG, Levin MJ. A short interspersed repetitive element provides a new 3 acceptor site for trans-splicing in certain ribosomal P2 beta protein genes of Trypanosoma cruzi. Mol Biochem Parasitol 1994;64:327-36. [ Links ]
29. Brisse S, Verhoef J, Tibayrenc M. Characterisation of large and small subunit rRNA and miniexon genes further support the distinction of six Trypanosoma cruzi lineages. Int J Parasitol 2001;31:1218-26. [ Links ]
30. Urrea DA, Carranza JC, Cuba CA, Gurgel-Goncalves R, Guhl F, Schofield CJ, et al. Molecular characterisation of Trypanosoma rangeli strains isolated from Rhodnius ecuadoriensis in Peru, R. colombiensis in Colombia and R. pallescens in Panama, supports a co-evolutionary association between parasites and vectors. Infect Genet Evol 2005;5:123-9. [ Links ]
31. Gutierrez R, Angulo VM, Tarazona Z, Britto C, Fernandes O. Comparison of four serological tests for the diagnosis of Chagas disease in a Colombian endemic area. Parasitology 2004;129:439-44. [ Links ]
32. Gomes ML, Galvao LM, Macedo AM, Pena SD, Chiari E. Chagas disease diagnosis: comparative analysis of parasitologic, molecular, and serologic methods. Am J Trop Med Hyg 1999;60:205-10. [ Links ]
33. Britto C, Cardoso MA, Vanni CM, Hasslocher-Moreno A, Xavier SS, Oelemann W, et al. Polymerase chain reaction detection of Trypanosoma cruzi in human blood samples as a tool for diagnosis and treatment evaluation. Parasitology 1995;110:241-7. [ Links ]
34. Wincker P, Bosseno MF, Britto C, Yaksic N, Cardoso MA, Morel CM, et al. High correlation between Chagas disease serology and PCR-based detection of Trypanosoma cruzi kinetoplast DNA in Bolivian children living in an endemic area. FEMS Microbiol Lett 1994;124:419-23. [ Links ]
35. Avila HA, Pereira JB, Thiemann O, De Paiva E, DeGrave W, Morel CM, et al. Detection of Trypanosoma cruzi in blood specimens of chronic chagasic patients by polymerase chain reaction amplification of kinetoplast minicircle DNA: comparison with serology and xenodiagnosis. J Clin Microbiol 1993;31:2421-6. [ Links ]
36. Junqueira AC, Chiari E, Wincker P. Comparison of the polymerase chain reaction with two classical parasitological methods for the diagnosis of Chagas disease in an endemic region of north-eastern Brazil. Trans R Soc Trop Med Hyg 1996;90:129-32. [ Links ]
37. Castro AM, Luquetti AO, Rassi A, Rassi GG, Chiari E, Galvao LM. Blood culture and polymerase chain reaction for the diagnosis of the chronic phase of human infection with Trypanosoma cruzi. Parasitol Res 2002;88:894-900. [ Links ]
38. Marcon GE, Andrade PD, de Albuquerque DM, Wanderley Jda S, de Almeida EA, Guariento ME, et al. Use of a nested polymerase chain reaction (N-PCR) to detect Trypanosoma cruzi in blood samples from chronic chagasic patients and patients with doubtful serologies. Diagn Microbiol Infect Dis 2002;43:39-43. [ Links ]
39. Luquetti AO. Megaesofago e anticorpos anti–Trypanosoma cruzi. Rev Goiana Med 1987;33:1-16. [ Links ]
40. Salomone OA, Basquiera AL, Sembaj A, Aguerri AM, Reyes ME, Omelianuk M, et al. Trypanosoma cruzi in persons without serologic evidence of disease, Argentina. Emerg Infect Dis 2003;9:1558-62. [ Links ]
41. Vacca M, Mercado M. Determinación de las características operativas de las pruebas serológicas con cepas colombianas de Trypanosoma cruzi utilizadas para el diagnóstico de la enfermedad de Chagas. (Tesis de Maestría en Epidemiología Clínica). Bogotá D.C.: Unidad de Epidemiología Clínica, Hospital Universitario San Ignacio, Pontificia Universidad Javeriana; 2006. [ Links ]














