Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.28 no.2 Bogotá Apr./June 2008
ARTÍCULO ORIGINAL
Recibido: 08/11/07; aceptado:06/03/08
Introducción. El complejo asociado al receptor de las células T está constituido por las moléculas CD3 (δ, γ, ε) y las cadenas ζ, todas proteínas transmembrana esenciales para la transducción de señales durante la activación del linfocito y la respuesta inmune, así como durante el desarrollo de los timocitos.
Objetivo. En este trabajo se buscaba determinar la estructura primaria de la cadena CD3ε, en el mono del nuevo mundo, Aotus nancymaae.
Materiales y métodos. A partir del ARN total obtenido de células mononucleares de sangre periférica, se amplificó la molécula de CD3ε, luego se clonó en un vector y, finalmente, se secuenció.
Resultados. Se presenta la secuencia deducida de aminoácidos de la cadena CD3ε de A. nancymaae. Se estableció una identidad de 84% y de 76% en la secuencia nucleotídica y la de aminoácidos, respectivamente, con su contraparte humana. La molécula de Aotus muestra bastante variabilidad en la región extracelular y bastante conservación en la intracelular.
Están conservadas subregiones del ectodominio que son importantes para el plegamiento de la molécula, así como para la asociación con las otras cadenas del complejo.
Conclusiones. La estructura primaria determinada aquí sugiere que la proteína de Aotus tiene una funcionalidad similar y que los pasos iniciales de activación de la célula T se suceden como en los humanos; por otra parte, la gran variabilidad encontrada en el ectodominio, permite explicar por qué algunos anticuerpos monoclonales dirigidos contra el complejo CD3 de humano no reconocen estas estructuras en el Aotus.
Palabras clave: sistema inmune, receptores del antígeno de célula T, antígenos CD3, proteínas adaptadoras transductoras de señales.
Characterizing the CD3 epsilon chain from the New World primate Aotus nancymaae
Introduction. The T-cell receptor (TCR)-associated complex, CD3 (d, g, e) and z-chains are essential transmembrane proteins for signal transduction during T cell activation and immune response, as well as during thymocyte development.
Objective. This work established the CD3ε-chain primary structure for the New World owl monkey Aotus nancymaae.
Materials and methods. Total RNA was isolated from peripheral blood mononuclear cells; CD3ε molecule was amplified, cloned and sequenced.
Results. The CD3ε amino acid sequence was deduced for the owl monkey Aotus nancymaae.
It has an identity for nucleotide and amino acid sequences with the human counterpart of 84% and 76%, respectively. As described in other species, the Aotus CD3-e molecule is very variable in the extracellular region and greatly conserved in the intracellular domain. Even though high variability occurs in the CD3ε-extracellular domain, the subregions involved in ectodomain folding are conserved.
Conclusions. The primary structure suggested that the Aotus protein has a functional role similar to that of humans, and that the initial T-cell activation steps are also similar. However, the great variation observed in CD3ε-extracellular region in humans in contrast to the Aotus (especially in areas that are surface-exposed) indicated that some monoclonal antibodies against the human CD3 complex will not recognize these Aotus determinants.
Key words: immune system; receptors, antigen, T-cell; CD3 antigens; adaptor proteins, signal transducing.
Los monos del nuevo mundo Aotus sp. son usados como modelo experimental para estudiar algunas enfermedades parasitarias humanas, como, por ejemplo, el paludismo producido por Plasmodium falciparum y Plasmodium vivax, debido a que estos monos son susceptibles a la infección (1,2).
El modelo tiene claras ventajas (3) pero, también, importantes aspectos desfavorables; los primates del viejo y del nuevo mundo divergieron hace, aproximadamente, 58 millones de años; durante ese tiempo se han venido acumulando cambios a diferentes niveles, incluido el molecular. Esto resulta relevante no sólo para las moléculas del complejo mayor de histocompatibilidad clases I y II, las cuales coevolucionan con los patógenos (4,5), sino también, para moléculas involucradas en la transducción de señales en las células del sistema inmune. Las variaciones a este nivel podrían ser responsables de inducir respuestas inmunitarias diferentes y sugerir que una vacuna que resultara exitosa en Aotus, pudiera no ser efectiva en humanos (6). Los puntos anteriores ilustran la importancia de caracterizar el sistema inmune de estos primates.
El reconocimiento específico del antígeno en el linfocito T es llevado a cabo por el receptor de las células T, mientras que el complejo CD3 (δ, γ, ε) y las cadenas ζ (complejo asociado a dicho receptor), son responsables de la transmisión de las señales (7,8). Todas las cadenas asociadas al receptor contienen en sus regiones intra-celulares los llamados motivos de activación de los inmunorreceptores basados en tirosina ( Immunoreceptor Tyrosine-based Activation Motif, ITAM); estos motivos son fosforilados por cinasas de proteínas de la familia Src durante las etapas iniciales de activación de la célula T (9,10).
Los ITAM fosforilados se constituyen en sitios de asociación para proteínas adaptadoras que portan los dominios SH2 y para otras enzimas (11). Estas señales son importantes, no solamente durante la activación del linfocito T, sino también durante el desarrollo de los timocitos, especialmente en la transición de la células doble
negativas a las doble positivas (12). En general, estos ITAM son responsables del adecuado funcionamiento de la célula T y, por ende, de la respuesta inmune adaptativa (8).
Recientemente se ha demostrado que se induce un cambio estructural en el complejo CD3 cuando las células T son estimuladas con anticuerpos anti-CD3; esta modificación expone una región en el dominio citoplasmático de la cadena e, que es rica en poliprolina y que se vuelve un sitio de asociación para la proteína adaptadora Nck. Este cambio estructural es inducido también en timocitos durante la activación del receptor. La asociación entre la proteína Nck y la cadena e precede a la fosforilación de los ITAM y es esencial para la activación del linfocito T (13-15).
La estructura primaria de la cadena CD3ε ha sido determinada en el humano (16), en ratones (17); primates del viejo mundo: Macaca fascicularis (18), Macaca mulatta ( GenBank, acceso número XM_001097204) y Pan troglodytes (GenBank, acceso número XM_001160645); en el primate del nuevo mundo Callitrix jaccus ( GenBank, acceso número DQ189218) y en otras especies. Las proteínas CD3 constituyen una familia de genes (cadenas d, g , e) que en humanos se han mapeado en el cromosoma 11 en la banda q23. Las comparaciones de secuencias indican que las moléculas δ, γ son altamente homólogas tanto en humanos como en ratones, mientras que e está menos relacionada (17,19-21).
La caracterización de los distintos componentes involucrados en la activación de la célula T, así como la evaluación de la función celular, son elementos importantes para entender la inmunidad del Aotus y, de esta forma, establecer la utilidad del modelo animal en el estudio de enfermedades como el paludismo. Recientemente hemos caracterizado algunas moléculas de Aotus sp. Que son importantes en la transducción de señales y hemos realizado estudios funcionales con células tendientes a comparar los respuestas inmunes(22-24).
En este reporte presentamos la estructura primaria de la proteína CD3ε de A. nancymaae, la cual se ha revelado como una pieza central durante los pasos iniciales de la activación de la célula T.
Materiales y métodos
Muestras biológicas
Las células mononucleares de sangre periférica, se obtuvieron de siete monos A. nancymaae capturados en la región de la amazonia colombiana; todas las muestras recibidas (12) se probaron para la presencia de filarias y solamente se procesaron aquellas muestras libres de parásitos. Las microfilarias se observaron directamente en el microscopio, colocando una gota de sangre total en un portaobjetos.
Las células mononucleares de sangre periférica se aislaron por un gradiente de separación en Ficoll Hypaque (Sigma, St Louis, Mo). La viabilidad celular se determinó por la exclusión del colorante azul de tripano. A muestras con más de dos millones de células se les extrajo el ARN total.
Todos los animales usados en esta investigación se manejaron de acuerdo con los protocolos del Ministerio de la Protección Social (8430/1993), bajo la supervisión de Corpoamazonía.
Extracción del ARN total y síntesis de cADN
El ARN total se aisló de las células mononucleares de sangre periférica de A. nancymaae usando el método del trizol (Invitrogen, Life Technologies, Carlsbad, CA) y se almacenó a -70 °C. En las mismas condiciones se aisló también ARN total de humano, para ser usado como control. El cADN fue sintetizado utilizando el sistema One-step RT-PCR (Invitrogen, Life Technologies, Carlsbad, CA) y los iniciadores específicos AGTTCTGGGCCTC-TGCCTCT (sentido) y ATGCGTCTCTGATTCAGG-CC (antisentido). El perfil de síntesis usado fue: 30 minutos a 55 °C; 2 minutos a 94 °C; 40 X (15 s a 94 °C, 30 s a 55 °C, 60 s a 68 °C); 5 minutos a 68 °C.
Ligación, secuenciación y análisis
Los productos de amplificación se extrajeron de geles de agarosa de bajo punto de fusión al 1% utilizando la enzima agar-ACE (Promega, Madison) y se ligaron en el vector pGEM-T easy (Promega, Madison). Posteriormente, los plásmidos recombinantes se extrajeron de las bacterias transformadas usando un sistema miniprep (Promega, Madison).
Todas las secuencias fueron realizadas por Macrogen Inc. (www.macrogen.com). Se llevaron a cabo por el método de Sanger, usando terminadores Bigdye, en condiciones cíclicas, los productos se purificaron por precipitación etanólica y se corrieron y leyeron en el secuenciador automatizado ABI3730XL; en la reacción se usaron iniciadores universales PUC-M13 dirigidos contra el vector.
Las secuencias de nucleótidos se editaron con el programa Chromas (versión 1.45) y la secuencia de aminoácidos se dedujo con el Generunner (versión 3.05); los alineamientos se hicieron con el ClustalX (versión 1.83) y se editaron con el Genedoc (versión 2.6.001); la matriz de sustitución PAM 250 fue usada para hacer los cálculos de homología entre las secuencias de aminoácidos. El árbol de Minimum-evolution (ME), Maximum likelihood (ML) y Parsimony (Pars) se construyó utilizando: TREEFINDER (versión mayo de 2006) y DNAPARS, que están incluidos en el paquete PHYLYP (versión 3.66, agosto de 2006).
Una aproximación bayesiana se utilizó para inferir las relaciones filogenéticas, programa Mr. Bayes (versión 3.1.2).
Resultados
Obtención del cADN de la cadena CD3ε de A. nancymaae
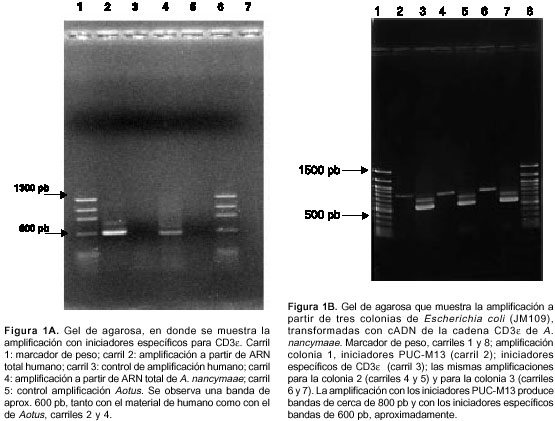
Los iniciadores utilizados para la amplificación de la cadena CD3ε de Aotus, se diseñaron con base en la secuencia de CD3ε de humano; el iniciador sentido está dirigido contra la región del péptido líder y el antisentido contra la región citoplasmática, empezando cuatro nucleótidos corriente arriba del codón de parada. Los productos de amplificación se observaron en gel de agarosa (figura 1A). Una banda de aproximadamente 600 pb se extrajo del gel y se ligó en el vector pGEM-T easy. Luego de la ligación, la presencia del inserto se confirmó por PCR, utilizando iniciadores universales contra el vector y los específicos contra la cadena ε (figura 1B).
Secuencia de la cadena CD3ε de A. nancymaae
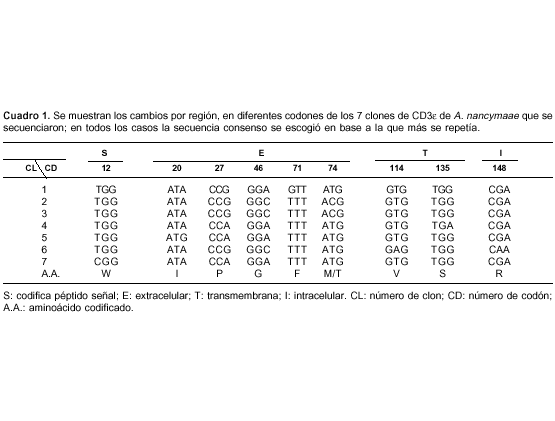
La secuencia de la cadena CD3ε de A. nancymaae correspondiente a un consenso de siete clones secuenciados independientes se reportó en el GenBank y recibió el número de acceso EF547186; en el cuadro 1 se muestran los cambios en los diferentes codones en los siete clones secuenciados. Con base en la secuencia consenso de nucleótidos, se predijo la secuencia de aminoácidos y se realizaron los respectivos alineamientos. Las comparaciones realizadas entre las secuencias de CD3ε de Aotus y las de otros primates mostraron los siguientes porcentajes de identidad en secuencia de nucleótidos y de aminoácidos respectivamente: 84% y 76% con Homo sapiens; 80% y 71% con Pan troglodytes; 86% y 77% con Macaca fascicularis; 87% y 78% con Macaca mulatta; 97% y 96% con Callitrix jaccus.
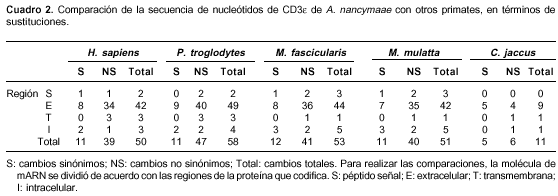
En el cuadro 2 se muestra la sustitución de codones entre estos mismos primates; para hacer esta comparación, la región codificadora de la molécula de mARN se dividió de acuerdo con las regiones en la proteína. Además de estos cambios, se encuentra que H. sapiens y P. troglodytes tienen una inserción de ocho codones y P. troglodytes una deleción de un codón con respecto a Aotus. Los otros tres primates presentan también la deleción de un codón; tanto la inserción como las deleciones se localizan en el segmento que determina el ectodominio.
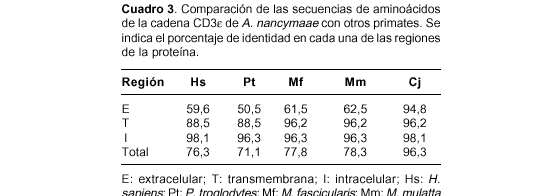
En el cuadro 3 se muestran los porcentajes de identidad por regiones al comparar las proteínas de los mismos primates. La proteína de Aotus tiene una gran variabilidad en la región extracelular con respecto a los primates del viejo mundo, mientras que las regiones transmembrana e intracelular exhiben poca variación.
Por otra parte, residuos que se han descrito como importantes para la funcionalidad y para la estructuración de la cadena CD3ε están muy conservados en todas las regiones de la proteína de Aotus; se pueden destacar (V, V, C, I, L, G, Y, C, R, C, N, C, E) en la región extracelular; (D) en la región transmembrana y (Y, Y) en el citoplasma.
En este mismo sentido, tanto el ITAM como la región rica en poliprolina de la cadena CD3ε tienen una identidad del 100% con respecto a los otros primates comparados; en la figura 2 se muestra un alineamiento entre las proteínas de humano y de Aotus, se indican los aminoácidos diferentes y se resaltan algunas subregiones y residuos importantes.

Discusión
En este trabajo se reporta la secuencia deducida de aminoácidos de la cadena CD3ε del mono del nuevo mundo, A. nancymaae; la secuencia de la proteína de Aotus mostró un porcentaje más alto de homología con la proteína de C. jaccus, el otro primate del nuevo mundo, en contraste con los del viejo mundo. Así mismo, la secuencia de aminoácidos de la cadena CD3ε de Aotus mostró una variabilidad significativa en la región extracelular y una gran conservación en la región intracelular (cuadros 2 y cuadro3). Este hallazgo está en concordancia con reportes previos en otros organismos (17-20) (número de acceso al GenBank XM_001097204; XM_001160645 y DQ189218) y también para otras moléculas transmembrana del Aotus (23,25-27). A pesar de la gran variabilidad encontrada en la porción extracelular de la proteína CD3ε de A. nancymaae, los residuos considerados esenciales para la estructura tridimensional están conservados (30-32).
Se ha demostrado experimentalmente que los dímeros eg de ratón se asocian con el receptor de las células T humano y la cadena d humana se asocia con la cadena e y el receptor de las células T de ratón (12,30), lo cual indica que sub-regiones en el ectodominio están conservadas entre estos organismos. Con un grado de homología más alto en el ectodominio entre humano y Aotus se puede suponer que estas asociaciones son también posibles. En el complejo receptor de las células T las superficies expuestas son limitadas; se ha visto que diferentes anticuerpos monoclonales específicos para el complejo CD3 humano pueden reconocer el mismo epítopo de conformación producido por los dímeros ε δ y εγ (31-33).
La predicción de la estructura secundaria para el ectodominio de la CD3ε de Aotus (28,29) indica una organización de hoja plegada β similar a las reportadas en humano y en ratón (30,31); en la figura 3 se muestra un alineamiento del ectodominio entre diferentes primates y el ratón; se puede observar la conservación de residuos en las subregiones responsables del plegamiento de la proteína (A, B, C, E, F y G) y variabilidad de residuos en las interregiones (-A, AB, BC, CE y FG), de acuerdo con las estructuras reportadas para los dímeros εγ y ε δ de humano (31,32) y εγ de ratón (30); las interregiones, especialmente BC, CE y FG son las zonas expuestas.
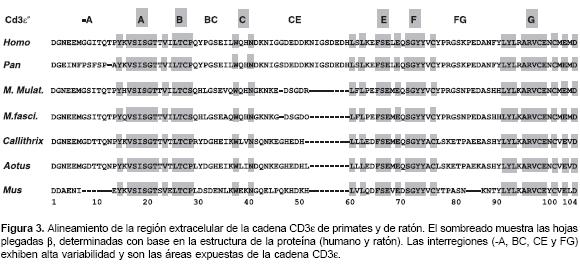
Los estudios en monos Cinomolgus y Rhesus han demostrado que la reactividad del anticuerpo monoclonal FN18, el cual es específico para primates no humanos, depende de los aminoácidos ubicados en las posiciones 35, 45 y 50 los cuales están ubicados en las interregiones BC y CE de la cadena CD3ε (18,34); los linfocitos T de monos con alta reactividad al FN18 tienen los aminoácidos V, E y R en estas posiciones, mientras que aquéllos con baja reactividad son homocigotos en CD3ε, con algunos de estos cambios: V35A, E45G y R50Q.
Es interesante notar que en los humanos en la posición 45 se encuentra un residuo G y en Aotus, un residuo E; en los humanos en la posición 50 hay un E y en Aotus esta posición corresponde al inicio de la deleción. Esto puede sugerir que los anticuerpos que reconozcan las zonas que involucren estos residuos en los humanos, podrían no reconocer la cadena CD3ε de Aotus. De hecho, en los estudios de citometría de flujo en los cuales se han utilizado células mononucleares de sangre periférica de Aotus y anticuerpos monoclonales contra CD3 de humano, no se ha podido encontrar reactividad del anticuerpo (no se presentan los resultados). Los anticuerpos monoclonales específicos contra el CD3 de humano, OKT3 y UCHT1 reconocen un epítopo de conformación producido por aminoácidos ubicados en las interregiones BC, CE y FG de la cadena CD3ε (31,32). La estructura primaria de CD3ε de A.
nancymaae que se ha determinado aquí, muestra una gran variación precisamente en estas interregiones, posiciones 29, 31, 33, 36, 42, 44, 45, 47, 50, 51, 52-59 ( gap), 77, 78, 79, 80, 81, 82, 83, 85, 87 y 88 (figura 3).
En la región transmembrana se encontró en todos los primates comparados un alto porcentaje de identidad en la cadena CD3ε, incluido el residuo ácido (D), esencial para la interacción con residuos básicos que se encuentran en las cadenas α y β del receptor de las células T y que son responsables de la organización del complejo multimérico (35,36).
El dominio intracelular de la cadena CD3ε también muestra un alto grado de conservación entre los diferentes organismos, incluido el Aotus. Reportes recientes han enfatizado los múltiples papeles que juega el complejo CD3, especialmente la cadena e, en la señalización del linfocito T. Se ha planteado que los dímeros ge/de permiten la señalización normal en la célula T en ausencia de los ITAM de la cadena ζ (37). La fosforilación selectiva de la cadena e por parte de la cinasa de proteínas p56Lck sugiere un papel potencial en la regulación de la señalización de la célula T a través del complejo receptor de las células T, probablemente por el reclutamiento diferencial de moléculas de señalización o de su activación (38).
Se ha planteado que un cambio estructural que está implicado en la señalización del linfocito T se induce en el complejo CD3 después de la activación del receptor de las células T (39); sin embargo, experimentalmente se ha demostrado también que la asociación Nck-e no se necesita para la funcionalidad ni para el desarrollo de la célula T (40). En conjunto, la evidencia experimental sustenta la importancia del complejo CD3 en la funcionalidad del linfocito T y la necesidad de su conservación.
Se han reportado variaciones intraespecíficas en algunos aminoácidos en la región extracelular de la cadena e de humano, chimpancé (interregión –A), M. fascicularis (interregión CE) (18) y en M.
mulatta (34), lo cual indica que CD3ε es una proteína polimórfica. Siete clones independientes de CD3ε de A. nancymaae se secuenciaron en el presente trabajo, encontrándose algunos cambios de nucleótidos principalmente en la región extracelular; sin embargo, en todos los casos estos cambios fueron sinónimos. Es probable que sea necesario secuenciar un mayor número de muestras de la cadena e de Aotus, para poder establecer si se trata también de una proteína polimórfica.
Un árbol filogenético con base en las secuencias de aminoácidos de CD3ε de diferentes vertebrados agrupó al Aotus con los otros primates y en estrecha cercanía de C. jaccus, el otro primate del nuevo mundo.
Los resultados presentados aquí muestran una estructura primaria muy similar para la cadena CD3ε de A. nancymaae, no solamente en la comparación con humanos, sino también con otros organismos. Esta similitud indica una importante restricción funcional para esta molécula y los eventos de señalización intracelular mediados por ella. Por otra parte, la gran variación encontrada en la región extracelular en la comparación entre humanos y Aotus, sugiere que el uso de anticuerpos contra células T humanas, para estudiar estas mismas células en Aotus, puede llevar a interpretaciones erróneas de los resultados obtenidos.
Conflicto de intereses
Los autores declaran que no tienen conflictos de intereses que puedan llegar a afectar la publicación de este manuscrito.
Financiación
La financiación para la presente investigación fue cubierta por la Universidad Nacional de Colombia, sede Bogotá.
Correspondencia:
Jean Paul Vernot, Laboratorio de Fisiología Celular y Molecular, Facultad de Medicina, Universidad Nacional de Colombia, Ciudad Universitaria, Bogotá, D. C., Colombia
Teléfono: 316 5000, extensión 15057; fax: 316 5308
Referencias
1. Collins WE. The owl monkey as a model for malaria. En: Baer JF, Weller RE, Kakoma J, editors. Aotus the owl monkey. San Diego: Academic Press; 1994. p. 217-44. [ Links ]
2. Gynsin J. Animal models: primates. En: Sherman IW, editor. Malaria: parasite biology, pathogenesis and pro-tection. Washington, DC: ASM Press; 1998. p. 419-39. [ Links ]
3. Stowers AW, Miller L H. Are trials in new world monkeys on the critical path for blood-stage malaria vaccine development? Trends Parasitol. 2001;17:415-9. [ Links ]
4. Vogel TU, Evans DT, Urvater JA, OConnor DH, Hughes AL, Watkins DI. Major histocompatibility complex class I genes in primates: co-evolution with pathogens. Immunol Rev. 1999;167:327-37. [ Links ]
5. Bontrop RE, Otting N, de Groot NG, Doxiadis GG. Major histocompatibility complex class II polymorphisms in primates. Immunol Rev. 1999;167:339-50. [ Links ]
6. Heppner DG, Cummings JF, Ockenhouse C, Kester KE, Lyon JA, Gordon DM. New world monkey efficacy trials for malaria vaccine development: critical path or detour? Trends Parasitol. 2001;17:419-25. [ Links ]
7. Cantrell D. T cell antigen receptor signal transduction pathways. Annu Rev Immunol. 1996;14:259-74. [ Links ]
8. Germain RN, tefanová I. The dynamics of T cell receptor signaling: complex orchestration and key roles of tempo and cooperation. Annu Rev Immunol. 1999;17:467-522. [ Links ]
9. Zamoyska R, Basson A, Filby A, Legname G, Lovatt M, Seddon B. The influence of the src-family kinases, Lck and Fyn, on T cell differentiation, survival and activation. Immunol Rev. 2003;191:107-18. [ Links ]
10. Pitcher LA, van Oers NS. T-cell receptor signal transmission: who gives an ITAM? Trends Immunol. 2003;24:554-60. [ Links ]
11. Werlen G, Palmer E. The TCR signalosome: a dynamic structure with expanding complexity. Curr Opin Immunol. 2002;14:299-305. [ Links ]
12. Pan Q, Brodeur JF, Drbal K, Dave VP. Different role for mouse and human CD3ä/å heterodimer in preT cell receptor (preTCR) function: Human CD3d/e heterodimer restores the defective preTCR function in CD3g- and CD3 gd-deficient mice. Mol Immunol. 2006;43:1741-50. [ Links ]
13. Gil D, Schamel WW, Montoya M, Sánchez-Madrid F, Alarcón B. Recruitment of Nck by CD3 reveals a ligand-induced conformational change essential for T cell receptor signaling and synapse formation. Cell. 2002;109:901-12. [ Links ]
14. Alarcón B, Gil D, Delgado P, Schamel WW. Initiation of TCR signaling: regulation within CD3 dimers. Immunol Rev. 2003;191:38-46. [ Links ]
15. Gil D, Schrum AG, Alarcón B, Palmer E. T cell receptor engagement by peptide-MHC ligands induces a conformational change in the CD3 complex of thymocytes. J Exp Med. 2005;201:517-22. [ Links ]
16. Gold DP, Puck JM, Pettey CL, Cho M, Coligan J, Woody JN, et al. Isolation of cDNA clones encoding the 20K non-glycosylated polypeptide chain of the human T-cell receptor/T3 complex. Nature. 1986;321: 431-4. [ Links ]
17. Gold DP, Clevers H, Alarcon B, Dunlap S, Novotny J, Williams AF, et al. Evolutionary relationship between the T3 chains of the T-cell receptor complex and the immunoglobulin supergene family. Proc Natl Acad Sci USA. 1987;84:7649-53. [ Links ]
18. Uda A, Tanabayashi K, Mukai R, Yachi M, Nam KH, Yamada A. CD3 polymorphism in cynomolgus monkeys (Macaca fascicularis). J Med Primatol. 2001;30:141-7. [ Links ]
19. Clevers H, Dunlap S, Saito H, Georgopoulos K, Wileman T, Terhorst C. Characterization and expression of the murine CD3-e gene. Proc Natl Acad Sci USA. 1988;85:8623-7. [ Links ]
20. Clevers HC, Dunlap S, Wileman TE, Terhorst C. Human CD3-e gene contains three miniexons and is transcribed from a non-TATA promoter. Proc Natl Acad Sci USA. 1988;85:8156-60. [ Links ]
21. Tunnacliffe A, Olsson C, Buluwela L, Rabbitts TH. Organization of the human CD3 locus on chromosome 11. Eur J Immunol. 1988;18:1639-42. [ Links ]
22. Pinzón-Charry A, Vernot JP, Rodríguez R, Patarroyo ME. Proliferative response of peripheral blood lymphocytes to mitogens in the owl monkey Aotus nancymae. J Med Primatol. 2003;32:31-8. [ Links ]
23. Montoya GE, Vernot JP, Patarroyo ME. Comparative analysis of CD45 proteins in primate context: owl monkeys vs humans. Tissue Antigens. 2004;64:165-72. [ Links ]
24. Vernot JP, Pérez-Quintero LA, Perdomo-Arciniegas AM, Quijano S, Patarroyo ME. Herpesvirus saimiri immortalization of Aotus T lymphocytes specific for an immunogenically modified peptide of Plasmodium falciparum merozoite surface antigen 2. Immunol Cell Biol. 2005;83:67-74. [ Links ]
25. Niño-Vásquez JJ, Vogel D, Rodríguez R, Moreno A, Patarroyo ME, Pluschke G, et al. Sequence and diversity of DRB genes of Aotus nancymaae, a primate model for human malaria parasites. Immunogenetics. 2000;51:219-30. [ Links ]
26. Díaz D, Naegeli M, Rodríguez R, Niño-Vásquez JJ, Moreno A, Patarroyo ME, et al. Sequence and diversity of MHC DQA and DQB genes of the owl monkey Aotus nancymaae. Immunogenetics. 2000;51:528-37. [ Links ]
27. Montoya GE, Vernot JP, Patarroyo ME. Partial characterization of the CD45 phosphatase cDNA in the owl monkey ( A. vociferans). Am J Primatol. 2002;57:1-11. [ Links ]
28. Rost B, Sander C. Prediction of protein secondary structure at better than 70% accuracy. J Mol Biol. 1993;232:584-99. [ Links ]
29. Rost B, Fariselli P, Casadio R. Topology prediction for helical transmembrane proteins at 86% accuracy. Protein Sci. 1996;5:1704-18. [ Links ]
30. Sun ZYJ, Kim KS, Wagner G, Reinherz EL. Mechanisms contributing to T cell receptor signaling and assembly revealed by the solution structure of an ectodomain fragment of the CD3 eg heterodimer. Cell. 2001;105:913-23. [ Links ]
31. Arnett KL, Harrison SC, Wiley DC. Crystal structure of a human CD3-ed dimer in complex with a UCHT1 single-chain antibody fragment. Proc Natl Acad Sci USA. 2004;101:16268-73. [ Links ]
32. Kjer-Nielsen L, Dunstone MA, Kostenko L, Ely LK, Beddoe T, Mifsud N, et al. Crystal structure of the human T cell receptor CD3ε g heterodimer complexed to the therapeutic mAb OKT3. Proc Natl Acad Sci USA. 2004;101:7675-80. [ Links ]
33. Salmerón A, Sánchez-Madrid F, Ursa MA, Fresno M, Alarcón B. A conformational epitope expressed upon association of CD3-e with either CD3-d or CD3-g is the main target for recognition by anti-CD3 monoclonal antibodies. J Immunol. 1991;147:3047-52. [ Links ]
34. Liu YY, Wang Z, Thomas J, Goodwin KJ, Stavrou S, Neville DM Jr. Polymorphisms of CD3ε in cynomolgus and rhesus monkeys and their relevance to anti-CD3 antibodies and immunotoxins. Immunol Cell Biol. 2007;85:357-62. [ Links ]
35. Call ME, Pyrdol J, Wiedmann M, Wucherpfennig KW. The organizing principle in the formation of the T cell receptor-CD3 complex. Cell. 2002;111:967-79. [ Links ]
36. Call ME, Wucherpfennig KW. Molecular mechanisms for the assembly of the T cell receptor–CD3 complex. Mol Immunol. 2004;40:1295-305. [ Links ]
37. Pitcher LA, Mathis MA, Young JA, DeFord LM, Purtic B, Wulfing C, et al. The CD3 ge/de signaling module provides normal T cell functions in the absence of the TCR z immunoreceptor tyrosine-based activation motifs. Eur J Immunol. 2005;35:3643-54. [ Links ]
38. Lysechko TL, Ostergaard HL. Differential src family kinase activity requirements for CD3z phosphorylation/ ZAP70 recruitment and CD3ε phosphorylation. J Immunol. 2005;174:7807-14. [ Links ]
39. Davis MM. A new trigger for T cells. Cell. 2002;110: 285-7. [ Links ]
40. Szymczak AL, Workman CJ, Gil D, Dilioglou S, Vignali KM, Palmer E, et al. The CD3 proline-rich sequence, and its interaction with Nck, is not required for T cell development and function. J Immunol. 2005;175:270-5. [ Links ]














