Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157versão On-line ISSN 2590-7379
Biomédica v.28 n.3 Bogotá jul./set. 2008
1 Instituto de Genética Humana, Facultad de Medicina, Pontificia Universidad Javeriana, Bogotá, D.C., Colombia
2 Grupo de Genética Forense, Instituto Nacional de Medicina Legal y Ciencias Forenses, Bogotá, D.C., Colombia
Recibido: 16/10/07; aceptado:12/05/08
Introducción. Es bien sabido que, entre los caracteres transmitidos por la línea paterna, el apellido se ha configurado en diferentes culturas como un carácter semejante a un alelo genético neutral asociado al cromosoma Y.
Objetivo. En este estudio se determinaron las frecuencias en la población de 17 STR del cromosoma Y en 308 individuos provenientes de las poblaciones de los departamentos del Valle del Cauca, Cauca y Nariño. Además, se propuso definir la correlación de los haplotipos obtenidos en cada individuo con su apellido paterno, para comparar estos dos códigos de identidad.
Materiales y métodos. Se extrajo el ADN de cada individuo a partir de sangre periférica y se utilizó el estuche comercial AmpFLSTR® Yfiler (Applied Biosystems) para tipificarlo. Los resultados de los haplotipos moleculares se compararon con los apellidos reportados por cada individuo y se asociaron apellidos amerindios y europeos con haplotipos que incluyeran o no el marcador DYS19/13, característico de la población amerindia.
Resultados. Se reportan las frecuencias alélicas de cada uno de los 17 marcadores del cromosoma Y analizados en esta región de Colombia, así como la diversidad génica y haplotípica hallada en los tres departamentos. Al comparar los resultados obtenidos a nivel molecular con los apellidos de origen europeo o amerindio reportados por cada uno de los individuos, se encontró cerca de 40% de inconsistencia de linaje. Conclusiones. La utilización del apellido como marcador de población debe hacerse con cautela, por cuanto las genealogías fundamentadas en éstos pueden no corresponder al origen biológico de sus portadores.
Palabras clave: genotipo, nombres, cromosoma Y, indios sudamericanos, Colombia.
Introduction. In Colombia, surnames are characters usually passed to the children by the father, and they have been compared to neutral alleles associated with the Y-chromosome.
Objective. Population frequencies were determined for 17 short tandem repeats (STR) DNA markers on the Y-chromosome to compare the two identity codes and define the correlation between haplotypes and surnames in each individual.
Materials and methods. DNA was extracted from blood samples from 308 male individuals in provinces of Valle del Cauca, Cauca and Nariño, all in southwestern Colombia. Sample DNA was analyzed with the commercial kit AmpFLSTR® Yfiler (Applied Biosystems) and examined for the following 17 Y-chromosome STR markers: DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393, DYS385a/b, DYS437, DYS438, DYS439, DYS448, DYS456, DYS458, DYS635 and Y-GATA-H4. The frequencies of molecular haplotypes were associated with the surname reported by each individual, and a correlation table was constructed. Amerindian and European surnames were associated with the presence of allele DYS19/13, a characteristic of Amerindian populations.
Results. Allele frequencies were reported for each of the 17 STR markers in the southwestern region of Colombiahigh genetic and haplotypic diversities were obtained. Approximately 40% of lineage inconsistencies were found when the molecular genotype was compared with the European or Amerindian surnames.
Conclusions. Surnames must be used as population markers with reservation. The genetic evidence indicates that traditional genealogies based on surnames with or without documental support, may be inconsistant with their biological provenance.
Key words: Genotype, names, Y chromosome; Indians, South American; Colombia.
Las variaciones en la secuencia del ADN entre individuos se conocen con el término de polimorfismos. Estos polimorfismos son útiles al examinar el ADN en la genética forense, pues muestran suficiente variabilidad entre individuos como para lograr un alto grado de discriminación cuando se realiza una prueba con múltiples loci, en especial, cuando se determina el número de repeticiones cortas en tándem (short tandem repeat, STR) presentes en diferentes cromosomas. Esto los convierte en una herramienta valiosa para hacer cartografía genética, análisis de ligamiento, estudios de población y asociación con enfermedades (1-5).
A diferencia de los marcadores autosómicos, los marcadores STR en el cromosoma Y (Y-STR) tienen cualidades particulares que los hacen excep-cionalmente útiles para estos propósitos. Los marcadores Y-STR se encuentran en la región no recombinante del cromosoma Y (nonrecombining portion of the Y chromosome, NRY) y producen un perfil haploide cuando se amplifican a partir de un ADN masculino. Puesto que el cromosoma Y se transmite de padre a hijo varón prácticamente sin recombinarse, estos marcadores individuales no se combinan usando la regla del producto, como se hace comúnmente con los STR autosómicos que se presentan en cromosomas homólogos. La herencia estrictamente paterna de estos marcadores los hace útiles en la resolución de delitos sexuales, estudios de paternidad y filiación.
En los laboratorios de genética forense es muy frecuente encontrar evidencias con mezcla de fluidos corporales de diferentes individuos; allí el análisis del cromosoma Y puede ser útil en la detección específica de la fracción masculina de ADN en muestras compuestas por ADN masculino y femenino. Esto permite una determinación directa del haplotipo implicado, sin necesidad de realizar una extracción diferencial. Actualmente, se dispone de un importante número de Y-STR que pueden amplificarse y tipificarse en una sola reacción múltiple (multiplex), configurándose así una herramienta valiosa en los laboratorios forenses. Para este propósito es ideal contar con un haplotipo de cromosoma Y que incluya tantos loci polimórficos como sea posible, para aumentar las posibilidades de excluir individuos que no estén implicados en el crimen del cual se les sindica. Estos multiplex han implicado la amplificación simultánea de ocho loci o más. La comunidad forense europea ha establecido para el cromosoma Y un haplotipo mínimo compuesto por nueve marcadores y un haplotipo extendido con dos marcadores adicionales, para la inclusión de loci comunes dentro de una base de datos de referencia de haplotipos de Y-STR (www.yhrd.org).
En Colombia, los estudios realizados con STR autosómicos han cubierto ya gran parte de las regiones geográficas, incluyendo poblaciones amerindias y no amerindias (6-9). Los estudios realizados con marcadores STR del cromosoma Y vienen ampliando su cobertura a varias regiones geográficas colombianas. Estos estudios incluyen los departamentos de Antioquia, Chocó, Cundinamarca, Boyacá, Santander, Norte de Santander, Valle y la región Caribe (10-16), así como los realizados en población amerindia (17-18, Gómez A, Lozano P, Briceño I, Umaña A, Bernal JE, Mitchell J, et al. Y chromosome microsatellite variation in Colombia: study of patrilineal lineages among amerindian tribes and mestizo populations. En: Genomes, the linkage to life, abstracts and posters. XIX International Congress of Genetics. Melbourne, Australia, 2003. p. 268) y en población considerada aislada como el Palenque de San Basilio en el departamento de Bolívar (19).
En el presente estudio se definió la composición genética de tres poblaciones colombianas ubicadas al suroccidente del país (Valle, Cauca y Nariño), con el ánimo de conocer mejor la estructura genética de la población colombiana y correlacionarla con datos históricos inherentes al proceso de mestizaje. Se tomó la región pacífica como un grupo de población representativo del país de acuerdo con el estudio previo de STR autosómicos en población colombiana (6). Teniendo en cuenta que existe una evidente diversidad étnica y cultural en los diferentes departamentos que conforman esta región, se consideró importante analizar la población a este nivel subregional.
Es bien sabido que entre los caracteres transmitidos por la línea paterna, el apellido se ha configurado en diferentes culturas como un carácter semejante a un alelo genético neutral asociado al cromosoma Y (20,21). Aunque los estudios de isonimia han buscado clasificar individuos en diferentes poblaciones de acuerdo con el apellido (22-24), en la mayoría de los casos con el complemento de los novedosos marcadores cromosómicos (25-27), fuera del estudio pionero del apellido Sykes (20) hay escasos análisis de correlación entre el apellido y el cromosoma Y en una población determinada. Si bien ambos marcadores (apellido y cromosoma Y) se heredan, en general, por vía "patrilineal" en las poblaciones estudiadas, se propuso en este estudio contrastar la estructura genética y patronímica de la población de estos tres departamentos del suroccidente colombiano.
Se incluyeron en este estudio 308 individuos de sexo masculino vinculados a pruebas de paternidad, excluyendo, en lo posible, el parentesco inmediato de los donantes, al tomar como indicadores de exclusión tanto el apellido paterno como la historia familiar reportada por los individuos participantes de cada departamento.
Se utilizaron 17 marcadores polimórficos del cromosoma Y (DYS19, DYS389 I/II, DYS390, DYS391, DYS392, DYS393, DYS438, DYS439, DYS437, DYS448, DYS456, DYS458, DYS635, YGATA-H4 y DYS385a/b), con el fin de tipificar los genotipos de los participantes y correlacionarlos con su apellido. Los apellidos se dividieron en dos grandes grupos (apellidos europeos y apellidos amerindios) de acuerdo con su etimología.
Materiales y métodos
Población
El presente estudio se realizó con muestras de sangre tomadas en tarjetas FTA® (Whatman) a 308 individuos de sexo masculino no emparentados, nacidos y residentes en los departamentos del Valle del Cauca, Cauca y Nariño en el suroccidente colombiano.
Las muestras se seleccionaron al azar del banco de muestras de ADN del convenio del Instituto Colombiano de Bienestar Familiar y el Instituto Nacional de Medicina Legal y Ciencias Forenses Regional Bogotá, y corresponden a muestras obtenidas de casos de paternidad (presuntos padres) clasificados como de rutina, con consentimiento escrito aprobado para utilizarlos en estudios de población. El número de individuos seleccionados y analizados para cada departamento se relaciona en el cuadro 1.

Extracción de ADN y marcadores moleculares utilizados
El ADN genómico fue extraído por el método de Chelex 100 al 20% (28). Las muestras se analizaron con el estuche AmpFLSTR® Yfiler, el cual tipifica 17 marcadores tipo STR del cromosoma Y, previamente caracterizados por otros laboratorios. Todos los loci analizados han sido secuenciados y localizados a lo largo de la secuencia del cromosoma Y (29).
Amplificación por PCR de 17 loci del cromosoma Y
Los 17 loci STR del cromosoma Y que se amplificaron utilizando el estuche AmpFLSTR® Yfiler, fueron los siguientes: DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393, DYS385a, DYS385b, DYS437, DYS438, DYS439, DYS448, DYS456, DYS458, DYS635 y GATA H4, los cuales tienen un rango de tamaño en pares de bases (pb) que va desde 95 hasta 330 pb, aproximadamente, (Applied Biosystems AmpFLSTR Yfiler Product Bulletin). La reacción se realizó en un solo paso y se llevó a cabo en un termociclador GeneAmp® 2720 (Applied Biosystems).
Las condiciones de reacción para cada una de las muestras fueron previamente validadas y estandarizadas en muestras de referencia conocidas, para utilizar la mitad del volumen de reacción recomendado por la casa fabricante (30). Como control positivo se utilizó ADN de la línea celular masculina 007 y, como control negativo, la línea celular femenina 9947A. Se adicionaron 5 µl del ADN (0,1 ng/µl) correspondiente a la mezcla de reacción, quedando cada una a una concentración final de 0,5 ng.
Análisis y tipificación de los productos de la PCR
Preparación y montaje de las muestras
Un volumen de 1 µl del producto de PCR se adicionó a un volumen de 9,7 µl de formamida HiDi junto con 0,3 µl de estándar interno LIZ (GeneScan® 500 LIZ® Internal Size Standard). Esta mezcla luego fue llevada a 95 °C para desnaturalizar el ADN e, inmediatamente, fue colocada en hielo durante tres minutos. Luego se realizó el montaje de las muestras en un analizador genético ABI® 3130, para separar electroforéticamente los fragmentos obtenidos. Posteriormente, los resultados se analizaron mediante el programa GeneMapper® para asignar los alelos a cada una de las muestras.
Análisis de alelos mediante el programa GeneMapper
Una vez finalizado el corrido en el equipo, se creó el proyecto mediante el programa GeneMapper® v.3.2.1, un programa de genotipificación automatizado, el cual combina de manera óptima los programas GeneScan® y Genotyper®, agilizando el análisis de las muestras. La escalera alélica se corrió en el equipo para verificar la asignación alélica en cada una de las muestras analizadas.
Se revisaron los pesos moleculares de los diferentes fragmentos de ADN que componen el estándar interno utilizado en cada muestra (50, 75, 100, 139, 150, 160, 200, 300, 340, 350, 400, 450 y 500 pares de bases). Luego se revisó la asignación alélica del control positivo y se revisaron el control negativo y el blanco de extracción para verificar que no presentaran señales de amplificación a una escala menor de 500 unidades relativas de fluorescencia en los electroferogramas, con el fin de detectar cualquier tipo de contaminación. Si el análisis cumplía con estos requisitos de calidad, se procedía a hacer el análisis de las muestras.
Análisis estadísticos
Los haplotipos obtenidos se numeraron en orden ascendente de acuerdo con la secuencia de tipificación de las muestras, iniciando con la población del Valle del Cauca, siguiendo con la población de Cauca y finalizando con la población de Nariño. Las frecuencias alélicas y haplotípicas se estimaron en una matriz de Excel y correspondieron al cociente que resultó de dividir el número de alelos iguales en una población por el número de alelos totales. Para los STR del cromosoma Y los valores de diversidad génica o haplotípica tienen la misma validez que la capacidad de discriminación (CD) y la probabilidad de exclusión (PE) (31). También se pueden calcular con el número total de haplotipos observados, divididos por el número total de individuos (32).
La probabilidad de coincidencia (PC) fue descrita por Jones (33) y se calcula como:
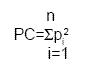
siendo pi la frecuencia del alelo o haplotipo i.
También puede calcularse como: PC=1-DH (diversidad haplotípica).
La diversidad génica (DG) y la diversidad haplotípica (DH) se calcularon siguiendo la siguiente ecuación: DG o DH = (n/n-1) (1-Σpi2), donde n es el número de muestras analizadas y "pi ", las frecuencias del alelo o haplotipo i (34). Los haplotipos únicos (HU) se definieron como el número de haplotipos que se presentan una sola vez en la población total.
Se efectuó el análisis molecular de varianzas (AMOVA) para el estudio de la variación dentro de la población, utilizando el programa ARLEQUIN de acuerdo con la descripción de Roewer et al. (1).
Asociación de haplotipos y apellidos
Las frecuencias de apellidos amerindios y españoles se contrastaron con la frecuencia de los haplotipos que incluyeran el marcador DYS19/13 o que no lo hicieran, utilizando una tabla de correlación simple.
Aspectos éticos
El presente estudio fue avalado por el Comité de Investigación y Ética de la Facultad de Medicina de la Pontificia Universidad Javeriana.
Resultados
Los 308 individuos vinculados al estudio representaron las poblaciones de los tres departamentos del suroccidente colombiano en proporciones iguales. Al evaluar la diversidad de apellidos en cada una de las tres cohortes, se encontró que en el Valle del Cauca había 88 apellidos en 104 individuos (diversidad=84,6), en Cauca, 70 apellidos en 102 individuos (diversidad=68,6) y en Nariño, 73 apellidos en 102 individuos (diversidad=71,5). Se llevó a cabo un análisis genético de la población utilizando 17 marcadores Y-STR en 308 individuos que se reportaron como no emparentados, nacidos y residentes en los departamentos que conforman la región suroccidental colombiana (Valle del Cauca, Cauca y Nariño).
Frecuencias alélicas
Se estimaron las distribuciones de las frecuencias alélicas para cada uno de los marcadores del cromosoma Y en cada uno de los departamentos, así como su diversidad génica. En los cuadro 2, cuadro 3, cuadro 4 y cuadro 5 se muestran las frecuencias alélicas calculadas para cada una de las poblaciones estudiadas y cada uno de los sistemas genéticos.
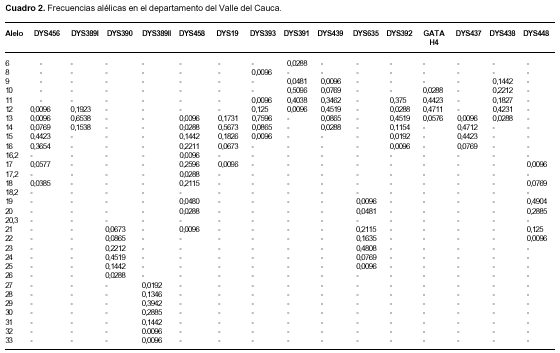
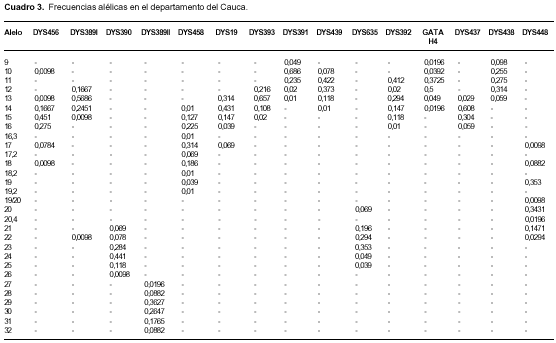
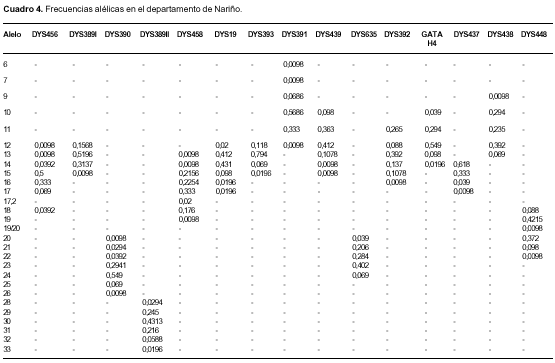
Determinación de haplotipos
Se determinaron los haplotipos de los 308 individuos para los 17 marcadores Y-STR contenidos en el estuche AmpFLSTR® Yfiler, teniendo en cuenta para su análisis, en primer lugar, los sistemas del haplotipo mínimo europeo (DYS19, DYS385a, DYS385b, DYS389I, DYS389II, DYS390, DYS391, DYS392 y DYS393), en segundo lugar, los del haplotipo mínimo extendido que comprenden los sistemas Y-STR del haplotipo mínimo europeo más los sistemas DYS438 y DYS439, y por último, los loci adicionales contenidos en el estuche AmpFLSTR® Yfiler (DYS437, DYS448, DYS456, DYS458, DYS635 (GATA-C4) y GATA-H4).
El orden en que se refieren es el siguiente: DYS19, DYS385a, DYS385b, DYS389I, DYS389II, DYS390, DYS391, DYS392, DYS393, DYS438, DYS439, DYS437, DYS448, DYS456, DYS458, DYS635 (GATA-C4) y GATA-H4. Se observaron 288 haplotipos en total, de los cuales, 268 son haplotipos únicos y los 20 restantes se encuentran representados dos o tres veces para los haplotipos más frecuentes.
El poder de discriminación haplotípica, determinado con base en el número de haplotipos individuales sobre el número total de individuos analizados, fue de 93,2%. Al efectuar un análisis molecular de varianzas (AMOVA) (1), se obtuvo un porcentaje de variación dentro de la población de 99,1%. Tanto el poder de discriminación haplotípica como la variación dentro de la población disminuyeron en la medida en que se utilizaron menos marcadores para comparar las tres cohortes.
Entre quienes comparten haplotipos construidos con base en los 17 marcadores utilizados, se encontraron seis individuos que comparten también el apellido (con tres haplotipos) y 34 individuos que no lo comparten. Entre estos últimos, el haplotipo más frecuente (H57:14-13-17-14-32-23-10-11-12-10-11-14-20-15-17,2-21-11) se encontró en dos individuos del Cauca y en un individuo del Valle del Cauca. La frecuencia para este haplotipo en la muestra de la región suroccidental corresponde a 0,0097 y los tres individuos que lo portaban tenían apellidos diferentes.
De los haplotipos observados como únicos, 103 se hallaron en la población del Valle correspondiendo al 99% de la población estudiada, 90 en el departamento de Cauca con un 88% y 95 en Nariño que equivale al 93% de la población analizada. Se obtuvo una diversidad haplotípica global de 0,9995.
Apellidos
Una descripción detallada de los 218 apellidos diferentes encontrados en las tres poblaciones estudiadas y sus coincidencias haplotípicas está a disposición de los lectores en el (cuadro 6). Entre éstos, 183 (84%) presentaban apellido de origen europeo y 35 (16%) apellido de origen amerindio. La relación E/A, de apellidos europeos (E) y amerindios (A), en los tres departamentos fue la siguiente: Valle del Cauca, 84/4, Cauca, 54/16, y Nariño, 58/15, con un claro predominio de los apellidos de origen europeo (>77%) en las tres cohortes. La suma total de individuos con apellido de origen europeo en la región suroccidental (n=196) superó la suma total de apellidos europeos (n=183), por cuanto se encontraron 13 apellidos entre éstos que se repetían en diferentes departamentos.
Entre los individuos con apellido amerindio, solamente 5/35 individuos presentaron el mismo apellido, sin reportar parentesco. De estos cinco, solamente uno compartía además el haplotipo con otro individuo con apellido diferente en el mismo departamento. El resto de los individuos con apellido amerindio (30/35) eran casos únicos, con la excepción del que, como se dijo, compartía su haplotipo con otro individuo. Entre estos individuos de apellido único, sólo dos compartían el haplotipo, pero con individuos de apellido de origen europeo.
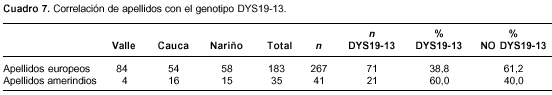
En el cuadro 7 se relacionan los porcentajes relativos de amerindios y europeos por vía paterna, de acuerdo con los dos sistemas utilizados en el estudio para definirlos y compararlos. De 183 individuos con apellido europeo, 71 (38,8%) presentaban el alelo DYS19/13, indicador de ancestro amerindio, y 112 (61,2%), un alelo diferente. Por el contrario, entre los 35 individuos con apellido amerindio, 21 (60%) presentaban el alelo DYS19/13, indicador de ancestro amerindio, y 14 (40%), un alelo diferente compatible con un ancestro europeo.
Discusión
Los STR descritos en este estudio conforman haplotipos de cromosoma Y informativos desde el punto de vista forense. La presencia de un gran número de individuos con haplotipos únicos (268/308) refleja la gran diversidad genética que presenta esta muestra tomada a partir de tres poblaciones en el suroccidente colombiano.
La inclusión del sistema DYS385 incrementa notablemente el poder de discriminación y la diversidad de los haplotipos construidos con STR del cromosoma Y (35). La distribución de las frecuencias alélicas obtenidas para los 17 marcadores en cada una de las poblaciones analizadas revela que, en 12 de los 17 marcadores analizados (DYS19, DYS389I, DYS390, DYS391, DYS393, DYS438, DYS437, DYS448, DYS456, DYS458, DYS635 y el Y-GATA-H4), el alelo más frecuente encontrado en cada una de ellas es el mismo para todas y que, en los cinco marcadores restantes, existen variaciones en cuanto al alelo más frecuente. Los alelos encontrados como los más frecuentes en cada uno de los departamentos, coinciden con los más frecuentes reportados para los sistemas DYS19, DYS389I, DYS389II, DYS390, DYS391, DYS392 y DYS393 en población europea (36), lo cual permite inferir que la población de la región suroccidental tiene, en general, un componente genético de cromosoma Y principalmente europeo.
Otras poblaciones colombianas han experimentado un flujo génico de origen principalmente caucásico a través del linaje masculino (9-11,16,17,27).
Para el marcador DYS19 se encontró que el alelo 13, característico por ser el más frecuente en población amerindia, estaba presente en todos los departamentos estudiados en frecuencias distribuidas en orden ascendente así: 17,1% en Valle del Cauca, 31,4% en Cauca y 41,2% en Nariño. Esta última frecuencia es levemente inferior a la del alelo 14 en esta población, que es de 43,1%. La frecuencia global para el alelo DYS19/13 en la región suroccidental es de 29,8%.
Estos hallazgos indicarían un bajo grado de mestizaje de las poblaciones de Valle del Cauca y Cauca que presentan este alelo amerindio en menor proporción, en comparación con lo hallado en la población de Nariño que presenta frecuencias equivalentes en los alelos 13 y 14 del DYS19.
Sin embargo, la sola presencia del alelo 13 en las poblaciones de este estudio no es indicación suficiente de componente amerindio. Para corroborar este hallazgo sería necesario analizar la substitución C:T en el DYS19/13 de cada uno. Dada la proporción de individuos con el DYS19/13 en individuos con apellidos europeos y amerindios y que se puede suponer que solamente una fracción de éstos presentarán la mutación C:T amerindia, tal y como lo demostró un trabajo previo en población mestiza chilena (37), la frecuencia de linajes paternos nativos americanos sería aún menor que la reportada en este estudio con base en la presencia del alelo 13. Otro estudio de nuestro grupo mostró cómo, aun en poblaciones consideradas amerindias, como los wayuu, el análisis de la sustitución C:T en el DYS19/13 revelaba que varios de los indígenas presentaban la citosina y no la timina, lo cual es consistente con un ancestro paterno europeo y no amerindio (Gómez A, Lozano P, Briceño I, Umaña A, Bernal JE, Mitchell J, et al. Y-chromosome microsatellite variation in Colombia: study of patrilineal lineages among amerindian tribes and mestizo populations. En: Genomes, the linkage to life. Abstracts and posters, XIX International Congress of Genetics. Melbourne, Australia, 2003. p. 268).
La proporción dominante de individuos considerados de linaje caucásico o europeo por su apellido en la región suroccidental de Colombia, ha sido confirmada por el Censo Nacional de 2005, en el que se reporta una proporción de indígenas en todo el país de 3,4% y, en cada uno de los departamentos de Valle del Cauca, Cauca y Nariño, de 1%, 21% y 10%, respectivamente (38). Sin embargo, tal y como se muestra en el presente estudio, hasta 39% de los individuos que reportan ser de ascendencia europea de acuerdo con su apellido, podrían ser en realidad descendientes de amerindios por línea paterna, al presentar el alelo 13 en el STR DYS19. Debe notarse que estas poblaciones presentan una frecuencia del alelo DYS19/13 cercana a 30%, lo cual contrasta con las bajas frecuencias (<1%) encontradas en las poblaciones españolas o europeas registradas actualmente en la base de datos YHRD (Y chromosome haplotype reference database) preparada por Willuweit y Roewer por comisión del International Forensic Y Chromosome User Group (39).
Puesto que este estudio utiliza muchos más marcadores de los que se han utilizado para la mayoría de los estudios a nivel mundial, no se pudo hacer la comparación de las frecuencias haplotípicas para los otros sistemas estudiados. En cuanto a la distribución de los alelos, se encontró que la mayoría de los STR de cromosoma Y tienen una distribución unimodal (un alelo muy frecuente con alelos adyacentes de forma decreciente), lo cual apoya el modelo mutacional paso a paso en el cual un alelo ancestral gana o pierde una sola unidad de repetición en cada evento. Algunos sistemas presentan una distribución diferente a la distribución simétrica, también conocida como distribución normal de Gauss. Las distribuciones más notorias para los diferentes alelos son: simétrica bimodal en el marcador DYS392, asimétrica sesgada hacia la izquierda (DYS19, y DYS437) o asimétrica sesgada hacia la derecha (DYS438, DYS390 y DYS635). Estos comportamientos de distribución podrían corresponder a mezclas recientes de dos poblaciones que inicialmente tenían frecuencias diferentes para los mismos alelos o, alternativamente, a efectos de muestreo por no vincular representantes de todos los alelos de ese marcador.
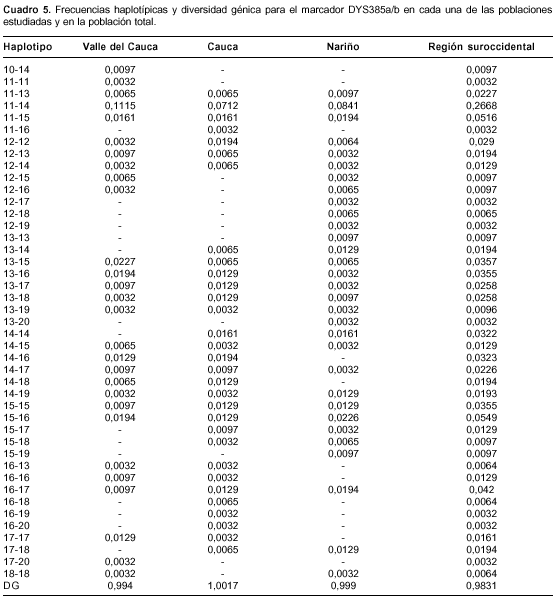
La diversidad génica por población para el sistema DYS385a/b se muestra en el cuadro 5 en donde, al igual que para los demás marcadores Y-STR analizados en este estudio, el haplotipo más frecuente para DYS385 a/b en todas las poblaciones coincide con el más frecuente reportado para la población europea. En cuanto a diversidad génica, este marcador es el que mayor diversidad presenta (>0,983) como es de esperarse al estar duplicado, comparado con los demás marcadores Y-STR analizados. Esta gran diversidad estaría relacionada con el hecho de que luego de que ocurre un evento de duplicación, generalmente, ocurre una mutación y las mutaciones, a su vez, están estrechamente relacionadas con la diversidad. En la población de Cauca se detectó el valor de diversidad más alto (1,0) para este marcador y en el Valle del Cauca, el más bajo (0,994).
Se encontraron 288 haplotipos, de los cuales, 268 son haplotipos únicos. Este resultado confirmaría que 93% de los individuos analizados no están emparentados por línea paterna y que el muestreo fue aleatorio y lo suficientemente heterogéneo como para validar estos marcadores del cromosoma Y en genética forense y disponer de una muestra adecuada para concluir sobre la correlación de los dos tipos de marcadores analizados en nuestro estudio (por ejemplo, el apellido y los haplotipos del cromosoma Y).
En cuanto a los haplotipos frecuentes, se encontraron 19 parejas y un trío en 308 individuos, es decir, que 13,3% de los individuos analizados compartían haplotipos, de los cuales, tres parejas también compartían el primer apellido. Los restantes compartían haplotipo pero no el primer apellido, lo cual seguiría confirmando que los individuos escogidos al azar no están emparentados por línea paterna.
La diversidad haplotípica encontrada para la región suroccidental supera las encontradas para otros estudios de población en ésta y en otras regiones del planeta (9,10,36,40). Este hallazgo puede explicarse porque en la gran mayoría de los estudios de población realizados en este tipo de marcadores, hasta el momento, sólo se han utilizado los marcadores que conforman el haplotipo mínimo europeo (nueve marcadores) y en otros, los del haplotipo mínimo extendido (11 marcadores).
Por otra parte, se ha visto que, a medida que se incrementa el número de marcadores Y-STR en una combinación, la diversidad haplotípica también aumenta y se acerca a 0,9999 (por ejemplo, con 20 marcadores), lo que a su vez hace que la probabilidad de coincidencias (matching) entre dos individuos tomados al azar disminuya (32). Cuando se reduce el número de marcadores para el análisis de haplotipos, es decir, al utilizar únicamente los 11 marcadores del haplotipo mínimo extendido, se incrementa el número de individuos que estarían emparentados por línea paterna, mientras que cuando se utiliza el haplotipo mínimo europeo con 9 marcadores, el incremento es más notorio, lo cual aumenta la posibilidad de falsas coincidencias de linaje. De igual forma, la diversidad haplotípica de cada subpoblación estudiada para la región suroccidental fue menor que la diversidad total hallada para toda la población (0,999) utilizando los 17 marcadores (0,998, 0,995 y 0,996, respectivamente). Estos resultados coinciden con los valores obtenidos en estudios previos utilizando estos mismos marcadores (14).
Uno de los objetivos de este estudio fue evaluar la utilidad forense de los haplotipos determinados con 17 marcadores y el hecho de haber obtenido diversidades superiores a 0,996 hace que los datos obtenidos sean una importante herramienta de población para ser utilizada en los estudios forenses con cromosoma Y, en particular, para individuos de la región suroccidental colombiana. Además, la utilización de un número reducido de marcadores haría que la probabilidad de coincidencia (matching) se incrementara al realizar pruebas de paternidad o estudios de mezclas forenses, lo cual aumentaría la probabilidad de encontrar ese haplotipo en la población y restaría valor al análisis, especialmente cuando el estudio no puede complementarse con otros marcadores, como los STR autosómicos.
Todas las poblaciones presentaron coincidencias haplotípicas con la base de datos de Applied Biosystems. Estas poblaciones presentan haplotipos de origen muy diverso, aunque el mayor número de coincidencias fue con respecto a los haplotipos caucásicos, seguido por los de origen hispano y afroamericano y con el menor número de coincidencias frente a los nativo-americanos. Este mayor número de coincidencias de estos datos con los caucásicos se explica por el posible origen europeo que presentan los haplotipos estudiados, sin olvidar que los hispanos son muy similares y comparten el mismo origen global de mestizaje europeo.
Para la población del Valle del Cauca se encontró el mayor número de coincidencias con población afroamericana, lo cual puede explicarse por su cercanía a la costa del Pacífico y la influencia africana de nuestra historia reciente, en la cual se refiere una migración que ingresó por el océano Pacífico, como base de asentamientos de esclavos traídos para labores de minería en esa región del país (13). Como dato interesante, se detectó que la única población que mostró coincidencia con haplotipos reportados en esta base de datos para la población nativo-americana fue la población de Nariño.
Los resultados anteriores indicarían que el componente genético del cromosoma Y predo-minante en las poblaciones que conforman la región suroccidental colombiana corresponden a linaje europeo, dado el alto porcentaje de haplotipos coincidentes con los estudiados para esta metapoblación. De igual manera, las coincidencias con la población colombiana, en especial con Antioquia y Bogotá -cuyo origen paterno predominante es el europeo-, corroboran una vez más este hallazgo.
No es, en realidad, sorprendente que el componente mayoritario patrilineal de la población estudiada sea europeo, ya que a través de la historia de la conquista del Nuevo Reino de Granada, especialmente en los siglos XVI y XVII, las diferentes expediciones de inmigrantes estuvieron conformadas por hombres provenientes de Europa y, en su mayor parte, de España. Ésta ha sido, como se sabe, la fuente principal de apellidos colombianos y, de acuerdo con el presente estudio, también de genotipos en esta región de Colombia.
Finalmente, de acuerdo con los resultados de este estudio, y de otros antecedentes reportados en la literatura (38,41-44), debe utilizarse con cautela el apellido como marcador de población (20-26) por cuanto, tal y como pudimos demostrar en la región suroccidental, cerca de 40% de los individuos que refirieron apellido de origen europeo tienen un cromosoma Y compatible con un linaje amerindio, y cerca de 40% de los individuos que refirieron apellido de origen amerindio, tienen un cromosoma Y compatible con un linaje europeo. La proporción amerindio/europeo debe definirse adecuadamente en los estudios que utilicen la isonimia en cualquier región de Colombia, bien sea en estudios clínicos o genéticos, con el fin de afinar el genérico de mestizos a la luz de las herramientas moleculares disponibles hoy. Servirse de frecuencias simples de apellidos para sugerir que no existe heterogeneidad genética en una población determinada y asegurar, con base en éstos, ausencia de estratificación en la muestra, podría conducir a conclusiones erróneas a la luz de los resultados del presente estudio.
Agradecimientos
A los participantes en el estudio y al Instituto Nacional de Medicina Legal y Ciencias Forenses, por habernos permitido desarrollar este trabajo en sus instalaciones. Este proyecto está registrado con el código 1889 en la Vicerrectoría Académica de la Pontificia Universidad Javeriana.Conflictos de interés
No se presentaron conflictos de interés
Financiación
El proyecto fue financiado por el Instituto Nacional de Medicina Legal y Ciencias Forenses y por la Pontificia Universidad Javeriana.
Correspondencia:
Alberto Gómez, Instituto de Genética Humana, Facultad de Medicina, Pontificia Universidad Javeriana, Bogotá, D.C., Colombia. Teléfono: (+571) 320 8320, extensión 2792; fax: (571) 320 8320, extensión 2793. agomez@javeriana.edu.co
Referencias
1. Roewer L, Kayser M, Dieltjes P, Nagy M, Bakker E, Krawezak M, et al. Analysis of molecular variance (AMOVA) of Y-chromosome-specific microsatellites in two closely related human populations. Hum Mol Genet. 1996;5:1029-33. [ Links ]
2. Jobling MA, Pandya A, Tyler-Smith C. The Y chromosome in forensic analysis and paternity testing. Int J Legal Med. 1997;110:118-24. [ Links ]
3. Prinz M, Boll K, Baum H, Shaler B. Multiplexing of Y chromosome specific STR and performance for mixed samples. Forensic Sci Int.1997;85:209-18. [ Links ]
4. Kayser M, Cagliá A, Corach D, Fretwell N, Gehrig C, Graziosi G, et al. Evaluation of Y-chromosomal STR: a multicenter study. Int J Legal Med. 1997;110: 125-33. [ Links ]
5. De Knijff P, Kayser M, Cagliá A, Corach D, Fretwell N, Gehrig C, et al. Chromosome Y microsatellites: population genetic and evolutionary aspects. Int J Legal Med. 1997;110:134-49. [ Links ]
6. Paredes M, Galindo A, Bernal M, Avila S, Andrade D, Vergara C, et al. Analysis of the CODIS autosomal STR loci in four main Colombian regions. Forensic Science Int. 2003;137:67-73. [ Links ]
7. Bravo ML, Moreno MA, Builes JJ, Salas A, Lareu MV, Carracedo A. Autosomal STR genetic variation in negroid Chocó and Bogotá populations. Int J Legal Med. 2001;115:102-4. [ Links ]
8. Cifuentes L, Bonilla V, Rondón F, Cárdenas H, Barreto G. Evaluación de la diversidad genética mediante el análisis de STR en poblaciones aisladas del centro y suroccidente colombiano. Revista de la División de Ciencias de la Salud Universidad del Norte. 2004;18:96. [ Links ]
9. Yunis JJ, Acevedo LE, Campo DS, Yunis EJ. Population data of Y-STR minimal haplotypes in a sample of Caucasian-Mestizo and African descent individuals of Colombia. Forensic Sci Int. 2005;151:307-13. [ Links ]
10. Gaviria AA, Ibarra AA, Palacio OD, Posada YC, Triana O, Ochoa LM, et al. Y-chromosome haplotype analysis in Antioquia (Colombia). Forensic Sci Int. 2005;151:85-91. [ Links ]
11. Builes JJ, Bravo ML Martínez-Pancorbo M, Moreno MA, Gómez CP. Discrimination index of Y-chromosomal haplotypes in an Antioquia (Colombia) population sample. Int Congr Ser. 2004;1261:275-7. [ Links ]
12. Yunis JJ, García O, Cuervo AG, Guío E, Pineda CR, Yunis EJ. Population data for PowerPlex 16 in thirteen departments and the capital city of Colombia. J Forensic Sci 2005; 50:685-702. [ Links ]
13. Torres MM. La variabilidad genética una herramienta útil en el estudio de poblaciones humanas. (Tesis doctoral). Bogotá D.C.: Universidad de los Andes; 2005. [ Links ]
14. Builes JJ, Castañeda SP, Espinal C, Aguirre D, Gómez MV, Villamarin D, et al. Analysis of 16 Y-chromosomal STR in a Valle (Colombia) population sample. Int Congr Ser. 2006;1288:219-21. [ Links ]
15. Romero RH. Determinación de haplotipos del cromosoma-Y en población de la Costa Caribe colombiana y su utilidad en el campo forense. (Tesis de Maestría). Bogotá D.C.: Pontificia Universidad Javeriana; 2006. [ Links ]
16. Builes JJ, Bravo ML, Montoya A, Caraballo L, Martinez B, Moreno MA. Population genetics of eight new Y-chromosomal STR haplotypes in three Colombian populations: Antioquia, Chocó and Cartagena. Int Congr Ser. 2004;1261:310-2. [ Links ]
17. Carvajal-Carmona LG, Soto ID, Pineda N, Ortiz-Barrientos D, Duque C, Ospina-Duque J, et al. Strong Amerind/white sex bias and a possible sephardic contribution among the founders of a population in north-west Colombia. Am J Hum Genet. 2000;67:1287-95. [ Links ]
18. Guarino FD, Federle RA, van Oorschot RAH, Briceño I, Bernal JE, Papiha SS, et al. Genetic diversity among five native american tribes of Colombia. En: Papiha SS, Deka R, Chakraborty R, editors. Genetic diversity: Applications in human population genetics. New York: Kluwer Academic/Plenum Publishers; 1999.p.33-51. [ Links ]
19. Lopera JA, Soto ID, Mondragón MC, Caraballo L, Bedoya BG, Ruiz-Linares A. Estructura genética de la población actual del Palenque de San Basilio (Bolívar, Colombia). Revista de la División de Ciencias de la Salud Universidad del Norte. 2004;18:97. [ Links ]
20. Sykes B, Irven C. Surnames and the Y chromosome. Am. J Hum Genet. 2000;66:1417-9. [ Links ]
21. Jobling MA. In the name of the father: surnames and genetics. Trends Genet. 2001;17:353-7. [ Links ]
22. Vernay M. Répartition géographique des patronymes et structure génétique: le département de l´Ardèche au début du XXe siècle. C R Acad Sci III. 2001;324:589-99. [ Links ]
23. Morelli L, Paoli G, Francalacci P. Surname analysis of the Corsican population reveals an agreement with geographical and linguistic structure. J Biosoc Sci. 2002;34:289-310. [ Links ]
24. Bedoya G, García J, Montoya P, Rojas W, Amézquita ME, Soto I, et al. Análisis de isonimia entre poblaciones del noroeste de Colombia. Biomédica. 2006;26:538-45. [ Links ]
25. Zei G, Lisa A, Fiorani O, Magri C, Quintana-Murci L, Semino O, et al. From surnames to the history of Y chromosome: the Sardinian population as a paradigm. Eur J Hum Genet. 2003;11:802-7. [ Links ]
26. Mikerezi I, Pizzetti P, Lucchetti E, Ekonomi M. Isonymy and the genetic structure of Albanian populations. Coll Anthropol. 2003;27:507-14. [ Links ]
27. Bedoya G, Montoya P, García J, Soto I, Bourgeois S, Carvajal L, et al. Admixture dynamics in Hispanics: A shift in the nuclear genetic ancestry of a South American population isolate. Proc Nat Acad Sci USA. 2006;103:7234-9. [ Links ]
28. Walsh PS, Metzger DA, Higuchi R. Chelex 100 as a medium for simple extraction of DNA for PCR-based typing from forensic material. Biotechniques. 1991;4:506-13. [ Links ]
29. Mulero J, Chang C, Calandro L, Green R, Li Y, Johnson C, et al. Development and validation of the AmpFLSTR® Yfiler PCR amplification kit: A male specific, single amplification 17 Y-STR multiplex system. J Forensic Sci. 2006;51:64-75. [ Links ]
30. Romero RH, Lizarazo R. Validation of the AmpFLSTR Yfiler kit. Int Congr Ser. 2006;1288:280-2. [ Links ]
31. Sensabaugh GF, Biochemical markers of individuality. En: Saferstein R, editor. Forensic Science Handbook. Englewood Cliffs NJ: Prentice Hall, Inc: 1982. p. 379-414. [ Links ]
32. Butler JM. State of the Y chromosome: New advances and state of the science for Y chromosome DNA testing. Consultado: 09/05/2008. Disponible en: http://www.cstl.nist.gov/biotech/strbase/pub_pres/TorontoJune2005_Y.pdf [ Links ]
33. Jones DA. Blood samples probability of discriminations. J Forensic Sci Soc. 1972;12:355-9. [ Links ]
34. Nei M. Molecular evolutionary genetics. New York: Columbia University Press; 1987. p. 176-9. [ Links ]
35. Cagliá A, Dobosz M, Boschi I, d´Aloja E, Pascali VL. Increased forensic efficiency of a STR-based Y-specific haplotype by addition of the highly polymorphic DYS385 locus. Int J Legal Med. 1998;111:142-6. [ Links ]
36. Kayser M, Krawczak M, Excoffier L, Dieltjes P, Corach D, Pascali V, et al. An extensive analysis on Y-chromosomal microsatellite haplotypes in globally dispersed human populations. Am J Hum Genet. 2001;68:990-1018. [ Links ]
37. Cifuentes L, Morales R, Sepúlveda D, Jorquera H, Acuña M. DYS19 and DYS199 loci in a Chilean population of mixed ancestry. Am J Phys Anthropol. 2004;125:85-9. [ Links ]
38. Departamento Administrativo Nacional de Estadística. Censo 2005. Consultado: 09/05/2008. Disponible en: http://www.dane.gov.co/censo/ [ Links ]
39. Willuweit S, Roewer L. Y chromosome haplotype reference database (YHRD): Update. Forensic Sci Int Genet. 2007;1:83-7. [ Links ]
40. Bosch E, Calafell F, Perez-Lezaun A, Comas D, Izaabel H, Akhayat O, et al. Y-chromosome STR haplotypes in four populations from northwest Africa. Int J Legal Med. 2000;114:36-40. [ Links ]
41. Dipierri JE, Alfaro E, Martínez-Marignac VL, Bailliet G, Bravi CM, Cejas S, et al. Paternal directional mating in two Amerindian subpopulations located at different altitudes in northwestern Argentina. Hum Biol. 1998;70:1001-10. [ Links ]
42. Bailliet G, Castilla EE, Adams JP, Orioli IM, Martínez-Marignac VL, Richard SM, et al. Correlation between molecular and conventional genealogies in Aicuña: a rural population from northwestern Argentina. Hum Hered. 2001;51:150-9. [ Links ]
43. Pettener D, Pastor S, Tarazona-Santos E. Surnames and genetic structure of a high-altitude Quechua community from the Ichu River Valley, Peruvian Central Andes, 1825-1914. Hum Biol. 1998; 70:865-87. [ Links ]
44. King TE, Ballereau SJ, Shürer KE, Jobling MA. Genetic signatures of coancestry within surnames. Curr Biol. 2006;16:384-8. [ Links ]














