Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.28 no.4 Bogotá Oct./Dec. 2008
1 Instituto de Genética Humana, Pontificia Universidad Javeriana, Bogotá, D.C., Colombia
2 Facultad de Medicina, Universidad de La Sabana, Chía, Cundinamarca, Colombia
3 Instituto de Estudios Ambientales, Universidad Nacional de Colombia, Bogotá, D.C., Colombia
4 Laboratorio de Antropología Física, Departamento de Antropología, Universidad Nacional de Colombia, Bogotá, D.C., Colombia
Recibido: 22/10/07; aceptado:08/09/08
Introducción. Los restos óseos arcaicos son fuente privilegiada de información biológica y su caracterización genética permite confirmar o descartar filiaciones propuestas por otras aproximaciones científicas. La historia precolombina de los Andes orientales se divide en tres periodos principales: i) un poblamiento temprano por parte de grupos cazadores-recolectores; ii) un periodo intermedio (Herrera) de pueblos con agricultura incipiente, y iii) un periodo tardío de pueblos chibchas, agrícolas y alfareros (agroalfarero).
Objetivo. Analizar el ADN mitocondrial de restos óseos del periodo Herrera.
Materiales y métodos. Se analizaron 11 individuos pertenecientes al yacimiento arqueológico Madrid 2-41, con una edad aproximada de 2.000 años. Un fragmento (192 pb) del segmento hipervariable I fue amplificado y secuenciado, siguiendo criterios estrictos de autenticidad de ADN arcaico. Las secuencias se compararon con las existentes en bases de datos de Norteamérica y Europa usando herramientas bioinformáticas.
Resultados. Todas las secuencias resultaron idénticas y fueron clasificadas como haplogrupo B. Esto puede relacionarse con el tipo de entierro ritual practicado en Madrid 2-41, es decir, probablemente los individuos analizados hagan parte de una familia jerárquicamente importante en la antigua sociedad Herrera. La búsqueda de secuencias homólogas en las bases de datos estadounidense y europea no arrojó coincidencias exactas, aunque existe el reporte de un individuo amazónico de ~4.000 años de antigüedad (Brasil) cuya secuencia coincide con la hallada en Madrid 2-41.
Conclusión. Los individuos del yacimiento arqueológico Madrid 2-41 están estrechamente emparentados entre sí por línea materna y presentan una secuencia aparentemente ausente en poblaciones actuales.
Palabras clave: ADN mitocondrial/análisis, polimorfismo de nucleótido simple, haplotipos, arqueología, población indígena, Colombia.
Introduction. Ancient bone remains constitute an important source of biological information, and their genetic characterization allows the confirmation or rebuttal of human affiliations proposed on the basis of non-molecular approaches. Pre-Columbian history of the Eastern Andes in Colombia has been divided into three main periods: (i) an early colonization by groups of hunter-gatherers, (ii) an intermediate period Herrera characterized by primitive agriculture and (iii) a late stage of Chibcha-speaking groups, with agriculture and ceramics (agroalfarero).
Objective. The mitochondrial DNA on ancient bone remains of the Herrera period were analyzed for comparison with modern and other ancient DNAs.
Materials and methods. Mitochondrial DNA was extracted from 11 Herrera individuals [~2,000 years before present (YBP)] found in the Madrid 2-41 archaeological site near Bogotá, Colombia. A 192 bp segment of the hypervariable segment I was amplified and sequenced, following stringent archaic DNA authenticity criteria. The sequences were compared with those in American and European databases using bioinformatics tools.
Results. All individuals had identical sequences and were classified as haplogroup B. This identity may be related to the type of ritual burial performed in the site, probably exclusively for members of a hierarchically important family of the ancient Herrera society. The search for homologous sequences in the American and European mtDNA data bases produced no identical coincidences, although a Brazilian Amazonic individual (~4,000 YBP) was recorded with a matching sequence.
Conclusion. Individuals buried in the Madrid 2-41 site were maternally closely related and showed a mtDNA sequence that is apparently absent in contemporary populations.
Key words: DNA, mitochondrial/analysis; polymorphism, single nucleotide; haplotypes, archaeology; indigenous population, Colombia.
Muchos de los estudios genéticos que buscan probar hipótesis relacionadas con el poblamiento de América y el mundo se basan en evidencias de ADN mitocondrial (ADNmt), debido a que el genoma de este organelo celular presenta una alta tasa de mutación en comparación con el genoma nuclear, lo cual permite mayor resolución en escalas cortas de tiempo, es heredado exclusiva-mente por línea materna y se asume una recombinación nula. Estas características permiten discriminar y cuantificar la relación genética entre individuos y poblaciones diferentes (1-3).
En las poblaciones nativas americanas se ha descrito la presencia de cuatro haplogrupos mitocondriales principales: A, B, C y D, definidos cada uno por una o más sustituciones exclusivas que los distinguen de otros haplogrupos en el mundo (2-5). Dichas poblaciones poseen, por lo general, una proporción definida de los cuatro haplogrupos, lo cual permite hacer inferencias sobre su origen y tiempo de divergencia de la población ancestral. Asimismo, es posible estudiar la presentación de eventos particulares tales como cuellos de botella, efectos fundadores o deriva génica (3,6).
Los haplogrupos antes mencionados están definidos con base en sitios de restricción ubicados dentro de la región codificante del ADN mitocondrial. Paralelamente, se han realizado análisis de las variaciones en la secuencia del segmento hipervariable I (HVS I), los cuales han demostrado la presencia de polimorfismos específicos de población que presentan correspondencia con aquéllos en la región codificante (1,6). Esto permite comparar los estudios realizados a partir de la secuenciación del HVS I con aquéllos basados en análisis de restricción de la región codificante (7,8).
En Colombia, los estudios de ADN mitocondrial en indígenas han demostrado que, a nivel general, las poblaciones presentan frecuencias relativamente homogéneas de los haplogrupos A (31,3%), B (30,5%) y C (28,5%), mientras que las frecuencias del haplogrupo D son normalmente muy bajas (6,6%). No obstante, se presentan variaciones regionales de las frecuencias que permiten hacer inferencias sobre el proceso de poblamiento y diversificación de los nativos en Colombia (9-14).
Por otra parte, Horai et al. (1993) estudiaron 65 individuos de Norteamérica, Centroamérica y Suramérica, incluidos 20 indígenas colombianos de distintas procedencias (una de la costa Caribe y tres de la región andina), y analizaron la secuencia del HVS I de su ADN mitocondrial (1). Estos investigadores hallaron que los haplotipos observados podían agruparse en cuatro haplogrupos (I, II, III y IV) y que el 50% de las muestras colombianas se clasificaban dentro del haplogrupo III. Estos resultados concuerdan con los reportados por Keyeux et al. en relación con las frecuencias de los haplogrupos A y D en indígenas colombianos (15).
La mayoría de las hipótesis y teorías planteadas sobre el poblamiento de América basadas en estudios genético-moleculares provienen de muestras de indígenas actuales. No obstante, una cantidad creciente de estudios de ADN arcaico, extraído de restos antiguos (principalmente, huesos y dientes), se han venido realizando en el mundo y en América, ampliando o refutando hipótesis planteadas con base en evidencias obtenidas a partir de ADN actual (16-28).
En el cuadro 1se presenta un resumen de los estudios más relevantes en ADN mitocondrial arcaico realizados en poblaciones suramericanas (19-26). Con base en dichos estudios, es posible observar que los haplogrupos más comunes en los periodos más antiguos fueron B y D, mientras que haplogrupos como el A y C sólo parecen estar presentes en épocas más recientes. Las excepciones a esta regla son los estudios de Ribeiro-Dos-Santos et al. y Moraga et al., en los que se encuentran individuos del haplogrupo A en épocas muy antiguas (>4.000 antes del presente) (23,26). La presencia de este haplogrupo en tiempos tan remotos pone en entredicho la propuesta ampliamente difundida sobre la migración inicial de poblaciones que poseían exclusivamente los haplogrupos B y D al continente suramericano, mientras que más adelante debieron entrar aquéllas con mayor frecuencia de haplogrupo A (1,3,15).
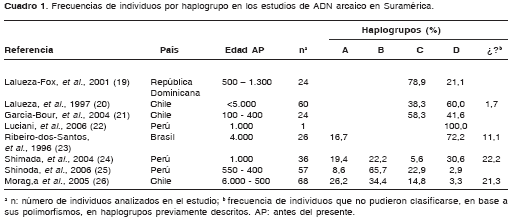
En Colombia se han realizado muy pocos estudios de ADN mitocondrial arcaico en humanos. Monsalve et al. analizaron el ADN mitocondrial de seis momias muiscas, incluyendo las regiones donde se encuentran las sustituciones que definen los haplogrupos amerindios y un segmento de la región control (HVS I) (27). Las momias estudiadas fueron halladas en territorio chibcha (Andes orientales), tres de las cuales no estaban asignadas específicamente a un grupo en particular. Las tres restantes pertenecían a los grupos tunebo, lache y muisca. Estos investigadores encontraron que tres de los individuos pertenecían al haplogrupo A, dos de ellos al haplogrupo C y, finalmente, un ejemplar presentó sustituciones características del haplogrupo B, aunque su ubicación en este grupo es incierta.
Por otro lado, Fernández (1999) realizó un análisis de los segmentos hipervariables I y II del ADN mitocondrial en restos óseos provenientes de diversas excavaciones arqueológicas, la mayoría de las cuales se encontraban en territorio chibcha, incluyendo Aguazuque, Checua, Herrera (vereda Chucua, Duitama, Boyacá) y San Agustín, y 14 muestras correspondientes al periodo agroalfarero (entre las cuales había representantes de los muiscas, guanes, laches y panches) (28). Este investigador analizó las muestras en relación con los tres periodos en los que se divide la historia del poblamiento del altiplano cundiboyacense: 1) lítico o paleoindio (checua entre ~8.000 y ~6.000 antes del presente y aguazuque entre ~5.000 y ~2.000 antes del presente); 2) formativo (Herrera entre ~3.000 y ~1.200 antes del presente); y 3) agroalfareros tardíos (muiscas,guanes, laches, panches, etc., desde el ~1.200 antes del presente hasta nuestros días). Sus resultados muestran que, de los cinco individuos del periodo lítico, cuatro se clasifican como haplogrupo B y uno como haplogrupo C; de las muestras del periodo formativo, una se clasificó como B (Herrera) y otra como D (San Agustín), y las muestras del periodo agroalfarero presentaron las siguientes frecuencias: A, 6,7%; B, 40%; C, 20%, y D, 26,7%.
Algunos de los individuos analizados por Fernández en 1999 (28) presentaron secuencias cuyos polimorfismos no pudieron ser clasificados en ninguno de los haplogrupos amerindios, por lo cual las frecuencias reportadas no suman 100%. La presencia de secuencias no pertenecientes específicamente a algún haplogrupo (que comúnmente se clasifican como E) es un fenómeno frecuente en este tipo de estudios (13-15,18,19,23).
Este investigador plantea que, a pesar de la presencia en los tres periodos del haplogrupo B, parece no haber existido un tronco genético ancestral de la población muisca, es decir, plantea la discontinuidad genética entre los periodos. No obstante, es imposible asegurar que no existieron otros haplotipos en las poblaciones que precedieron a los agroalfareros tardíos, dado el muestreo tan reducido. La falta de una muestra representativa de los dos primeros periodos impide analizar la diversidad genética a nivel mitocondrial y las frecuencias de los haplogrupos en dichas poblaciones, de tal forma que las hipótesis sobre la continuidad o discontinuidad de los pobladores a nivel genético carecen de sustento.
Es posible que los pobladores de los periodos precedentes al agroalfarero hayan evolucionado influidos tanto cultural como genéticamente por sociedades adyacentes, hasta alcanzar, por un lado, los desarrollos tecnológicos y culturales de los grupos humanos hallados por los conquista-dores en el siglo XVI y, por el otro, el genotipo característico del periodo agroalfarero. Este modelo de ocupación del territorio, así como cualquier otro que se haya desarrollado, se apoya en evidencias provenientes de diversas disciplinas, pero aún falta incluir un análisis de la información genética disponible, para lo cual es esencial ampliar el muestreo del periodo Herrera, del cual Fernández sólo estudió un ejemplar (28).
Materiales y métodos
Sujetos de estudio
Las muestras empleadas en este estudio corres-ponden a fragmentos óseos de los 11 individuos provenientes del yacimiento arqueológico denominado Madrid 2-41, hallado en marzo de 2003, el cual corresponde al periodo arqueológico Herrera, gracias a la asociación de los restos a material cerámico característico del periodo y a la datación por análisis de colágeno (29).
Maceración y digestión del tejido óseo
De acuerdo con el protocolo sugerido por Kalmar et al. (2000), inicialmente se elimina la superficie del fragmento óseo (~2 g) frotándolo suavemente con una lija estéril de tal forma que se eliminan las posibles contaminaciones de ADN actual por contacto. A continuación, se limpia con una solución de hipoclorito de sodio al 5% (30).
Este procedimiento ha demostrado destruir preferentemente el ADN contaminante, debido aparentemente a que el ADN arcaico se une a la hidroxiapatita del tejido óseo mientras el contaminante no (31). De esta forma, se elimina el posible ADN contaminante proveniente de las personas que manipularon los huesos con anterioridad a los procedimientos de extracción.
A continuación, el fragmento se envuelve en papel aluminio estéril y se pulveriza golpeándolo fuerte-mente. El hueso pulverizado se coloca en un tubo Falcon® estéril de 15 ml y se adicionan 5 ml de una solución jabonosa compuesta por 4,7 ml de solución tampón de lisis celular (EDTA 0,5 M, pH 8,0 y SDS al 10%) y 300 µl de solución de proteina-sa K en una concentración de 0,75 mg/ml. La reacción se incuba durante 24 horas en agitación constante a 37 °C y luego se coloca en baño serológico a 55 °C por 24 horas adicionales (30).
Extracción
Se hacen dos lavados sucesivos del tejido para remover la porción proteica mediante precipitación con acetato de amonio 10M. A continuación se precipitan los ácidos nucleicos con isopropanol absoluto, que ha demostrado, además, ser útil en la eliminación de compuestos inhibidores de PCR, frecuentes en este tipo de estudios (30).
El ADN así obtenido es sometido a una purificación adicional con un estuche comercial de extracción salina (Corpogen).
Amplificación
Las condiciones de amplificación se estandarizaron con base en el protocolo propuesto por Vigilant et al. (32). Se utilizó ADN extraído, empleando el protocolo antes mencionado, de restos óseos del yacimiento arqueológico Candelaria en la sabana de Bogotá (periodo agroalfarero), con el fin de reservar el material específico del estudio únicamente para cuando las condiciones de amplificación hubieran sido exitosamente probadas.
La solución de amplificación (solución tampón 1X, 2,0mM MgCl, 0,2mM dNTPs, 0,3µM de cada iniciador y 1,0 U/µl de Taq polimerasa en un volumen final de 25 µl) con 2 µl del ADN extraído fue sometida a 33 ciclos de desnaturalización a 95 °C por 1 minuto, anillamiento a 55 °C por 1 minuto y elongación a 72 °C por 1 minuto. Inicialmente se empleó un par de iniciadores que amplificaban una gran parte del segmento hipervariable I (359 pb) pero la ausencia total de productos amplificados obligó a un cambio en la estrategia de amplificación, pues se sabe que en ADN arcaico se encuentra fraccionado. Por ello, se empleó otro par de iniciadores que amplificaban un fragmento de 192 pb, un tamaño de fragmento más factible de ser amplificado. Estos últimos iniciadores fueron: a- L16209: 5-CCATGCTTACAA GCAAGT-3 y b- H16401: 5-TGATTTCACGGAGG ATGGTG-3.
Fue necesaria la implementación de un sistema de dos amplificaciones sucesivas, en la segunda de las cuales se emplea el producto de la amplificación de la primera como ADN molde (reamplificación).
Secuenciación
La secuenciación de los fragmentos amplificados de 192 pb en este estudio se realizó en el secuenciador automático ABI PRISM 3100 Avant® del Instituto de Genética Humana de la Pontificia Universidad Javeriana, empleando el kit BigDye Terminator® de Applied Biosystems.
Durante todo el proceso se siguieron de la manera más estricta posible las recomendaciones comúnmente aplicadas al trabajo con ADN arcaico (33):
1) el manejo del material óseo se realizó con guantes previamente lavados con hipoclorito de sodio al 5% y para cada individuo se utilizaron guantes diferentes;
2) se empleó material estéril para cada procedimiento en cada uno de los pasos (extracción, amplificación y secuenciación);
3) se colocaron blancos negativos en las amplificaciones;
4) se secuenció el mismo segmento del ADN mitocondrial de los investigadores que estuvieron en contacto directo con el material óseo, y
5) se realizaron extracciones, amplificaciones y secuenciaciones independientes de dos huesos de un mismo individuo para confirmar la coincidencia de la secuencia.
Estas medidas y otras evidencias explicadas más adelante permiten tener una gran confianza en que el ADN estudiado es efectivamente de los indivi-duos pertenecientes al yacimiento Madrid 2-41.
Análisis de los datos
Las secuencias obtenidas se compararon con secuencias homólogas consignadas en la base de datos de ADN mitocondrial humano de la Federal Bureau of Investigation (FBI) de los Estados Unidos y con aquéllas en la base de datos EMPOP de la Unión Europea.
La comparación de las secuencias en la primera de estas bases de datos se realizó con el programa de distribución gratuita mtDNA en el cual se incorporan las secuencias del HVS I y HVS II reportadas por laboratorios de investigación a nivel global, así como las secuencias obtenidas en investigaciones forenses (34).
La comparación de las secuencias en la base de datos europea, conocida como EMPOP, se realizó mediante el aplicativo web montado en el sitio de internet de dicha organización (www.empop.org).
Resultados
A pesar de que el ADN extraído de varios individuos no presentó una banda clara en el gel de agarosa al 2%, o que algunos de ellos mostraban evidencias de degradación (fragmentación) molecular, se logró la amplificación exitosa a partir de nueve de los once individuos estudiados. Inicialmente se intentó amplificar el HVS I completo (359 pb), pero todos los intentos fueron fallidos a pesar de que los controles positivos amplificaban correcta-mente. Esto permitió concluir que muy probablemente los extractos de ADN arcaico carecían o contenían una muy baja concentración de fragmentos de ADN mitocondrial de dicha longitud que sirvieran adecuadamente de molde para la amplificación, lo cual puede resultar del hecho de que el ADN sufre un proceso de fragmentación a través del tiempo. En este tipo de estudios se espera encontrar fragmentos de ADN de 100 a 200 pb, aproximadamente (30).
La secuenciación de los fragmentos de 192 pb se logró en seis de los nueve individuos cuyo ADN pudo ser amplificado y los resultados de ésta se presentan en el cuadro 2. La coincidencia de la secuencia del individuo A11 en la extracción, amplificación y secuenciación independiente a partir de dos fragmentos óseos distintos agrega una evidencia adicional a la autenticidad de los resultados de este estudio.
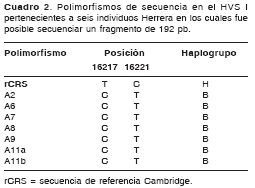
Cabe aclarar que las secuencias encontradas en los restos óseos no coincidieron con las de los investigadores que manipularon el material durante la experimentación.
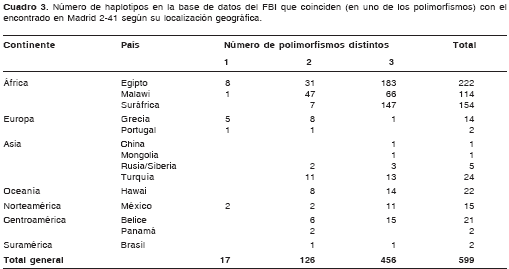
La comparación de las secuencias con aquéllas consignadas en la base de datos del FBI se presenta en el cuadro 3. La comparación con las secuencias de la base de datos EMPOP no generó información adicional a la encontrada con la del FBI, por lo cual sus resultados no se presentan en el presente artículo.
Discusión
A primera vista resalta el hecho de que todos los individuos analizados presentaron el mismo haplotipo, con dos polimorfismos evidentes: una transición T-C en la posición 16.217 y otro C-T en la posición 16.221 respecto a la secuencia de referencia rCRS (revised Cambridge Reference Sequence). El primero de estos polimorfismos fue reportado inicialmente por Horai y Hayasaka en 1990, aunque no se le asignó relación con algún grupo étnico ni con alguna ubicación geográfica en particular (35).
Torroni et al. (1993), por su parte, realizaron una comparación entre los haplogrupos mitocondriales basados en fragmentos de restricción definidos como A, B, C y D, y las secuencias de la región control de 321 nativos americanos pertenecientes a 17 etnias diferentes representantes de todo el continente (14). Uno de sus hallazgos más significativos fue una segregación coincidente de las secuencias de la región control con los haplogrupos establecidos para la región codificante. Es decir, se presentaron polimorfismos característicos en la secuencia de la región control que reiteraron la clasificación de los grupos mitocondriales ya establecida con base en la región codificante. Uno de estos polimorfismos característicos fue precisamente T16217C que, junto con una transición T-C en la posición 16.189, se presentaba inequívocamente sólo en individuos previamente definidos como haplogrupo B.
Con base en esto, es posible clasificar los individuos Herrera estudiados como pertenecientes al haplo-grupo B y, al ser todos coincidentes, es probable que se trate de un grupo humano estrechamente emparentado, con mucha endogamia. No obstante, es posible que la muestra de individuos analizada no sea realmente representativa de la población asentada en las márgenes de lo que hoy se conoce como la laguna de la Herrera hace, aproximada-mente, 2.000 años. En este caso, es posible formular la hipótesis de que los individuos analizados pertenecen a un grupo de parentesco dentro de la sociedad Herrera, y que los cuerpos corresponden a entierros reservados a distintas generaciones de una misma familia dominante. Esto concuerda con el tipo de entierro ritual y la utilería asociada a los cuerpos hallados en Madrid 2-41 (29).
La homogeneidad temporal del linaje mitocondrial encontrado en Madrid 2-41 puede estar relacionada con la práctica de sucesión del poder en el grupo de parentesco dominante de aquella sociedad. Se sabe que entre los muiscas, cultura posterior a los Herrera, la herencia del poder y de la propiedad se daba entre hermanos y sobrinos hijos de las herma-nas, lo cual garantizaba que un mismo linaje materno mantendría el estatus social y la propiedad (36).
La existencia de una élite o grupo de parentesco dominante sugiere que la sociedad Herrera había alcanzado un nivel de desarrollo en el que existía jerarquización social, probablemente gracias a una producción de alimentos avanzada que permitía la especialización del trabajo, tal y como ha sido planteado por Langebaek (37).
Por otro lado, el haplotipo hallado en Madrid 2-41 fue comparado con secuencias homólogas de todo el mundo, consignadas en la base de datos mtDNA Population Database del FBI (34). Si bien no se halla ningún haplotipo idéntico en toda la base de datos, existen varias coincidencias en distintas localidades geográficas. Ningún haplotipo de la base de datos presentó ambos polimorfismos simultáneamente. En el cuadro 3 se muestra, por ejemplo, que en África nueve haplotipos presentaron sólo un polimorfismo de diferencia con el haplotipo Madrid 2-41, ocho de ellos en Egipto (de una muestra de ADN mitocondrial arcaico) y uno en Malawi. En otras palabras, nueve haplotipos presentaron el polimorfismo T16217C o el C16221T, pero ninguno presentó ambos a la vez. En el continente americano, sólo en México, más específicamente en nativos de la península de Yucatán, se encuentran haplotipos que presentan un solo polimorfismo de diferencia con la secuencia Madrid 2-41. Este mismo ejercicio se hizo con la base de datos EMPOP de la Unión Europea, con resultados muy similares.
No obstante la ausencia de este haplotipo en la base de datos, Ribeiro-Dos-Santos et al. reportan un haplotipo que presenta ambos polimorfismos en un individuo amazónico de 4.000 antes del presente del Museo Paraense Emilio Goeldi, Pará, Brasil (23). A pesar de que el individuo brasileño presenta otros polimorfismos adicionales, es paradójico que este haplotipo haya sido encontrado en un individuo del periodo paleoindio, hecho que sugiere que el linaje encontrado en Madrid 2-41 pudo haber estado dentro de los individuos que colonizaron inicialmente el continente suramericano.
Estas observaciones permiten conjeturar sobre el origen del haplotipo encontrado en Madrid 2-41. Al parecer, no hay reportes de un haplotipo igual en el mundo: la base de datos del FBI cuenta con, al menos, 10.828 secuencias de todo el mundo, recolectadas de la literatura científica, de GeneBank y de una base de datos del SWGDAM (Scientific Working Group on DNA Analysis Methods), y se actualiza periódicamente (34). Este hecho podría significar que el haplotipo reportado en el presente estudio corresponde a un linaje que se extinguió una vez que los indígenas pasaron por el cuello de botella asociado a la llegada de los españoles. No obstante, cabe formular la hipótesis, asimismo, que el linaje pudo haberse perdido por eventos de mutación que revirtieron los polimorfismos característicos de este haplotipo.
Agradecimientos
Los autores a Elizabeth Vargas y Patricia Jara por su colaboración en la manipulación experimental. También agradecen a Marta Roa y Cristina Sánchez por la investigación bibliográfica.
Conflictos de intereses
Los autores declaramos que durante la realización de este estudio no incurrimos en conflicto de interés alguno que pudiera haber afectado sus resultados.
Financiación
Este trabajo fue financiado en su totalidad por el Instituto de Genética Humana de la Pontificia Universidad Javeriana.
Correspondencia:
Alejandro Silva, Instituto de Genética Humana, Pontificia Universidad Javeriana, Carrera 7 Nº 40-62, Bogotá, D.C., Colômbia. Teléfono: (571) 320 8320, extensión 2792; fax: (571) 320 8320, extensión 2793Referencias
1. Horai S, Kondo R, Nakagawa-Hattori Y, Hayashi S, Sonoda S, Tajima K. Peopling of the Americas, founded by four major lineages of mitochondrial DNA. Mol Biol Evol. 1993;10:23-47. [ Links ]
2. Schurr TG, Ballinger SW, Gan YY, Hodge JA, Merriwether DA, Lawrence DN, et al. Amerindian mitochondrial DNAs have rare Asian mutations at high frequencies, suggesting they derived from four primary maternal lineages. Am J Hum Genet. 1990;46:613-23. [ Links ]
3. Schurr TG. The peopling of the new world: Perspectives from molecular anthropology. Annu Rev Anthropol. 2004;33:551-83. [ Links ]
4. Torroni A, Schurr TG, Yang CC, Szathmary EJ, Williams RC, Schanfield MS, et al. Native American mitochondrial DNA analysis indicates that the Amerind and the Nadene populations were founded by two independent migrations. Genetics. 1992;130:153-62. [ Links ]
5. Melton PE, Briceño I, Gómez A, Devor EJ, Bernal JE, Crawford MH. Biological relationship between Central and South American Chibchan speaking populations: evidence from DNA. Am J Phys Anthropol. 2007;133:753-70. [ Links ]
6. Wallace DC, Garrison K y Knowler WC. Dramatic founder effects in Amerindian mitochondrial DNA. Am J Phys Anthropol. 1985;68:149-55. [ Links ]
7. Bailliet G, Rothhammer F, Carnese FR, Bravi CM, Bianchi NO. Founder mitochondrial haplotypes in Amerindian populations. Am J Hum Genet. 1994; 55: 27-33. [ Links ]
8. Macaulay V, Richards M, Hickey E, Vega E, Cruciani F, Guida V, et al. The emerging tree of West Eurasian mtDNAs: A synthesis of control-region sequences and RFLPs. Am J Hum Genet. 1999;64:232-49. [ Links ]
9. Rondón-González F, Cifuentes L, Cárdenas H, Barreto G. Evaluación de la diversidad genética mediante el análisis de mtDNA en poblaciones aisladas del centro y suroccidente colombiano. Salud Uninorte. 2004;18:74. [ Links ]
10. Rondón-González F, Torres AM, Barreto G. Relaciones filéticas de comunidades indígenas colombianas a partir de la comparación de secuencias de mtDNA y sus implicaciones en el poblamiento del continente americano. Salud Uninorte. 2004;18:88. [ Links ]
11. Briceño I, Umaña A, Bernal JE, Torres DM. Estudios moleculares en poblaciones amerindias. Salud Uninorte. 2004;18:76. [ Links ]
12. Acosta MA, Salas A, Álvarez V, Lareu MV, Carracedo A. El ADN mitocondrial revela la condición multiétnica de las poblaciones del Cauca (Colombia). Salud Uninorte. 2004;18:78. [ Links ]
13. Roa M. Polimorfismos de la región control del ADN mitocondrial humano en una muestra de población mestiza del altiplano cundiboyacense colombiano. (tesis). Bogotá, D.C.: Universidad Nacional de Colombia; 2005. p. 140. [ Links ]
14. Torroni A, Schurr TG, Cabell MF, Brown MD, Neel JV, Larsen M, et al. Asian affinities and continental radiation of the four founding native American mtDNAs. Am J Hum Genet. 1993;53:563-90. [ Links ]
15. Keyeux G, Rodas C, Gelvez N, Carter D. Possible migration routes into South America deduced from mitochondrial DNA studies in Colombian Amerindian populations. Hum Biol. 2002;74:211-33. [ Links ]
16. Jones M. Ancient DNA in pre-Columbian archaeology: a review. J Archaeol Sci. 2003;30:629-35. [ Links ]
17. Lleonart R, Riego E, Rodríguez R, Travieso R, de la Fuente J. Analyses of DNA from ancient bones of a pre-columbian Cuban woman and a child. Genet Mol Biol. 1999;22:285-89. [ Links ]
18. Stone A, Stoneking M. mtDNA analysis of a prehistoric Oneota population: implications for the peopling of the New World. Am J Hum Genet. 1998;62:1153-70. [ Links ]
19. Lalueza C, Perez-Perez A, Prats E, Cornudella L, Turbon D. Lack of founding American mitochondrial DNA lineages in extinct aborigines from Tierra del Fuego-Patagonia. Hum Mol Genet. 1997;6:41-6. [ Links ]
20. Lalueza-Fox C, Luna Calderon F, Calafell F, Morera B, Bertranpetit J. MtDNA from extinct Tainos and the peopling of the Caribbean. Ann Hum Genet. 2001;65:137-51. [ Links ]
21. García-Bour J, Pérez-Pérez A, Álvarez S, Fernández E, López-Parra AM, Arroyo-Pardo E, et al. Early population differentiation in extinct aborigines from Tierra del Fuego-Patagonia: Ancient mtDNA sequences and Y-chromosome STR characterization. Am J Phys Anthropol. 2004;123:361-70. [ Links ]
22. Luciani S, Fornaciari G, Rickards O, Martínez C, Rollo F. Molecular characterization of a Pre-Columbian mummy and in situ coprolite. Am J Phys Anthropol. 2006;129:620-9. [ Links ]
23. Ribeiro-Dos-Santos AK, Santos SE, Machado AL, Guapindaia V, Zago M. Heterogeneity of mitochondrial DNA haplotypes in Pre-Columbian natives of the Amazon region. Am J Phys Anthropol. 1996;101:29-37. [ Links ]
24. Shimada I, Shinoda K, Farnum J, Corruccini R, Watanabe H. An integrated analysis of Pre-Hispanic mortuary practices: A middle Sicán case study. Curr Anthropol. 2004;45:369-402. [ Links ]
25. Shinoda K, Adachi N, Guillen S, Shimada I. Mitochondrial DNA analysis of ancient Peruvian highlanders. Am J Phys Anthropol. 2006;131:98-107. [ Links ]
26. Moraga M, Santoro CM, Standen VG, Carvallo P, Rothhammer F. Microevolution in prehistoric Andean populations: Chronologic mtDNA variation in the desert valleys of northern Chile. Am J Phys Anthropol. 2005; 127:170-81. [ Links ]
27. Monsalve MV, Cárdenas F, Guhl F, Delaney AD, Devine DV. Phylogenetic analysis of mtDNA lineages in South American mummies. Ann Hum Genet. 1996;60:293-303. [ Links ]
28. Fernández C. La arqueología molecular aplicada a la solución de problemas prehistóricos: análisis de ADN mitocondrial en momias y restos óseos prehispánicos (tesis). Bogotá: Universidad Nacional de Colombia; 1999. [ Links ]
29. Rodríguez JV, Cifuentes A. Un yacimiento formativo ritual en el entorno de la antigua laguna de La Herrera, Madrid, Cundinamarca. Maguaré. 2005;19:103-31. [ Links ]
30. Kalmár T, Bachrati CZ, Marcsik A, Raskó I. A simple and efficient method for PCR amplifiable DNA extraction from ancient bones. Nucleic Acids Res. 2000;28:e67. [ Links ]
31. Kemp BM, Smith DG. Use of bleach to eliminate contaminating DNA from the surface of bones and teeth. Forensic Sci Int. 2005;154:53-61. [ Links ]
32. Vigilant L, Pennington R, Harpending H, Kocher TD, Wilson AC. Mitochondrial DNA sequences in single hairs from a southern African population. Proc Natl Acad Sci USA. 1989;86:9350-4. [ Links ]
33. Cooper A, Poinar HN. Ancient DNA: Do it right or not at all. Science. 2000;289:1139. [ Links ]
34. Monson KL, Miller KW, Wilson MR, DiZinno JA, Budowle B. The mtDNA population database: An integrated software and database resource for forensic comparison. Forensic Sci Commun. 2002;4. [Fecha de consulta: agosto de 2007]. Disponible en: http://www.fbi.gov/hq/lab/fsc/backissu/april2002/miller1.htm [ Links ]
35. Horai S, Hayasaka K. Intraspecific nucleotide sequence differences in the major noncoding region of human mitochondrial DNA. Am J Hum Genet. 1990;46:828-42. [ Links ]
35. Correa F. El sol del poder. Bogotá D.C.: Unibiblos, Universidad Nacional de Colombia; 2004. [ Links ]
37. Langebaek CH. Regional archaeology in the Muisca territory: a study or the Fúquene and Susa Valleys. Memoirs in Latin American Archaeology No. 9, Bogotá, University of Pittsburgh Latin American Archaeology Publications and Universidad de los Andes; 1995. [ Links ]














