Servicios Personalizados
Revista
Articulo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Accesos
Accesos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares en
SciELO
Similares en
SciELO -
 Similares en Google
Similares en Google
Compartir
Biomédica
versión impresa ISSN 0120-4157versión On-line ISSN 2590-7379
Biomédica v.28 n.4 Bogotá oct./dic. 2008
1 Departamento de Parasitología, Instituto Pedro Kourí, La Habana, Cuba
2 Programa de Estudio y Control de Enfermedades Tropicales (PECET), Universidad de Antioquia, Medellín, Colombia
3 Institute of Tropical Medicine, Molecular Parasitology Lab, Nationaalstraat, Antwerp, Belgium
Recibido: 25/02/08; aceptado:04/08/08
Introducción. El análisis de la longitud de los fragmentos de restricción del producto amplificado y el estudio del ADN polimórfico amplificado al azar han demostrado ser herramientas útiles para la tipificación de Leishmania.
Objetivos. Estudiar la utilidad de las técnicas moleculares para la identificación y tipificación de cepas de referencia de Leishmania spp. del Nuevo Mundo y valorar su aplicabilidad a muestras clínicas.
Materiales y métodos. Se aplicó PCR para amplificar el gen que codifica la cisteíno-proteinasa B, y el análisis de la longitud de los fragmentos de restricción del producto amplificado utilizando ácido desoxirribonucleico de 16 cepas de referencia de Latinoamérica y de muestras clínicas de pacientes colombianos con leishmaniasis, y la técnica del ácido desoxirribonucleico polimórfico amplificado al azar utilizando ocho cepas de referencia. Se establecieron los patrones de bandas en cada caso.
Resultados. Se obtuvo producto de amplificación en la PCR para Leishmania braziliensis, L. peruviana, L. panamensis y L. guyanensis. Para el resto, no fue posible amplificar el gen con los cebadores utilizados. La restricción mostró un patrón de bandas común para L. peruviana, L. guyanensis y L. panamensis, mientras L. braziliensis, presentaba un perfil individual único. El análisis de restricción del producto amplificado generó un patrón de bandas similar en los cinco pacientes estudiados, que se correspondía con el patrón generado por L. peruviana, L. guyanensis o L. panamensis. Mediante la amplificación al azar se obtuvieron patrones de bandas reproducibles con todas las cepas estudiadas, que posibilitaron la diferenciación. Se discuten las ventajas y limitaciones de ambos procederes.
Conclusiones. El combinar ambas metodologías resultaría útil para identificar especies de importancia médica, tomando en cuenta sus ventajas y desventajas.
Palabras clave: Leishmania, leishmaniasis/diagnóstico, reacción en cadena de la polimerasa, polimorfismo de longitud del fragmento de restricción.
Introduction. The analysis of the PCR-restriction fragment length polymorphism and random amplified polymorphic DNA have been useful tools for Leishmania identification.
Objectives. Molecular procedures were demonstrated for identification and typing of reference strains of New World Leishmania and their applicability was validated for clinical samples.
Materials and methods. DNA was extracted from 16 reference strains of Latin American Leishmania as well as from clinical samples of leishmaniasis patients. A sequence coding for cysteine proteinase B was amplified by PCR and subjected to restriction fragment length polymorphism analysis. The enzyme used was Taq1. For eight of the reference strains, the random amplified polymorphic desoxyribonucleic acid technique (RAPD) was applied. Band patterns for Leishmania species differentiation were established each each method. The sample size of the clinical sample was of 5.
Results. PCR products of the cysteine proteinase B gene were obtained for L. braziliensis, L. peruviana, L. panamensis and L. guyanensis. For the other species, L. mexicana, L. amazonensis, L. garnhami, L. lainsoni, L. chagasi, L. naiffi, no amplification occurred. The patterns of restriction fragments revealed band patterns in common for L. peruviana, L. guyanensis and L. panamensis, whereas L. braziliensis had a distinctive pattern. When human samples were examined, amplification occurred for all cases, and the profiles corresponded to the common profile of L. peruviana, L. guyanensis and L. panamensis. The RAPD technique demonstrated reproducible and distinctive patterns for each of the 8 reference strains, L. mexicana, L. amazonensis, L. garnhami, L. lainsoni, L. chagasi, L. naiffi, making possible to differentiate all them. The advantages and limitations of each procedure are discussed.
Conclusions. The combination of RFP and RAPD methodologies provide useful tools to identify medical important species of Leishmania by recognizing DNA sequences characteristic of each species.
Key words: Leishmania, leishmaniasis/diagnosis, polymerase chain reaction; polymorphism, restriction fragment length.
La leishmaniasis, enfermedad causada por parásitos protozoos del género Leishmania, se caracteriza por un considerable pleomorfismo clínico y epidemiológico, muy relacionado con la diversidad de especies involucradas y sus vectores, además de los factores ligados al huésped (1).
La enfermedad se presenta en 88 países de todas las latitudes y 368 millones de personas se encuentran en riesgo de contraerla (2), por lo que el diagnóstico seguro y una conducta epidemiológica adecuada son cruciales como vías para lograr su control.
La tipificación de especies de Leishmania causan-tes de infecciones clínicas en areas geográficas donde varias especies se transmiten de forma simpátrica es importante por razones clínicas y epidemiológicas, entre otras: las lesiones pueden ser causadas por otros patógenos, diferentes especies pueden dar lugar a una misma presentación clínica, así como diferentes especies pueden requerir distintos tratamientos (3).
Tanto el análisis de la longitud de los fragmentos de restricción del producto amplificado (PCR-RFLP) (4,5), como el estudio del ADN polimórfico amplificado al azar (RAPD) (6) han demostrado ser herramientas útiles para la tipificación de parásitos, aunque ambas metodologías tienen ven-tajas y desventajas particulares para su aplicación.
Basándonos en lo anterior, amplificamos el gen que codifica para la cisteíno-proteinasa B (Cpb) de Leishmania utilizando ADN puro de un grupo de cepas de referencia y desarrollamos la PCR-RFLP/Cpb, así como el RAPD empleando cebadores OPA, con el fin de estudiar la utilidad de ambas metodologías, tanto en la detección como en la tipificación de Leishmania spp. Asimismo, se verificó la aplicabilidad de la PCR-RFLP/Cpb empleando ADN aislado de biopsias de cinco pacientes con leishmaniasis cutánea confirmada.
Materiales y métodos
Cepas
Las cepas de referencia utilizadas provienen del criobanco del Instituto de Medicina Tropical de Amberes y aparecen listadas en el cuadro 1.
Los parásitos se mantuvieron en su forma promastigote a 26 °C en medio RPMI (Gibco) o Schneider (SIGMA), con suplemento de 10% de suero fetal bovino, 250 µg/ml de estreptomicina y 100 UI de penicilina.
Extracción del ADN genómico
Se obtuvo ADN genómico de parásitos en su fase estacionaria de crecimiento por extracción rutinaria con fenol-cloroformo (7); la calidad del ADN se verificó por electroforesis en gel de agarosa al 0,8% en tampón TBE 0,5%, (45 mM Tris, 45 mM borato y 1 mM EDTA, pH 8,0) y bromuro de etidio (0,5 µg/ml), mediante corrida electroforética a 150 V, por 30 minutos. La visualización se realizó en un transiluminador UV.
Además, se evaluó el ADN aislado de las biopsias de cinco pacientes con leishmaniasis cutánea, en los cuales se había confirmado previamente la presencia del parásito por examen microscópico directo, cultivo o PCR, en el Programa de Estudio y Control de Enfermedades Tropicales (PECET) de la Universidad de Antioquia, Colombia. El ADN obtenido de las biopsias se purificó empleando el mini estuche de extracción QIAamp DNA (Qiagen®), siguiendo las instrucciones del fabricante. A todos los pacientes de quienes se obtuvieron las biopsias se les solicitó consentimiento informado y las muestras fueron tomadas siguiendo las indicaciones institucionales para ese fin, así como lo expresado en la Declaración de Helsinski.
Cebadores
Los cebadores utilizados para la PCR/Cpb fueron reportados por García y colaboradores (1) y sintetizados por Sigma Genosys (Inglaterra). La selección de los cebadores empleados en el RAPD, se hizo con base en una tamización previa realizada en el laboratorio (8) de varios cebadores comerciales de Operon Technologies (Kit OPA®, Operon Technologies, USA). Con base en esta tamización, se seleccionaron aquéllos con patrones reproducibles para la amplificación arbitraria de marcadores de ADN. La secuencia (5-3) de todos los cebadores son: Cpb fwd: TGTGCTATTCGAGGAGTTCAA; Cpb rvs: TTACCCTCAGGAATCACTTTGT; OPA-3: agtcagccac; OPA-4: aatcgggctg; OPA-8: gtgacgtagg
PCR/Cpb
La reacción en cadena de la polimerasa para amplificar el gen que codifica para la Cpb de Leishmania fue optimizada, evaluando las concentraciones más adecuadas de cada componente de la reacción. La mezcla (50 µl) contenía,1X de tampón de la Taq polimerasa (Promega, USA), 1X tampón de amplificación de la enzima (50 mM tris-HCl pH=8,0; 100 mM NaCl; 0,1 mM EDTA; 1 mM DTT; 50% glicerol y 1% Tritón X-100) (Promega, USA), 200 µM de cada dNTP; 1,5 mM MgCl2; 1,0 U Taq polimerasa, 0,4µM de cebadores, 0,25% de DMSO y 50 ng de ADN molde de las cepas de referencia, o 2,5 µl de ADN extraído de las biopsias.
El procedimiento utilizado fue el siguiente: desnaturalización inicial a 94 °C por 5 minutos, seguido de 33 ciclos cada uno consistente en: 94 °C por 30 s, 53 °C por 1 minuto y 72 °C por 3 minutos. Finalmente, se realizó una extensión de 8 minutos a 72 °C. Un control negativo que contenía todos los componentes de la reacción, excepto ADN, se incluyó en cada experimento. Los resultados se verificaron por electroforesis en gel de agarosa al 2% teñido con bromuro de etidio 0,5 µg/ml. Este protocolo se siguió utilizando como molde ADN de todas las cepas de referencia utilizadas, así como ADN de las biopsias de los cinco pacientes.
RFLP/Cpb
Los productos de amplificación obtenidos de cada muestra se concentraron por precipitación en etanol y se resuspendieron en 20 µl de agua desionizada. Posteriormente, se digirieron con TaqI a 65 °C toda la noche, en un volumen final de 10 µl, siguiendo las recomendaciones de los productores. El tamaño de los fragmentos productos de las digestiones (RFLP) se determinó por electroforesis en gel de agarosa para pequeños fragmentos (Promega, USA) al 3%, a 50 voltios constantes. El análisis del tamaño de los fragmentos generados permitió establecer los patrones de bandas característicos para cada especie o grupo de especies, registrando la presencia o ausencia de bandas en cada caso.
RAPD
Las concentraciones de los reactivos empleados en la reacción de RAPD fueron previamente optimizadas. Se realizó el RAPD utilizando como molde ADN extraído de ocho especies de referencia de Leishmania, con el fin de conocer la posible utilización de esta técnica para la identificación de las mismas, así como para revelar la presencia de polimorfismo genético entre ellas. La amplificación se llevó a cabo en una mezcla de reacción de 25 µl con 2,5 µl de tampón 10X de la PCR (50 mM tris-HCl pH 8,3,100 mM NaCl, 0,1 mM EDTA, 1 mM DTT, 50 % glycerol, 1% tritón X-100) (Promega, USA), 200 µM de cada dNTP (Promega, USA), 2 mM MgCl2, 5 pmol de los cebadores, 2 U de Taq polimerasa (Promega, USA) y, aproximadamente, 75 ng de ADN molde. Se incluyeron controles negativos para las reacciones con cada cebador OPA utilizado, la cual contenía todos los componentes de la reacción excepto ADN molde. La amplificación consistió en: desnaturalización inicial a 94 °C por 5 minutos, seguida de 40 ciclos de 1 minuto a 94 °C, 1 minuto a 35 °C y 2 minutos a 72 °C, así como un paso de extensión final a 72 °C por 15 minutos. Los productos amplificados fueron analizados por electroforesis en gel de agarosa al 1,2% en tampón TBE con bromuro de etidio (0,5 µg/ml) y visualizados por medio de un transiluminador UV.
Análisis del polimorfismo genético
La presencia o ausencia de bandas individuales para cada especie fueron registradas (1 o 0) y el coeficiente de similaridad de Jaccard (9) se utilizó en la fórmula siguiente: Sj= 1-a/(a+b+c), donde a representa el número de bandas compartidas entre dos especies, b representa el número de bandas presentes en la especie 1 y ausentes en la 2, mientras que c representa el número de bandas ausentes en la especie 1 y presentes en la 2.
La relación filogenética se determinó mediante la estrategia de agrupamiento UPGMA (9), utilizando el paquete de Programas FreeTree, versión 0.9.1.59 (10). El dendrograma se construyó con base en los resultados obtenidos con los tres cebadores y el análisis de bootstrap se realizó con 1.000 réplicas.
Consideraciones éticas
La realización de esta investigación estuvo avalada por el Comité de Ética del Instituto Pedro Kourí (CEIPK), La Habana, Cuba, y en lo concerniente a la toma de muestras clínicas, por el Comité de Ética de la Universidad de Antioquia, en Medellín, Colombia.
Resultados
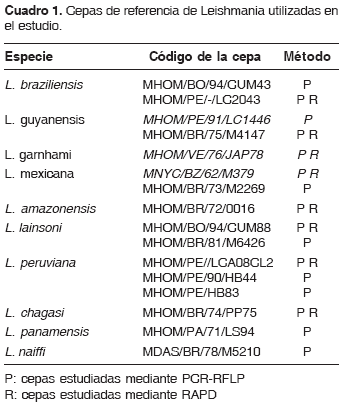
Se realizó la PCR/Cpb utilizando ADN de 15 cepas de referencia de 10 diferentes especies de Leishmania del Nuevo Mundo. La electroforesis en gel de agarosa (figura 1) muestra que los cebadores empleados fueron capaces de amplificar ADN sólo en 4 de las 10 especies estudiadas, generando un amplicón de 1.170 pb, aproximadamente, en: L. braziliensis, L. peruviana, L. panamensis y L. guyanensis. No fue posible amplificar el gen que codifica para la Cpb B con los cebadores utilizados en el resto de las especies estudiadas: L. mexicana, L. amazonensis, L. chagasi, L. naiffi, L. garnhami y L. lainsoni. No se encontraron diferencias en los resultados cuando se estudiaron distintas cepas de una misma especie.
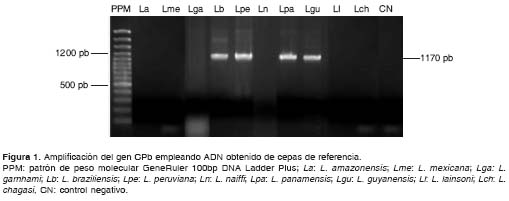
Los productos de amplificación digeridos con la enzima TaqI, mostraron distintos patrones de bandas (figura 2). Mientras L. peruviana, L. guyanensis y L. panamensis comparten un patrón de bandas común, L. braziliensis presenta un perfil individual único. Fue evidente la digestion total, dado que la suma del tamaño de los distintos fragmentos obtenidos representan la totalidad del tamaño del amplicón.
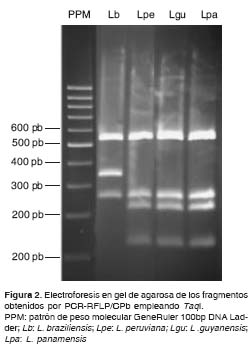
Cuando utilizamos ADN proveniente de biopsias, con diagnóstico confirmado de leishmaniasis, se obtuvo amplificación específica en todos los casos, así como en el control positivo, constituido por 100 ng de ADN de una cepa de referencia de L. braziliensis (figura 3). El análisis de restricción del producto amplificado, generó un patrón de bandas similar en todos los pacientes analizados y similar al perfil generado con las cepas de referen-cia de L. peruviana, L. guyanensis o L. panamensis, pero diferente del obtenido en el control positivo de la reacción, cuyas bandas corresponden a L. braziliensis (figura 4).
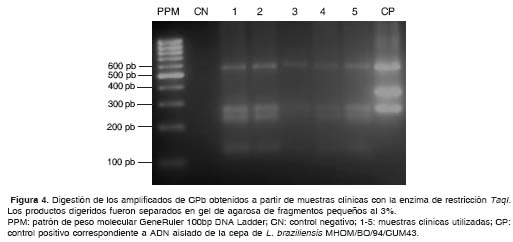
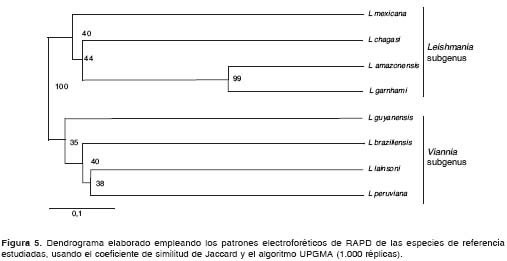
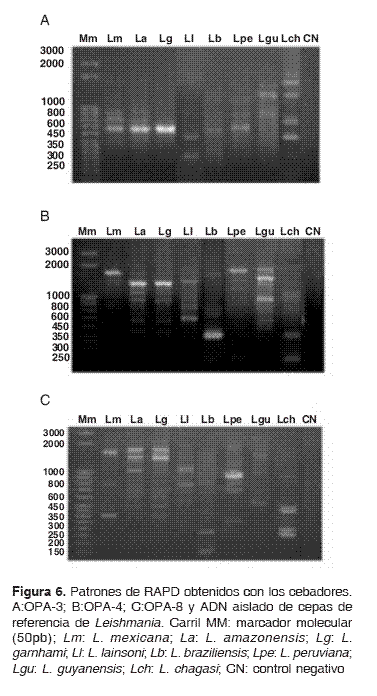
Empleando la RAPD, se obtuvieron patrones de bandas reproducibles utilizando como molde ADN puro de todas las cepas de referencia estudiadas (figura 5), que posibilitan la diferenciación de cada una de las especies evaluadas. El análisis filogenético realizado con estos patrones, muestra la relación entre las especies estudiadas (figura 6), donde se observan dos agrupaciones claramente distinguibles correspondientes a las especies que pertenecen a los subgéneros Leishmania y Viannia, con valor de distancia SJ entre los dos grupos de 0,54.
Discusión
Los datos de vigilancia epidemiológica a nivel global indican que el número de casos de leishmaniasis se ha incrementado en las últimas décadas. Esto puede explicarse, en parte, por el desarrollo de nuevos métodos diagnósticos y, por tanto, el reporte de más casos, pero también por otras razones como el control inadecuado de vectores o de reservorios, la detección asociada a infecciones oportunistas, la urbanización del vector, la deforestación, la resistencia a los medica-mentos, los conflictos armados y el turismo (11).
Ninguno de estos factores resulta ajeno en el caso de la leishmaniasis tegumentaria americana, en la que la tipificación temprana y segura de los parásitos puede ser de importancia crucial para seguir el curso clínico de la enfermedad, indicar el tratamiento adecuado (12) y evaluar los riesgos epidemiológicos (13), considerando la diversidad de especies y reservorios implicados en la transmisión. Por esta razón, nos propusimos identificar posibles marcadores moleculares para la tipificación útil en nuestras condiciones.
Aunque el análisis de isoenzimas continúa siendo la técnica de referencia para la tipificación, es un proceso demorado y muy laborioso, por lo que otros métodos, basados en la PCR y sus variantes, se han utilizado para discriminar entre las especies causantes de las lesiones cutáneas (14,15).
En el caso de la PCR-RFLP, la posibilidad de aplicar la técnica a muestras clínicas fue un factor decisivo para nuestros propósitos. Desde que Cupolillo y colaboradores, en 1995, reportaron el uso del gen que codifica para los espaciadores internos de la transcripción del ADNr como blanco de sus estudios de tipificación (16), varios genes han sido evaluados mediante la PCR-RFLP para discriminar entre las especies de Leishmania de la región neotropical (1).
Entre ellos, el gen que codifica para la Cpb nos pareció adecuado por cuanto la PCR-RFLP posibilita identificar L. braziliensis, la especie mayormente implicada en la forma mucocutánea de la enfermedad y considerada la más grave entre las presentaciones cutáneas, de una manera rápida e inconfundible, pues el patrón de bandas obtenido para esta especie es único.
En laboratorios de referencia como el nuestro, donde se pueden recibir casos importados de cualquier procedencia, fundamentalmente de un área geográfica determinada, y en otros países de Latinoamérica donde L. braziliensis es causa común de leishmaniasis, es muy conveniente contar con un método seguro para detectar e identificar las especies que pueden causar mayor perjuicio al hombre y, sin duda, L. braziliensis es la más importante en nuestra región.
Sin embargo, existen otras especies cuya significación clínica y epidemiológicica puede ser importante, por lo que en nuestro trabajo utilizamos ADN obtenido de especies nunca antes estudiadas en relación con el gen que codifica para la Cpb con estos cebadores, con la finalidad de explorar su posible utilidad en la tipificación.
Tal como lo reportaron García y colaboradores (1), la digestión con TaqI del producto amplificado para la Cpb, dió lugar a un patrón de bandas común para L. guyanenis, L. panamensis y L. peruviana, y un patrón único para L. braziliensis.
Previamente, se había cuestionado la diferenciación genética entre L. panamensis y L. guyanensis, ambas pertenecientes al subgénero Viannia. En un estudio realizado en Ecuador, en 1999, utilizando RAPD (17), no se encontraron fragmentos de amplificación específicos para una u otra especie, ni se agruparon en conglomerados diferentes, lo que estuvo apoyado también por resultados obtenidos mediante el análisis de isoenzimas.
En relación con el resto de las especies analizadas, todo parece indicar que, tal como antes se reportó para L. lainsoni y L. amazonensis, existe un impor-tante polimorfismo en la zona correspondiente al sitio de unión con los cebadores, que impidió amplificar el gen deseado, por lo que se deben analizar nuevas variantes en futuros trabajos para identificar estas especies.
En cuanto a la utilidad de la PCR-RFLP empleando muestras clínicas, en este caso, biopsias, se pudo comprobar que este método puede emplearse directamente en ADN obtenido del tejido de la lesión, lo que posibilita no sólo el diagnóstico rápido, sino que permite la identificación de la especie presente en la lesión, al menos en aquellas especies que amplificaron con los cebadores empleados. En todos los casos, el examen parasitológico mediante examen directo con tinción por Giemsa había sido positivo previamente y, a excepción de una sola muestra cuyo cultivo se contaminó, en el resto se aislaron los parásitos en medio de cultivo NNN.
En todo el ADN aislado de las biopsias se comprobó la infección por Leishmania spp. al obtenerse un producto de amplificación de 1.170 pb, el cual, luego de la digestion con TaqI, generó un patrón de restricción compatible con infecciones por L. panamensis, L. guyanensis o L. peruviana. Con base en este patrón de restricción y teniendo en cuenta los criterios epidemiológicos de donde provenían las muestras, es posible incriminar a L. panamensis como la especie responsable de estas infecciones, puesto que L. peruviana no está reportada en la zona de procedencia de los pacientes estudiados, y L. guyanensis en Colombia se presenta en focos muy localizados del territorio y ninguna de las muestras provenían de estos focos. La identificación posterior de especies empleando anticuerpos monoclonales y PCR en tiempo real, confirmó que se trataba de infecciones causadas por L. panamensis (no se mustran los datos), aunque la utilización de una segunda PCR-RFLP empleando el gen HSP70 pudiera resultar útil para discriminar entre estas especies (18).
Bhattacharyya et al. (19) y Tibayrenc et al. (20) aplicaron por primera vez la técnica de RAPD para identificar o detectar variantes genéticas en este parásito digénico, y encontraron un grado conside-rable de polimorfismo entre cepas del Viejo Mundo, que han sido las más estudiadas por este método. Ambos recomiendan su utilidad para reconstruir filogenias, identificar subespecies y linajes.
Más tarde, diferentes grupos han demostrado el polimorfismo genético entre parásitos del Viejo Mundo (19,21-23) y especies neotropicales (24,25) siendo la principal característica la posibilidad de obtener patrones claros para distinguir cada especie estudiada.
Como ya se señaló, obtuvimos patrones reproducibles de amplificación con el empleo de los oligonucleótidos OPA 3, 4 y 8, y se analizó por primera vez el polimorfismo de L. garnhami, junto al resto de las otras especies del Nuevo Mundo estudiadas. De igual forma, quedó demostrado el gran polimorfismo existente entre los subgéneros Viannia y Leishmania, mediante una clara diferenciación de las especies pertenecientes a cada uno de ellos.
El RAPD es un método molecular relativamente simple para identificar Leishmania, requiere de poca cantidad de ADN, permite distinguir directamente una especie de otra, y los resultados, al igual que ocurre en la PCR-RFLP, se basan en la observación directa de las bandas en un gel de agarosa.
Pero la arbitrariedad y el tamaño de los cebadores limita su especificidad y restringe su uso a parásitos aislados, lo que ha dificultado su aplicabilidad a muestras clínicas. Sin embargo, la posibilidad de obtener ADN de promastigotes en etapas tempranas de aislamiento in vitro, sin necesidad de un mantenimiento de larga duración (19), subsana este inconveniente, por lo que no se descarta la posibilidad de utilizar la técnica en determinadas situaciones.
Otro inconveniente que se ha señalado al RAPD es su alta sensibilidad a las variaciones experimentales, debido al pequeño tamaño de los cebadores u oligos empleados en la técnica, así como la baja temperatura de hibridación, podría resultar en una falta de reproducibilidad (26). Sin embargo, en nuestro trabajo logramos patrones reproducibles con los OPA mencionados, al repetir los experimentos en tres ocasiones diferentes, sin variaciones en los patrones de bandas obtenidos.
Aunque se ha revelado la inconsistencia del RAPD para la tipificación genética dentro del mismo zimodema, (26) sugerimos la posibilidad de emplear esta metodología en la tipificación de subgénero y, de manera cautelosa, en la diferenciación interespecífica, siempre teniendo en cuenta que nuestros resultados están basados en el estudio de una cepa por cada especie estudiada, y las desventajas previamente citadas.
A pesar de que consideramos necesaria la exploración de otros genes cuya conservación y polimorfismo (16,26) garanticen la tipificación de un mayor número de representantes de ambos subgéneros de este complejo parásito, la PCR-RFLP/CPb resulta de gran ayuda en el reconocimiento de especies vitales en nuestra área geográfica y de aplicación directa a muestras clínicas, mientras el RAPD con los cebadores OPA citados, pudiera constituir una alternativa en casos de parásitos aislados primariamente.
El uso combinado de ambos procedimientos sería, entonces, una herramienta útil en la necesaria identificación y tipificación de especies de Leishmania en nuestra región neotropical.
Conflictos de interés
No existen conflictos de intereses entre los autores del presente trabajo.
Financiación
Este trabajo ha sido financiado por el Programa DGOS, de la Cooperación Belga con Cuba, mediante la colaboración entre el Instituto Pedro Kourí y el Instituto de Medicina Tropical de Amberes, Bélgica.
Correspondencia:
Ana Margarita Montalvo, Departamento de Parasitología, Instituto Pedro Kourí, autopista Novia del Mediodía Km 6 y ½ La Lisa, apartado postal 601, Ciudad de La Habana, Cuba. Teléfono: (537) 202 0650; fax: (537) 204 6051 amontalvo@ipk.sld.cu, mmontal@infomed.sld.cuReferencias
1. García AL, Kindt A, Quispe-Tintaya KW, Bermúdez H, Llanos A, Arévalo J, et al. American tegumentary leishmaniasis: antigen gene polymorphism, taxonomy and clinical pleomorphism. Inf Genet Evol. 2005;5:109-16. [ Links ]
2. World Health Organization. Tropical disease research. Progress 1975-94, highlights 1993-94. Twelfth Programme Report of the UNDP/World Bank/ WHO Special Programme for Research and Training in Tropical Disease (TDR). Geneva: WHO; 1995. p.135-46. [ Links ]
3. Rotureau B, Ravel C, Couppie P, Pratlong F, Nacher M, Dedet JP, et al. Use of PCR-restriction fragment length polymorphism analysis to identify the main New World Leishmania species and analyze their taxonomic properties and polymorphism by application of the assay to clinical samples. J Clin Microbiol. 2006;44:459-67. [ Links ]
4. Victoir K, De Doncker S, Cabrera L, Álvarez E, Arévalo J, Llanos-Cuentas A, et al. Direct identification of Leishmania species in biopsies from patients with American tegumentary leishmaniasis. Trans R Soc Trop Med Hyg. 2003;97:80-7. [ Links ]
5. Serin MS, Daglioglu K, Bagirova M, Allahverdiyev A, Uzun S, Vural Z, et al. Rapid diagnosis and genotyping of Leishmania isolates from cutaneous and visceral leishmaniasis by microcapillary cultivation and polymerase chain reaction-restriction fragment length polymorphism of miniexon region. Diagn Microbiol Infect Dis. 2005; 53:209-14. [ Links ]
6. Noyes HA, Belli AA, Maingon R. Appraisal of various random amplified polymorphic DNA polymerase Chain reaction primers for Leishmania identification. Am J Trop Med Hyg. 1996;55:98-105. [ Links ]
7. Sambrook J, Fritsch EF, Maniatis T. Molecular cloning. A laboratory manual. Second edition. Cold Spring Harbor: Cold Spring Harbor Laboratory; 1989. [ Links ]
8. Ordeñana-Pilotos R. Optimización de la técnica del ADN polimórfico amplificado al azar (RAPD), para la identificación de especies de Leishmania (tesis). Ciudad de La Habana: Instituto Pedro Kourí; 2005. [ Links ]
9. Sneath PH, Sokal RR. Numerical taxonomy. San Francisco: Freeman WH & Co.; 1973. [ Links ]
10. Pavlícèk A, Hrdá S, Flegr J. Free tree-freeware program for construction of phylogenetic trees on the basis of distance data and for bootstrap/jackknife analysis of the threes robustness. Application in the RAPD analysis of genus Frenkelina. Folia Biol (Praha). 1999;45:97-9. [ Links ]
11. Reithinger R, Dujardin JC. Molecular diagnosis of leishmaniasis: Current status and future applications. J Clin Microbiol. 2007;45:21-5. [ Links ]
12. Croft SL, Yardley V, Kendrick H. Drug sensitivity of Leishmania species: some unresolved problems. Trans R Soc Trop Med Hyg. 2002;96(Suppl.1):127-9. [ Links ]
13. Berman JD. Human leishmaniasis: clinical, diagnostic, and chemotherapeutic developments in the last 10 years. Clin Infect Dis. 1997;24:684-703. [ Links ]
14. Singh S, Sivakumar R. Recent advances in the diagnosis of leishmaniasis. J Postgrad Med. 2003; 49:55-60. [ Links ]
15. Tavares CA, Fernandes AP, Melo MN. Molecular diagnosis of leishmaniasis. Expert Rev Mol Diagn. 2003;3:657-67. [ Links ]
16. Cupolillo E, Grimaldi G Jr, Momen H, Beverly SM. Intergenic region typing (IRT):a rapid molecular approach to the characterization and evolution of Leishmania. Mol Biochem Parasitol. 1995;73:145-55. [ Links ]
17. Bañuls AL, Jonquieres R, Guerrini F, LePont F, Barrera C, Espinel I, et al. Genetic analysis of Leishmania parasites in Ecuador: are Leishmania (Viannia) panamensis and Leishmania (V) guyanensis distinct taxa? Am J Trop Med Hyg. 1999;61:828-45. [ Links ]
18. Montalvo AM, Fraga J, Romero JA, Monzote L, Montano I, Dujardin JC. PCR-RFLP/HsP70 para identificar y tipificar Leishmania de la región neotropical. Rev Cub Med Trop. 2006;58(3). [Fecha de consulta: 24 de octubre de 2008]. Disponible en: http://scielo.sld.cu/scielo.php?script=sci_arttext&pid=S0375-07602006000300009&lng=es&nrm=iso. [ Links ]
19. Bhattacharyya R, Singh R, Hazra TK, Majumder HK. Application of polymerase chain reaction with specific and arbitrary primers to identification and differentiation of Leishmania parasites. FEMS Microbiol Lett. 1993;114:99-104. [ Links ]
20. Tibayrenc M, Neubauer K, Barnabe C, Guerrini F, Skarechy D, Ayala FJ. Genetic characterization of six parasitic protozoa: a parity between random-primer DNA typing and multilocus enzyme electrophoresis. Proc Natl Acad Sci USA. 1993;90:1335-9. [ Links ]
21. Diakou A, Dovas CI. Optimization of random amplified polymorphic DNA producing amplicons of 8500 bp and revealing intraspecies polymorphism in Leishmania infantum isolates. Anal Biochem. 2001;288:195-200. [ Links ]
22. Hanafi R, Barhoumi M, Ali SB, Guizani I. Molecular analyses of Old World Leishmania RAPD markers and development of a PCR assay selective for parasites of the L. donovani species complex. Exp Parasitol. 2001;98:90-9. [ Links ]
23. Zemanova E, Jirku M, Mauruicio IL, Miles MA, Lukes J. Genetic polymorphism within the Leishmania donovani complex: correlation with geographic origin. Am J Trop Med Hyg. 2004;70:613-7. [ Links ]
24. Martínez E, Alonso V, Quispe A, Thomas MC, Alonso R, Pinero JE, et al. RAPD method useful for distinguishing Leishmania species: design of specific primers for L. braziliensis. Parasitology. 2003;127:513-7. [ Links ]
25- Rodríguez-Bonfante C, Bonfante-Garrido R, Grimaldi G Jr, Momen H, Cupolillo E. Genotypically distinct Leishmania colombiensis isolates from Venezuela cause both cutaneous and visceral Leishmaniasis in humans. Infect Genet Evol. 2003;3:119-24. [ Links ]
26- García L, Kindt A, Bermúdez H, Llanos-Cuentas A, De Doncker S, Arévalo J, et al. Culture- independent species typing of neotropical Leishmania for clinical validation of a PCR-based assay targeting heat shock protein 70 genes. J Clin Microbiol. 2004;42:2294-7. [ Links ]














