Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.30 no.3 Bogotá July/Sept. 2010
ARTÍCULO ORIGINAL
1 Facultad de Ciencias de la Salud, Universidad Tecnológica de Pereira, Pereira, Colombia
2 Laboratorio de Genética Médica, Universidad Tecnológica de Pereira-ESE Salud Pereira, Pereira, Colombia
3 Clínica de Anticoagulación, Hospital Universitario La Samaritana, Bogotá D.C., Colombia
4 Grupo de Investigación en Farmacoepidemiología y Farmacovigilancia, Audifarma S.A., Pereira, Colombia
Institución donde se llevó a cabo el trabajo: Facultad de Ciencias de la Salud, Universidad Tecnológica de Pereira
Recibido: 09/11/09; aceptado:12/05/10
Introducción. La warfarina es un anticoagulante de difícil manejo por su estrecho margen terapéutico y los numerosos factores que influyen en la respuesta.
Objetivo. Determinar la contribución de variables demográficas, clínicas y genéticas sobre las dosis de mantenimiento de warfarina en pacientes colombianos.
Materiales y métodos. Se incluyeron 145 adultos de ambos sexos, en anticoagulación estable con International Normalized Ratio (INR) entre 2 y 3, al menos, durante dos meses, sin cambio en las dosis de warfarina ni en el preparado comercial. Previa firma del consentimiento informado de cada voluntario, se registró su edad, sexo, peso, talla, tabaquismo, enfermedades concomitantes, medicación simultánea, INR, dosis de warfarina, su indicación y nombre comercial.
Cada paciente se tipificó para los genes CYP2C9, VKORC1, CYP4F2 y PROC, y en 59 de ellos, se cuantificaron las concentraciones séricas de warfarina. La genotipificación y la cuantificación sanguínea se hicieron mediante minisecuenciación y HPLC, respectivamente.
Resultados. Los factores de edad, medicación simultánea con inhibidores (amiodarona, sertralina, fluoxetina) o inductores enzimáticos (fenitoína, carbamacepina), los alelos rs1799853 (*2) y rs1057910 (*3) del gen CYP2C9, así como rs9923231 del gen VKORC1, se asociaron con las dosis de warfarina requeridas para conseguir anticoagulación con INR de 2 a 3.
Dichas variables se incluyeron en un modelo de regresión lineal múltiple que permitiera predecir la dosis semanal de warfarina, y se obtuvo un algoritmo que explica el 47,4% de la variabilidad en las dosis.
Conclusión. La consideración de las variables clínicas y las farmacogenéticas puede mejorar la relación entre seguridad y eficacia de la warfarina, aunque la adopción de un algoritmo de dosificación farmacogenético requiere información obtenida con ensayos clínicos.
Palabras clave: warfarina/farmacología, farmacogenética, anticoagulantes, coagulación sanguínea, polimorfismo genético, tiempo de protrombina, vitamina K.
Genetic and bioenvironmental factors associated with warfarin response in Colombian patients
Introduction.Warfarin is an anticoagulant that is difficult to administer because of its narrow therapeutic margin and the numerous factors that influence patient response.
Objective. Demographic, clinical and genetic variables were characterized to establish the appropriate maintenance dosages of warfarin.
Materials and methods. The Colombian patients consisted of 145 adults of both sexes. They were in stable anticoagulation status with international normalized ratio between 2 and 3 for at least two months, and without changes in the warfarin commercial preparation or in the dosage. After signing the informed consent, the following data was recorded for each volunteer: age, gender, weight, height, smoker status, co-morbidity, co-medication, International Normalized Ratio (INR), warfarin dose, and commercial brand. Each patient was typed for genes CYP2C9, VKORC1, CYP4F2 and PROC; for 59 patients, the serum levels of warfarin were quantified. The genotyping and the blood quantification were performed by mini-sequencing and HPLC methods, respectively.
Results. Age, co-medication with enzymatic inhibitors (amiodarone, sertraline, fluoxetine) or inducers (phenytoin, carbamazepine), and the alleles rs1799853 (*2) and rs1057910 (*3) of the CYP2C9 gene, as well as rs9923231 of the VKORC1 gene were associated with warfarin dose required to achieve anticoagulation with INR of 2-3. These variables were included in a multiple linear regression model for predicting the optimum dose/week of warfarin. This resulted in an algorithm that explained 47.4% of the variability in the dose responses.
Conclusion: Clinical and pharmacogenetic variables provided a basis for improving the safety and effective dosage of warfarin; however, the use of a pharmacogenetic algorithm will require patient data obtained during clinical trials.
Key words: warfarin/pharmacology, pharmacogenetics, anticoagulants, blood coagulation; polymorphism, genetic; prothrombin time, vitamin K.
La warfarina (4-hidroxicoumadina) es el anti-coagulante oral más prescrito para la prevención y el tratamiento de alteraciones tromboembólicas arteriales y venosas, pese a estar entre los medicamentos asociados con alta incidencia de efectos indeseables; su reacción adversa más común y peligrosa es el sangrado, que puede llegar a ser fatal (1).
Al estrecho margen terapéutico del fármaco se agregan la variabilidad entre individuos en la respuesta y sus numerosas interacciones farmacológicas, factores que se conjugan para hacer de la warfarina un medicamento difícil de manejar, sobre todo en ancianos en quienes su empleo está en expansión. Los reportes coinciden en que un importante número de pacientes usuarios de warfarina oscila entre el riesgo de sangrado y la insuficiente anticoagulación (2,3).
La warfarina inhibe la enzima epóxido reductasa de la vitamina K (VKORC1), impidiendo la activación de los factores de la coagulación II, VII, IX y X, que dependen de la vitamina K reducida; su efecto anticoagulante tarda 4 a 5 días en instalarse, mientras se agotan dichos factores (3).
Las personas tratadas con este agente se deben controlar con el tiempo de protrombina, expresado como el International Normalized Ratio (INR), el cual mide la suma de la actividad de los mencionados factores de coagulación dependientes de la vitamina K. Para la mayoría de las indicaciones clínicas (fibrilación auricular, accidente cerebro-vascular recurrente, trombosis venosa profunda, embolismo pulmonar, cardiomiopatía, enfermedad coronaria), un INR entre 2 y 3 brinda la mejor relación entre riesgo y beneficio (3).
Las dosis requeridas para alcanzar un adecuado estado de anticoagulación con warfarina dependen de variables como edad, sexo, índice de masa corporal (IMC), contenido alimentario de vitamina K, enfermedades concomitantes y medicación simultánea con fármacos que inducen o inhiben la ruta metabólica del medicamento o que alteran la coagulación por otros mecanismos (4,5).
Con respecto a la eliminación del fármaco, administrado como mezcla racémica de R-warfarina y S-warfarina, la isoenzima CYP2C9 es responsable del metabolismo del enantiómero S- (principal forma activa), mientras el isómero R-warfarina, menos activo, es degradado principalmente por vía de la CYP3A4 (3). Por lo tanto, los factores que modulan la expresión y la actividad de estas enzimas influyen en la depuración hepática y en las concentraciones séricas del fármaco.
Por consiguiente, los factores genéticos relacionados con polimorfismos de los genes que tienen que ver con la acción de la warfarina o con su biotransformación, también inciden en sus efectos farmacológicos; en efecto, algunas variantes alélicas de los genes VKORC1 y CYP2C9 se han asociado claramente con las dosis anticoagulantes de warfarina, aunque el papel de otros genes (PROC, CYP4F2, GGCX, FVII) también parece importante en determinados grupos étnicos (6-12).
En un esfuerzo por mejorar la seguridad y la eficacia de la warfarina, se han buscado los factores asociados con la variabilidad en las dosis entre individuos y entre grupos raciales, y se han diseñado algoritmos de dosificación que incluyen variables demográficas, clínicas y genéticas de diversas poblaciones, incluido el mestizo colombiano (13).
Dichos protocolos tienen aceptable poder pronóstico en los grupos étnicos donde son comunes los alelos incluidos en el algoritmo y, por lo tanto, deben ser ajustados para otras poblaciones, en el sentido de incorporar en el modelo los alelos relevantes en cada grupo étnico (14). A modo de ejemplo, entre euroamericanos ciertos polimorfismos CYP2C9 y VKORC1 explican hasta 30% de la variabilidad en las dosis de warfarina, mientras en afroamericanos esos mismos polimorfismos sólo explican 8% de la variabilidad (15).
Otra limitación de la mayoría de los algoritmos de dosificación propuestos, consiste en que incluyen únicamente variantes alélicas relacionadas con la sensibilidad a la warfarina y dejan por fuera del modelo otras asociadas con la resistencia al fármaco (14,16). Debido a la falta de potencia de los estudios individuales para hacer estimativos confiables de dosis en diferentes poblaciones, varios autores han expresado dudas con respecto al beneficio clínico y la costo-efectividad de tales algoritmos farmacogenéticos de la dosificación de la warfarina (3,17,18).
En este estudio nos propusimos determinar la contribución de algunas variables demográficas (edad, sexo, IMC), clínicas (enfermedades concomitantes, medicación simultánea, dosis e indicación de warfarina) y de polimorfismos de los genes VKORC1 (rs9923231, rs2359612 y 5417G>T), CYP2C9 (rs1799853 y rs1057910), CYP4F2 (rs2108622) y PROC (rs2069919), sobre las dosis de warfarina requeridas para mantener una anticoagulación terapéutica y derivar un algoritmo de dosificación del fármaco en los pacientes colombianos.
Materiales y métodos
Pacientes
Este estudio se condujo con pacientes colombianos de rasgos mestizos, no emparentados, que recibían warfarina como terapia de mantenimiento a largo plazo en cuatro centros de atención ambulatoria situados en las ciudades de Bogotá (Hospital La Samaritana e IPS Virrey Solís) y Pereira (EPS Salud Total y Comfamiliar).
Se incluyeron 145 personas de ambos sexos, con edades entre 20 y 92 años, en quienes el estado de anticoagulación se había estabilizado con INR entre 2 y 3, por lo menos durante dos meses, sin cambio en las dosis del medicamento ni en el preparado comercial utilizado (warfarina de marca o genérica). Mediante entrevista personal se obtuvo información con respecto a edad, sexo, peso, talla, tabaquismo actual, indicación para warfarina, su marca comercial, dosis en mg por semana y valor del último INR, enfermedades concomitantes y medicación simultánea.
Luego se procedió a la toma de la muestra de sangre o de mucosa bucal para la genotipificación. Además, por limitaciones de orden presupuestal, en forma aleatoria se les tomó muestra de sangre sólo a 59 voluntarios, para la cuantificación de las concentraciones séricas de enantiómeros de warfarina, a fin de comparar la actividad de la enzima CYP2C9.
Se registró toda la medicación simultánea que el paciente recibía en forma crónica y, posteriormente, se analizó el potencial de interacción con el INR de cada uno de los fármacos prescritos concomitantemente con mayor frecuencia (acetaminofén, ácido acetilsalicílico, amiodarona, amlodipino, fluoxetina, gemfibrozil, glibenclamida, digital, losartán, lovastatina, metformin, metoprolol, omeprazol, sertralina y verapamilo).
En las enfermedades concomitantes se incluyó la cardiovascular (insuficiencia cardíaca, hipertensión arterial, cardiopatía isquémica, arritmia) y la metabólica (dislipidemia, diabetes mellitus). Se excluyeron pacientes con enfermedad hepática, renal o tiroidea no compensada, síndrome de mala absorción, alcoholismo crónico y cáncer.
El protocolo fue aprobado en la categoría de investigación con riesgo mínimo por el Comité de Bioética de la Universidad Tecnológica de Pereira, y por los comités técnico-científicos de cada una de las instituciones de salud participantes.
Genotipificación
Se aplicó la técnica de minisecuenciación o ddNTP primer extension. Previa extracción del ADN genómico de las células obtenidas de sangre o mucosa bucal, se procedió a la amplificación de dos fragmentos del gen CYP2C9, tres del gen VKORC1,uno del gen CYP4F2 y uno del gen PROC, en los cuales quedaron incluidos los SNP (single-nucleotide polymorphism) que se iban a estudiar. La reacción en cadena de la polimerasa (PCR) se llevó a cabo en un volumen total de 23 µl, los cuales contienen 1-10 ng de ADN genómico, 1X PCR solución tampón, 180 µM de cada dNTP, 0,8 mM de MgCl2, 2 U de Taq ADN polimerasa (Invitrogen®) y 0,5 a 9 µM de cada uno de los iniciadores (cuadro 1).
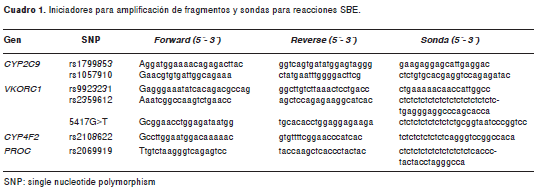
La amplificación de los fragmentos se llevó a cabo en un termociclador automático (DNA Thermal Cycler, MJ Research) con los siguientes parámetros: un paso inicial de desnaturalización por 2 minutos a 96oC, seguido por 30 ciclos de amplificación, desnaturalización a 94oC por 1 minuto, annealing a 55oC por 1 minuto y extensión a 72oC por 1 minuto, con una extensión final a 60 oC por 1 hora. El exceso de iniciadores y de nucleótidos fue removido por adición de 1 U de ExoSAP-IT (Amersham Pharmacia) e incubación a 37oC durante 80 minutos, seguido por 15 minutos a 85oC.
Usando este producto de preamplificación como plantilla, se efectuaron las reacciones múltiple para la detección de los SNP con el método de minisecuenciación (SBE, single base extension).
Las reacciones de SBE se realizaron en un volumen total de 10 ul:1 ul de agua desionizada, 2 ul del producto de preamplificación, 5 ul de SNaPshot Reaction Mix (Applied Biosystems) y 2 ul de la mezcla de sondas SBE con concentración 0,2 uM (cuadro 1). Los tubos se llevaron a termociclador para su amplificación en 35 ciclos con las siguientes condiciones: desnaturalización a 96oC por 10 s, annealing a 50oC por 5 s y extensión a 60oC por 30 s.
El exceso de nucleótidos fue removido por adición de 1 U de SAP (Shrim Alkaline Phosphatase, Amersham Pharmacia) e incubación a 37°C durante 2 horas, seguido por 15 minutos a 85°C. Se mezcló 1 ul del producto de la reacción SBE con 9 ul de Hi-Di formamida y 0,5 ul de GeneScan-120 Liz internal size Standard (Applied Biosystems).
La detección se llevó a cabo en el secuenciador ABI Prism 3100-Avant Genetic Analyzers (Applied Biosystems) por electroforesis con capilar de 36 cm de longitud y polímero POP-4, en el módulo de corrida SNP36-POP4. Los datos se analizaron de acuerdo con el color de los picos y el tamaño de los fragmentos, mediante el software Genemapper, versión 3.2 (PE Applied Biosystems). La presencia de los polimorfismos se confirmó por secuenciación directa de muestras seleccionadas.
Niveles séricos de warfarina
Mínimo 6 horas después de haber consumido la warfarina (con el fármaco en fase de eliminación), a 59 voluntarios se les tomaron muestras de sangre (10 ml) en tubos al vacío con EDTA y se centrifugaron a 2.500g por 15 minutos para obtener el plasma, el cual se conservó a -20°C hasta el momento del análisis.
Las concentraciones séricas de S- y R-warfarina se cuantificaron por HPLC mediante un método previamente descrito (19), con algunas modificaciones. Se utilizaron un HPLC System 2000 plus, marca JASCO, equipado con bomba de gradiente cuaternario, automuestreador, horno para columnas y detector de arreglo de diodos MD 2015 controlado por EZChrom Elite, versión 3.16. Se utilizó una columna quiral analítica tipo Pirkle (R,R) Whelk-O1 [250 mm de largo x 4,6 mm de diámetro interno, 5 µm tamaño de partícula (Regis Technologies, USA)]. El sistema de elusión fue isocrático con una mezcla de metanol y agua en proporción 70:30, más 0,1% de ácido acético glacial a 1 ml/minuto. El detector se programó en el rango de 240-400 nm y la cuantificación se realizó a 305 nm.
Los estándares se prepararon adicionando a plasma humano libre del fármaco una solución madre de 1 mg/ml de cada enantiómero, para obtener 2 ml de estándar en el rango de concentración esperada (0,1 a 1,1 µg/ml) en los pacientes a quienes se les administraba warfarina. Los estándares y las muestras se extrajeron en fase sólida en cartuchos RP-18, 500 mg (Merck), previamente activados con metanol (3 ml x 3) seguido de ácido acético al 5% (3 ml x 3).
Los estándares o las muestras (2 ml) se acidificaron con 1 ml de ácido acético al 5% y se aplicaron al cartucho, el cual se secó por succión. El lavado se realizó con tres porciones de 0,5 ml de ácido acético al 5%, secando entre lavados. Se hizo un lavado final con 0,5 ml de ácido acético 5%-metanol (80:20), secando por succión. La elusión se realizó con dos porciones de 0,25 ml de isopropanol, de la cual se inyectaron 50 ul para la detección con las condiciones mencionadas anteriormente.
Con el fin de establecer los tiempos de elusión de cada uno de los enantiómeros, éstos fueron corridos inicialmente por separado. Para la cuantificación se generaron curvas lineales de calibración en el rango de 0,2 a 1,5 ug/ml; cada uno de los estándares fue preparado e inyectado en el HPLC por triplicado, lo que permitió obtener el porcentaje de desviación estándar relativa, el límite de detección y el límite de cuantificación, para lo cual se utilizó el software EZChrom Elite v3.16.
El límite de detección se definió como tres veces la señal de ruido y, el límite de cuantificación, como 3,3 veces el límite de detección.
Con la finalidad de determinar el porcentaje de recuperación de warfarina durante el proceso de extracción en fase sólida, se prepararon tres estándares por triplicado en plasma libre de warfarina en concentraciones de 0,25 ug/ml, 0,5 ug/ml y 1 ug/ml.
Análisis estadístico
Las variables continuas se compararon con ANOVA o t de Student. La prueba de ji al cuadrado se empleó para comparar variables categóricas y para verificar el equilibrio de Hardy-Weinberg. Se utilizaron los análisis de regresión simple y múltiple para estimar la influencia de las variables estudiadas en las dosis de mantenimiento de warfarina y construir el algoritmo de dosificación propuesto. Se utilizaron IC95% y los valores de p<0,05 se consideraron significativos en test de dos colas. Los análisis estadísticos se hicieron con los software SPSS 15.0 para Windows y Stata 10.1.
Resultados
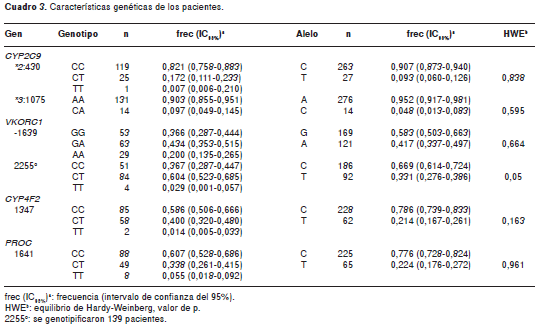
Las características demográficas y clínicas de los 145 pacientes del estudio se resumen en el cuadro 2. En el cuadro 3 se presentan las frecuencias alélicas y genotípicas halladas. La mutación 5417G>T del gen VKORC1, asociada con resistencia a warfarina y de alta prevalencia en etíopes, no se encontró en nuestra cohorte y tampoco se hallaron individuos homocigotos mutados *3 ni con la doble mutación *2/*3 del gen CYP2C9. Todos los genotipos estudiados estuvieron en equilibrio de Hardy-Weinberg (cuadro 3).

En los análisis univariados los factores no genéticos que se correlacionaron con las dosis de warfarina requeridas para conseguir anticoagulación tera-péutica estable con INR entre 2 y 3, fueron: edad (Pearson=-0,22, p=0,007), medicación simultánea con amiodarona (sí: 21,3±8 mg/semana Vs. no: 33,4±11 mg/semana, p<0,0001), con antidepresivos (uso de sertralina o fluoxetina: 20±5,1 mg/semana Vs. no uso: 33,4±11 mg/semana, p<0,0001) o con inductores enzimáticos (uso de fenitoína o carbamacepina: 56,1±30 mg/semana Vs. no uso: 33,4±11 mg/semana, p<0,0001).
Por el contrario, no tienen efectos significativos en las dosis terapéuticas de warfarina, las variables sexo (p=0,695), peso (p=0,938), talla (p=0,611), IMC (p=0,574), indicación de la warfarina (p=0,092), enfermedades concomitantes (p=0,459) y si el medicamento es de marca o genérico (p=0,416). Se encontró tabaquismo actual solamente en tres pacientes; por lo tanto, esta variable fue excluida de los análisis.
Con respecto a las variables genéticas potencial-mente influyentes en la respuesta a la warfarina, los SNP rs1799853 (*2) y rs1057910 (*3) del gen CYP2C9, y rs9923231 y rs2359616 del gen VKORC1,tienen efecto significativo sobre la dosis de mantenimiento de warfarina: CYP2C9*1/*1=35,7± 14,4 mg/semana Vs.*1/*2=26,5±10,3 mg/semana Vs. *1/*3=22,8±9 mg/semana, p=0,001; VKORC1-1639GG=40,5±15,2 mg/semana Vs. GA=30,4±10,3 mg/semana Vs. AA=24,5±13 mg/semana, p<0,0001; VKORC1 2255CC=40,7±15,5 mg/semana Vs. CT=29,2±11,7 mg/semana Vs. TT=25,1±5 mg/semana, p<0,0001. Sólo encontramos un paciente homocigoto *2/*2, con 25 mg/semana de warfarina; los SNP rs9923231 y rs2359616 del gen VKORC1 están en fuerte desequilibrio de ligamiento (D´=0,93), en tanto que los alelos rs2108622 y rs2069919 de los genes CYP4F2 y PROC, respectivamente, no mostraron asociación con las dosis de warfarina.
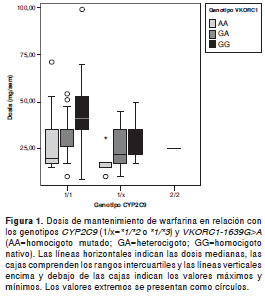
Para el análisis de las influencias combinadas de los genes influyentes en las dosis de warfarina, por un lado, agrupamos los pacientes en homocigotos nativos (*1/*1), heterocigotos (*1/*2 o *1/*3) y homocigotos mutados (*2/*2) con respecto al gen CYP2C9 y, por otro lado, consideramos el alelo -1639G>A del gen VKORC1. La figura 1 muestra cómo las mutaciones de ambos genes suman sus efectos individuales para aumentar la sensibilidad de los pacientes a la warfarina.
Las variables edad, medicación simultánea con inhibidores (amiodarona, sertralina, fluoxetina) o con inductores enzimáticos (fenitoína, carbamacepina) y los genotipos CYP2C9*2, CYP2C9*3, VKORC1 -1639G>A y VKORC1 2255C>T, cuyas influencias resultaron significativas en los análisis univariados, se incluyeron en un modelo de regresión lineal múltiple por pasos que permitiera predecir la dosis diaria de mantenimiento de warfarina.
Puesto que los SNP rs9923231 y rs2359616 del gen VKORC1 se encuentran en fuerte desequilibrio de ligamiento y al reemplazar el genotipo VKORC1 -1639G>A por el diplotipo conformado por VKORC1 -1639G>A y VKORC1 2255C>T no se producen cambios en el valor predictivo del modelo, asumimos que el SNP rs2359616 no es factor predictor independiente de las dosis de warfarina (cuadro 4).
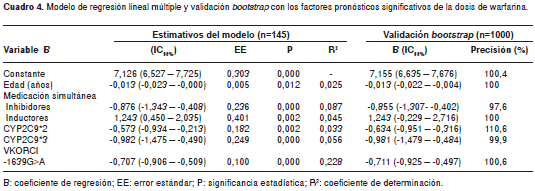
Además, en la construcción del modelo encontramos que la transformación de la variable dependiente en la raíz cuadrada de la dosis era la que mejor se ajustaba a la distribución normal; por lo tanto, la ecuación de regresión propuesta para la predicción de las dosis de mantenimiento de warfarina es la siguiente: raíz cuadrada de la dosis de warfarina (mg/semana)=7,126 - 0,013 x edad (en años) - 0,876 x inhibidor enzimático (sí=1, no=0) + 1,243 x inductor enzimático (sí=1, no=0) - 0,573 x CYP2C9 430C>T (0=homocigoto nativo, 1=heterocigoto, 2=homocigoto mutado) - 0,982 x CYP2C9 1075A>C (0=homocigoto nativo, 1=heterocigoto) - 0,707 x VKORC1 -1639G>A (0=homocigoto nativo, 1=heterocigoto, 2=homocigoto mutado).
Este algoritmo explica el 47,4% de la variabilidad en las dosis diarias de mantenimiento de warfarina en los pacientes del estudio. La estabilidad del modelo fue confirmada con 1.000 replicaciones de los datos originales mediante el método bootstrap sampling, el cual explica un porcentaje de variación de 48,5%, lo que indica que nuestro modelo es confiable (cuadro 4).
La figura 2 muestra que existe significativa correlación entre la dosis de mantenimiento observada y la estimada por el algoritmo obtenido a partir de la regresión múltiple (R2=0,46, p<0,0001).
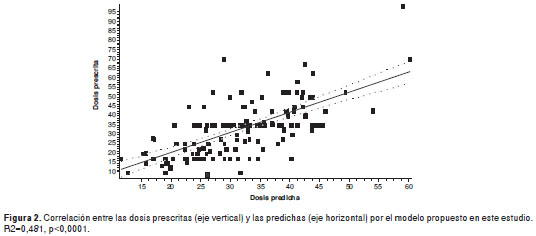
A fin de evaluar el desempeño del algoritmo en diferentes rangos de dosis, lo aplicamos al subgrupo de pacientes que requieren dosis bajas (&≤21 mg/semana) o altas (≥49 mg/semana) de warfarina para conseguir el INR entre 2 y 3. En este subgrupo (n=57) el poder predictivo del modelo ascendió a 62,7%.
Por último, de acuerdo con el hecho de que la relación S/R de enantiómeros de warfarina en plasma varía con el genotipo CYP2C9 y sirve para evaluar la actividad de la enzima que metaboliza la S-warfarina, encontramos entre los pacientes con metabolismo "normal" (EM: CYP2C9*1/*1) una relación S/R de 0,295±0,08, mientras los pacientes con metabolismo "lento" (PM: *2 o *3) exhiben relaciones S/R significativamente superiores (*1/*2=0,460±0,25, p=0,001; *1/*3=0,470±0,20, p=0,001).
Discusión
En este estudio se investigó la influencia de variables demográficas, clínicas y genéticas en la respuesta a la warfarina de 145 pacientes mestizos en terapia de anticoagulación estable, con INR entre 2 y 3. Encontramos que la edad es un factor que contribuye a la variabilidad en la respuesta al medicamento, lo cual coincide con la mayoría de las investigaciones encaminadas a identificar los factores que inciden en la respuesta a la warfarina (12-14,20-24); esta correlación inversa se explica porque los fármacos que deben ser biotransformados en el hígado se depuran con mayor dificultad a medida que avanza la edad, debido a la declinación de la actividad enzimática (24).
Por otro lado, se sabe que el tabaquismo también es uno de los factores ambientales que contribuye a la variabilidad entre individuos en la respuesta a los medicamentos, al parecer por inducción de enzimas del metabolismo de fármacos (25); sin embargo, puesto que el número de pacientes fumadores de nuestro estudio fue muy bajo (n=3) y dos de ellos recibían además agentes inductores enzimáticos, esta variable no se incluyó en los análisis.
Las interacciones de la warfarina con inhibidores o inductores de enzimas del citocromo P-450, están suficientemente estudiadas. La farmacocinética de la amiodarona es compleja, porque este agente interactúa con numerosos medicamentos de uso común, ya que es un potente inhibidor de varias enzimas del citocromo P-450, en particular de la CYP3A4 y la CYP2C9, justamente las isoenzimas encargadas del metabolismo de los dos isómeros de la warfarina (3,26). Creemos que ésta es la explicación de que los pacientes de nuestro estudio tratados concomitantemente con amiodarona (n=13) recibieran dosis de warfarina 36% menores que los individuos sin medicación simultánea (n=93). La sertralina y la fluoxetina también actúan como inhibidores de enzimas CYP, potenciando la acción de la warfarina (12,26).
De otra parte, la fenitoína y la carbamacepina son dos neurofármacos reconocidos inductores de varias enzimas del metabolismo de los xenobióticos, entre las cuales se encuentran las isoenzimas CYP3A4 y CYP2C9, razón por la cual pueden incrementar la depuración hepática de una gran cantidad de medicamentos, incluyendo la warfarina (3,26), lo que explica por qué las dosis de warfarina necesarias para alcanzar INR en el rango de 2 a 3 para quienes se prescriben simultáneamente con fenitoína o carbamacepina (n=5) son superiores en 65% a las de los pacientes sin medicación simultánea.
Resulta interesante que el paciente con la dosis más alta de warfarina (95 mg/semana) es portador de alelos nativos para todos los genes estudiados, pero, además de carbamacepina consumía azatio-prina, otro fármaco inductor de resistencia a la warfarina, por un mecanismo aún no definido y raramente reportado (12,27).
Existen diferencias entre las estatinas con respecto a su capacidad de interaccionar con los anticoagulantes coumarínicos, siendo fluvastatina y simvastatina las más implicadas en ocasionar desajustes del INR y riesgo de sangrado por warfarina (3,28,29). Los pacientes de nuestro estudio tratados de manera simultánea con lovastatina (n=34), utilizan dosis de warfarina similares a las de los individuos sin medicación simultánea (32±12,5 mg/semana Vs. 33,4+11 mg/semana, respectivamente; p=0,44), lo cual podría deberse a las bajas dosis de lovastatina utilizadas entre nosotros (20 mg al día) y a un menor impacto de este agente sobre el INR, de acuerdo con la clasificación hecha por Holbrook AM et al. (26) quienes consideran el potencial de interacción warfarina-lovastatina sólo como "posible".
Otro caso de resistencia a la warfarina merece comentario. Corresponde a una mujer de 26 años con trastorno trombofílico hereditario, medicada con 70 mg semanales de warfarina más danazol, este último reportado como agente androgénico que potencia los efectos de la warfarina e incrementa el riesgo de sangrado (30); además, esta paciente es homocigota mutada para el alelo -1639G>A, lo que la haría aún más sensible a la warfarina. A este resultado contrario a lo que podría esperarse de la interacción warfarina-danazol y del genotipo VKORC1 -1639AA, no le encontramos explicación, pero podríamos especular que la resistencia al anticoagulante en este caso es causada por su trastorno trombofílico de base, el cual aumenta el riesgo de recurrencia de eventos tromboembólicos (31).
Pese a que factores como sexo, talla, peso, enfermedades concomitantes e indicación de la warfarina, se han considerado tradicionalmente como influyentes en las dosis necesarias para lograr anticoagulación terapéutica, en concordancia con nuestros hallazgos ya ha sido reportada en diferentes grupos étnicos la falta de asociación de las dosis de warfarina con las variables sexo (13,20,21), IMC (12,13), indicación para el uso de warfarina (12,23) y enfermedades concomitantes (12,20-23).
Tampoco encontramos diferencia entre la warfarina de marca y las genéricas con respecto a las dosis requeridas para alcanzar un INR en el rango terapéutico de 2 a 3 [Coumadin® (n=16): 31,3±11 mg/semana Vs. genéricas (n=129): 33,4±14 mg/semana, p=0,57]. Aunque la bioequivalencia entre la warfarina de marca y las genéricas se ha ratificado mediante ensayos clínicos y metanálisis (32), queremos destacar este hallazgo dadas las grandes diferencias de precios entre el producto de marca y los genéricos, asunto crítico en países en desarrollo con recursos limitados.
Las frecuencias alélicas y genotípicas halladas se asemejan a las reportadas en grupos étnicos con características de mestizaje similares a las nuestras (20,33). De los tres polimorfismos estudiados del gen VKORC1, uno de ellos (5417G>T), asociado con resistencia a la warfarina (34,35), no se halló en nuestra muestra y no ha sido reportado entre mestizos, lo que respalda la recomendación de aplicar modelos que se ajusten al perfil genético relevante en cada grupo étnico. El fuerte desequilibrio de ligamiento hallado entre los alelos rs9923231 y rs2359616 del gen VKORC1 (D´=0,93) y el hecho de que este último no sea un predictor independiente de respuesta a la warfarina, coincide con lo reportado por otros autores (12,14,21).
Diferentes algoritmos propuestos para estimar las dosis de warfarina que tienen en cuenta variables demográficas, clínicas y genéticas, tienen valores predictivos entre 31% y 60% de la variabilidad de las dosis (13,21,22). El modelo propuesto en este estudio para nuestro propio grupo étnico cae en este rango (47,4%) y, además, confirma que los más importantes factores de predicción son los polimorfismos -1639G>A del gen VKORC1 y el CYP2C9*3. Las asociaciones entre genes y dosis, según las cuales la dosis de warfarina disminuye a medida que aparecen los alelos mutados de los genes CYP2C9 (*2 y *3) y VKORC1 (-1639A) (figura 1), hacen un aporte mayor a la variabilidad entre individuos en las dosis efectivas de warfarina que el que hacen otras variables demográficas y clínicas, y está de acuerdo con numerosos reportes en diversos grupos étnicos (12,20-23,36). En efecto, en nuestro modelo las variables genéticas contribuyen con 31,7%, en tanto que la edad y la medicación simultánea aportan 15,7% a la variabilidad en las dosis (cuadro 4).
En forma similar a como se demostró en el estudio del Warfarin Pharmacogenetics Consortium (14), nuestro algoritmo farmacogenético se desempeña mejor entre quienes requieren dosis bajas (≤21 mg/semana) o altas (≥49 mg/semana), al elevar el R2 de 47,4% a 62,7% en el subgrupo de pacientes con dosis altas o bajas, que representan 39,3% de la cohorte completa.
Aunque algunos autores señalan que el uso de test farmacogenéticos para la iniciación del tratamiento con warfarina no muestra mejores resultados en seguridad y eficacia y, por lo tanto, no apoyan el uso de rutina de estas pruebas en la iniciación del tratamiento (3,37,38), otros estudios prospectivos sí demuestran que estos algoritmos contribuyen a mejorar la seguridad y la eficacia de la warfarina (6-8,12,14,39).
Dado el estrecho margen terapéutico del fármaco y el alto riesgo de sangrado como principal reacción adversa, la adopción de un algoritmo de dosificación que contribuya a reducir el riesgo de hemorragia y a predecir interacciones potencialmente peligrosas aportaría una medida preventiva a los programas de farmacovigilancia. Sin embargo, la incorporación de marcadores farmacogenéticos en la dosificación de warfarina debe estar justificada en información obtenida mediante ensayos clínicos controlados y de asignación aleatoria (18) y en estudios de costo-efectividad (40).
Otros alelos con influencia significativa en la respuesta a la warfarina no fueron considerados por nosotros; tal podría ser el caso de algunos polimorfismos de los genes CYP2C9 (*5, *6, *11) (20,41,42) y FVII (11), cuyas asociaciones con la sensibilidad a la warfarina fueron recientemente demostradas.
Tampoco se consideraron en esta investigación variables potencialmente influyentes, como la ingestión de vitamina K y de productos naturales, aunque prácticamente todos los pacientes habían recibido educación sobre una dieta estandarizada de vitamina K.
Al dejar por fuera de este estudio los pacientes cuyos INR no habían podido estabilizarse en el rango terapéutico de 2 a 3, es evidente que se introduce un sesgo en la búsqueda de variables determinantes de las dosis de warfarina.
El tamaño de la muestra es probablemente la limitación más importante de esta investigación, ya que variables, sin duda, influyentes en las dosis de warfarina, como el tabaquismo y el genotipo CYP4F2, no alcanzaron a resultar significativas por el escaso número de pacientes fumadores o con mutaciones; por ejemplo, tal como se ha reportado en otros estudios (43), nuestros pacientes con el genotipo CYP4F2 TT usaban 1,37 mg más de warfarina diaria que los pacientes nativos con los dos alelos CC, pero el número de homocigotos mutados encontrados (n=2) no permitió alcanzar significancia estadística.
Agradecimientos
A los pacientes y a las siguientes instituciones de salud que nos colaboraron: Hospital Universitario La Samaritana de Bogotá, IPS Virrey Solís de Bogotá, EPS Salud Total de Pereira, Comfamiliar de Pereira y Empresa Social del Estado Salud Pereira.
Conflicto de intereses
Álvaro Vallejos y Jorge Machado tienen relación laboral con Audifarma, S.A. Los demás autores consideran y declaran que no existe conflicto de intereses y que no hubo injerencia externa alguna en la marcha del estudio o en la preparación del manuscrito.
Financiación
Esta investigación fue financiada por Colciencias (proyecto No. 1110-408-20392), la Universidad Tecnológica de Pereira y Audifarma, S.A.
Correspondencia: Carlos A. Isaza, Facultad de Ciencias de la Salud, Universidad Tecnológica de Pereira, La Julita, Pereira, Colombia. Telefax: (57) 313 7125 caisaza@utp.edu.co
Referencias
1. Gurwitz JH, Field TS, Radford MJ, Harrold LR, Becker R, Reed G, et al. The safety of warfarin therapy in the nursing home setting. Am J Med. 2007;120:539-44. [ Links ]
2. Sconce EA, Kamali F. Appraisal of current vitamin K dosing algorithms for the reversal of over-anticoagulation with warfarin: the need for a more tailored dosing regimen. Eur J Haematol. 2006;77:457-62. [ Links ]
3. Ansell J, Hirsh J, Hylek E, Jacobson A, Crowther M, Palareti G. Pharmacology and management of the vitamin K antagonists: American College of Chest Physicians Evidence-Based Clinical Practice Guidelines (8th edition). Chest. 2008;133:160S-98S. [ Links ]
4. Greenblatt DJ, von Moltke LL. Interaction of warfarin with drugs, natural substances, and foods. J Clin Pharmacol. 2005;45:127-32. [ Links ]
5. Rettie AE, Jones JP. Clinical and toxicological relevance of CYP2C9: drug-drug interactions and pharmacogenetics. Annu Rev Pharmacol Toxicol. 2005;45:477-94. [ Links ]
6. Sconce EA, Khan TI, Wynne HA, Avery P, Monkhouse L, King BP, et al. The impact of CYP2C9 and VKORC1 genetic polymorphism and patient characteristics upon warfarin dose requirements: proposal for a new dosing regimen. Blood. 2005;106:2329-33. [ Links ]
7. Wadelius M, Chen LY, Eriksson N, Bumpstead S, Ghori J, Wadelius C, et al. Association of warfarin dose with genes involved in its action and metabolism. Hum Genet. 2007;121:23-34. [ Links ]
8. Lindh JD, Holm L, Andersson ML, Rane A. Influence of CYP2C9 genotype on warfarin dose requirements-a systematic review and meta-analysis. Eur J Clin Pharmacol. 2008;65:365-75. [ Links ]
9. Takeuchi F, McGinnis R, Bourgeois S, Barnes C, Eriksson N, Soranzo N, et al. A genome-wide association study confirms VKORC1, CYP2C9, and CYP4F2 as principal genetic determinants of warfarin dose. PLoS Genet. 2009;5:e1000433. [ Links ]
10. Kimura R, Miyashita K, Kokubo Y, Akaiwa Y, Otsubo R, Nagatsuka K, et al. Genotypes of vitamin K epoxide reductase, gamma-glutamyl carboxylase, and cytochrome P450 2C9 as determinants of daily warfarin dose in Japanese patients. Thromb Res. 2007;120:181-6. [ Links ]
11. Fuchshuber-Moraes M, Perini JA, Rosskopf D, Suarez-Kurtz G. Exploring warfarin pharmacogenomics with the extreme-discordant-phenotype methodology: impact of FVII polymorphisms on stable anticoagulation with warfarin. Eur J Clin Pharmacol 2009;65:789-93. [ Links ]
12. Wadelius M, Chen LY, Lindh JD, Eriksson N, Ghori MJ, Bumpstead S, et al. The largest prospective warfarin-treated cohort supports genetic forecasting. Blood. 2009;113:784-92. [ Links ]
13. Palacio L, Falla D, Tobón I, Mejía F, Lewis J, Martínez A, et al. Pharmacogenetic impact of VKORC1 and CYP2C9 allelic variants on warfarin dose requirements in a Hispanic population isolate. Clin Appl Thromb Hemost. 2009;16:83-90. [ Links ]
14. International Warfarin Pharmacogenetics Consortium, Klein TE, Altman RB, Eriksson N, Gage BF, Kimmel SE, et al. Estimation of the warfarin dose with clinical and pharmacogenetic data. N Engl J Med. 2009;360:753-64. [ Links ]
15. Limdi NA, Beasley TM, Crowley MR, Goldstein JA, Rieder MJ, Flockhart DA, et al. VKORC1 polymorphisms, haplotypes and haplotype groups on warfarin dose among African-Americans and European-Americans. Pharmacogenomics. 2008;9:1445-58. [ Links ]
16. Loebstein R, Dvoskin I, Halkin H, Vecsler M, Lubetsky A, Rechavi G, et al. A coding VKORC1 Asp36Tyr polymorphism predisposes to warfarin resistance. Blood. 2007;109:2477-80. [ Links ]
17. Garcia DA, Hylek E. Warfarin pharmacogenetics. N Engl J Med. 2009;360:2474. [ Links ]
18. Gulseth MP, Grice GR, Dager WE. Pharmacogenomics of warfarin: uncovering a piece of the warfarin mystery. Am J Health Syst Pharm. 2009;66:123-33. [ Links ]
19. Osman A, Arbring K, Lindahl TL. A new high-performance liquid chromatographic method for determination of warfarin enantiomers. J Chromatogr B Analyt Technol Biomed Life Sci. 2005;826:75-80. [ Links ]
20. Perini JA, Struchiner CJ, Silva-Assunção E, Santana IS, Rangel F, Ojopi EB, et al. Pharmacogenetics of warfarin: development of a dosing algorithm for brazilian patients. Clin Pharmacol Ther. 2008;84:722-8. [ Links ]
21. Gage BF, Eby C, Johnson JA, Deych E, Rieder MJ, Ridker PM, et al. Use of pharmacogenetic and clinical factors to predict the therapeutic dose of warfarin. Clin Pharmacol Ther. 2008;84:326-31. [ Links ]
22. Ohno M, Yamamoto A, Ono A, Miura G, Funamoto M, Takemoto Y, et al. Influence of clinical and genetic factors on warfarin dose requirements among Japanese patients. Eur J Clin Pharmacol. 2009;65:1097-103. [ Links ]
23. Sandanaraj E, Lal S, Cheung YB, Xiang X, Kong MC, Lee LH, et al. VKORC1 diplotype-derived dosing model to explain variability in warfarin dose requirements in Asian patients. Drug Metab Pharmacokinet. 2009;24:365-75. [ Links ]
24. Majerus PW, Tollefsen DM. Blood coagulation and anticoagulant, thrombolytic, and antiplatelet drugs. In: Brunton LL, Lazo JS, Parker KL, editors. The pharmacological basis of therapeutics. 11 ed. Mexico D.F.: Mc Graw-Hill; 2006. p. 1475-80. [ Links ]
25. Schein JR. Cigarette smoking and clinically significant drug interactions. Ann Pharmacother. 1995;29:1139-48. [ Links ]
26. Holbrook AM, Pereira JA, Labiris R, McDonald H, Douketis JD, Crowther M, et al. Systematic overview of warfarin and its drug and food interactions. Arch Intern Med. 2005;165:1095-106. [ Links ]
27. Vázquez SR, Rondina MT, Pendleton RC. Azathioprine-induced warfarin resistance. Ann Pharmacother. 2008; 42:1118-23. [ Links ]
28. Sconce EA, Khan TI, Daly AK, Wynne HA, Kamali F. The impact of simvastatin on warfarin disposition and dose requirements. J Thromb Haemost. 2006;4:1422-4. [ Links ]
29. Einarson TR, Metge CJ, Iskedjian M, Mukherjee J. An examination of the effect of cytochrome P450 drug interactions of hydroxymethylglutaryl-coenzyme A reductase inhibitors on health care utilization: a Canadian population-based study. Clin Ther. 2002;24:2126-36. [ Links ]
30. Meeks ML, Mahaffey KW, Katz MD. Danazol increases the anticoagulant effect of warfarin. Ann Pharmacother. 1992;26:641-2. [ Links ]
31. Santamaría MG, Agnelli G, Taliani MR, Prandoni P, Moia M, Bazzan M, et al. Thrombophilic abnormalities and recurrence of venous thromboembolism in patients treated with standardized anticoagulant treatment. Thromb Res. 2005;116:301-6. [ Links ]
32. Kesselheim AS, Misono AS, Lee JL, Stedman MR, Brookhart MA, Choudhry NK, et al. Clinical equivalence of generic and brand-name drugs used in cardiovascular disease: a systematic review and meta-analysis. JAMA. 2008;300:2514-26. [ Links ]
33. Llerena A, Dorado P, O´Kirwan F, Jepson R, Licinio J, Wong ML. Lower frequency of CYP2C9*2 in Mexican-Americans compared to Spaniards. Pharmacogenomics J. 2004; 4: 403-6. [ Links ]
34. Aklillu E, Leong C, Loebstein R, Halkin H, Gak E. VKORC1 Asp36Tyr warfarin resistance marker is common in Ethiopian individuals. Blood. 2008;111:3903-4. [ Links ]
35. Sinxadi P, Blockman M. Warfarin resistance. Pharmacogenomics J Cardiovasc J Afr. 2008; 19: 215-7. [ Links ]
36. Kim MJ, Huang SM, Meyer UA, Rahman A, Lesko LJ. A regulatory science perspective on warfarin therapy: a pharmacogenetic opportunity. J Clin Pharmacol. 2009;49:138-46. [ Links ]
37. Hynicka LM, Cahoon WD Jr, Bukaveckas BL. Genetic testing for warfarin therapy initiation. Ann Pharmacother. 2008;42:1298-303. [ Links ]
38. Kangelaris KN, Bent S, Nussbaum RL, Garcia DA, Tice JA. Genetic testing before anticoagulation? A systematic review of pharmacogenetic dosing of warfarin. J Gen Intern Med. 2009;24:656-64. [ Links ]
39. Huang SW, Chen HS, Wang XQ, Huang L, Xu DL, Hu XJ, et al. Validation of VKORC1 and CYP2C9 genotypes on interindividual warfarin maintenance dose: a prospective study in Chinese patients. Pharmacogenet Genomics. 2009;19:226-34. [ Links ]
40. You JH, Tsui KK, Wong RS, Cheng G. Potential clinical and economic outcomes of CYP2C9 and VKORC1 genotype-guided dosing in patients starting warfarin therapy. Clin Pharmacol Ther. 2009:86:540-7. [ Links ]
41. Scott SA, Jaremko M, Lubitz SA, Kornreich R, Halperin JL, Desnick RJ. CYP2C9*8 is prevalent among African-Americans: implications for pharmacogenetic dosing. Pharmacogenomics. 2009;10:1243-55. [ Links ]
42. Limdi NA, Wiener H, Goldstein JA, Acton RT, Beasley TM. Influence of CYP2C9 and VKORC1 on warfarin response during initiation of therapy. Blood Cells Mol Dis. 2009;43:119-28. [ Links ]
43. Caldwell MD, Awad T, Johnson JA, Gage BF, Falkowski M, Gardina P, et al. CYP4F2 genetic variant alters required warfarin dose. Blood. 2008;111:4106-12. [ Links ]














