Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.31 no.4 Bogotá Oct./Dec. 2011
ARTÍCULO ORIGINAL
Expresión diferencial entre estadios de Trypanosoma cruzi I en el aislamiento de un paciente con cardiomiopatía chagásica crónica de zona endémica de Santander, Colombia
1Grupo de Inmunología y Epidemiología Molecular, GIEM, Facultad de Salud, Universidad Industrial deSantander, Bucaramanga, Colombia
2Grupo de Bioquímica y Microbiología, Escuela de Química, Universidad Industrial de Santander, Bucaramanga,Colombia
Recibido: 06/12/10; aceptado:07/07/11
Introducción. Trypanosoma cruzi es el agente causal de la enfermedad de Chagas. Durante la infección en los huéspedes mamíferos, se observan dos formas del parásito: tripomastigotes y amastigotes. En el curso de la diferenciación del parásito cada estadio expresa un patrón de proteínas específicas de fase, las cuales son responsables de sus características morfológicas, bioquímicas y biológicas, que podrían estar determinando un papel importante en la capacidad infecciosa, virulencia y supervivencia del parásito.
Objetivo. Analizar la expresión diferencial entre los estadios tripomastigote y amastigote de un aislamiento de T. cruzi I, utilizando la electroforesis en dos dimensiones y la identificación de las proteínas diferencialmente expresadas mediante espectrometría de masas.
Materiales y métodos. Se utilizó un clon del aislamiento MHOM/07/338 de T. cruzi I y, mediante electroforesis en dos dimensiones, se compararon los perfiles proteicos de los estadios tripomastigote y amastigote del parásito. Las imágenes se analizaron con el software PDQuest y las proteínas diferencialmente expresadas se identificaron por MALDI TOF o LC MS/MS.
Resultados. Los geles bidimensionales mostraron un promedio de 325 manchas proteicas en cada estadio. En los análisis comparativos se detectaron 21 manchas "sobre expresadas" en el estadiotripomastigote y 30, en el estadio amastigote. Se seleccionaron 16 proteínas para identificación por espectrometría de masas y se clasificaron en diferentes categorías funcionales.
Conclusiones. Las proteínas exclusivas de T. cruzi relacionadas, principalmente, con metabolismo glucolítico y ensamble del citoesqueleto, fueron las que presentaron una mayor expresión diferencial entre los estadios tripomastigote y amastigote del parásito. Estas proteínas podrían ser utilizadas para el diseño de fármacos.
Palabras clave: Trypanosoma cruzi, enfermedad de Chagas, proteómica, cardiomiopatía chagásica,Colombia.
Differential protein expression in developmental stages of Trypanosoma cruzi I isolated from a patient with chronic chagasic cardiomyopathy
Introduction. Trypanosoma cruzi is the causative agent of Chagas disease. During infection inmammalian hosts, two main forms of the parasite are observed: trypomastigotes and amastigotes.
During differentiation, each stage of the parasite expresses a pattern of proteins specific to each phase-proteins which are responsible for the cell’s morphological, biochemical and biological properties.
These properties ultimately govern the infectivity, virulence and survival of the parasite.
Objective. A differential expression analysis was conducted to compare trypomastigote and amastigote stages of T. cruzi I isolate, and to identify proteins differentially expressed by means of mass spectrometry.
Materials and methods. A T. cruzi clone of the strain MHOM/07/338 was used to analyze the differential expression between trypomastigote stages of a T. cruzi isolate, using two-dimensional electrophoresis and identification of diferentially expressed proteins by mass spectrometry. The protein profiles of the stages of the parasite were obtained by two-dimensional gel electrophoresis and visualized in gels dyed with Coomassie blue. The images were analyzed with PDQuest software and the differential expression of the proteins was identified by MALDI TOF or LC MS/MS.
Results. The two-dimensional gels revealed an average of 325 protein spots in each stage. The comparative analyses detected 21 spots that were over expressed in the trypomastigote stage and 30 in the amastigote stage. Sixteen of the over expressed proteins were selected for identification by mass spectrometry and classified in several functional categories. Mass spectrophotometry determined that the proteins were associated mainly with glucolytic metabolism and assembly of the cytoskeleton constituents.
Conclusions. The differential expression between trypomastigote and amastigote stages consisted of proteins specific to T. cruzi and are potential targets for the design of treatment drugs.
Key words: Trypanosoma cruzi, Chagas disease, proteomics, chagasic cardiomyopathy, Colombia.
Trypanosoma cruzi, el agente causal de la enfermedad de Chagas, es un parásito protozoario hemoflagelado. Esta enfermedad es un problema importante de salud pública en Centroamérica y Suramérica; actualmente se estima que entre 8 y 15 millones de personas están infectadas con este parásito y cerca de 28 millones se encuentran en riesgo de adquirir la infección (1). Hasta el momento, no existen vacunas disponibles y los fármacos en uso, nifurtimox y beznidazol, presentan poca eficacia para el tratamiento de la fase crónica de la enfermedad y graves efectos colaterales.
Trypanosoma cruzi cumple su ciclo de vida en la naturaleza entre dos huéspedes diferentes: uno invertebrado, representado por insectos reduvídeos de la subfamilia Triatominae, y uno vertebrado, representado por mamíferos como el hombre, animales domésticos y silvestres. Durante su ciclo,T. cruzi presenta cuatro estadios: epimastigotes, tripomastigotes sanguíneos, tripomastigotes metacíclicos y amastigotes.
Los epimastigotes se encuentran en el intestino medio del insecto y son las formas de replicación, las cuales se diferencian en tripomastigotes metacíclicos, las formas infecciosas. Cuando estas formas alcanzan un huésped vertebrado como el hombre, rápidamente invaden las células huésped y en el citoplasma se convierten en amastigotes, los cuales se multiplican hasta llenar casi completamente la célula; en este momento, los amastigotes se transforman en tripomastigotes muy móviles que rompen la célula y son liberados a la sangre, desde donde pueden invadir otras células (2).
Este ciclo de vida tan complejo exige, por parte del parásito, una rápida adaptación a los diferentes ambientes; por lo tanto, se espera que en el curso de la diferenciación del parásito cada estadio exprese un patrón de proteínas específicas de fase, responsables de sus características morfológicas, bioquímicas y biológicas, que pudieran jugar un papel importante en la capacidad infecciosa y la supervivencia dentro del ambiente hostil del huésped vertebrado.
En este sentido, la identificación de estas proteínas específicas permitiría un mejor entendimiento de los mecanismos involucrados en la interacción entre el huésped y el parásito que, entre otros aspectos, podría facilitar la identificación de potenciales blancos moleculares para el desarrollo de nuevos fármacos o el diseño de vacunas.
En el 2005, se publicó la secuencia total del genoma de T. cruzi (3). Sin embargo, por sí solo este paso fundamental que aportó nueva información del parásito, no es suficiente para conocer las proteínas involucradas en los mecanismos de interacción entre huésped y parásito. En los tripanosomátidos esto es particularmente cierto, dado que la regulación de la expresión génica se da posterior a la transcripción, por mecanismos de transplicing, control de la estabilidad o traducción de los ARNm específicos (4). Además, cabe señalar que las modificaciones posteriores a la traducción juegan un papel importante en la modulación de la función de las proteínas en estos parásitos.
Teniendo en cuenta lo anterior, la cuantificación del ARNm no siempre refleja el nivel de proteína correspondiente y ello hace que un análisis proteómico sea la herramienta ideal para evaluar el nivel de expresión de proteínas. En el caso de T.cruzi I, no existen reportes de análisis de expresión génica en aislamientos de humanos; en este trabajo se analizó la expresión diferencial entre los estadios tripomastigote y amastigote del grupo T.cruzi I, comparando los perfiles proteicos obtenidos por electroforesis en dos dimensiones y posterior identificación de las proteínas por espectrometría de masas.
Materiales y métodos
Parásitos
En este trabajo se utilizó un aislamiento de T. cruzi denominado MHOM/CO/0005/338, perteneciente al grupo I, obtenido de un paciente con enfermedad crónica de Chagas proveniente de la zona endémica del departamento de Santander, Colombia. Se hizo la clonación del aislamiento por el método de plaqueo sobre medio sólido, para asegurar una población homogénea (5).
Obtención de los estadios
Para obtener los estadios de tripomastigote y amastigote, se utilizó la línea celular Vero ATCC149. Estas células fueron infectadas con tripomastigotes resuspendidos en medio DMEM (Sigma, St. Louis, USA), con suplemento de 25 mM HEPES, a pH 7,4, 25 mM Na2HCO3, 100 UI/ml penicilina, 100 µg/ml estreptomicina y 7,5 % suero bovino fetal, a 37°C en una atmósfera con 5 % de CO2. Transcurridos cinco días, se obtuvieron los tripomastigotes en el sobrenadante, los cuales fueron purificados (6) y usados para infectar una nueva monocapa de células Vero, o contados y recogidos en un pellet. Los amastigotes se obtuvieron por incubación de los tripomastigotes (5 x 106 células por ml) en medio LIT a pH 7,0 por 24 horas, en las mismas condiciones.
Extracción de proteínas
Los parásitos de cada estadio se recolectaron por centrifugación y los precipitados se lavaron tres veces con PBS a pH 7,4 y 0,9 % de NaCl a 4 oC. Las proteínas se extrajeron por incubación de los parásitos en solución tampón de lisis que contenían 40 mM de Tris base, 7 M de urea, 2 M de tiourea, 4 % de CHAPS and 1 mM de PMSF. Después de 30 minutos de incubación a temperatura ambiente con agitación constante, los parásitos lisados se centrifugaron por 30 minutos a 13.000 rpm, para remover todo el material en partículas y el sobrenadante resultante se utilizó para el isoelectroenfoque. Las proteínas se cuantificaron siguiendo el método de Bradford (7).
Electroforesis en dos dimensiones
Los extractos solubles de proteínas (180 µg) se aplicaron a tiras con gradiente de pH inmovilizado de 7 cm, con un rango de pH de 5 a 8. Las tiras se rehidrataron durante 16 horas a temperatura ambiente. Después de la rehidratación, se llevó a cabo el isoelectro enfoque sobre un sistema IEF (BioRad) a 22°C, usando el siguiente protocolo: 20 minutos a 250 V y 2 horas a 4.000 V, hasta alcanzar 80.000 V/hora. Antes de la electroforesis en poliacrilamida, las tiras IPG se sometieron a reducción y alquilación, incubándolas durante 15 minutos en una solución con 6 M de urea, 30 % de glicerol, 2 % de SDS y 125 mM de DTT, y 15 minutos en la misma solución tampón en la que se reemplazó el DTT por 125 mM de iodoacetamida.
Posteriormente, las proteínas se separaron en función de su masa molecular (segunda dimensión), colocando las tiras equilibradas sobre geles de poliacrilamida al 12 % en presencia de SDS. La electroforesis se llevó a cabo usando el sistema Miniprotean® (BioRad, Hércules, CA, USA), a un voltaje de 150 V a 20°C. Los geles se tiñeron con azul de Coomassie coloidal R-250 y se documentaron usando un densitómetro (BioRad).
Visualización de las proteínas y análisis de imagen
Para el análisis comparativo de los geles teñidos, se digitalizaron en el escáner (UMAX, Power Look 2100XL®) a una resolución de 300 dpi (pixeles por pulgada). Posteriormente, se analizaron las imágenes usando el software PDQuest 2D®, versión 8.1 (BioRad). Se compararon las manchas proteicas de tres geles distintos por cada uno de los estadios analizados (tripomastigotes y amastigotes) y se creó un gel virtual usando las tres réplicas de cada muestra.
La intensidad de la señal se calculó teniendo en cuenta el área y la intensidad de cada mancha seguida por normalización, y se expresó como la intensidad total en pixeles de cada mancha, respecto a la intensidad total de todas las manchas presentes en el gel.
Ãnicamente las manchas presentes en el promedio de los geles de cada estadio que presentaban valores de desviaciones estándar menores de 20 %, se compararon con las manchas de los geles promedio del otro estadio. Se consideró la expresión diferencial cuando el radio de la intensidad de la mancha era mayor de 2 y se consideró significativa una p<0,05. De éstas, se seleccionaron 12 en tripomastigotes y 4 en amastigotes para la identificación por espectrometría de masas.
Digestión de proteínas
Después del secado, las manchas seleccionadas se rehidrataron y se digirieron in situ con tripsina como lo describe Shevchenko (8), con modificaciones menores. Las manchas proteicas coloreadas se incubaron en 50 mM NH4HCO3 con tripsina (5 ng/µl) por una hora en baño de hielo.
La solución tampón de digestión se eliminó, y los geles se cubrieron con 50 mM de NH4HCO3 y se incubaron a 37°C por 12 horas. Los sobrenadantes se secaron y almacenaron a 20°C hasta el análisis de espectrometría de masas.
Análisis de espectrometría de masas: MALDITOF (matrix-assisted laser desorption ionization/time-of-flight mass spectrometry) El análisis de huella peptídica se hizo como se ha descrito anteriormente (9), usando un espectrómetro de masas Autoflex⢠(Bruker Daltonics, Bremen, Germany), con el reflectrón en modo de iones positivos y empleando ácido 2,5-dihidroxibenzoico como matriz y un placa AnchorChip⢠(Bruker Daltonics). La identificación y asignación de los espectros obtenidos se hicieron automáticamente, usando el software Flexanalysisâ¢, version 2.2 (Bruker Daltonics).
La búsqueda en las bases de datos se realizó mediante MASCOT http://matrixscience.com (10), contra la base de datos del National Center forBiotechnology Information (NCBI). Los parámetros de búsqueda seleccionados, fueron: tolerancia de dos sitios de clivaje, carbamidometilación (Cys) y oxidación (Met) como modificaciones fijas y variables, respectivamente, y dando una tolerancia de error al péptido de 100 ppm después de la calibración externa cerrada. Un puntaje de probabilidad de MASCOT significativo (p<0,05) se consideró una condición para la identificación de la proteína.
Análisis de espectrometría de masas: RP-LC/ MS/MS (cromatografía líquida en fase inversaespectrometría de masas) Cuando la huella de los péptidos falló en la identificación de la mancha, la proteína digerida se secó, resupendió en 7 µl de de ácido fórmico al 0,1 % y analizó por RP-LC-MS/MS, en un espectrómetro de masas acoplado a trampa iónica modelo Deca XP™ (Thermo Fisher Scientific, Waltham, MA, USA). Los péptidos se separaron por cromatografía en fase inversa, usando una columna de 0,18 mm x 150 mm Bio-Basic C18 RP™ (Thermo Fisher Scientific), operando a 1,8 µl/minuto. Los péptidos fueron eluidos usando, durante 50 minutos, un gradiente de 5 a 40 % de solvente B (solvente A: ácido fórmico al 0,1 % en agua; solvente B: ácido fórmico al 0,1 %, acetonitrilo al 80 % en agua). La ionización ESI se hizo usando un micro-spray con una interfase Metal Needle-Kit™ (Thermo Fisher Scientific).
Los péptidos se identificaron utilizando el algoritmo SEQUEST™ (Bioworks Browser 3.2, Thermo Fisher Scientific). La búsqueda se hizo en las bases de datos de Apicomplexa, a-proteobacteria y metazoan, bajadas de Protein Knowledgebase (UniProtKB) http://www.uniprot.org.
Las siguientes restricciones se usaron para la búsqueda: clivaje tríptico después de Arg y Lys, más de dos sitios de clivaje y tolerancias de 2 Da para iones precursores y de 0,8 Da para iones de fragmentos MS/MS; las búsquedas se hicieron siguiendo la oxidación opcional Met y la carbamidometilación Cys fija. Si la búsqueda de SEQUEST no daba ningún resultado positivo, se seleccionaban los espectros de alta calidad no identificados y se hacía una interpretación manual.
Estos resultados se confirmaron con el software PEAKS Studio 5.2® (Bioinformatics Solutions Inc., Waterloo, ON, Canada).
Resultados
Los perfiles proteicos para cada uno de los estadios se muestran en las imágenes de los geles de dos dimensiones en la figura 1. Los perfiles proteicos obtenidos a partir de preparaciones de proteínas individuales, fueron fácilmente reproducibles en términos del número de manchas proteicas y sus posiciones e intensidades relativas. Cada gel de electroforesis en dos dimensiones mostró un promedio de 325 manchas para el aislamiento de tripomastigote y para el de amastigote (figuras 1A y B).
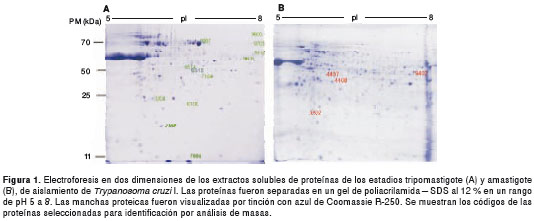
Los análisis comparativos de ambos estadios mostraron 33 manchas de péptidos detectadas específicamente en tripomastigotes y 28 presentes únicamente en el estadio amastigote. Además, otras 21 manchas se expresaron con mayor intensidad en tripomastigotes que en amastigotes, mientras 30 manchas lo hicieron en amastigotes (figura 2).
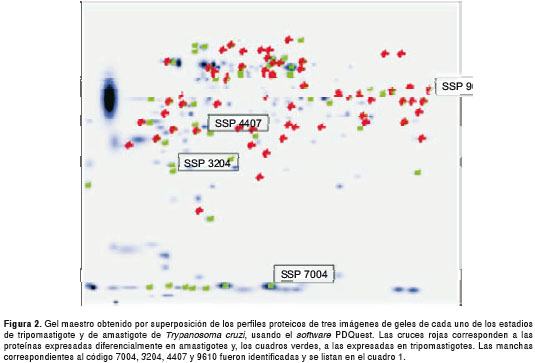
Para la identificación por espectrometría de masas MALDITOF o LC MS/MS, se seleccionaron 12 manchas de péptidos expresadas diferencialmente en el estadio de tripomastigote y 4 en el amastigote (figura 1), según los criterios previamente definidos.
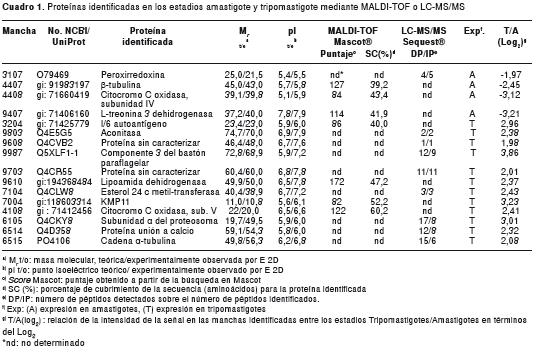
Mediante la búsqueda en las bases MASCOT y UNIPROT, se identificaron 16 proteínas (cuadro 1). Todas las identificaciones se confirmaron por la interpretación manual de los espectros de masas.
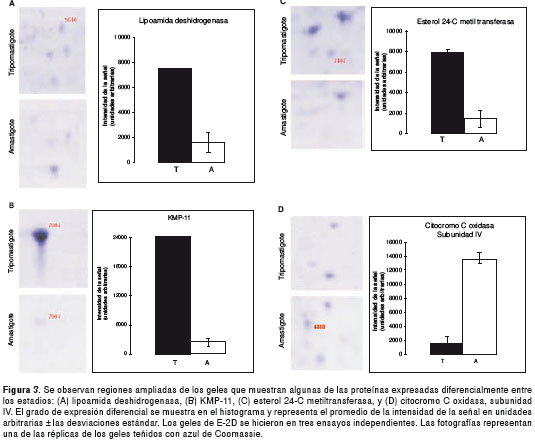
Las regiones ampliadas de los geles que mostraron algunas de las proteínas expresadas diferencialmente entre los estadios y su correspondiente análisis de intensidad relativa, se presentan en la figura 3.
Las proteínas identificadas por MALDITOF o RPLC MS/MS se agruparon en categorías funcionales de acuerdo con su función principal, anotada en Gene Ontology GO (cuadro 2).
Discusión
La expresión diferencial en los estadios de T. cruzi permite la identificación de proteínas relevantes en el proceso de adaptación a los diferentes ambientes a los que se ve sometido el parásito.
Su identificación es fundamental para entender los procesos de interacción entre huésped y parásito y, al mismo tiempo, determinar si podrían ser usados como potenciales blancos de fármacos o antígenos relevantes en el diseño de vacunas.
Cabe resaltar que en T. cruzi se han reportado algunos estudios sobre expresión diferencial entre estadios en aislamientos pertenecientes a T. cruzi II (11,12), grupo asociado principalmente con las diferentes formas clínicas de la enfermedad en países del Cono Sur de Suramérica. Sin embargo, aún no se cuenta con estudios proteómicos en aislamientos de humanos pertenecientes al grupo I, principal responsable de la cardiomiopatía chagásica en países del norte de Suramérica, como Colombia (13), y países de Centroamérica, que podrían presentar perfiles particulares relacionados con la presentación clínica de la enfermedad y su gravedad.
Los tripomastigotes presentan mecanismos metabólicos para contrarrestar los cambios abruptos en el ambiente (por ejemplo, nutrientes y temperatura) cuando alternan entre los dos tipos de huésped.
Estas formas están adaptadas para sobrevivir en ambientes con un nivel constante de glucosa; por lo tanto, los requisitos energéticos son proporcionados por un tipo de glucólisis aerobia en que la glucosa es convertida a piruvato (14).
Posteriormente, el piruvato experimenta un proceso de descarboxilación oxidativa, el cual involucra la deshidrogenación de la dihidrolipoamida mediante la enzima lipoamida deshidrogenasa (LipDH), componente esencial de los complejos mitocondriales piruvato deshidrogenasa y 2 oxoglutarato deshidrogenasa (15).
En este estudio, la LipDH se sobreexpresó en el estadio tripomastigote (figura 3A), igual a lo observado por Parodi, et al., al estudiar la cepa Dm28c (16); sin embargo, se ha reportado que esta enzima está presente en todas las fases del ciclo de vida de T. cruzi (17). Además, se ha demostrado que la LipDH cataliza la reducción de los electrones de los nitrofuranos, generando concomitantemente radicales superóxido y peróxido de hidrógeno (18); por ello, la LipDH se ha señalado como un blanco potencial para el desarrollo de nuevos fármacos.
En procariontes y eucariontes inferiores, las deshidro-genasas de isocitrato dependientes de NADP son el resultado de un proceso de adaptación ancestral que les permite a estos organismos crecer en ambientes muy oxidativos y pobres en nutrientes. En estas condiciones, no hay intermediarios derivados de la vía de las pentosas y, por lo tanto, se requieren fuentes alternativas de NADPH para los procesos de biosíntesis (19). Por ello, las enzimas málicas y las deshidrogenasas de isocitrato son las responsables de proveer el NADPH necesario, no sólo en la biosíntesis, sino también en la defensa contra el daño oxidativo.
En T. cruzi, no hay evidencia de un ciclo de Krebs clásico, como el de los eucariontes superiores, relacionado con la producción de NADPH dependiente de NAD. Los resultados recientes muestran que una aconitasa putativa le daría el sustrato a las deshidrogenasas de isocitrato para la producción de NADPH; así, el citrato producido en la mitocondria podría convertirse en isómero de isocitrato en el citosol y en la mitocondria por acción de esta enzima. En T. cruzi, la aconitasa putativa tiene gran similitud con la descrita para T. brucei y se ha encontrado que es más abundante en los estadios tripomastigote y amastigote que en el epimastigote (19). En este estudio se encontró una mayor expresión en el estadio tripomastigote en comparación con el amastigote, que no sólo estaría relacionada con la capacidad de crecer en medios pobres de nutrientes sino que sería capaz de sobrevivir en el ambiente oxidativo del macrófago.
Los macrófagos actúan en la primera línea de defensa frente al ingreso de patógenos intracelulares y son una de las células blanco de T. cruzi. Entre los principales mecanismos para la eliminación de estos parásitos, la célula cuenta con las especies reactivas derivadas del oxígeno y el nitrógeno; en este sentido, la efectividad del sistema de defensa antioxidante es crítico para la supervivencia del parásito dentro de la célula huésped y para que se establezca la infección.
Por ello, los tripanosomátidos cuentan con un sistema muy sofisticado de desintoxicación, en el cual participan numerosas enzimas entre las que se cuentan diferentes peroxidasas, que incluyen triparredoxinas, tripanotión sintetasas y tripanotión reductasas (20). Las peroxidasas son la segunda clase de enzimas desintoxicantes de hidroperóxido en tripanosomátidos; en T. cruzi se han identificado cinco peroxidasas diferentes con distinta localización subcelular y especificidad de sustrato (20).
Dada su relevancia en los mecanismos de evasión de la respuesta inmunitaria, estas enzimas se han considerado factores de virulencia importantes y están siendo objeto de estudio para utilizarlas en el diseño racional de fármacos (21). En un estudio de análisis de expresión diferencial entre parásitos obtenidos a partir de un caso agudo comparados con los de uno crónico, obtenidos en la zona endémica de Santander en Colombia, se encontró sobreexpresión de la peroxidasa triparredoxina, la cual podría estar asociada con el desarrollo de la forma aguda de la enfermedad (22).
Un rasgo característico de las formas de epimastigote y tripomastigote es la presencia del flagelo; en los tripanosomátidos está formado por el bastón paraflagelar y el axonema. El bastón paraflagelar es una estructura filamentosa parecida a una malla que corre paralela al axonema dentro de la membrana flagelar (23). A diferencia del axonema, el cual se conserva en los eucariontes, el bastón paraflagelar está restringido a los Kinetoplastidos. En el presente estudio se observó expresión diferencial de las proteínas de unión al calcio flagelar (PUFCa) y el componente 3 del bastón paraflagelar (BPF-3) en las formas de tripomastigote en relación con las de amastigote; se obtuvieron resultados similares en los niveles de expresión de BPF-3, utilizando reacción en cadena de la polimerasa (PCR) en tiempo real con la cepa Perú (T. cruzi I) (24). Por su localización en la membrana flagelar, estas proteínas son importantes en la movilidad y, además, cumplen funciones metabólicas, reguladoras, de señalamiento y adherencia (25-27). En el caso de la proteína BPF-3, se ha demostrado su propiedad inmunógena en T. cruzi (28,29).
Igualmente, en relación con el estadio de tripomastigote y la presencia de flagelo, se observó una mayor expresión de la proteína KMP11 en este estadio (figura 3B). Esta proteína de 11 kDa está muy conservada en Kinetoplastidos con características comunes, entre las que se destacan el peso molecular, el elevado número de copias, su localización en el flagelo, el bolsillo flagelar y la superficie de la membrana del parásito (30-32). Aunque se expresa en todos los estadios del ciclo de vida del parásito, lo hace con mayor abundancia en los estadios desarrollados en el insecto, como son tripomastigotes y epimastigotes, como se demostró mediante Western blot e inmunomicroscopía electrónica (33). Los resultados del presente estudio, mediante la electroforesis en dos dimensiones, confirman su sobreexpresión en la forma tripomastigota.
En el mismo estadio de tripomastigotes se observó expresión diferencial de proteínas asociadas al metabolismo de los lípidos, como la metiltransferasa de esterol 24-C (figura 3C). Esta proteína es una enzima que se encuentra en hongos, levaduras, plantas y protozoos, la cual cataliza un paso esencial en la biosíntesis de esteroles (34).
En los tripanosomátidos, los esteroles como el ergosterol, son componentes esenciales de las membranas biológicas y juegan un papel integral en la organización dinámica y función de la bicapa lipídica (35); algunos datos recientes muestran que esta proteína ha sido identificada en los reservosomas de los tripomastigotes (36).
En el presente estudio, esta proteína se expresó exclusivamente en el estadio de tripomastigote, lo cual se correlaciona con el hecho de que estas formas sintetizan cantidades mínimas pero esenciales de ergosterol a partir de los esteroles precursores derivados del huésped los cuales son metilados por esta enzima (37). Cabe señalar que esta proteína ha sido identificada como blanco en el diseño de inhibidores para el tratamiento de la enfermedad de Chagas (38).
Por otra parte, durante el proceso de infección, las formas de amastigote se localizan en el citoplasma de las células huésped, donde los niveles de glucosa libre son muy bajos; por lo tanto, estas formas recurren al catabolismo de los aminoácidos aromáticos como fuente de energía y carbón (39).
Una de las vías metabólicas que emplea T. cruzi es el clivaje de L-treonina para producir acetato y glicina mediante la acción de la deshidrogenasa de L-treonina. En el presente estudio se detectó que esta proteína se expresaba exclusivamente en el estadio de amastigote, lo que concuerda con lo hallado en T. cruzi II. Asimismo, en el modelo de T. brucei se ha demostrado la utilización y excreción de aminoácidos y otros metabolitos como fuente de energía (40).
Otro de los mecanismos utilizados por los tripanosomátidos digenéticos en su proceso adaptativo a huéspedes, vertebrados e invertebrados, es la inactivación reversible del ciclo de Krebs (41).
Dado que las subunidades de los complejos respiratorios mitocondriales están codificadas en el genoma nuclear y en el de la mitocondria, se espera que ambos tipos de genes experimenten una regulación específica de fase (42).
En este estudio, la subunidad IV de la enzima citocromooxidasa C presentó una mayor expresión en el estadio de amastigote (figura 3D), lo que está de acuerdo con este resultado; los datos preliminares indican que en T. brucei esta subunidad tiene poca expresión en los tripomastigotes (43). Esta subunidad cataliza la transferencia de electrones de citocromo C reducido a oxígeno (44). Otros estudios sugieren un papel regulador para este polipéptido, por su localización extramembranosa y la presencia de un posible dominio de unión a ATP.
La transición de tripomastigotes a amastigotes se acompaña de algunos cambios morfológicos, como la pérdida del flagelo externo, la remodelación del citoesqueleto, y la reducción del tamaño y el volumen del parásito. Varios estudios previos han encontrado que la expresión de los genes de tubulinas en T. cruzi, es regulada durante el ciclo de vida del parásito (45). Generalmente, las tubulinas se encuentran en abundancia en la membrana de los tripomastigotes, mientras que en el estadio de amastigote, a pesar de la disminución del tamaño y volumen, la concentración relativa de tubulinas permanece constante.
En este estudio, las variaciones en la expresión de las tubulinas estuvo relacionada con el tipo de tubulina y la β tubulina se expresó exclusivamente en el estadio de amastigote, mientras que la α tubulina lo hizo en el estadio de tripomastigote. En concordancia con estos resultados, se ha reportado reducción significativa en los niveles de ARNm de α y β tubulinas en epimastigotes y de α tubulinas en amastigotes (46). Las dos proteínas constituyen un componente clave de los microtúbulos, ya que forman polímeros conformados por dímeros con subunidades de 55 kDa.
Si bien el análisis comparativo del perfil proteico de los estadios de tripomastigotes y amastigotes del aislamiento de un paciente crónico del grupo Tc I, mostró diferencias cualitativas y cuantitativas en el perfil de proteínas de este parásito, cabe resaltar que la mayoría de las proteínas identificadas corresponden a proteínas del citosol involucradas en el ensamblaje del citoesqueleto y enzimas relacionadas con el metabolismo glucolítico; dos de estas proteínas, la lipoamida deshidrogenasa y la esterol metil transferasa, han sido sugeridas por otros autores como blancos potenciales para el diseño de fármacos. Sin embargo, se requiere continuar con el estudio de estas proteínas para ratificar su papel en T. cruzi y su relevancia en la interacción entre huésped y parásito y en la supervivencia del parásito, para poderlas usar posteriormente como blancos terapéuticos o en la generación de vacunas.
Agradecimientos Los autores de este estudio desean agradecer al Servicio de Proteómica del Centro de Biología Molecular Severo Ochoa, CBMSO, miembro del Instituto Nacional de Proteómica (ProteoRed), Madrid, España, por la identificación de proteínas por MALDITOF Y LC MS/MS, y a Rocío Niño Moyano, por su apoyo técnico.
Conflicto de intereses Los autores manifiestan que no existe, de manera directa o indirecta, ningún tipo de conflicto de intereses, financieros, académicos ni personales para la publicación de este artículo.
Financiación Los recursos económicos necesarios para la realización de este trabajo fueron aportados por Colciencias, en el marco del proyecto 1102- 343-19320, y por la Universidad Industrial de Santander.
Correspondencia: Clara Isabel González, Escuela de Bacteriología, Facultad de Salud, Universidad Industrial de Santander, Carrera 32 N° 29- 31, oficina 419, Bucaramanga, Colombia.
Teléfono: (577) 6322429; telefax: (577) 6322429.
Referencias
1. Lannes-Vieira J, De Araujo-Jorge T, Correia-Soeiro M, Gadelha P, Correa-Oliveira R. The centennial of the discovery of Chagas disease: Facing the current challenges. PLoS Negl Trop Dis. 2010;4:e645. [ Links ]
2. De Souza W. Basic cell biology of Trypanosoma cruzi. Curr Pharm Des. 2002;8:269-85. [ Links ]
3. El-Sayed N, Myler P, Bartholomeu DC, Nilsson D, Aggarwal G, Tran AN, et al. The genome sequence of Trypanosoma cruzi, etiologic agent of Chagas disease. Science. 2005;309:409-15. [ Links ]
4. Clayton C, Shapira M. Post-transcriptional regulation of gene expression in trypanosomes and leishmanias. Mol Biochem Parasitol. 2007;156:93-101. [ Links ]
5. Goldberg SS, Chiari E. Growth and isolation of single colonies of Trypanosoma cruzi on solid medium. J Parasitol. 1980;66:677-9. [ Links ]
6. Velazco C, Puentes F, Moreno A, Patarroyo M, Puerta C. Adaptación de la cepa Munantá de Trypanosoma cruzi al cultivo in vitro en células Vero. Universitas Scientiarum. 1997;4:83-94. [ Links ]
7. Bradford MM. A rapid and sensitive method for quantitation of microgram quantities of protein utilizing the principle of protein-dye-binding. Anal Biochem. 1976;72:248-54. [ Links ]
8. Shevchenko A, Wilm M, Vorm O, Mann M. Mass spectrometric sequencing of proteins silver-stained polyacrylamide gels. Anal Chem. 1996;68:850-8. [ Links ]
9. Naranjo V, Villar M, Martín-Hernando MP, Vidal D, Höfle U, Gortazar C, et al. Proteomic and transcriptomic analyses of differential stress/inflammatory responses in mandibular lymph nodes and oropharyngeal tonsils of European wild boars naturally infected with Mycobacterium bovis. Proteomics. 2007;7:220-31. [ Links ]
10. Perkins DN, Pappin DJ, Creasy DM, Cottrell JS. Probability-based protein identification by searching sequence databases using mass spectrometry data. Electrophoresis. 1999;20:3551-67. [ Links ]
11. Paba J, Santana JM, Teixeira AR, Fontes W, Sousa MV, Ricart CA. Proteomic analysis of the human pathogen Trypanosoma cruzi. Proteomics. 2004;4:1052-9. [ Links ]
12. Atwood J, Weatherly D, Minning T, Bundy B, Cavola C, Opperdoes F, et al. The Trypanosoma cruzi proteome. Science. 2005;309:473-6. [ Links ]
13. Zafra G, Mantilla JC, Valadares HM, Macedo AM, González CI. Evidence of Trypanosoma cruzi II infection in Colombian chagasic patients. Parasitol Res. 2008;103:731-4. [ Links ]
14. Vertommen D, Van Roy J, Szikora JP, Rider M, Opperdoes FR. Differential expression of glycosomal and mitochondrial proteins in the two major life-cycle stages of Trypanosoma brucei. Mol Biochem Parasitol. 2008;158:189-201. [ Links ]
15. Blumenstiel K, Schoneck R, Yardley V, Croft S, Krauth-Siegel L. Nitrofuran drugs as common subversive substrates of Trypanosoma cruzi lipoamide dehydrogenase and trypanothione reductase. Biochem Pharmacol. 1999;58:1791-9. [ Links ]
16. Parodi-Talice A, Monteiro-Goes V, Arrambide N, Ávila A, Durán R, Correa A, et al. Proteomic analysis of metacyclic trypomastigotes undergoing Trypanosoma cruzi metacyclogenesis. J Mass Spectrom. 2007;42:1422- 32. [ Links ]
17. Schoneck R, Billaut-mui O, Numrich P, Ouaissi´ MA, Krauth-Siegel R. Cloning, sequencing and functional expression of dihydrolipoamide dehydrogenase from the human pathogen Trypanosoma cruzi. Eur J Biochem. 1997;243:739-47. [ Links ]
18. Sreider C, Grinblat L, Stoppani A. Reduction of nitrofuran compounds by heart lipoamide dehydrogenase: Role of flavin and the reactive disulfide groups. Biochem Int. 1992;28:323-34. [ Links ]
19. Leroux AE, Maugeri DA, Cazzulo JJ, Nowicki C. Functional characterization of NADP-dependent isocitrate dehydrogenase isozymes from Trypanosoma cruzi. Mol Biochem Parasitol. 2011;177:61-4. [ Links ]
20. Piacenza L, Peluffo G, Álvarez MN, Kelly JM, Wilkinson SR, Radi R. Peroxiredoxins play a major role in protecting Trypanosoma cruzi against macrophage- and endogenouslyderived peroxynitrite. Biochem J. 2008;410:359-68. [ Links ]
21. Piñeyro MD, Parodi-Talice A, Portela M, Árias DG, Guerrero SA, Robello C. Molecular characterization and interactome analysis of Trypanosoma cruzi Tryparedoxin 1. J Proteomics. 2011;74:1683-92. [ Links ]
22. Díaz ML, Solari A, González CI. Differential expression of Trypanosoma cruzi I associated with clinical forms of Chagas disease: over expression of oxidative stress proteins in acute patient isolate. J Proteomics. 2011;74:1671-82. [ Links ]
23. Souto-Padron T, De Carvalho TU, Chiari E, De Souza W. Further studies on the cell surface charge of Trypanosoma cruzi. Acta Trop. 1984;41:215-25. [ Links ]
24. Clark AK, Kovtunovych G, Kandlikar S, Lal S, Stryker GA. Cloning and expression analysis of two novel paraflagellar rod domain genes found in Trypanosoma cruzi. Parasitol Res. 2005;96:312-20. [ Links ]
25. Landfear S, Ignatushchenko M. The flagellum and flagellar pocket of trypanosomatids. Mol Biochem Parasitol. 2001;115:11-7. [ Links ]
26. Wingard J, Ladner J, Vanarotti M, Fisher A, Robinson H, Buchanan K, et al. Structural insights into membranetargeting by the flagellar calcium-binding protein (FCaBP), a myristoylated and palmitoylated calcium sensor in T. cruzi. J Biol Chem. 2008;283:23388-96. [ Links ]
27. Miranda G, Teixeira E, Miranda K, Weissmuller G, Mascarello P, De Souza W. Structural changes of the paraflagellar rod during flagellar beating in Trypanosoma cruzi. PLoS ONE. 2010;5:e11407. 28. Miller MJ, Wrightsman RA, Stryker GA, Manning JE. Protection of mice against Trypanosoma cruzi by immunization with paraflagellar rod proteins requires T cell, but not B cell, function. J Immunol. 1997;158:5330-7. [ Links ] [ Links ]
29. Wrightsman RA, Miller MJ, Saborio JL, Manning JE. Pure paraflagellar rod protein protects mice against Trypanosoma cruzi infection. Infect Immun. 1995;63:122-5. [ Links ]
30. Stebeck CE, Beecroft RP, Singh BN, Jardim A, Olafson RW, Tuckey C, et al. Kinetoplastid membrane protein-11 (KMP- 11) is differentially expressed during the life cycle of African trypanosomes and is found in a wide variety of kinetoplastid parasites. Mol Biochem Parasitol. 1995;71:1-13. [ Links ]
31. Fuertes MA, Pérez JM, Soto M, López MC, Alonso C. Calcium-induced conformational changes in Leishmania infantum kinetoplastid membrane protein-11. J Biol Inorg Chem. 2001;6:107-17. [ Links ]
32. Thomas MC, García-Pérez JL, Alonso C, López MC. Molecular characterization of KMP11 from Trypanosoma cruzi: A cytoskeleton-associated protein regulated at the translational level. DNA Cell Biol. 2000;19:47-57. [ Links ]
33. Diez H, López MC, Thomas M, Puerta C. KMP-11: proteína 11 de membrana de kinetoplástidos. Univérsitas Scientiarum. 2004;9:29-44. [ Links ]
34. Palmié-Peixoto IV, Rocha M, Urbina JA, De Souza W, Einicker-Lamas M, Machado MC. Effects of sterol biosynthesis inhibitors on endosymbiont-bearing trypanosomatids. FEMS Microbiol Lett. 2006;255:33-42. [ Links ]
35. De Vas M, Portal P, Alonso G, Schlesinger M, Flawiá M, Torres H, et al. The NADPH-cytochrome P450 reductase family in Trypanosoma cruzi is involved in the sterol biosynthesis pathway. Int J Parasitol. 2011;41:99-108. [ Links ]
36. Sant´Anna C, Nakayasu E, Pereira M, Lourenço D, De Souza W, Almeida IC, et al. Subcellular proteomics of Trypanosoma cruzi reservosomes. Proteomics. 2009;9:1782-94. [ Links ]
37. van Hellemond JJ, Tielens AG. Adaptations in the lipid metabolism of the protozoan parasite Trypanosoma brucei. FEBS Lett. 2006;580:5552-8. [ Links ]
38. Urbina JA. Chemotherapy of Chagas disease: The how and the why. J Mol Med. 1999;77:332-8. [ Links ]
39. Silber AM, Tonelli R, Lopes C, Cunha-e-Silva N, Torrecilhas A, Schumacher R, et al. Glucose uptake in the mammalian stages of Trypanosoma cruzi. Mol Biochem Parasitol. 2009;168:102-8. [ Links ]
40. Nowicki C, Cazzulo JJ. Aromatic amino acid catabolism in trypanosomatids. Comp Biochem Physiol A Mol Integr Physiol. 2008;151:381-90. [ Links ]
41. Tielens A, Van Hellemond J. Differences in energy metabolism between Trypanosomatidae. Parasitol Today. 1998;14:265-70. [ Links ]
42. Maslov D, Zíkova A, Kyselova I, Lukes J. A putative novel nuclear-encoded subunit of the cytochrome C oxidase complex in trypanosomatids. Mol Biochem Parasitol. 2002;125:113-25. [ Links ]
43. NeboháÄová M, Kim CE, Simpson L, Maslov D. RNA editing and mitochondrial activity in promastigotes and amastigotes of Leishmania donovani. Int J Parasitol. 2009;39:635-44. [ Links ]
44. Schneider A. Unique aspects of mitochondrial biogenesis in trypanosomatids. Int J Parasitol. 2001;31:1403-15. [ Links ]
45. Da silva R, Bartholomeu D,Teixeira S. Control mechanisms of tubulin gene expression in Trypanosoma cruzi. Int J Parasitol. 2006;36:87-96. [ Links ]
46. González-Pino MJ, Rangel-Aldao R, Slezynger TC. Cloning and sequence analysis of a Trypanosoma cruzi a-tubulin cDNA. Biol Res. 1997;30:161-6. [ Links ]














