Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157
Biomédica vol.32 no.3 Bogotá Jul./Sept. 2012
https://doi.org/10.7705/biomedica.v32i3.392
ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v32i3.392
1Programa de Estudio y Control de Enfermedades Tropicales, PECET, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia
2Laboratório de Pesquisa em Leishmaniose, Instituto Oswaldo Cruz-FIOCRUZ, Rio de Janeiro, Brasil
Contribución de los autores:
Ronald Guillermo Peláez: contribuyó al desarrollo de todos los ensayos de laboratorio, al análisis de los datos y a la escritura del artículo.
Carlos Enrique Muskus y Patricia Cuervo: contribuyeron al diseño y desarrollo del proyecto, al entrenamiento del estudiante de maestría (Ronaldo Guillermo Peláez) y al análisis de los datos.
Marcel Marín-Villa: contribuyó al diseño y desarrollo del proyecto, análisis de los datos, escritura y corrección del artículo.
Recibido: 01/07/11; aceptado:27/04/12
Introducción. Los mecanismos de resistencia al antimonio pentavalente conocidos hasta el momento, se han descrito ampliamente en cepas del subgénero Leishmania, pero poco se sabe sobre las proteínas involucradas en los mecanismos de resistencia presentes en cepas del subgénero Viannia, como Leishmania panamensis.
Objetivo. Identificar proteínas diferencialmente expresadas entre las cepas de L. panamensis (UA140), sensible y resistente al antimonio pentavalente, y analizar el posible papel de estas proteínas en mecanismos de resistencia.
Materiales y métodos. Las proteínas de las cepas, sensible y resistente al antimonio pentavalente, se compararon usando electroforesis bidimensional. Las proteínas con aumento de la expresión fueron aisladas e identificadas por espectrometría de masas mediante MALDI-TOF/TOF (Matrix Assisted Laser Desorption Ionization/Time of Flight). La expresión del ARNm de cinco de estas proteínas se cuantificó mediante PCR en tiempo real.
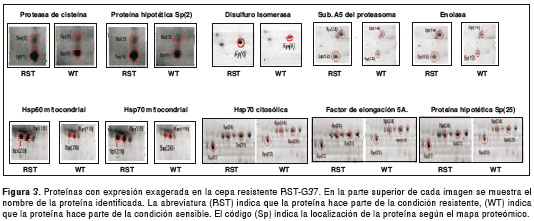
Resultados. Los geles bidimensionales de las cepas sensible y resistente detectaron 532±39 y 541±43 manchas proteicas. Se encontraron 10 manchas con aumento de la expresión en la cepa resistente, identificadas como proteínas de choque térmico (Hsp60 mitocondrial, Hsp70 mitocondrial y citosólica), isomerasa de disulfuro, proteasa de cisteína, enolasa, factor de elongación 5-α, la subunidad 5-α del proteasoma y dos proteínas hipotéticas nombradas como Sp(2) y Sp(25).
Conclusión. Este es el primer estudio llevado a cabo con una cepa resistente al antimonio pentavalente en L. panamensis, en el cual se han identificado proteínas que están relacionadas con el mecanismo de resistencia del parásito frente al medicamento, abriendo el camino para futuros estudios de estas proteínas como blancos terapéuticos.
Palabras clave: Leishmania, proteómica, electroforesis, espectrometría de masas, reacción en cadena de la polimerasa.
doi: http://dx.doi.org/10.7705/biomedica.v32i3.392
Differential expression of proteins in Leishmania (Viannia) panamensis associated with mechanisms of resistance to meglumine antimoniate.
Introduction. The well-known drug resistance mechanisms to pentavalent antimony have been widely described in strains of the Leishmania subgenus, but little is known about the mechanisms of resistance and the proteins associated with it in strains of the Viannia subgenus such as Leishmania panamensis.
Objective. Differentially expressed proteins were identified between pentavalent antimonial sensitive and resistant L. panamensis (UA140) strains, and the role of these proteins was analyzed as possible resistance mechanisms.
Materials and methods. The protein lysates of pentavalent antimony sensitive and resistant strains were separated by two-dimensional gel electrophoresis,and the protein patterns compared. The proteins identified as overexpressed were separated and analyzed using MALDI-TOF/TOF (Matrix Assisted Laser Desorption Ionization/Time of Flight). The level of mRNA expression of five of these proteins was quantified using real-time PCR.
Results. On the 2-dimensional gels, 532 ± 39 protein spots were identified for the sensitive strains, and 541 ± 43 spots for the resistant strains. Ten spots were overexpressed in the resistant strain and identified as heat shock protein (Hsp60 mitochondrial, Hsp70 cytosolic and mitochondrial), disulfide isomerase, cysteine protease, enolase, elongation factor 5-alpha, the proteasome alpha-5 subunit and two hypothetical proteins named as Sp(2) and Sp(25).
Conclusion. This is the first proteomic study conducted with a L. panamensis resistant strain where several proteins were identified and related with the parasite resistance mechanism to pentavalent antimony. This opens the way for future studies aimed at modulating the drug resistance or at evaluating these proteins as therapeutic targets.
Key words: Leishmania, proteomics, electrophoresis, mass spectrometry, polymerase chain reaction.
doi: http://dx.doi.org/10.7705/biomedica.v32i3.392
La leishmaniasis es una enfermedad de distribución mundial que afecta a 98 países en cuatro continentes, con un reporte de 12 millones de personas infectadas en el mundo y 2 millones de casos nuevos anuales, de los cuales, un cuarto corresponde a la forma visceral de la enfermedad (1,2).
Las fallas en la producción de una vacuna efectiva y los innumerables inconvenientes en el control del vector, han hecho que el control de la enfermedad se enfoque principalmente en la quimioterapia (3,4). En la actualidad, cada vez son más los estudios que reportan el hallazgo de Leishmania spp. resistente al antimonio pentavalente alrededor del mundo y en Colombia (5-18). Como tratamientos de segunda línea se utilizan la anfotericina B, la pentamidina y la miltefosina, que son medicamentos efectivos contra la leishmaniasis, pero, debido a sus altos costos y los efectos secundarios que pueden ocasionar, estos son poco utilizados (18).
El antimonio pentavalente es suministrado como un promedicamento que requiere ser activado a antimonio trivalente; no se tiene claro si su activación se produce por una vía enzimática o de manera espontánea (19). Se han reportado varios blancos celulares para el antimonio pentavalente y el antimonio trivalente , incluyendo el tripanotión, que es el principal agente reductor de tioles en el parásito (20-22).
Los mecanismos por los cuales Leishmania spp. adquiere resistencia, han sido motivo de discusión durante varias décadas. Se propone que los altos niveles de resistencia pueden deberse a la pérdida en la reducción de los metales, disminución en el ingreso del medicamento, incremento en la síntesis de tripanotión-glutatión e incremento en el transporte (secuestro o eflujo) del conjugado metal-tiol (23).
Dado que son pocos los estudios sobre la especie Leishmania panamensis, el tener una aproximación al proteoma de una cepa resistente al antimonio pentavalente, es un paso importante para el descubrimiento de proteínas y mecanismos de resistencia que pueden ser extrapolables a cepas naturalmente resistentes, dando indicios de cómo actúan los medicamentos y por qué algunas veces fallan, lo cual puede ser útil para el desarrollo de nuevos tratamientos.
En el presente estudio se compararon los perfiles de proteínas citosólicas en una cepa de L. panamensis resistente y otra sensible al antimonio pentavalente, identificando las proteínas con aumento de la expresión en la cepa resistente y, de acuerdo con sus funciones biológicas, se proponen algunos posibles papeles en el mecanismo de resistencia al antimonio pentavalente para estas moléculas.
Materiales y métodos
Inducción de resistencia in vitro
Para inducir la resistencia in vitro al antimonio pentavalente en promastigotes de L. panamensis, cepa (MHOM/87/CO/UA140), los promastigotes se cultivaron en medio Schneider (Sigma®) con 10 % de suplemento de suero fetal bovino en presencia de penicilina y estreptomicina (10.000 U/ml -10.000 µg/ml) a 26 °C. Los parásitos se lavaron dos veces en PBS, y se centrifugaron a 2.500g durante 10 minutos a temperatura ambiente. Los promastigotes se ajustaron a una concentración de 2 x 106 por ml en medio de cultivo y se adicionó un mezcla de antimonio pentavalente (Glucantime®, Aventis Pharma Ltda., Brasil) de los lotes L203890 y L203891 a su concentración efectiva 50 (CE50), correspondiente a 4 mg/ml. Esta concentración se determinó previamente utilizando la técnica de bromuro 3-(4,5-dimetil tiazol-2-il)-2,5-difenil tetrazolio (MTT) (24). Los promastigotes se incubaron a 26 °C por cinco días, haciendo cambio de medio con medicamento; en cada cambio, los parásitos se lavaron con PBS a pH 7,0 ajustando de nuevo la concentración de parásitos a 2 x 106 promastigotes/ml de medio.
Este procedimiento se hizo hasta obtener una viabilidad de los parásitos en presencia del medicamento mayor de 80 %, por recuento en cámara de Neubauer. Se utilizaron concentraciones crecientes y graduales de antimonio (8, 12, 16, 20, 24, 26, 28, 32, 33 mg/ml), hasta alcanzar una concentración final de 37 mg/ml. En un trabajo previo, para demostrar un comportamiento diferencial frente al medicamento entre la cepa susceptible y la resistente al antimonio pentavalente, se evaluó la resistencia in vivo infectando hámsters con las dos cepas y midiendo el tamaño de la nariz antes de la inoculación, después del desarrollo de la lesión (día 42), terminado el tratamiento con antimonio pentavalente (día 52) y cinco días después del tratamiento (día 57) (no se presentan los datos).
Extracción de proteínas
Para la obtención de proteínas de promastigotes de L. panamensis sensible y resistente al antimonio pentavalente, se utilizó el método descrito por Cuervo, et al. (25). Se tomaron cultivos de promastigotessensibles y resistentesen fase logarítmicacrecidos durante cuatro días en botellas en una banda giratoria, recolectados por centrifugación a 2.000g durante 10 minutos a 4 °C y lavados dos veces con PBS a pH 7,2, resuspendidos en 2 ml de PBS hipotónico (13,6 mM NaCl, 0,27 mM KCl, 0,4 mM Na2HPO4, 0,15 mM KH2PO4) en presencia de 1X de cóctel de inhibidores de proteasas (Roche), para obtener una concentración de 5 x 109 parásitos/ml. Los parásitos se lisaron por 15 ciclos de congelamiento y descongelamiento, mediante pasos sucesivos en nitrógeno líquido y periodos de cinco minutos en baño con ultrasonido a temperatura ambiente, respectivamente. Los lisados se centrifugaron a 14.000g por siete minutos a 4 °C, en una centrífuga Thermo IEC MicroMax® (Harlow Scientific), para remover el material insoluble y se recuperó el sobrenadante que contenía las proteínas solubles. Las proteínas se precipitaron por incubación con 10 % (v/v) de ácido tricloroacético durante una hora a 4 °C; luego se centrifugaron a 14.000g por 15 minutos a 4 °C y se lavaron con acetona fría (Sigma), bajo las mismas condiciones.
El sedimento de proteínas se dejó secar a temperatura ambiente y luego se volvió a suspender en solución tampón de isoelectroenfoque compuesta de 9 M de urea, 4 % (p/v) CHAPS, 40 mM DTT y 1 % (v/v) de anfolitos 3-10 (GE Healthcare), según el protocolo empleado por Cuervo, et al. (25). Posteriormente, se determinó la concentración de las proteínas con el 2D Quant Kit™ (GE Healthcare), siguiendo las recomendaciones de la casa comercial. Las proteínas cuantificadas se guardaron a -20 °C hasta su uso.
Electroforesis en dos dimensiones
Para el isoelectroenfoque se utilizaron alícuotas de 600 µg de proteínas en un volumen final de 350 µl de solución de rehidratación (9 M de urea, 4 % CHAPS, 40 mM DTT y 1 % de anfolitos 3-10). Las proteínas se separaron por punto isoeléctrico (pI), en tiras de poliacrilamida (IPG) de 18 cm de longitud con un rango de pH de 4 a 7 (GE Healthcare), utilizando el sistema Ettan IPGphor 3 a una temperatura de 20 °C a 50 µA/tirilla.
Los parámetros de isoelectroenfoque fueron los siguientes: rehidratación pasiva por 14 horas; constante de 1 a 200 V por cuatro horas; gradiente desde 200 a 500 V por una hora; gradiente desde 500 a 1.000 V por una hora; gradiente desde 1.000 a 8.000 V por 30 minutos; 8.000 V constantes por seis horas o hasta completar un voltaje igual o superior a 32.000. Antes de la segunda dimensión, las tiras se redujeron (10 mg/ml de DTT) y alquilaron (25 mg/ml de iodoacetamida) en 5 ml de solución tampón de equilibrio (urea 6M, SDS 6%, Tris/Cl 0,375M, glicerol 20 %) durante 15 minutos.
La segunda dimensión se hizo en geles de poliacrilamida al 12 % de 1,5 mm de espesor. Las tiras fueron selladas con agarosa al 1 % (p/v) que contenía azul de bromofenol al 0,001 %. La electroforesis se llevó a cabo en un sistema Ettan DALT Six™ (GE Healthcare) a 10 °C en solución tampón estándar Tris/Glicina/SDS, empleando 2,5 W/gel por 30 minutos, seguido de 15 W/gel por cuatro horas o hasta que el azul de bromofenol alcanzara la parte inferior del gel. Para la visualización de las proteínas, los geles se tiñeron con coloración de azul de Coomassie coloidal (G-250), según el protocolo estándar descrito por Neuhoff, et al. (26).
Se hicieron nueve réplicas de geles bidimensionales para cada uno de los perfiles proteicos de la línea de L. panamensis sensible y la resistente a antimonio pentavalente, empleando muestras proteicas de cultivos independientes. Las imágenes de los geles se adquirieron en el escáner ImageScannerTM III™ y el análisis diferencial de las imágenes se llevó a cabo en el software ImageMaster 2D Platinum 7.0™ (GE Healthcare). Para determinar si una proteína se expresaba de manera diferencial, se consideró que el cambio relativo de expresión era estadísticamente significativo si el análisis de varianza (ANOVA) arrojaba un valor de p<0,05 y el valor de proporcionalidad era igual o superior a 1,5.
Identificación de proteínas por espectrometría de masas y análisis bioinformáticos
Las manchas proteicas que mostraron una expresión diferencial en los geles bidimensionales se cortaron manualmente; los trozos de gel se almacenaron en ácido acético al 7 % y se enviaron al System Proteomics Center, University of North Carolina-Chapel Hill, en Estados Unidos, para su análisis por espectrometría de masas. Las proteínas se digirieron con tripsina y los péptidos resultantes se analizaron en un espectrómetro de masas ABI 4800 MALDI-TOF/TOFTM (Applied Biosystems), trabajando en un modo de reflectrón positivo.
Se obtuvo un espectro de barrido completo en el rango de m/z de 700 a 4.000, seguido de máximo 45 espectros MS/MS por mancha proteica. Los espectros se calibraron externamente utilizando una mezcla estándar 4.700 Proteomics Analyzer Calibration Mixture (Applied Biosystems) e, internamente, utilizando iones derivados de los péptidos de autodigestión de la tripsina. Los espectros de masa se interpretaron usando el programa GPS ExplorerTM Protein Analysis™, versión 3.6 (Applied Biosystems), el cual permite búsquedas no redundantes en la base de datos NCBInr del National Center for Biotechnology Information, mediante el motor de búsqueda MASCOT® (Matrix Science).
Para las búsquedas no se restringió la masa molecular ni el punto isoeléctrico (pI)de las proteínas; no se hizo restricción de grupo taxonómico, se eligió tripsina como enzima de digestión especificando dos cortes parciales, y se tuvieron en cuenta modificaciones como la oxidación de las metioninas y la acetilación de las lisinas.
Una tolerancia de masa de 80 ppm y 0,5 Da se asignaron para la comparación de los espectros de masa (modo MS) y sus fragmentos correspondientes (modo MS/MS), respectivamente.
Finalmente, todos los péptidos identificados automáticamente por homología con especies de Leishmania, se confirmaron: i) buscando la homología proteica en la base de datos GeneDB Leishmania braziliensis, y ii) haciendo secuenciación manual de todos los péptidos no identificados por el programa GPS ExplorerTM. Las funciones biológicas de las proteínas de L. panamensis identificadas se asignaron de acuerdo con la ontología de genes y se determinó la probable vía metabólica en la cual participan las proteínas por medio de la librería de genes y genomas de Kyoto (KEGG); ambos análisis se basaron en búsqueda de información de las proteínas de L. braziliensis y L. major anotadas en la base de datos GeneDB.
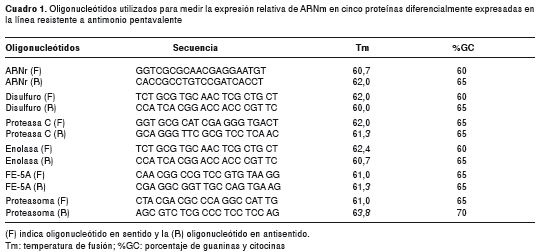
Cuantificación del ARNm en proteínas con aumento de expresión
Se diseñaron seis juegos de cebadores para los genes 16S (gen normalizador), proteasa de cisteína, isomerasa de disulfuro, enolasa, factor de iniciación 5-α, subunidad 5-α del proteasoma (cuadro 1). El ARN total se extrajo con Trizol Reagent™ (Invitrogen), según recomendaciones de la casa comercial. Los extractos de ARN total de las líneas sensibles y resistentes, se cuantificaron mediante el espectrofotómetro NanoDrop 1000™ (Thermo Scientific) y se tomaron volúmenes equivalentes a 2 µg de ARN para el proceso de transcripción inversa, el cual se practicó con el estuche comercial QuantiTect Reverse Transcription™ (Qiagen).
Para cada reacción de PCR en tiempo real, las concentraciones de los reactivos fueron las siguientes: 12,5 µl de 2x QuantiFast SYBR Green PCR Master Mix (1x), 1 µl de cada cebador (1 µM), 2 µl de ADNc, 8,5 µl de agua libre de ARNsa, para completar un volumen final de 25 µl.
El perfil térmico utilizado para todos los genes fue de cinco minutos a 95 °C para la activación inicial de la Taq polimerasa; luego se usaron 35 ciclos con una temperatura de desnaturalización de 95 °C por 10 segundos y una temperatura de hibridación-extensión de 60 °C por 30 segundos. Una vez terminados los 35 ciclos, se hizo un análisis de melting, dejando las reacciones a 72 °C durante cinco minutos y con un rampa de temperatura de 60 a 95 °C, obteniendo los datos de fluorescencia cada 0,4 segundos.
El cálculo del número de veces que un transcripto estaba diferencialmente expresado entre la cepa sensible y la resistente, se estimó mediante el método del delta-delta Ct (27), teniendo en cuenta la eficiencia de amplificación para cada gen. La eficiencia de amplificación se calculó mediante el programa LinReg, empleando, al menos, cuatro o cinco puntos de los valores de fluorescencia de la fase de amplificación exponencial.
Resultados
Leishmania panamensis resistente a antimonio pentavalente
Mediante un proceso gradual de selección in vitro, fue posible obtener parásitos de L. panamensis resistentes al antimonio pentavalente, los cuales presentaban la capacidad de sobrevivir y multiplicarse en medio de cultivo con altas concentraciones del medicamento (37 mg/ml). En la evaluación del tamaño de la lesión de hámsters infectados con la cepa silvestre y la resistente de L. panamensis y tratados con antimonio pentavalente, se pudo observar una diferencia estadísticamente significativa (Mann-Whitney de dos colas; p=0,0016), en el tamaño de la lesión entre los dos grupos de animales después de diez días de tratamiento con antimonio pentavalente (no se presenta el dato), lo cual indica diferencias en la sensibilidad al medicamento entre las dos cepas. En los cultivos en masa crecieron 1 x 1010 promastigotes/100 ml, tanto en la cepa sensible como en la resistente, obteniéndose entre 2 y 4 mg de proteínas que fueron disueltas en solución tampón IEF y almacenadas a -20 °C.
Análisis comparativo del proteoma de las cepas sensible y resistente
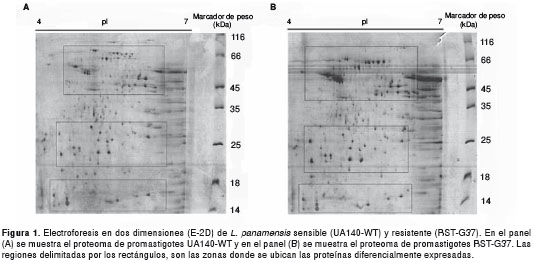
Los dos perfiles proteicos obtenidos para las cepas sensible y resistente presentaron, 532±39 manchas proteicas en la cepa sensible y 541±43 manchas proteicas en la cepa resistente. Estas proteínas tuvieron puntos isoeléctricos entre 4,09 y 7,00, y masas moleculares correspondientes a un intervalo de 10 kDa y 116 kDa, con la mayoría de las manchas localizadas por encima de los 23 kDa (figura 1).
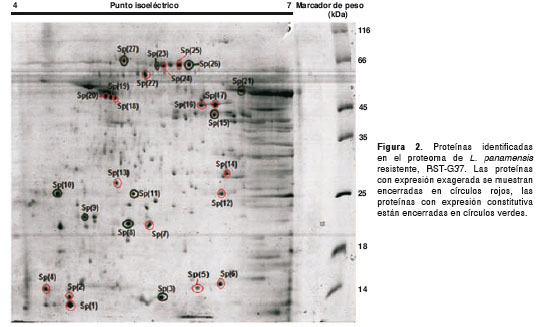
Una vez hecho el análisis de imágenes, al comparar los perfiles proteicos de las cepas sensible y resistente, se encontraron 16 proteínas que presentaban un aumento en la expresión mayor o igual a 1,5 veces y con un valor p (ANOVA) menor o igual a 0,05. De manera paralela, se eligieron 10 proteínas con expresión constitutiva entre la cepa resistente y la sensible al antimonio pentavalente, con valores de p (ANOVA) mayores de 0,05 y con diferencias en la expresión menores de 1,5 veces, con el objetivo de validar su uso posterior como posibles genes normalizadores en estudios de transcriptoma.
Entre las manchas proteicas que tuvieron un aumento en la expresión, estuvieron: Sp(1), Sp(2), Sp(5) , Sp(6) , Sp(7), Sp(12), Sp(13), Sp(14), Sp(16), Sp(17), Sp(18), Sp(19), Sp(20), Sp(22), Sp(24) y Sp(25); mientras que las manchas proteicas que no tuvieron variaciones de expresión entre las cepas sensible y resistente al antimonio pentavalente, fueron nombradas como: Sp(3), Sp(8), Sp(9), Sp(10), Sp(11), Sp(15), Sp(21), Sp(23), Sp(26) y Sp(27) (figura 2).
Identificación de las proteínas seleccionadas
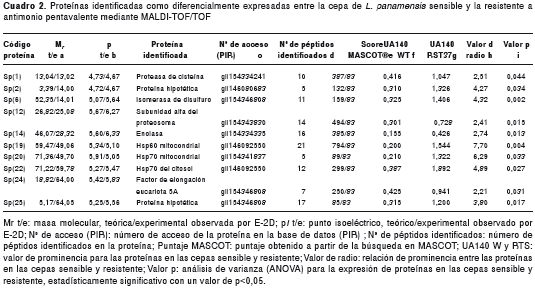
En la mayoría de las proteínas analizadas, la correlación del punto isoeléctrico (pI) teórico con el experimental fue la esperada, abarcando puntos isoeléctricos entre 4 y 6,8. Solo algunas proteínas de masa molecular de 13 a 67 kDa tuvieron variaciones significativas en los valores teórico y experimental de masa, debido a la fragmentación de algunas de ellas.
De las 26 proteínas que se identificaron, 16 correspondían a proteínas con aumento de la expresión en la cepa resistente. Sin embargo, una vez caracterizadas por espectrometría de masas, se evidenció que varias de estas proteínas correspondían a péptidos fragmentados de tres proteínas, la Hsp70 mitocondrial, la isomerasa de disulfuro y la enolasa.
Teniendo en cuenta lo anterior, el número de proteínas con aumento de la expresión se redujo a 10 (cuadro 2, figura 3). En las proteínas con expresión constitutiva en la cepa sensible y la resistente, se identificaron siete de ellas como: endorribonucleasa Sp(3), complejo proteolítico (hs1vu) Sp(9), posible polipéptido naciente Sp(11), proteína ligasa de biotina Sp(15), proteína de unión a GTP Sp(21), proteína reguladora de glucosa Sp(23) y s-adenosilhomocisteína hidrolasa Sp(27). El número total de péptidos encontrados para cada proteína estuvo entre 3 y 26. La identificación de las proteínas se aceptó como válida, ya que la puntuación del coeficiente Mowse fue superior a 83 (p<0,05) y la cobertura de secuencia para los péptidos correspondió, al menos, a 10 % del tamaño de la proteína.
Clasificación de las proteínas según su función biológica
Las proteínas identificadas mediante espectrometría de masas, se agruparon en seis categorías:
1) proteólisis (proteasa de cisteína, complejo proteolítico Hs1vu, subunidad 5 alfa del proteasoma),
2) plegamiento/chaperonas (isomerasa de disulfuro, Hsp70m, Hsp70c, Hsp60m, proteína reguladora glucosa),
3) síntesis de proteínas (posible polipéptido naciente, proteína de unión a GTP, factor de elongación eucariota 5 alfa),
4) metabolismo de carbohidratos (enolasa),
5) metabolismo de aminoácidos (S-adenosil homocisteína hidrolasa), y
6) función desconocida (Sp2, Sp25, endorribo-nucleasa, proteína ligasa de biotina).
Cuantificación del ARNm por PCR en tiempo real de cinco proteínas con aumento de expresión por electroforesis bidimensional
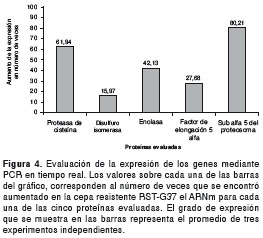
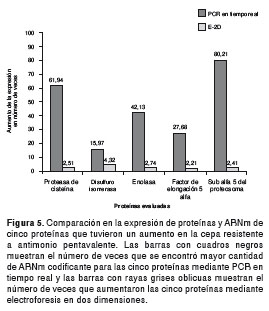
Utilizando diferentes cantidades de parásitos, se logró determinar que el número óptimo para los procesos de extracción de ARN es de 5 x 106 parásitos, lográndose extraer entre 533 y 975 ng/µl de ARN total de la cepa resistente y entre 500 y 950 ng/µl de la cepa sensible. El número de veces que se encontró expresión exagerada de la cantidad de ARNm en la cepa resistente a antimonio pentavalente con respecto a la sensible, fue de 61,9 veces para el gen de la proteasa de cisteína, 15,9 en la isomerasa de disulfuro, 42,1 en la enolasa, 27,6 en el factor de elongación 5 alfa y 80,2 en la subunidad 5 alfa del proteasoma (figura 4). Al hacer un análisis comparativo entre las veces que se expresan en forma aumentada las proteínas cuantificadas mediante electroforesis en dos dimensiones y el ARNm de las proteínas cuantificado por PCR en tiempo real, se encontró que con ambas técnicas se evidenció un incremento en la expresión de ambos tipos de moléculas en la cepa resistente (figura 5).
Discusión
La aparición de cepas de Leishmania resistentes a los medicamentos antimoniales en varias partes del mundo, ha venido en incremento como consecuencia de la mala utilización del antimonio pentavalente como fármaco de primera línea por más de 50 años para el tratamiento de las diferentes formas clínicas de la leishmaniasis (28). Inicialmente, una pequeña dosis del medicamento (10 mg/kg) por un corto tiempo (6 a 10 días) era suficiente para la resolución de la enfermedad, pero, la aparición de reportes de fallas clínicas al tratamiento, obligó al aumento de la dosis a 20 mg/kg por 30 a 40 días (23).
En el distrito norte de Bihar, en la India, se ha reportado falla clínica al tratamiento con antimonio pentavalente en 56 a 60 % de los pacientes (28). De igual manera, en países de Suramérica, como Brasil (9), Venezuela (6) y Perú (16), se han evidenciado fallas clínicas al tratamiento con antimonio pentavalente, lo cual sugiere que pueden ser ocasionadas por parásitos resistentes al medicamento.
En Colombia, se tienen pocos informes de resistencia, aunque se ha documentado que las fallas clínicas al tratamiento con antimonio cada vez son más frecuentes, lo cual indica que la falla clínica al antimonio pentavalente en casos de leishmaniasis cutánea puede deberse a la resistencia de L. panamensis al antimonio pentavalente (10-12).
La resistencia de Leishmania spp. a los antimoniales parece estar mediada principalmente por mecanismos de amplificación y coamplificación génica, y sistemas de eflujo del medicamento asociados a proteínas de membrana (29-31). Kumar, et al., caracterizaron en el 2010 el proteoma de un aislamiento clínico de L. donovani resistente a estibogluconato de sodio, identificando proteínas con aumento de expresión (32).
No obstante el trabajo anterior, nuestra investigación es uno de los pocos estudios proteómicos en una cepa resistente a antimonio pentavalente y es el primer trabajo llevado a cabo en L. panamensis resistente a este medicamento. La cepa fue seleccionada hasta 37 mg/ml de antimonio pentavalente, debido a que se buscó tener un espectro de resistencia bastante amplio entre la cepa sensible y la resistente (CE50 de 4 mg/ml versus 37 mg/ml) y con mayor probabilidad de detectar proteínas asociadas al fenotipo resistente. En esta cepa resistente, se encontró expresión exagerada de las proteínas Hsp60 mitocondrial, Hsp70 mitocondrial y citosólica, proteína de isomerasa de disulfuro y dos proteínas hipotéticas nombradas como Sp(2) y Sp(25), las cuales aparentemente podrían estar participando en una respuesta inicial del parásito para evadir la acción leishmanicida del antimonio pentavalente de manera indirecta, contribuyendo al mecanismo primario de desintoxicación mediado por las moléculas de tripanotión-glutatión al eliminar las formas disulfuro de estos compuestos (19). Además, estas proteínas podrían contribuir a evitar la pérdida del potencial de membrana mitocondrial, controlar el potencial de oxidorreducción de la célula y evitar los procesos de apoptosis de fragmentación del ADN inducido por los antimoniales pentavalentes.
En varios estudios se ha demostrado que los medicamentos antimoniales inducen un proceso similar a la muerte celular programada de tipo apoptosis, en promastigotes y amastigotes de Leishmania spp. (17,33,34). El proceso de muerte de tipo apoptosis en Leishmania spp. ha sido demostrado claramente a nivel fenotípico, con el estudio de varias características citoplásmicas, mitocondriales y nucleares, como la contracción de las células, la liberación de fosfatidilserina mitocondrial, una disminución en el potencial de la membrana mitocondrial, la liberación de citocromo C, la condensación del ADN y la activación de las proteasas citoplásmicas (35-37).
En concordancia con nuestros hallazgos, las Hsp70, en sus versiones citosólicas y mitocondriales, se han reportado con aumento de la expresión después de estimular promastigotes de Leishmania spp. con antimonio trivalente, lo que sugiere su papel protector en un posible mecanismo de tolerancia primario a los antimoniales. Igualmente, se ha postulado que la proteína Hsp83 mitocondrial podría estar confiriendo un mecanismo protector contra la muerte celular programada inducida por el antimonio pentavalente, evitando la disminución en el potencial de membrana mitocondrial (37,38). Lo anterior nos permite sugerir que el aumento de la expresión de los precursores mitocondriales Hsp60 y Hsp70, presentes en la matriz mitocondrial de L. panamensis, puede participar de igual manera en el mantenimiento del potencial de membrana mitocondrial que está siendo afectado por el antimonio pentavalente en los promastigotes resistentes al medicamento.
La proteína hipotética Sp(2) presenta un dominio Alba, cuya principal función es la unión reversible de proteínas al ADN (39,40). Algunas proteínas que poseen este dominio están distribuidas uniforme y abundantemente sobre el cromosoma, al parecer, participando en el establecimiento o mantenimiento de la arquitectura de la cromatina, y posiblemente protegiendo al ADN de un proceso de fragmentación durante la apoptosis. En nuestro estudio se encontraron dos proteínas con el potencial de reemplazar la función de la reductasa de tripanotión, la proteína hipotética Sp(25) y la isomerasa de disulfuro.
La proteína Sp(25), al poseer un dominio tiolasa, juega un papel clave en el mecanismo de resistencia al antimonio, ya que presenta la función potencial de escindir las formas disulfuro de tripanotión y glutatión, reemplazando o complementando de esta manera la función de la reductasa de tripanotión, la cual se encuentra inhibida por el antimonio trivalente, dándose de esta manera la producción normal de tioles dentro del parásito.
La isomerasa de disulfuro es un miembro de la superfamilia tiorredoxina, enzima capaz de catalizar la oxidación, reducción e isomerización de los puentes disulfuro según el potencial de oxidorreducción in vitro (41); esta enzima podría convertir las formas disulfuro del tripanotión y el glutatión a sus formas de tioles libres, contribuyendo de esta manera al secuestro y excreción del antimonio trivalente, mediante transportadores de membrana dependientes del conjugado antimonio trivalentetiol, como la glucoproteína P (Pgpa) o los transportadores de resistencia múltiple a fármacos (42).
Además de las proteínas mencionadas anteriormente, otras como la proteasa de cisteína, la subunidad 5 alfa del proteasoma, la enolasa y el factor de elongación 5 alfa eucariota, también tuvieron un aumento significativo en la cepa resistente y, a pesar de que no se logró vincularlas directamente con el mecanismo de resistencia a antimonio pentavalente, creemos que posiblemente son el reflejo de un cambio metabólico que sufre el parásito para poder mantener el mecanismo de resistencia, debido a que estas proteínas están involucradas en procesos como la síntesis de proteínas, la proteólisis y el metabolismo de carbohidratos.
Cabe anotar que solo en cinco proteínas analizadas por electroforesis en dos dimensiones, se cuantificó la expresión de ARNm mediante PCR en tiempo real (figura 5). Sin embargo, se encontró que la expresión difiere bastante entre estas dos técnicas, debido a que la cuantificación de la expresión por PCR en tiempo real presenta una sensibilidad superior a la cuantificación observada mediante la electroforesis en dos dimensiones. A pesar de ello, lo que se encontró exageradamente expresado en las proteínas, también lo fue en el ARNm. Una explicación lógica de las diferencias en el nivel de aumento de expresión detectado por ambas técnicas, puede ser que la traducción de proteínas en Leishmania spp. no conserva una relación lineal con la transcripción de ARNm debido a los diferentes mecanismos de regulación postranscripcionales que existen en este parásito (43-45).
En este trabajo se encontró que tres de las proteínas con expresión exagerada (las proteínas Hsp70, la enolasa y la subunidad 5 alfa del proteasoma) también se reportaron con aumento de la expresión por Kumar, et al., en un aislamiento clínico de L. donovani, lo cual confirma y apoya nuestros resultados.
Este trabajo es la primera aproximación proteómica para identificar proteínas involucradas en mecanismos de resistencia al antimonio pentavalente en L. panamensis, lo cual valida la utilidad de la electroforesis en dos dimensiones en combinación con la espectrometría de masas. Posteriores estudios de análisis funcional de genes y establecimiento de la función enzimática de estas proteínas, podrían ayudar a dilucidar el verdadero papel de estas proteínas en los mecanismos de resistencia que presentan los promastigotes de L. panamensis para evadir el efecto tóxico de los antimoniales.
Los autores de este estudio agradecen a Óscar Álzate, director del System Proteomics Center, University of North Carolina-Chapel Hill, U. S. A., por los servicios prestados en la identificación de las proteínas por MALDI-TOF/TOF.
Declaramos que en este trabajo no se presenta ningún conflicto de intereses.
Este estudio fue financiado por el proyecto 111540820533 de Colciencias, CODI: Universidad de Antioquia y el Programa para la Formación de Jóvenes Investigadores de Colciencias.
Correspondencia: Marcel Marín-Villa, Programa de Estudio y Control de Enfermedades Tropicales, PECET, Facultad de Medicina, Universidad de Antioquia, Calle 62 Nº 52-59, laboratorio 632, Medellín, Colombia Teléfono: (574) 219 6507; telefax: (574) 219 6511 mvmarcel@gmail.commvmarcel@gmail.com
1. WHO. Control of the leishmaniases: Report of a meeting of the WHO Expert Committee on the Control of Leishmaniases, Geneva, March 22-26, 2010. WHO technical report series. Geneva: WHO; 2010. p. 1-202. [ Links ]
2. Desjeux P. Leishmaniasis: Current situation and new perspectives. Comp Immunol Microbiol Infect Dis. 2004;27:305-18. http://dx.doi.org/10.1016/j.cimid.2004.03.004 [ Links ]
3. Davies CR, Kaye P, Croft SL, Sundar S. Leishmaniasis: New approaches to disease control. BMJ. 2003;326:377-82. http://dx.doi.org/10.1136/bmj.326.7385.377 [ Links ]
4. Requena JM, Iborra S, Carrion J, Alonso C, Soto M. Recent advances in vaccines for leishmaniasis. Expert Opin Biol Ther. 2004;65:1505-17. http://dx.doi.org/10.1517/14712598.4.9.1505 [ Links ]
5. Rocha RA, Sampaio RN, Guerra M, Magalhaes A, Cuba CC, Barreto AC, et al. Apparent glucantime failure in five patients with mucocutaneous leishmaniasis. Am J Trop Med Hyg. 1980;83:131-9. [ Links ]
6. El-Masum MA, Evans DA. Characterization of Leishmania isolated from patients with Kala-azar and post Kala-azar dermal leishmaniasis in Bangladesh. Trans R Soc Trop Med Hyg. 1995;89:331-2. [ Links ]
7. Bonfante GR, Barroeta S, Mejía de los Alejos MA, Meléndez E, Torrealba J, Valdivia O, et al. Disseminated American cutaneous leishmaniasis. Int J Dermatol. 1996;35:561-5. [ Links ]
8. Costa JM, Marsden PM, Llanos-Cuentas EA, Netto EM, Carvalho EM, Barral A, et al. Disseminated cutaneous leishmaniasis in a field clinic in Bahia, Brazil: A report of eight cases. J Trop Med Hyg. 1986;89:319-23. [ Links ]
9. Ribeiro RN, Marsden PD. Mucosal leishmaniasis unresponsive to glucantime therapy successfully treated with AmBisomeTM. Trans R Soc Trop Med Hyg. 1997;91:77. [ Links ]
10.Vélez I, Agudelo S, Hendrickx E, Puerta J, Grogl M, Modabber F, et al. Inefficacy of allopurinol as monotherapy for Colombian cutaneous leishmaniasis: A randomized, controlled trial. Ann Intern Med. 1997;126:232-6. [ Links ]
11. Palacios R, Osorio LE, Grajalew LF, Ochoa MT. Treatment failure in children in a randomized clinical trial with 10 and 20 days of meglumine antimonate for cutaneous leishmaniasis due to Leishmania Viannia species. Am J Trop Med Hyg. 2001;64:187-93. [ Links ]
12.Rojas R, Valderrama L, Valderrama M, Varona MX, Ouellette M, Saravia NG. Resistance to antimony and treatment failure in human Leishmania (Viannia) infection. J Infect Dis. 2006;193:1375-83. http://dx.doi.org/10.1086/503371 [ Links ]
13.Faraut-Gambarelli F, Piarroux R, Deniau M, Giusiano B, Marty P, Michel G, et al. In vitro and in vivo resistance of Leishmania infantum to meglumine antimoniate: A study of 37 strains collected from patients with visceral leishmaniasis. Antimicrob Agents Chemother. 1997;41:827-30. [ Links ]
14. Lira R, Sundar S, Makharia A, Kenney R, Gam A, Saraiva E, et al. Evidence that the high incidence of treatment failures in Indian Kala-azar is due to the emergence of antimony-resistant strains of Leishmania donovani. J Infect Dis. 1999;180:564-7. http://dx.doi.org/10.1086/314896 [ Links ]
15.Moreira ES, Guerra JB, Petrilló-Peixoto M de L. Glucantime resistant Leishmania promastigotes are sensitive to pentostam. Rev Soc Bras Med Trop. 1992;25:247-50. [ Links ]
16. Grogl M, Thomason TN, Franke ED. Drug resistance in leishmaniasis: Its implication in systemic chemotherapy of cutaneous and mucocutaneous disease. Am J Trop Med Hyg. 1992;47:117-26. [ Links ]
17. Sereno D, Holzmuller P, Mangot I, Cuny G, Ouaissi A, Lemesre J. Antimonial-mediated DNA fragmentation in Leishmania infantum amastigotes. Antimicrob Agents Chemother. 2001;45:2064-9. http://dx.doi.org/10.1128/AAC.45.7.2064-2069.2001 [ Links ]
18. Croft SL, Coombs GH. Leishmaniasis -current chemotherapy and recent advances in the search for novel drugs.Trends Parasitol. 2003;19:502-8. http://dx.doi.org/10.1016/j.pt.2003.09.008 [ Links ]
19. Wyllie S, Cunningham ML, Fairlamb AH. Dual action of antimonial drugs on thiol redox metabolism in the human pathogen Leishmania donovani. J Biol Chem. 2004;279:39925-32. http://dx.doi.org/10.1074/jbc.M405635200 [ Links ]
20. Berman JD, Waddel D, Hanson BD. Biochemical mechanisms of the antileishmanial activity of sodium stibogluconate. Antimicrob Agents Chemother. 1985;27:916-20. [ Links ]
21. Berman JD, Gallalee JV, Best JM. Sodium stibogluconate (Pentostam) inhibition of glucose catabolism via the glycolytic pathway and fatty acid beta-oxidation in Leishmania mexicana amastigotes. Biochem Pharmacol. 1987;36:197-201. http://dx.doi.org/10.1016/0006-2952(87)90689-7 [ Links ] Tovar J, Cunningham ML, Smith AC, Croft SL, Fairlamb AH. Down-regulation of Leishmania donovani trypanothione reductase by heterologous expression of a trans-dominant homologue: Effect on parasite intracellular survival. Proc Natl Acad Sci. 1998;95:5311-6. [ Links ]
23. Croft SL, Sundar S, Fairlamb AH. Drug resistance in leishmaniasis. Clin Microbiol Rev. 2006;19:111-26. http://dx.doi.org/10.1128/CMR.19.1.111-126.2006 [ Links ]
24. Muñoz DL, Cardona DP, Cardona A, Carrillo LM, Quiñones W, Echeverri F, et al. Effect of hydrazones against intracellular amastigotes of Leishmania panamensis and a parasitic cystein protease. Vitae. 2006;12:5-12. [ Links ]
25.Cuervo P, Batista de Jesus J, Junqueira M, Mendonça-Lima L, Gonzalez LJ, Betancourt L, et al. Proteome analysis of Leishmania (Viannia) braziliensis by two-dimensional gel electrophoresis and mass spectrometry. Mol Biochem Parasitol. 2007;154:6-21. http://dx.doi.org/10.1016/j.molbiopara.2007.03.013 [ Links ]
26.Neuhoff V, Arold N, Taube D, Ehrhardt W. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250. Electrophoresis. 1988;9:255-62. http://dx.doi.org/10.1002/elps.1150090603 [ Links ]
27. Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR. Nucleic Acids Research. 2001;29:2002-7. [ Links ]
28. Sundar S. Drug resistance in Indian visceral leishmaniasis. Trop Med Int Health. 2001;6:849-54. http://dx.doi.org/ 10.1046/j.1365-3156.2001.00778.x [ Links ]
29.Grondin K, Roy G, Ouellette M. Formation of extrachromosomal circular amplicons with direct or inverted duplications in drug-resistant Leishmania tarentolae. Mol Cell Biol. 1996;16:3587-95. [ Links ]
30.Haimeur A, Ouellete M. Gene amplification in Leishmania tarentolae selected for resistance to sodium stibogluconate. Antimicrob Agent Chemoter. 1998;42:1689-94. [ Links ]
31.Haimeur A, Brochu C, Genest P, Papadopoulou B, Ouellette M. Amplification of the ABC transporter gene PGPA and increased trypanothione levels in potassium antimonyl tartrate (SbIII) resistant Leishmania tarentolae. Mol Biochem Parasitol. 2000;108:131-5. http://dx.doi.org/10.1016/S0166-6851(00)00187-0 [ Links ]
32.Kumar A, Sisodia B, Misra P, Sundar S, Shasany AK, Dube A. Proteome mapping of overexpressed membrane-enriched and cytosolic proteins in sodium antimony gluconate (SAG) resistant clinical isolate of Leishmania donovani. Br J Clin Pharmacol. 2010;70:609-17. http://dx.doi.org/10.1111/j.1365-2125.2010.03716.x [ Links ]
33.Lee N, Bertholet S, Debrabant A, Muller J, Duncan R, Nakhasi H. Programmed cell death in the unicellular protozoan parasite Leishmania. Cell Death Differ. 2002;9:53-64. http://dx.doi.org/10.1038/sj/cdd/4400952 [ Links ]
34.Sudhandiran G, Shaha C. Antimonial-induced increase in intracellular Ca2+ through non-selective cation channels in the host and the parasite is responsible for apoptosis of intracellular Leishmania donovani amastigotes. J Biol Chem. 2003;278:25120-32. http://dx.doi.org/10.1074/jbc.M301975200 [ Links ]Arnoult D, Akarid K, Grodet A, Petit PX, Estaquier J, Ameisen, JC. On the evolution of programmed cell death: Apoptosis of the unicellular eukaryote Leishmania major involves cysteine proteinase activation and mitochondrion permeabilization. Cell Death Differ. 2002;9:65-81. http://dx.doi.org/10.1038/sj/cdd/4400951 [ Links ]
36. Zangger H, Mottram JC, Fasel N. Cell death in Leishmania induced by stress and differentiation: Programmed cell death or necrosis? Cell Death Diff. 2002;9:1126-39. http://dx.doi.org/10.1038/sj.cdd.4401071 [ Links ]
37. Brochu C, Haimeur A, Ouellette M. The heat shock protein HSP70 and heat shock cognate protein HSC70 contribute to antimony tolerance in the protozoan parasite Leishmania. Cell Stress Chaperones. 2004;9:294-303. http://dx.doi.org/10.1379/CSC-15R1.1 [ Links ]Baptiste V, Benjamin G, Isabelle G, Sundar S, Drummelsmith J, Ouellette M. A proteomics screen implicates HSP83 and a small kinetoplastid calpain-related protein in drug resistance in Leishmania donovani clinical field isolates by modulating drug-induced programmed cell death. Mol Cell Proteomics. 2007;6:88-101. http://dx.doi.org/10.1074/mcp.M600319-MCP200 [ Links ]
39. Wardleworth BN, Russell RJ, Bell SD, Taylor GL, White MF. Structure of Alba: An archaeal chromatin protein modulated by acetylation. EMBO J. 2002;21:4654-62. http://dx.doi.org/10.1093/emboj/cdf465 [ Links ]
40.Bell SD, Botting CH, Wardleworth BN, Jackson SP, White MF. The interaction of Alba, a conserved archaeal chromatin protein, with Sir2 and its regulation by acetylation. Science. 2002;296:148-51. http://dx.doi.org/10.1126/science.1070506 [ Links ]
41.Halliwell B, Gutteridge JM. Free radicals in biology and medicine. Third edition. Oxford: Oxford University Press; 1999. p. 1-899. [ Links ]
42.Cortés-Selva F, Muñoz-Martínez M, Attila I, Jiménez AJ, Varadi A, Gamarro F, et al. Functional expression of a multidrug P-glycoprotein transporter of Leishmania. Biochem Biophys Res Commun. 2005;329:502-7. http://dx.doi.org/10.1016/j.bbrc.2005.02.009 [ Links ]
43.Haile S, Papadopoulou B. Developmental regulation of gene expression in trypanosomatid parasitic protozoa. Curr Opin Microbiol. 2007;10:569-77. http://dx.doi.org/10.1016/j.mib.2007.10.001 [ Links ]
44. Requena JM. Lights and shadows on gene organization and regulation of gene expression in Leishmania. Front Biosci. 2011;17:2069-85. http://dx.doi.org/10.2741/3840 [ Links ]
45.Walker J, Vásquez J, Gómez MA, Drummelsmith J, Burchmore R, Girard I, et al. Identification of developmentally-regulated proteins in Leishmania panamensis by proteome profiling of promastigotes and axenic amastigotes. Mol Biochem Parasitol. 2006;147:64-73. http://dx.doi.org/10.1016/j.molbiopara.2006.01.008 [ Links ]













