Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157
Biomédica vol.33 supl.1 Bogotá Sept. 2013
https://doi.org/10.7705/biomedica.v33i0.1573
ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v33i0.1573
1 Grupo de Investigaciones Biomédicas, Universidad de Sucre, Sincelejo, Colombia
2 Secretaría de Salud de Sucre Dasssalud, Sincelejo, Colombia
Contribución de los autores:
Sandy Milena Caldera, María Cristina Jaramillo y Suljey Cochero: recolección del material biológico, cría e identificación taxonómica, trabajo molecular y análisis de resultados.
Alveiro Pérez-Doria y Eduar Elías Bejarano: asesoría en metodología molecular y análisis de resultados.
Recibido: 17/01/12; aceptado:15/02/13
Introducción. Aedes aegypti es el principal vector del dengue en zonas urbanas. A pesar de su importancia epidemiológica, se desconoce la variabilidad genética de las poblaciones del vector en Colombia.
Objetivo. Determinar la variabilidad genética del gen mitocondrial ND4 , que codifica para la subunidad 4 de la enzima NADH-deshidrogenasa , entre poblaciones de Ae. aegypti de los municipios de Sincelejo y Guaranda, donde se registra alta y baja incidencia de dengue, respectivamente.
Materiales y métodos. A partir del material genético extraído de 36 hembras de Ae. aegypti , se determinó la secuencia parcial del gen mitocondrial ND4 y se estimaron los parámetros de diversidad de nucleótidos, diversidad haplotípica, estructura genética y flujo de genes entre las poblaciones de Sincelejo y Guaranda. También, se analizó la varianza molecular y se construyó una red haplotípica.
Resultados. Se obtuvieron 36 secuencias de nucleótidos de 282 pb; éstas presentaron doce sitios polimórficos y se agruparon en diez haplotipos, dos presentes en ambas poblaciones, tres exclusivos de la población de Sincelejo y cinco de la población de Guaranda. Los estimadores de estructura genética ( F ST =0,15) y de flujo de genes ( Nm =1,40) evidencian diferenciación genética y un limitado intercambio de genes entre las poblaciones.
Conclusión. Las poblaciones de Ae. aegypti de Sincelejo y Guaranda son genéticamente divergentes.
Palabras clave: Aedes , dengue, vectores de enfermedades, genética de población, ADN mitocondrial, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v33i0.1573
Genetic differences between populations of Aedes aegypti from municipalities in northern Colombia, with high and low dengue incidence
Introduction: Aedes aegypti is the principal vector of dengue in urban areas. Despite its epidemiological importance, the genetic variability of Colombian populations of this species is unknown.
Objetive: To determine the genetic variability of mitocondrial gene ND4, which codes for subunit 4 of the enzyme NADH deshydrogenase, between populations of Ae. aegypti from municipalities of Sincelejo and Guaranda. The incidences of dengue reported from these two localities are high and low, respectively.
Materials and methods: Genetic material extracted from 36 females of Ae. aegypti was used to determine the partial sequence of the mitocondrial gene ND4 as well as to estimate the parameters of nucleotidic and haplotypic diversities, genetic structure and gene flow between the Sincelejo and Guaranda populations. The molecular variance was also analysed and a haplotypic network constructed.
Results: In all 36 nucleotide sequences of 282pb were obtained. These presented 12 polymorphic sites and could grouped into 10 haplotypes, two of them present in both populations, three exclusive to the Sincelejo population and five to that of Guaranda. The estimators of genetic structure ( F ST = 0.15) and gene flow ( Nm = 1.40) are both indicative of genetic differentiation and a limited exchange of genes between the populations.
Conclusions: The Sincelejo and Guaranda populations of Ae. aegypti are genetically divergent.
Key words: Aedes , dengue, disease vectors; genetics, population; DNA, mitochondrial; Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v33i0.1573
Aedes ( Stegomyia ) aegypti (Linnaeus, 1762) es un mosquito de hábitos antropofílicos y domésticos que se encuentra ampliamente distribuido en regiones tropicales y subtropicales del mundo. Es uno de los insectos de mayor importancia médico-epidemiológica por el papel que desempeña en la transmisión de algunos arbovirus, incluido el agente etiológico de la fiebre amarilla y los virus causantes de los diferentes cuadros clínicos del dengue, que constituye la enfermedad transmitida por insectos vectores con mayor impacto en salud pública a nivel mundial. La incidencia de esta enfermedad se ha incrementado de forma notoria en la última década, con el registro en América de 50 millones de infecciones anuales por dengue (1).
En Colombia también se ha observado un aumento en la incidencia del dengue en los últimos años, al pasar de 36.732 casos en 2008 a 130.571casos en 2010 (2). Entre los diversos actores del ciclo epidemiológico del dengue que favorecen al aumento en la incidencia de la enfermedad, se destacan los altos índices de infestación por Ae. aegypti registrados en el país , especialmente, en municipios situados por debajo de los 2.000 msnm, considerados como zonas de riesgo (3).
Dentro de estas áreas de riesgo, llama la atención la situación epidemiológica del dengue en los municipios de Sincelejo y Guaranda, del departamento de Sucre, donde a pesar de los altos índices aédicos de infestación, 37,3 % y 35 % respectivamente, se observa un notable contraste en la incidencia de la enfermedad en humanos, mientras que Sincelejo aportó el 54,9 % de los casos de la enfermedad registrados en el año 2009 en el departamento, el municipio de Guaranda registró un bajo número de casos (4).
Las diferencias en el comportamiento de la enfer-medad en estos municipios, podrían atribuirse a diversos factores epidemiológicos y entomológicos tales como variaciones en la estructura genética del vector, aspectos que han sido escasamente estudiados en poblaciones colombianas de Ae. aegypti , donde los estudios entomológicos se han orientado hacia vigilancia de las densidades del vector y de la sensibilidad o resistencia a los insecticidas (5,6).
El conocimiento de la dinámica de la población de Ae. aegypti podría contribuir al diseño de estrategias más efectivas de control vectorial y ayudaría a comprender, además, la historia evolu- tiva de las poblaciones del mosquito, su adaptación ambiental, la propagación de cepas resistentes a los insecticidas, sus nuevos comportamientos ecológicos y la epidemiología de la enfermedad (7,8). Considerando lo anterior, el objetivo principal de este estudio fue determinar la variabilidad genética que exhiben las poblaciones de Ae. aegypti de los municipios de Guaranda y Sincelejo, Sucre.
Materiales y métodos
Recolección de ejemplares de Aedes aegypti
Para este estudio se analizaron dos poblaciones con un número total de 36 hembras de Ae. aegypti, 18 pertenecientes a la población de Sincelejo y 18 a la población de Guaranda (cuadro 1). En Sincelejo, capital del departamento de Sucre (figura 1), los muestreos se realizaron en dos localidades, El Cortijo y Botero, barrios considerados como de alto riesgo para adquirir la enfermedad, según el informe de casos del año 2009 suministrado por la Secretaría de Salud de Sucre (DASSSALUD), y por poseer altos índices de infestación del vector. De igual manera, se recolectaron ejemplares en dos localidades, San Camilo y Calle Tercera, del municipio de Guaranda, localizado al sureste del departamento de Sucre (figura 1), en la subregión La Mojana, donde se ha registrado un bajo número de casos de dengue y los índices de infestación del mosquito son altos (4).
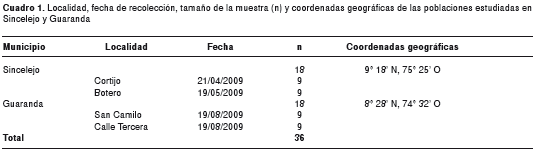
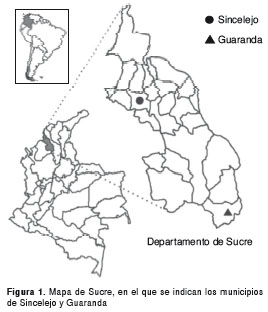
Las condiciones ecológicas de Sincelejo (09°18' N, 75°25' O), subregión sabanas, corresponden a bosque seco tropical, con promedios anuales de temperatura y humedad relativa de 27,5 °C y 80 %, respectivamente, y una precipitación media que oscila entre los 1.000 y 1.200 mm por año. Por sus características climáticas, el municipio de Guaranda (08°28' N, 74°32° O), subregión La Mojana, es clasificado en la zona de vida de bosque húmedo tropical, con promedios anuales de temperatura, humedad relativa y pluviosidad de 28 °C, 85 % y 2.800 mm, respectivamente. La distancia geográfica entre estas dos poblaciones es de 136 km, aproximadamente, estimada con el programa Google Earth 4.2.0198 Beta (9).
En cada municipio se hizo un muestreo de tipo aleatorio, y se inspeccionaron 30 viviendas por localidad, de las cuales, en promedio, nueve fueron positivas para la presencia del mosquito durante la revisión de los depósitos de almacenamiento de agua encontrados en el interior y alrededor de las viviendas. Las larvas de tercer y cuarto estadio de Ae. aegypti se recolectaron con pipetas de succión y se transportaron al Laboratorio de Entomología de DASSSALUD, donde se conservaron en bandejas con agua reposada. Al llegar al estado de pupa se transfirieron a jaulas de cría, en las que tuvo lugar la eclosión de los adultos. Estos fueron examinados bajo un estereomicroscopio con el fin de establecer la especie, mediante la visualización de las características morfológicas utilizadas rutinariamente para la determinación taxonómica (10-12). Los ejemplares fueron conservados a -70 °C hasta su análisis molecular.
Extracción y amplificación del gen mitocondrial ND4
Como marcador molecular se seleccionó el gen mitocondrial ND4 que codifica para la proteína NADH–deshidrogenasa, subunidad cuatro, el cual ha sido útil en estudios de dinámica de poblaciones en mosquitos (13-17). Para extraer el ADN genómico, se modificó el protocolo descrito por Pérez-Doria, et al ., (18), macerando los especímenes con 3 µl de proteinasa K e incubando por tres horas. La reacción en cadena de la polimerasa (PCR) se llevó a cabo con los cebadores ND4F (5'-ATTGCCTAAGGCTCATGTAG-3') y ND4R (5'- TCGGCTTCCTAGTCGTTCAT-3'), siguiendo las condiciones de reacción y el perfil descrito por Bracco, et al ., (13), se ajustaron la concentración del MgCl 2 (5 mM) y la cantidad de cebadores (0,4 µM de cada uno).
El producto de la PCR se sometió a electroforesis en gel de agarosa al 2 %. Además de las muestras, se incluyeron un control positivo, un control negativo y un marcador de peso molecular GeneRuler de 1 kb DNA Ladder (Fermentas). Éstas se visualizaron en un transiluminador de luz ultravioleta Visi-Blue UVP.
Secuencias de nucleótidos
La secuencia de nucleótidos de los fragmentos amplificados se determinó en un secuenciador automático de electroforesis capilar ABI 3730xl y los electroferogramas se editaron con el programa MEGA, versión 4.1 (19). La similitud y posición relativa de las secuencias obtenidas, con relación al genoma mitocondrial de Ae. aegypti , se determinaron con el programa en línea Blast (20). El alineamiento múltiple de las secuencias se efectúo usando el programa CLUSTAL W (21) y, como parte del análisis, se incluyeron secuencias homólogas de Ae. aegypti de Brasil (14,22), México (15), Perú (16), Camerún (23), Kenia (24), Venezuela, Guatemala, Estados Unidos, Guinea, Senegal, Uganda, Singapur, Camboya y Tahití (13). Todas las secuencias de nucleótidos derivadas del presente estudio fueron depositadas en el GenBank con los números de accesos JN896652 al JN896687.
Análisis de variabilidad genética
Para efectos del análisis, los mosquitos recolecta-dos en las localidades de El Cortijo y Botero fueron considerados como pertenecientes a la población de Sincelejo, teniendo en cuenta que estas localidades se encuentran a menos de 500 m de distancia entre sí. Por la misma causa, los especímenes de las localidades de San Camilo y Calle Tercera se consideraron como representantes de la población de Guaranda. El programa DnaSP, versión 4.1 (25), se usó para calcular el número de haplotipos, la frecuencia haplotípica, el número de sitios polimórficos (S) y el número promedio de diferencias de nucleótidos (k), en las dos poblaciones, así como para estimar parámetros de diversidad genética, entre ellos diversidad haplotípica (DH) y diversidad de nucleótidos (p), y para las pruebas de neutralidad D de Tajima (26) y D* y F* de Fu y Li (27), que permiten evaluar si el patrón de polimorfismo observado en las secuencias corresponde con lo esperado bajo la teoría neutral de la evolución molecular.
El programa DnaSP también fue utilizado para establecer los estimadores de estructura genética y flujo de genes con base en el valor estadístico F ST de Wright (28), que mide el nivel de diferenciación genética entre las poblaciones, y el Nm obtenido a partir del F ST que corresponde al número efectivo de migrantes por generación. La red haplotípica fue inferida con el programa TCS, versión 1.2 (29), en el que cada haplotipo se relacionó con otro por cambios en la secuencia de ADN, usando el algoritmo de máxima parsimonia descrito por Templeton, et al ., (30).
Además, se realizó un análisis de la varianza molecular, AMOVA, con el programa GenAlex 6.3 (31), que involucra la varianza en la frecuencia haplotípica pero a su vez considera el número de mutaciones ( q ) entre los haplotipos (32); este programa calcula el valor estadístico phi, que es una versión modificada del F ST de Wright (33). Finalmente, para estimar si las distancias genéticas entre las cuatro localidades se correlacionaban con las distancias geográficas, se hizo una prueba de Mantel con el programa Xlstat, versión 3.9 (34).
Resultados
Sitios polimórficos
Se obtuvieron 36 secuencias de 282 nucleótidos; en el alineamiento múltiple se observaron 270 sitios conservados y 12 sitios variables, y entre estos se encontraron nueve transiciones y tres transversiones. El inicio del fragmento analizado corresponde a la posición 8677 del genoma mitocondrial de Ae. aegypti (número de acceso EU352212.1), mientras que el último nucleótido se sitúa en la posición 8383 del mismo genoma. En las poblaciones naturales de Ae. aegypti de Sincelejo y Guaranda se encontraron 10 haplotipos. En el cuadro 2 se muestran los sitios polimórficos característicos de cada haplotipo, algunos de los sitios corresponden a bases mixtas.

Distribución y frecuencia haplotípica
El haplotipo 1 (H1) fue hallado en Sincelejo y Guaranda con alta frecuencia, equivalente al 55,55 %. El haplotipo 3 (H3) fue el segundo más frecuente, con un porcentaje del 11,11 %, seguido de los haplotipos 2 (H2) y 5 (H5) con 8,33 %, en tanto que cada uno de los demás haplotipos mostró una frecuencia de 2,77 %. Los haplotipos 2, 3 y 4 (H4) fueron exclusivos de la población de Sincelejo. El H4 sólo se halló en la localidad de El Cortijo, mientras que el H5 estuvo presente en tres de las cuatro localidades estudiadas, con excepción de Botero. En la población de Guaranda se encontraron cinco haplotipos únicos, que incluyen el 6 (H6), 7 (H7) y 8 (H8) observados en mosquitos de la localidad de San Camilo, y los haplotipos 9 (H9) y 10 (H10) que corresponden a ejemplares de la localidad Calle Tercera (figura 2).
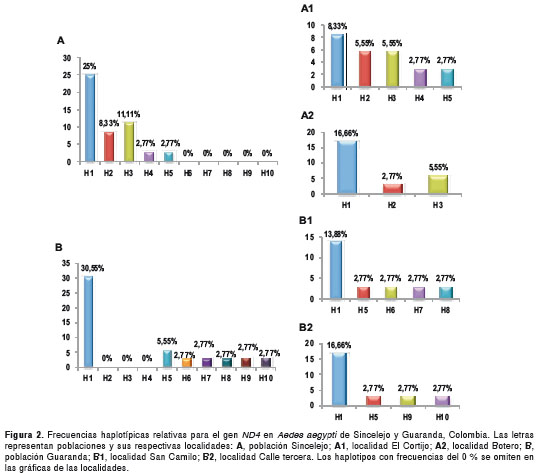
La comparación de los 10 haplotipos con secuencias homólogas de Ae. aegypti disponibles en el GenBank , evidenció que todos los haplotipos encontrados en los municipios de Sincelejo y Guaranda también han sido registrados en otros países. Con relación a la genealogía haplotípica en la que los haplotipos se relacionan entre sí mediante sustituciones o cambios en la secuencia de nucleótidos, el H6 se separó del H4 y del H2 por tres y seis pasos de mutación, respectivamente, mientras que el H7 está correlacionado con el H5 y separado del H3 por un paso de mutación (figura 3).

Diversidad genética y pruebas de neutralidad
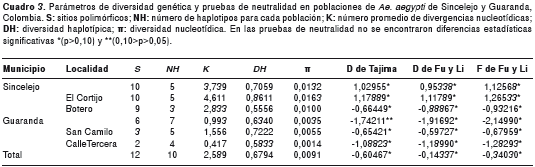
Los índices de diversidad genética y las pruebas de neutralidad, estimados para cada una de las poblaciones estudiadas, se muestran en el cuadro 3. Las pruebas de neutralidad D de Tajima (26), D* y F* de Fu y Li (27), aplicadas dentro y entre las poblaciones de Ae. aegypti de Sincelejo y Guaranda , arrojaron valores sin diferencia estadística significativa (p>0,10) y (0,10>p>0,05), lo cual indica que las mutaciones detectadas en las secuencias de nucleótidos son selectivamente neutrales. En la población de Ae. aegypti de Guaranda todos los valores fueron negativos, mientras que en la de Sincelejo se obtuvieron valores tanto positivos como negativos, lo cual no afecta las inferencias realizadas a partir del gen ND4 por cuanto no fueron significativos (26,35,36).
Varianza molecular y estimadores de flujo de genes
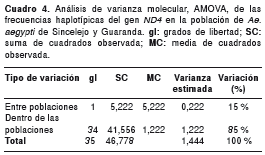
El AMOVA mostró que un gran componente de la variación haplotípica total, equivalente al 85 %, es atribuible a diferencias genéticas dentro de las poblaciones, mientras que el 15 % restante es producto de diferencias entre poblaciones (cuadro 4). El valor de PhiRT=0,154, calculado entre las poblaciones, fue estadísticamente significativo (p>0,015), lo cual evidencia una considerable diferenciación genética entre ellas. Los valores de los estimadores de flujo de genes entre las poblaciones de Ae. aegypti de Sincelejo y Guaranda fueron de 0,15 y 1,40 para F ST y Nm , respectivamente, lo que pone de manifiesto la existencia de diferenciación genética y un limitado flujo de genes entre ambas poblaciones. En el cuadro 5 se muestran los valores pareados de los parámetros estadísticos F ST y Nm entre las cuatro localidades estudiadas. El análisis de aislamiento por distancia calculado con la prueba de Mantel, mostró una correlación positiva (r=0,269) pero carente de significancia estadística (p=0,35), entre la distancia genética y la geográfica.

Discusión
A pesar de los altos índices de infestación por Ae. aegypti y del aumento en la incidencia y la prevalencia del dengue en Colombia, existe un escaso conocimiento de la variabilidad genética de este insecto en el país, sobre el cual se ejercen constantes presiones selectivas con insecticidas (37,38). El presente estudio constituye el primer análisis genético de las poblaciones del vector del dengue en la costa Caribe colombiana. No obstante el limitado tamaño muestral, la variación haplotípica representada en el hallazgo de 10 haplotipos dentro de las 36 muestras estudiadas, es superior a la de estudios previos en los que se incluyó un mayor número de individuos (13,14,39). Por ejemplo, Lima, et al ., (14) analizaron 123 especímenes en Brasil pero sólo encontraron 13 haplotipos mitocondriales.
Según la red haplotípica y teniendo en cuenta la alta frecuencia del H1 en las dos poblaciones, es posible inferir que este fue uno de los primeros haplotipos introducidos y posteriormente disemi-nado en el departamento de Sucre (figura 2). La presencia de este haplotipo en Brasil (14), Perú (16), México (39) y Kenia (24), permite suponer que este linaje geográfico fue introducido al país desde el continente africano, posiblemente a través de Europa cuando se llevaron a cabo las primeras exploraciones y colonizaciones europeas (40). El H5 registrado en muy baja frecuencia en Sincelejo y Guaranda, no se encontró en la localidad de Botero; esta última ha estado sometida a presión constante con insecticidas organofosforados y piretroides, que se han utilizado en la localidad durante brotes epidémicos, y además, en ella se reporta resistencia al temefós (38).
El H3 solo se halló en Sincelejo y fue el segundo haplotipo más abundante, con una frecuencia del 11,11 %. Sin embargo, por la ruta de eventos evolutivos de la genealogía haplotípica (figura 3), se deduce que este haplotipo estuvo presente en Guaranda, considerando que se encuentra relacionado a través de cambios por mutación con el H6 y H7 exclusivos de esta última población. Cabe mencionar que tanto en la población de Sincelejo como en la de Guaranda se encontraron varios haplotipos únicos (figura 2), todos derivados evolutivamente del H1, tal como se aprecia en la genealogía haplotípica (figura 3).
Estos resultados sugieren una pérdida de varia-bilidad haplotípica en la población de Sincelejo al compararse con la de Guaranda, si se asume que provienen de un linaje común. Es necesario tener en cuenta que en Guaranda el control vectorial con insecticidas ha sido escaso o inexistente, lo que explicaría la mayor variabilidad haplotípica observada, sin descartar la posibilidad de que se trate de nuevas cepas de mosquitos introducidas a la región desde otras áreas del país, de los cuales se desconoce su genética de población, o a cambios evolutivos recientes que originaron variantes haplotípicas que se fijaron en la población después de los procesos de colonización y recolonización favorecidos por las inundaciones periódicas a las que está sometida esta subregión (41).
Los resultados del AMOVA, que atribuyen la mayor parte de la variación observada a diferencias dentro de las poblaciones, concuerdan con la diversidad haplotípica hallada en Sincelejo y Guaranda (cuadro 4), y con la presencia de haplotipos exclusivos en cada una de estas poblaciones. La mayor variación dentro de las poblaciones constituye un indicativo de que tanto la población de Guaranda como la de Sincelejo podrían estar subestructuradas, lo cual deberá resolverse en futuros estudios mediante el aumento del tamaño muestral y la inclusión de otras localidades. El bajo porcentaje con el que las diferencias entre las dos poblaciones contribuyen a la varianza molecular, puede deberse a la alta frecuencia del H1 y, en menor proporción del H5, hallados en ambas poblaciones. No obstante, el valor del índice PhiRT (0,154) es significativo (p>0,015), lo que demuestra que las poblaciones de Ae. aegypti de Sincelejo y Guaranda son diferentes entre sí.
Con relación a los estimadores tradicionales de flujo de genes, el F ST de 0,15 indica que las poblaciones están moderadamente diferenciadas, lo que concuerda con los resultados del AMOVA , mientras que el Nm de 1,40 sugiere un intercambio limitado de genes entre éstas. Es oportuno resaltar que el municipio de Sincelejo pertenece a la subregión sabanas, que se caracteriza por sus ambientes secos, con predominio de sequía estacional, mientras que el municipio de Guaranda está ubicado en La Mojana, subregión húmeda sometida a constantes inundaciones. Estas disímiles condiciones ambientales podrían favorecer cambios genéticos en el vector, situación que ha sido documentada en otros países (42).
Otro de los probables factores que podrían propi-ciar el mantenimiento de las diferencias genéticas entre las poblaciones de Ae. aegypti de Sincelejo y Guaranda, es el limitado flujo de personas entre los dos municipios, producto de las difíciles condiciones de acceso geográfico al municipio de Guaranda. Es ampliamente conocido que la migración de este vector en el mundo ha sido pasiva y estrechamente dependiente de la migración humana (43), en particular, de las operaciones comerciales entre territorios y de los medios de transporte empleados (44). Debido a que las precarias vías de acceso terrestre a Guaranda permanecen inutilizables durante parte del año por las inundaciones, un porcentaje importante del transporte se efectúa por vía acuática, con transbordos en diferentes poblados, lo cual haría difícil que, por dispersión pasiva, se mantuviera el intercambio genético entre ambas poblaciones. Por otro lado, la dispersión activa en este caso es improbable por cuanto, tradicionalmente, Ae. aegypti distribuye sus huevos entre varios sitios de oviposición y una dispersión mayor de 100 metros es infrecuente, aunque se han registrado distancias de 580 (45) y 840 metros (46), e incluso se ha demostrado que una hembra grávida puede volar hasta 3 km para encontrar un lugar adecuado donde depositar sus huevos (47).
Además, como se había mencionado, no se puede desestimar el efecto de la presión que ejercen los insecticidas sobre las poblaciones urbanas del vector, lo cual es considerado como un factor selectivo en Ae. aegypti . En un estudio desarrollado por Ocampo y Wesson (37) en la zona urbana de Cali, se señala que las actividades locales de control de vectores podrían constituir una de las principales causas de heterogeneidad en las poblaciones del mosquito, factor que también podría contribuir a las diferencias encontradas entre Sincelejo y Guaranda.
En conclusión, las diferencias observadas en cuanto a la estructura y composición genética de las poblaciones de Ae. aegypti de Sincelejo y Guaranda, podrían estar relacionadas con las condiciones ambientales, la heterogeneidad del hábitat, la presión selectiva ejercida con insecticidas y la limitada migración humana. Se necesitan más estudios para establecer el potencial efecto que las diferencias genéticas entre las poblaciones del vector tendrían en la epidemiología de la enfermedad en la región.
A los residentes de las localidades de El Cortijo y Botero del municipio de Sincelejo, y de San Camilo y Calle Tercera del municipio de Guaranda, departamento de Sucre.
No existe conflicto de interés.
Este trabajo fue realizado con el apoyo logístico de la Secretaría de Salud de Sucre (DASSSALUD), con recursos aportados por el Grupo de Investigaciones Biomédicas de la Universidad de Sucre y del Departamento Administrativo de Ciencia, Tecnología e Innovación – Colciencias (proyecto 112951929257).
Correspondencia: Sandy Caldera, Centro de Diagnóstico Médico, Universidad de Sucre, Carrera 14 N° 16B-32, Sincelejo, Colombia Teléfono: (575) 282 0830; fax: (575) 282 1240 scaldera80@hotmail.com
1. San Martín JL, Brathwaite O, Zambrano B, Solórzano JO, Bouckenooghe A, Dayan G, et al. The epidemiology of dengue in the Americas over the last three decades: A worrisome reality. Am J Trop Med Hyg. 2010;82:128-35. http://dx.doi.org/10.4269/ajtmh.2010.09-0346 [ Links ]
2. Ministerio de la Protección Social, Subdirección de Vigilancia y Control en Salud Pública (SIVIGILA). Boletín de vigilancia epidemia por dengue en Colombia. Boletín 29. Bogotá: Minprotección; 2010. [ Links ]
3. Ministerio de la Protección Social, Subdirección de Vigilancia y Control en Salud Pública (SIVIGILA). Situación de las enfermedades transmisibles objeto de vigilancia intensificada en salud pública. Colombia. Bogotá: Minprotección; 2010. [ Links ]
4. Departamento Administrativo de Seguridad Social en Salud de Sucre (DASSSALUD). Enfermedades transmitidas por vectores ETV. Total índices aédicos en los municipios del departamento de Sucre. Sincelejo: Secretaría de Salud de Sucre; 2009. [ Links ]
5. Ocampo CB , Salazar-Terreros MJ , Mina NJ , McAllister J , Brogdon W. Insecticide resistance status of Aedes aegypti in 10 localities in Colombia. Acta Trop. 2011;118:37-44. http://dx.doi.org/10.1016/j.actatropica.2011.01.007 [ Links ]
6. Fonseca-González I, Quiñones ML, Lenhart A, Brogdon WG. Insecticide resistance status of Aedes aegypti (L.) from Colombia. Pest Manag Sci. 2011;67:430-7. http://dx.doi.org/10.1002/ps.2081 [ Links ]
7. Hiragi C, Simões K, Martins E, Queiroz P, Lima L, Monnerat R. Variabilidade genética em populações de Aedes aegypti (L.) (Diptera: Culicidae) utilizando marcadores de RAPD. Neotrop Entomol. 2009;38:542-47. http://dx.doi.org/10.1590/S1519-566X2009000400018 [ Links ]
8. Yan G, Chadee DD, Severson DW. Evidence for genetic hitchhiking effect associated with insecticide resistance in Aedes aegypti . Genetics. 1998;148:793-800. [ Links ]
9. Google. Google Earth 4.2.0198 Beta. Fecha de consulta: 14 de septiembre del 2009. Disponible en: http://google-earth.brothersoft.com/google-earth4.2.0198-beta [ Links ]
10. Rueda LM. Pictorial keys for the identification of mosquitoes (Diptera: Culicidae) associated with dengue virus transmission. Auckland, New Zealand: Magnolia Press; 2004. p. 60. [ Links ]
11. Chaverri LG. Clave fotográfica para hembras de zancudos (Diptera: Culicidae) presentes en Centroamérica y Panamá. 2003. Fecha de consulta: 18 de marzo de 2009. Disponible en: http://www.inbio.ac.cr/EN/papers/culicidae_hembra/Clave.pdf [ Links ]
12. Carpenter SJ, LaCasse WJ. Mosquitoes of North America. Berkeley: University of California California University Press Fiocruz; 1995. p. 262-5. [ Links ]
13. Bracco JE, Lara CM, Lourenço-de-Oliveira R, Mureb MA. Genetic variability of Aedes aegypti in the Americas using a mitochondrial gene: Evidence of multiple introductions. Mem Inst Oswaldo Cruz. 2007;102:573-80. http://dx.doi.org/10.1590/S0074-02762007005000062 [ Links ]
14. Lima RS, Scarpassa VM. Evidence of two lineages of the dengue vector Aedes aegypti in the Brazilian Amazon, based on mitochondrial DNA ND4 gene sequences. Genet Mol Biol. 2009;32,2:414-22. http://dx.doi.org/10.1590/S1415-47572009005000036 [ Links ]
15. Gorrochotegui-Escalante N, Gómez-Machorro C, Lozano-Fuentes S, Fernandez-Salas I, Muñoz ML, Farfan-Ale JA, et al. Breeding structure of Aedes aegypti populations in Mexico varies by region. Am J Trop Med Hyg. 2002;66:213-22. [ Links ]
16. Costa-da-Silva AL, Capurro ML, Bracco JE. Genetic linajes in yellow fever mosquito Aedes (stegomyia) aegypti (Diptera: Culicidae) from Peru. Mem Inst Oswaldo Cruz. 2005;100:639-44. http://dx.doi.org/10.1590/S0074-02762005000600007 [ Links ]
17. Herrera F, Urdaneta L, Rivero J, Zoghbi N, Ruiz J, Carrasquel G, et al . Population genetic structure of the dengue mosquito Aedes aegypti in Venezuela. Mem Inst Oswaldo Cruz. 2006;101:625-33. http://dx.doi.org/10.1590/S0074-02762006000600008 [ Links ]
18. Pérez-Doria AJ. Caracterización molecular de Lutzomyia tihuiliensis y Lutzomyia pia (Diptera: Psychodidae), potenciales vectores de Leishmania en la región andina (tesis). Sincelejo: Universidad de Sucre; 2005. [ Links ]
19. Kumar S, Tamura K, Nei M. MEGA 4: Integrated software for molecular evolutionary genetics analysis and sequence alignment. Brief Bioinform. 2004;5:150-63. http://dx.doi.org/10.1093/bib/5.2.150 [ Links ]
20. Altschul S, Gish W, Miller W, Myers E, Lipman D. Blast. Basic local alignment search tool. J Mol Biol. 1990;215:403-10. [ Links ]
21. Thompson JD, Higgins DG, Gibson TJ. CLUSTAL W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice. Nucleic Acids Res. 1994;22:4673-80. http://dx.doi.org/10.1093/nar/22.22.4673 [ Links ]
22. Costa CV, Paduan KS, Ribolla EM, Lourenco-de-Oliveira R. Temporal analysis of mitochondrial gene (NDH4) in Aedes aegypti populations from endemic and non-endemic areas in Brazil. Rio de Janeiro: Fiocruz; 2006. [ Links ]
23. Paupy C, Brengues C, Kamgang B, Hervé JP, Fontenille D, Simard F. Gene flow between domestic and sylvan populations of Aedes aegypti (Diptera: Culicidae) in North Cameroon. J Med Entomol. 2008;45:391-400. http://dx.doi.org/10.1603/0022-2585(2008)45[391:GFBDAS]2.0.CO;2 [ Links ]
24. Burugu MW, Sang RC, Kamau LW, Kenya EU. Genetic structure of Aedes aegypti populations in coastal and inland Kenya using mitochondrial DNA. Kenya: Kenya Medical Research Institute; 2008. [ Links ]
25. Rozas J, Sánchez-Del Barrio J, Messeguer X, Rozas R. DnaSP 4.5: DNA polymorphism analyses by the coalescent and other methods. Bioinformatics. 2003;19:2496-7. http://dx.doi.org/10.1093/bioinformatics/btg359 [ Links ]
26. Tajima F. Evolutionary relationship of ADN sequences in finite populations. Genetics. 1983;105:437-60. [ Links ]
27. Fu YX, Li WH. Statistical tests of neutrality mutations. Genetics. 1993;133:693-709. [ Links ]
28. Wright S. The genetical structure of populations. Annals of Eugenics. 1951;15:323-54. http://dx.doi.org/10.1111/j.1469-1809.1949.tb02451.x [ Links ]
29. Clement M, Posada D, Crandall KA. TCS: A computer program to estimate gene genealogies. Mol Ecol. 2000;9:1657-60. http://dx.doi.org/10.1046/j.1365-294x.2000.01020.x [ Links ]
30. Templeton AR, Routman E, Phillips C. Separating population structure from population history: A cladistic analysis of the geographical distribution of mitochondrial DNA haplotypes in the tiger salamander, Ambystoma tigrinum. Genetics. 1995;140:767-82. [ Links ]
31. Peakall R, Smouse P. GENALEX 6.3: Genetic analysis in Excel, population genetic software for teaching and research. Mol Ecol Notes. 2006;6:288-95. http://dx.doi.org/10.1093/bioinformatics/bts460 [ Links ]
32. Excoffier L, Smouse PE, Quattro JM. Analysis of molecular variance inferred from metric distances among DNA haplotypes: Application to human mitochondrial DNA restriction data. Genetics. 1992;131:479-91. [ Links ]
33. Wright S. Evolution and the genetics of populations, Variability within and among natural populations. Chicago: Chicago University Press; 1978. p. 4. [ Links ]
34. Addinsoft. Statistical Software for MS Excel - XLSTAT. 2007. Fecha de consulta: 12 de agosto de 2010. Disponible en: http://www.xlstat.com/ [ Links ]
35. Watterson GA. On the number of segregating sites in genetical models without recombination. Theor Popul Biol. 1975;7:256-76. http://dx.doi.org/10.1016/0040-5809(75)90020-9, [ Links ]
36. Tajima, F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism. Genetics. 1989;123:585-95. [ Links ]
37. Ocampo C, Wesson D. Population dynamics of Aedes aegypti from a dengue hyperendemic urban setting in Colombia. Am J Trop Med Hyg. 2004;71:506-13. [ Links ]
38. Anaya YP. Evaluación de la susceptibilidad a insecticidas en Aedes aegypti capturados en el municipio de Sincelejo, departamento de Sucre, Colombia. (tesis). Sincelejo: Universidad de Sucre; 2008. [ Links ]
39. Gorrochotegui-Escalante N, Muñoz ML, Fernández-Salas I, Beaty BJ. Black WC IV. Genetic isolation by distance among Aedes aegypti populations along the northeastern coast of Mexico. Am J Trop Med Hyg . 2000;62:200-9. [ Links ]
40. Gast-Galvis A. Historia de la fiebre amarilla en Colombia. Historia de los vectores. Santafé de Bogotá: Instituto Nacional de Salud; 1982. p. 95. [ Links ]
41. Caraballo P, De la Ossa J. Inundaciones en La Mojana: ¿Vía crucis social o condición ambiental? Revista Colombiana de Ciencias Animales. 2011;3:198-210. [ Links ]
42. Leiva N, Caceres O. Variabilidad genética de Aedes aegypti en algunas áreas del Perú usando Single Stranded Conformational Polymorphism (SSCP). Rev Perú Med Exp Salud Pública. 2004;21:157-66. [ Links ]
43. Nelson MJ. Aedes aegypti: Biology and ecology. Washington D.C.: Pan American Health Organization; 1986. [ Links ]
44. Scarpassa VM, Bacry CT, Cardoso JR. Population genetics and phylogeography of Aedes aegypti (Diptera: Culicidae) from Brazil. Am J Trop Med Hyg. 2008 ;78 : 895-903. [ Links ]
45. Hausermann W, Fay RW, Hacker CS. Dispersal of genetically marked female Aedes aegypti in Mississippi. Mosq News. 1971;32:37-51. [ Links ]
46. Reiter P, Amador MA, Anderson RA, Clark GG. Short report: Dispersal of Aedes aegypti in an urban area after blood feeding as demonstrated by rubidium-marked eggs. Am J Trop Med Hyg. 1995;52:177-9. [ Links ]
47. Organización Panamericana de la Salud (OPS). Dengue y dengue hemorrágico en las Américas: su prevención y control. Washington, D.C.: OPS; 1995. p. 548. [ Links ]













