Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157
Biomédica vol.34 no.3 Bogotá July/Sept. 2014
https://doi.org/10.7705/biomedica.v34i3.2240
doi: http://dx.doi.org/10.7705/biomedica.v34i3.2240
1 Grupo de Fisiología Molecular, Subdirección de Investigación Científica y Tecnológica, Dirección de Investigación en Salud Pública, Instituto Nacional de Salud, Bogotá, D.C., Colombia
2 Programa de Doctorado Interfacultades en Biotecnología, Universidad Nacional de Colombia, Bogotá, D.C., Colombia
3 Departamento de Ciencias Fisiológicas, Facultad de Medicina, Universidad Nacional de Colombia, Bogotá, D.C., Colombia
Contribución de los autores: María Luz Gunturiz: formulación del proyecto de investigación de tesis de doctorado, diseño experimental, actividades experimentales, análisis de resultados y escritura del artículo
Luis Alberto Gómez: dirección y formulación del proyecto de investigación, diseño experimental, actividades experimentales, análisis, discusión e interpretación de los resultados, escritura y corrección del artículo
Recibido: 14/01/14; aceptado
Introducción. El factor de transcripción asociado a la microftalmia ( Microphtalmia-Associated Transcription Factor , MITF) regula la expresión de genes específicos, pero no se conoce su expresión y su función a nivel cardiaco.
Objetivos. Identificar la expresión del MITF en corazón y en cardiomiocitos aislados de cobayo, describir los cambios morfológicos asociados con su disminución y evaluar los niveles relativos de su expresión en cardiomiocitos aislados en condiciones de preacondicionamiento isquémico.
Materiales y métodos. El análisis de la expresión relativa de la isoforma específica de tejido cardiaco ( heart-type MITF, MITF-H), se determinó mediante reacción en cadena de la polimerasa (PCR) en tiempo real semicuantitativa, secuenciación y Western blot . La disminución del ARNm del MITF se indujo con un ARN pequeño de interferencia ( short hairpin RNA interference , shRNAi) específico. El tamaño, el diámetro y el número de fibras musculares se evaluaron por observación directa con microscopía de luz.
Resultados. Se amplificó un fragmento de 281 pb de ADNc; el análisis de la secuencia confirmó la identidad del exón 1 y la isoforma H del MITF. La interferencia del ARNm del MITF se asoció con un mayor índice cardiaco (peso corazón/peso corporal: 5,46 x 10 -3 Vs. 4,6 x 10 -3 ) y un incremento del diámetro de las fibras cardiacas (50,2±16 µm Vs. 38,7±14,7 µm; p<0,05, n=150). En los cardiomiocitos aislados en condiciones de preacondicionamiento isquémico, se observó una expresión relativa del MITF-H mayor que en los miocitos en normoxia y expuestos a lesión por isquemia simulada (80 y 100 veces más, n=5, p<0,05, n=3).
Conclusión. Los resultados sugieren que el MITF-H podría estar involucrado en la hipertrofia, la respuesta al estrés por isquemia y la supervivencia de cardiomiocitos de cobayo.
Palabras clave: isquemia, preacondicionamiento isquémico, miocitos cardíacos, microftalmia, corazón, expresión génica, hipertrofia.
http://dx.doi.org/10.7705/biomedica.v34i3.2240
Identification and differential expression of the microphthalmia-associated transcription factor in heart and isolated cardiomyocytes from Guinea pigs: Possible role in hypertrophy and viability
Introduction: The microphthalmia -associated transcription factor ( MITF ) regulates the expression of specific genes and its cardiac expression and function is not known.
Objectives: To identify the expression of MITF in hearts and isolated cardiomyocytes from Guinea pigs, to describe morphological changes associated with mRNA interference of MITF and to evaluate their relative changes in expression in isolated cardiomyocytes under ischemic preconditioning.
Materials and methods: The cardiac specific isoform, MITF-H, and relative expression level analysis, was determined by semi-quantitative real time PCR, sequencing and Western blotting. Reduction of mRNA-MITF-H was induced by transduction of specific-MITF-shRNAi interference. The cardiac morphological changes, diameter and number of cardiac fibers were evaluated by direct observation and light microscopy.
Results: A cDNA fragment of 281 bp was amplified from heart and isolated ventricular cardiac myocytes. Sequence analysis confirmed the identity of the isoform MITF-H, exon 1. The MITF silencing was associated with an increase in cardiac index (heart weight/body weight vs . 5.46 x 10 -3 vs 4.6 x 10 -3 ) and higher diameter of cardiac fibers (50.2±16 µ m vs 38,7±14,7 µ m p<0.05, n=150). In isolated cardiac myocytes under ischemic preconditioning we observed a higher relative expression compared with that measured in myocytes exposed to normoxia and simulated ischemia (eighty and one hundred times, p <0.05, n = 5).
Conclusion. The results suggest that MITF-H isoform may be involved in Guinea pig cardiac hypertrophy, response to stress by ischemia and cardiomyocytes survival.
Key words: ischemia, ischemic preconditioning; myocytes, cardiac; microphthalmos, heart, gene expression, hypertrophy .
http://dx.doi.org/10.7705/biomedica.v34i3.2240
Las enfermedades cardiovasculares responden a muchos factores ambientales y genéticos, afectan a personas de todas las edades y tienen una alta tasa de mortalidad en nuestro país, en tanto que a nivel mundial constituyen la primera causa de muerte; además, son muy costosas en términos de discapacidad y de su manejo médico y quirúrgico (1-3). Aunque en los últimos 20 años se ha avanzado en el conocimiento de la biología molecular de las células cardiacas y su potencial implicación en la cardiomiopatía dilatada, la hipertrófica y la isquémica, se conoce muy poco sobre la identidad y la función de las moléculas cardiacas que regulan su estabilidad funcional en condiciones de estrés metabólico y físico (3). Por lo tanto, se requieren investigaciones básicas y aplicadas para entender e intervenir los mecanismos moleculares asociados con la enfermedad cardiaca isquémica, ya que es la responsable de más del 50 % de la morbilidad y la mortalidad de origen cardiovascular (2,3).
Se ha sugerido que la desregulación' de genes estructurales (cadena pesada de la miosina, actina, tropomiosina, complejo troponina y titina, entre otros) reguladores del estrés biomecánico (4) y del metabolismo cardiaco, así como los genes involucrados en diferentes vías de señalización (endotelina 1 (5), angiotensina II, cardiotrofina 1 o factor de crecimiento insulínico de tipo 1, entre otros), pueden influir en la aparición, el desarrollo y la progresión de las enfermedades cardiacas más frecuentes (6,7). Sin embargo, poco se sabe sobre los factores de transcripción implicados en la enfermedad cardiaca isquémica.
El gen del factor de transcripción asociado a microftalmia ( Microphthalmia-Associated Transcription Factor , MITF) ha sido clonado en varias especies, incluidos seres humanos, ratones, ratas, primates, hámsteres, aves, bovinos, équidos, perros, ranas del género Xenopus y peces, entre otros (8-13), pero no se ha identificado en cobayos. En todas estas especies, el MITF muestra una gran homología en la estructura, la cual contiene el dominio bHLHZip, que permite el reconocimiento específico de secuencias conocidas como cajas M y E en las regiones promotoras de sus genes diana, así como dos dominios de activación y una secuencia diana de proteína cinasa asociada a microtúbulos ( Microtubule-Associated Protein Kinase, MAPK) (14,15). Además, las secuencias diana de la glucógeno sintasa 3B y Rsk1 (cinasa ribosómica S6 de 90 kDa) se conservan en todas las especies, excepto en el pez cebra. Por otro lado, se ha demostrado que después de la transcripción y de la traducción (16,17), el MITF puede ser regulado por ARN pequeños y por fosforilación en, al menos, tres sitios, Ser73, Ser298 y Ser409, que modulan su estabilidad, mantenimiento, degradación y actividad (14,15).
El MITF es una proteína de unión a ADN que regula la expresión de genes específicos y ha sido ampliamente estudiado en melanocitos (8). Este factor pertenece a la superfamilia de factores de transcripción con una estructura hélice-bucle-hélice básica y cremallera de leucina (bHLH-Zip), los cuales regulan la expresión de genes específicos involucrados en la supervivencia y el desarrollo normal de los melanocitos, los osteoclastos y otros tipos de células (9-12). No obstante, poco se sabe sobre su expresión a nivel cardiaco.
La expresión del MITF también es regulada por varios promotores alternativos (M, H, A), gene rando diversas isoformas (18-22) con funciones biológicas potencialmente distintas (12,13). De esta manera, por ejemplo, el exón 1M es específico de melanocitos, el 1H se expresa en corazón y otro, el 1A, se expresa en más de un tipo celular. Cada uno de estos exones aparece ampliamente disperso en el ADN genómico. La regulación de la expresión del MITF ha sido estudiada extensamente en melanocitos y en el melanoma, pero se sabe muy poco de su expresión y regulación en otros modelos celulares (18-24). El MITF reconoce las cajas M en los promotores de genes que intervienen en procesos celulares como la apoptosis (Bcl2) (25), la proliferación celular, el ciclo celular (p16, Rb, p21, CDk2) (26-30), la diferenciación (tirosinasa, TRP1, TRP2) (31), etc., poniendo de manifiesto que este factor de transcripción puede tener varias funciones dependiendo del contexto celular y de los modelos experimentales empleados.
Se ha propuesto recientemente la existencia de una relación entre el MITF y el crecimiento y la hipertrofia cardiaca (32,33). En este sentido, Tshori, et al. , reportaron que los ratones con mutación en el gen del MITF mostraban una disminución en la respuesta hipertrófica ante la estimulación beta-adrenérgica, así como una disminución en la función cardiaca y tendencia a la muerte súbita, y concluyeron que el MITF puede jugar un papel importante en la hipertrofia cardiaca inducida por beta-adrenérgicos. Un elemento característico de la hipertrofia es el aumento del tamaño de los miocitos, el cual se desencadena a través de la activación producida por estímulos externos de diferentes señales intracelulares que, actuando sobre el núcleo, favorecen una mayor síntesis de proteínas y producen una reduplicación de sarcómeros, los cuales determinan el incremento del tamaño de la célula
Aunque se sabe que el MITF podría estar invo lucrado en la hipertrofia cardiaca inducida por beta-adrenérgicos en corazones de ratón, no se conoce su nivel de expresión en el corazón de la mayoría de las especies, incluido el cobayo, ni en células cardiacas aisladas en condiciones de lesión por isquemia o de protección cardiaca inducida por el preacondicionamiento isquémico (PCI). La información experimental disponible sugiere que las vías beta-adrenérgicas se acoplan diferencialmente y participan en la señalización que regula la función y la remodelación cardiacas y la viabilidad de los cardiomiocitos, y pueden tener un efecto de protección contra la isquemia (34-41). Sin embargo, no se conocen los factores de transcripción implicados, ni los genes relevantes en la respuesta de las células cardiacas aisladas sometidas a diferentes condiciones de lesión isquémica o de protección contra esta.
Debido a que las evidencias experimentales existentes son insuficientes para asignar un papel al MITF en la regulación y la fisiología cardiacas, y a que no se conoce su función en la patogenia de la enfermedad cardiaca isquémica, el objetivo de este trabajo fue evaluar la expresión del MITF en corazón y cardiomiocitos de cobayo, describir los efectos en el tamaño cardiaco asociados con la interferencia génica y determinar los cambios relativos de su expresión en cardiomiocitos aislados sometidos a condiciones de pérdida y protección de la viabilidad por isquemia y preacondicionamiento isquémico, respectivamente.
Materiales y métodos
Aislamiento de miocitos cardiacos
Se aislaron células ventriculares de corazones de hembras adultas de cobayos Hartley ( Cavia porcellus ) de 250 a 300 g de peso, empleando el sistema de perfusión retrógrada de tipo Langendorff y disociación enzimática con colagenasa, pronasa (Sigma, St Louis, MO, Estados Unidos) y proteinasa K (Invitrogen, Carlsbad, CA, Estados Unidos) descrito por Gómez, et al., (42,43). Después de aislar los cardiomiocitos se hizo el recuento celular mediante el ensayo de exclusión de azul tripano. Luego, las células se separaron en tres grupos experimentales (entre 100.000 y 250.000 células cardiacas ventriculares adultas por grupo experimental para la extracción de ARN) y se sometieron a lesión y protección por preacondicionamiento isquémico, de acuerdo con lo descrito previamente (43,44).
Disminución de la expresión del MITF inducida por ARN pequeños de interferencia
Se hizo la interferencia génica empleando ARN pequeños de interferencia para obtener una secuencia diferente a la del gen MITF ( short hairpin Scramble , shScramble) y para el gen del MITF (shMITFi). Descrita brevemente, esta se realizó con el método hidrodinámico (44) así: los cobayos hembra de 250 a 300 g se inyectaron por vía intraperitoneal con 3 ml de solución salina libre de pirógenos, 2 µl de las respectivas partículas virales, 1 µl de polibreno 10 mg/ml (sc-134220, Santa Cruz Biotechnology Inc., Delaware, CA, Estados Unidos) y 2 µl de partículas lentivirales diseñadas para expresar el ARN de interferencia específico del MITF, shMITFi (sc-35934-V, Santa Cruz Biotechnology Inc., Estados Unidos), o un ARN de interferencia inespecífico, shScramble (sc-108080, Santa Cruz Biotechnology Inc., Delaware, CA, Estados Unidos), y la copGFP (sc-108084, Santa Cruz Biotechnology Inc., Delaware, CA, Estados Unidos), utilizando la tecnología de ARN pequeños como herramienta para inducir la interferencia específica de un gen (44-47). Como control adicional se inyectó un cobayo con 3 ml de solución salina, sin vector de expresión, llamado control sin ARN de interferencia.
Los animales se mantuvieron en jaulas individuales de policarbonato durante 30 días; se les proporcionó alimento (Rodentina ® ) y agua a voluntad. Se les controló diariamente midiendo la temperatura y la frecuencia cardiaca y observando posibles efectos de toxicidad y cambios fenotípicos y de comportamiento. A los 30 días de la transducción, los animales se anestesiaron con una dosis de 70 mg/kg de pentobarbital (anestesia profunda) por vía intraperitoneal y a continuación se hizo la toracotomía y la cardiotomía; se tomaron registros fotográficos de los corazones aislados y se obtuvo el peso de cada uno de ellos. Se estableció la relación entre el peso del corazón y el peso corporal, y cada corazón se fragmentó para la extracción de ARN, ADN y proteínas, para la histopatología, el análisis de fluorescencia (GFP) y la microscopía de luz. La verificación de la transfección y la evaluación de los niveles relativos de expresión de ARNm y de proteínas se realizó mediante PCR semicuantitativa y Western blot , respectivamente. El protocolo para el manejo de los animales fue aprobado por el Comité de Ética del Instituto Nacional de Salud.
Extracción de ARN y síntesis de ADN complementario
Para la extracción de ARN se empleó el protocolo descrito por Chomczynski y Sacchi (48) y el reactivo trizol (Invitrogen, Carlsbad, California, Estados Unidos). La calidad e integridad del ARN obtenido se verificaron mediante espectrofotometría a 260 nm (relación 260/280) y electroforesis en condiciones de desnaturalización en gel de agarosa al 1,5 %. La síntesis de ADN complementario (ADNc) se llevó a cabo a partir de 1 a 3 µ g de ARN total, utilizando oligo-dT y transcriptasa inversa Superscript II (Invitrogen, Carlsbad, California, Estados Unidos). La calidad e integridad del ADNc obtenido se verificó mediante la amplificación del gen de expresión constitutiva GAPDH . Los productos de amplificación se fraccionaron por medio de electroforesis en gel de poliacrilamida y se visualizaron mediante tinción con nitrato de plata.
Identificación de la isoforma H del MITF
Se hizo la caracterización de la isoforma H del MITF mediante la amplificación por PCR de un fragmento del gen que codifica para dicha isoforma en tejido cardiaco. Se emplearon los oligonucleótidos 5´-ATGGAGGCGCTTAGAGTTCAG-´3 y 5´-GTGGCAGAGGGAAGGGTGGTG-´3. Además, se amplificó la región carboxilo terminal (MITF 9-10e) del gen, que es común en varios tipos celulares, como control adicional (5´-CAGGAACTTGAAATGCAGGCTCGA-´3; 5´-GATCAGTGACACCGACGGGAGAA-´3) (no se presentan los datos). El fragmento de amplificación obtenido se clonó en el vector pGEM-T (Promega, Madison, WI, Estados Unidos) y se secuenció empleando el método de Sanger, el sistema Big Dye Terminator, versión 3.1 (Applied Biosystems, Foster City, California, Estados Unidos) y electroforesis capilar con el analizador genético ABI Prism 310 (Perkin Elmer, Estados Unidos). Para verificar la identidad de la isoforma, las secuencias obtenidas se compararon con las secuencias disponibles en las bases de datos de dominio público (GenBank, EMBL) con la plataforma BLAST (49).
PCR semicuantitativa en tiempo real
Se hicieron análisis de expresión semicuantitativa a nivel de ARNm en células cardiacas sometidas a lesión por isquemia simulada y a protección por preacondicionamiento isquémico, así como en células en condición de normoxia. Como control de expresión positiva se empleó ADNc obtenido de corazón completo de cobayo. La expresión de los productos de la transcripción del MITF-H y el GAPDH se evaluó mediante PCR cuantitativa con transcriptasa inversa ( quantitative Reverse Transcriptase PCR, qRT-PCR) en corazones completos sometidos o no a interferencia génica y en células cardiacas sometidas a lesión y protección in vitro. Como controles positivos y estándares de calibración, se emplearon plásmidos recombinantes que contenían el ADNc del MITF previamente caracterizados mediante clonación y secuenciación de ácidos nucleicos con, al menos, seis diluciones. Los ensayos se hicieron por triplicado utilizando el estuche DyNAmo HS SYBR Green (Thermo Scientific, Rockford, Illinois, Estados Unidos) y el equipo Chromo 4 (BioRad Laboratories Inc., Irvine, California, Estados Unidos). Como control de reacción de la PCR, se empleó agua libre de nucleasas en lugar de ácido nucleico. Las condiciones de amplificación fueron: un ciclo a 95 °C durante 15 minutos seguido de 39 ciclos de 10 s a 96 °C, 57 ciclos de 30 s a 2 °C, uno de 30 s a 72 °C, y una extensión final de 10 minutos a 72 °C, y una curva de fusión de 65 a 95 °C con lecturas cada 0,2 °C por segundo.
Para la normalización de los datos y para deter minar la expresión relativa del MITF, se empleó la expresión del gen de expresión constitutiva GAPDH una vez que se demostró que la expresión no cambiaba de manera significativa en las diferentes condiciones experimentales. Además, con el fin de discriminar con suficiente fiabilidad la expresión diferencial, en el análisis semicuantitativo de la PCR se tuvieron en cuenta las eficiencias de la amplificación y se construyó una curva de calibración a partir de cantidades conocidas y crecientes del ADNc del MITF (0,39- 50 ng) que permitió interpolar los resultados obtenidos en las reacciones de amplificación de cada una de las muestras y controles, los cuales también incluyeron diferentes cantidades de ADN complementario, ADNc, (10-100 ng). En todos los casos la fluorescencia se graficó en unidades arbitrarias y se estableció la línea de base (0,02 unidades de fluorescencia).
Los análisis se hicieron con el software Opticon 3 (BioRad Laboratories Inc., Irvine, California, Estados Unidos) y la metodología descrita por Schefe, et al. (50). La curva de calibración permitió interpolar los resultados obtenidos en las reacciones de amplificación para cada una de las muestras y controles, que también incluyeron diferentes cantidades de ADN plasmídico (50-200 ng). Para cada muestra, el software calculó el número de ciclo en el que el lector empezó a detectar un incremento de fluorescencia significativo con respecto a la señal de base.
El ciclo en el que se empezó a detectar el aumento de fluorescencia se estableció como el punto de corte ( crossing point ) o ciclo umbral ( threshold cycle C(t)) que, por efecto de la cinética de amplifi-ación, se esperaba que fuera inversamente proporcional a la concentración inicial del ADNc presente en la muestra.
A fin de evaluar la expresión relativa del ADNc, fue preciso comparar los valores obtenidos después de normalizarlos y controlar el efecto causado por los cambios en las eficiencias de las reacciones de amplificación. Las muestras de ADNc de concentraciones iniciales decrecientes (un orden de magnitud cada vez), se amplificaron mediante PCR en un mismo experimento. Cada cinética permitió determinar un C(t) para cada uno (es decir, un número en referencia a un ciclo concreto). Las concentraciones de ADNc se representaron como el número de copias de ADNc por tubo. La fluorescencia se graficó en unidades arbitrarias y el ruido de fondo se sustrajo. La recta de calibración para los valores medios de los ciclos umbral se definió en una regresión lineal con un coeficiente de correlación (r 2 ) que, para ser considerado un valor de calidad, debía ser de 0,998. No obstante, los errores se representaron según el rango de valores obtenidos para un punto (n=3). Los cálculos de la expresión relativa de los ADNc se determinaron utilizando el método del cálculo de las diferencias en los ciclos umbral y del log 2 (ciclo umbral de tratamiento/de referencia). Para la comparación indirecta, los valores se generaron de la siguiente manera: log 2 (tratamiento/referencia)-log 2 (normoxia/referencia)=log 2 (tratamiento/referencia), n=3, barras DE * diferencia estadísticamente significativa, p=0,05.
Ensayos de Western blot
Las células cardiacas aisladas y sometidas a isquemia experimental y los fragmentos de corazón sometidos a interferencia génica, se lavaron con solución de Tyrode, pH 7,4, se homogeneizaron en una solución de lisis que contenía PBS 1X, Nonidet P-40 1 %, deoxicolato de sodio 0,5 %, SDS 1 %, 2 ug/ml de leupeptina, 2 ug/ml de pestatina y 1 mM de fluoruro de fenilmetilsulfonilo ( phenylmethylsulfonyl fluoride , PMFS) (Sigma, St. Louis, Missouri, Estados Unidos), y se centrifugaron a 13.000 g durante 15 minutos a 4 °C. La cuantificación de proteínas se hizo utilizando el método del ácido bicinconínico (Pierce, Thermo Scientific, Rockford, Illinois, Estados Unidos) con albúmina sérica bovina (BSA), en diferentes cantidades (200-25 µg) para la curva de calibración. De cada grupo experimental se fraccionaron 100 µ g de proteína en SDS-PAGE 10-12 % y se hizo la transferencia capilar a membranas de PVDF (Inmobilon-P IPVH00010, Millipore, Sedford, Massachusetts, Estados Unidos).
Para el bloqueo de sitios inespecíficos, se empleó leche descremada al 5 %, 1 % de albúmina sérica bovina, fracción V, y 0,05 % de Tween 20 (TBST) (Sigma, St. Louis, Missouri, Estados Unidos) en solución tampón TBS ( Tris-Buffered Saline ). Las membranas se incubaron con anticuerpos primarios diluidos (1:1.000) en TBST y albúmina al 1 % por una hora o durante la noche a temperatura ambiente y en agitación y, luego, con anticuerpos secundarios acoplados a peroxidasa diluidos igualmente en TBST más albúmina al 1 % a temperatura ambiente y en agitación durante una hora
Para la detección del MITF se utilizó como anticuerpo primario el anticuerpo policlonal anti-MITF (N-15), (sc-10999, Santa Cruz Biotechnology Inc., Estados Unidos), cuyo epítopo de reconocimiento es la región amino de la proteína MITF humana. Para la detección de lámina beta, se empleó el anticuerpo policlonal anti-Lamin ß (M-20), (sc-6217, Santa Cruz Biotechnology Inc., Estados Unidos) y, como anticuerpo secundario, un anti-IgG de cabra (HRP) ( Anti-IgG Goat-HRP , PI-9500, Vector Laboratories Inc., Burlingame, California, Estados Unidos). La detección se hizo con un sustrato quimioluminiscente ( ECL + plus Western blotting detection system RPN 2132, GE Healthcare, Buckinghamshire, Inglaterra) y, los análisis densitométricos, con el programa Kodak 1D 3.5 (Eastman, Kodak, Inglaterra).
Resultados
Identificación y análisis de secuencia del ADNc que codifica para el MITF
A partir de miocitos cardiacos aislados y de corazón completo de cobayos, se extrajo el ARN y se sintetizó el ADNc. Se estandarizó la PCR para la amplificación de un fragmento de 281 pb de la isoforma H del gen MITF , el cual se clonó y se secuenció (figura 1A). A partir del análisis de la secuencia de los fragmentos de ADNc obtenidos, se observó que la isoforma H de cobayo (número de acceso al GenBank: JF_309109.1) fue diferente a las secuencias reportadas para los genes descritos en humano (número de acceso al Gen Bank: NP_937820.1), en rata (número de acceso al GenBank: XM_001065584) y en ratón (número de acceso al GenBank: NM_001113198), con 6/281 cambios en los nucleótidos, como se muestra en las figura 1B y figura 1C.
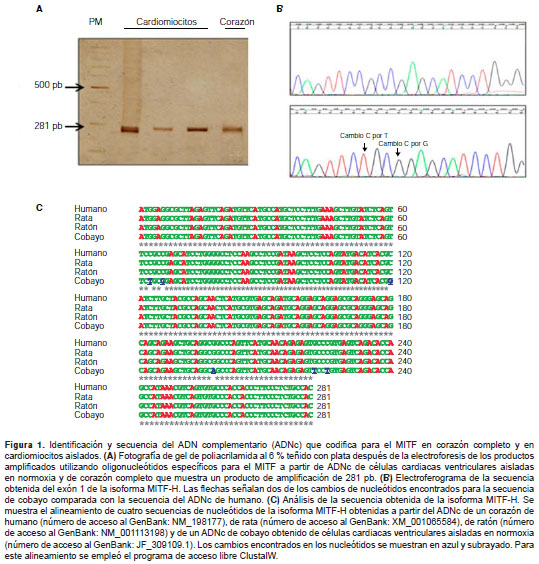
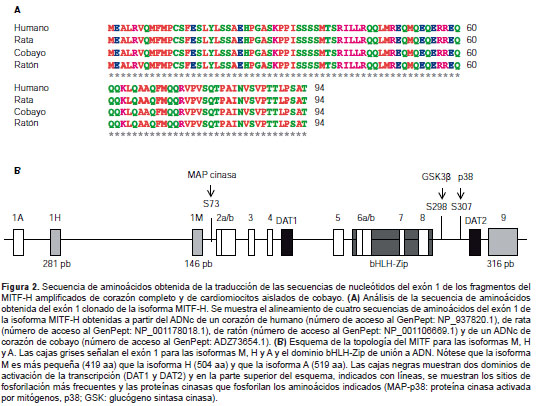
Estos cambios no implicaron diferencias en la secuencia de aminoácidos, lo cual sugiere una alta conservación de la secuencia de la proteína entre estas especies (figura 2A). Se comparó la topología de dominios del MITF entre las isoformas M, H y A, y, como se muestra en el esquema (figura 2B), la principal diferencia se encontró en el exón 1 para las isoformas mencionadas, las cuales están identificadas por cajas grises. La isoforma M es más pequeña (419 aa) que la isoforma H (504 aa) y que la isoforma A (519 aa). Como ya se ha reportado en otros trabajos, se conservaron los dominios bHLH-Zip de unión a ADN y dos dominios de activación de la transcripción (DAT1 y DAT2) (figura 2B, cajas negras). También se conservaron los sitios más frecuentes de fosforilación por las proteínas cinasas que fosforilan los aminoácidos indicados (MAP-p38: proteína cinasa activada por mitógenos, p38; GSK: glucógeno sintasa cinasa) (figura 2B, parte superior del esquema indicado con líneas verticales).
Disminución del ARNm y de la proteína MITF inducida por ARN pequeños de interferencia específicos para el MITF y cambios morfológicos cardiacos
Una vez identificada la expresión de la isoforma MITF-H en corazón y en los cardiomiocitos aislados, se examinaron los efectos de la disminución de la expresión del MITF inducida en el corazón por medio de la transducción de lentivirus que expresan el ARN pequeño de interferencia específico para el MITF (shRNAi-MITF); como controles se utilizaron un ARN pequeño inespecífico (shRNAi-Scramble) y solución salina sin ARN de interferencia. El efecto de la transducción con el shRNAi específico del MITF en la disminución de su expresión, se determinó examinando los niveles de expresión relativos del ARNm del MITF y comparándolos con los niveles de expresión de ARNm del GAPDH por medio de la qRT-PCR y de la proteína nuclear de lámina B, utilizando Western blot .
Los ensayos de qRT-PCR mostraron la amplificación diferencial de un producto de 281 pb para el MITF-H (figura 3A, parte superior) y de 415 pb para el GAPDH (figura 3A, parte inferior). El control positivo correspondió a la amplificación del MITF-H y del GAPDH clonados en el plásmido pGEM-T y los controles negativos correspondieron a la misma mezcla de reacción pero sin ADN complementario.
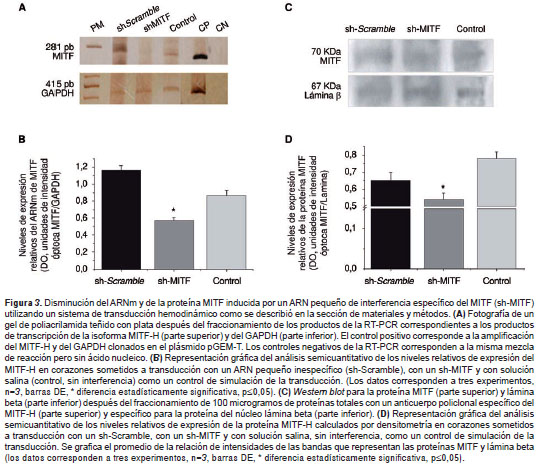
A partir del promedio de intensidades para cada banda amplificada (unidades de densidad óptica, DO), el nivel relativo de expresión del ARNm del MITF normalizado con la expresión del GAPDH (MITF/GAPDH) resultó en unos niveles relativos de expresión de 1,6±0,06 en el tejido sometido a transducción con un ARN pequeño de interferencia inespecífico (sh-Scramble): 0,57±0,03 en el tejido sometido a transducción con un ARN pequeño de interferencia específico para el MITF y 0,86±0,07 en el tejido sin ARN pequeño de interferencia. Estos resultados indicaron una reducción relativa en la expresión del producto de la transcripción del MITF de 2 y 1,5 veces, aproximadamente, en la expresión del transcrito del MITF-H en corazón sometido a transducción con el ARN pequeño de interferencia específico del MITF comparada con la del control sin interferencia y la del sometido a transducción con el ARN de interferencia no específico (shScramble), respectivamente (figura 3B) . Estas diferencias fueron estadísticamente significativas (n=3, p=0,05).
Los ensayos de Western blot con un anticuerpo específico para la proteína MITF, detectados por quimioluminiscencia, mostraron una banda de aproximadamente 70 kDa (figura 3C, parte superior), mientras que los ensayos de Western blot con un anticuerpo específico para la proteína lámina beta mostraron una banda de 66 kDa, aproximadamente (figura 3C, parte inferior).
De forma similar a los cambios detectados en los productos de la transcripción, el ensayo resultó en unos niveles relativos de expresión de la proteína MITF de 0,65±0,05 en el tejido sometido a transducción con un ARN pequeño de interferencia inespecífico (sh-Scramble), de 0,54±0,04 en el tejido sometido a transducción con un ARN pequeño de interferencia específico para el MITF (sh-MITF) y de 0,78±0,04 en el tejido sin ARN de interferencia. Estos resultados indicaron una reducción relativa de la expresión de la proteína MITF de aproximadamente 17 y 31 % en la expresión del producto de la transcripción del MITF-H en corazón sometido a transducción con el sh-MITF con respecto al control sin inter ferencia y al producto de la transducción con el shScramble, respectivamente (figura 3C y figura 3D). Dichas diferencias fueron estadísticamente significativas (n=3, p=0,05). Estos resultados se validaron con la expresión de las proteínas a-tubulina, b -actina y a-actina sarcomérica como controles de normalización (datos no mostrados).
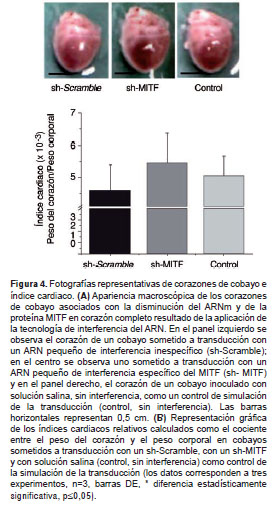
Para observar los efectos asociados a la disminución de la expresión del MITF en corazón, se calcularon los índices cardiacos y se hizo un análisis histopatológico. Macroscópicamente se observó que el corazón sometido a transducción con el sh-MITF era de mayor tamaño y peso aparentes que los otros dos corazones (2,2±0,02 g Vs. 2,0±0,01 g) (figura 4). Concordando con estos incrementos, en la figura 4D se presentan gráficamente los índices cardiacos relativos calculados como el cociente entre el peso del corazón y el peso corporal en cobayos sometidos a transducción con un sh-Scramble, con un sh-MITF y con solución salina (control, sin ARN de interferencia) como control de la simulación de la transducción. Se observó un mayor índice cardiaco en los cobayos tratados con el sh-MITF (5,46 x 10 -3 ) comparado con el índice cardiaco de los corazones sometidos a transducción con el sh-Scramble (4,6 x 10 -3 ) y con el control sin interferencia (5,06 x 10 -3 ). Estas diferencias fueron estadísticamente significativas (n=3, p=0,05) (figura 4A y B).
En los cortes histológicos se cuantificaron el diámetro y el número promedio de las fibras musculares de los corazones a los que se les indujo a la disminución del ARNm y de la proteína MITF ( knockdown ) por medio de la tecnología de interferencia del ARN, así como de aquellos a los que no se les indujo dicha disminución. En el panel superior de la figura 4 se presentan tres microfotografías representativas de los cortes transversales de los corazones de cobayo teñidos con azul de toluidina después de haber sido sometidos a transducción con el sh-Scramble (figura 5A), de los sometidos a transducción con un sh-MITF (figura 5B), y de los inoculados con solución salina y sin interferencia utilizados como control de simulación de la transducción (figura 5C). Se observó que las fibras musculares que fueron interferidas para el MITF eran de mayor tamaño y con mayores espacios intercelulares (figura 4B, indicado con flechas) que las observadas en los otros dos cortes (figura 5A y C).
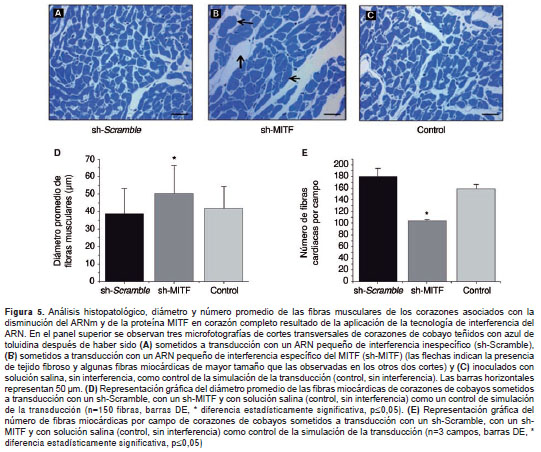
Para confirmar el aparente efecto en el crecimiento del corazón asociado con la disminución de la expresión del MITF, se midió el diámetro y se contó el número de fibras cardiacas de los corazones en las diferentes condiciones experimentales. En la figura 5D se muestra la representación gráfica del diámetro promedio de las fibras miocárdicas de corazones de cobayos sometidos a transducción con el sh-Scramble (38,7±14,7 µm), con un sh-MITF (50,2±16,1 µm) y con solución salina (control, sin interferencia) como control de la simulación de la transducción (41,6±12,8 µm). La comparación de los diámetros máximos y mínimos de algunas fibras cardiacas también reveló un aumento relativo en las fibras musculares tratadas con el sh-MITF con respecto a los diám etros máximos y mínimos de las fibras tratadas con el sh-Scramble y las del control sin interferencia (mínimos: 22,9±3,5 Vs. 18,5±4,1 Vs . 20,9±3,2 µm; y máximos: 101,6±11,8 Vs. 88,77±13,7 Vs. 85,9±13,8 µm, respectivamente). Estos resultados indicaron que hubo un incre mento estadísticamente significativo (n=150 fibras analizadas, p=0,05) en el diámetro de los miocitos que tenían menor expresión de MITF.
Es interesante señalar que cuando se cuantificó el número de fibras miocárdicas de los cortes histológicos de los corazones de cobayos sometidos a transducción con un sh-Scramble, con un sh-MITF y con solución salina (control, sin interferencia) como control de la simulación de la transducción, se observó que había 178±16, 102±4 y 156±4 fibras por campo microscópico, respectivamente (n=3 cortes analizados, p=0,05) (figura 5F). Este resultado sugiere cambios en el número de células asociado con los diferentes niveles de expresión del MITF.
Niveles relativos de expresión del MITF en cardiomiocitos aislados sometidos a diferentes condiciones de supervivencia
Para establecer una posible asociación entre la expresión del MITF y la supervivencia de las células cardiacas, se examinó la expresión del MITF en cardiomiocitos ventriculares adultos aislados que fueron sometidos a condiciones de pérdida y protección de la viabilidad por isquemia y preacondicionamiento isquémico, respectivamente, según el protocolo presentado en las figura 6A, B y C (41-44). Las células cardiacas aisladas en condiciones de normoxia (21 % de oxígeno) (figura 6A y D), presentaron morfología de bastón con la presencia de sarcómeras morfológicamente bien definidas (en más del 90 % de las células), que son características propias de los cardiomiocitos viables.
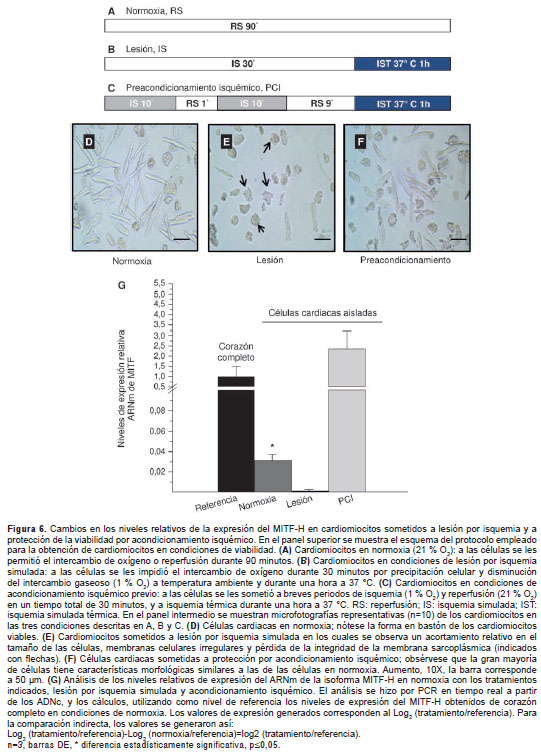
Por el contrario, en los cardiomiocitos sometidos a lesión por isquemia simulada (1 % de oxígeno) (figura 6B y E), se observó acortamiento (hipercontractura') en el tamaño de las células, membranas celulares irregulares, pérdida de la integridad de las sarcómeras y disminución de la viabilidad celular entre 30 y 50 %, comparados con el grupo en condiciones de normoxia. Estas características indicaron que la exposición de las células cardiacas aisladas a estas condiciones de isquemia simulada (figura 6B) resultó en lesión celular (figura 6E, flechas).
En cuanto al grupo de células cardiacas sometidas a protección por preacondicionamiento isquémico (figura 6C y F), se observó que la gran mayoría de ellas (entre 65 y 75 %) tenía características morfológicas similares a las de las células en condiciones de normoxia (figura 6F), lo que confirmó que el protocolo empleado para inducir lesión y protección, evaluado por las características de supervivencia celular en respuesta a la isquemia experimental, fue efectivo y es reproducible (42-44).
Se extrajo el ARN y se sintetizó el ADNc de cada grupo celular experimental descrito arriba, y mediante PCR en tiempo real se evaluó el nivel de expresión del ARNm para el MITF-H empleando el GAPDH como gen de normalización. El análisis se hizo por PCR en tiempo real a partir de los ADNc; para la comparación de los niveles relativos de expresión, se tomaron los valores de los ciclos umbral medidos en función de la cantidad de ADNc inicial de todas las réplicas por cada grupo experimental amplificadas mediante PCR en tiempo real en un mismo experimento y considerando las diferencias en las eficiencias de amplificación. Los cálculos se hicieron mediante una curva de calibración estándar (con R 2 mayor de 0,998 para todos los experimentos) y mediante el análisis ?? C(t), utilizando como valor de referencia el nivel de expresión del MITF-H del ADNc obtenido del corazón completo (1,0±0,5)(figura 6G), de manera similar a como se obtuvieron los resultados graficados en las figura 3A y B.
Mediante estos ensayos se evidenció una expresión diferencial de la isoforma H del MITF en cardiomiocitos ventriculares aislados en condiciones de isquemia experimental (lesión por isquemia simulada) (0,01±0,001), de normoxia (0,03±0,006) y de preacondicionamiento isquémico (2,3±0,9) (figura 6G). Este resultado sugiere que los niveles relativos de expresión del MITF se incrementan de manera significativa (entre 2 y 100 veces, n=3, p=0,05) en cardiomiocitos aislados y sometidos a condiciones de protección contra la isquemia por acondicionamiento inducido durante periodos breves de isquemia y reoxigenación, de acuerdo con el protocolo de la figura 6C (42-44).
Discusión
El factor de transcripción MITF juega un papel esencial en el desarrollo y en la función de varios tipos de células, incluidos los melanocitos, los mastocitos y los osteoclastos, entre otros . S e han descrito varias isoformas del MITF, pero la relevancia de esta diversidad y sus funciones biológicas aún no se han dilucidado completamente (8,9,23). Además, muy poco se sabe sobre su expresión y su función en el corazón y, en especial, en las células cardiacas.
En este trabajo se logró amplificar e identificar el exón 1 de la isoforma del MITF-H en corazón y cardiomiocitos aislados de cobayo. Se describieron algunos cambios morfológicos cardiacos aso ciados con el knockdown inducido por ARN de interferencia específico del MITF y se analizó la expresión del MITF-H en cardiomiocitos sometidos a condiciones de lesión y preacondicionamiento isquémico. La amplificación específica y el análisis de la secuencia de nucleótidos obtenida, demostraron que la isoforma H del MITF de corazón y células cardiacas aisladas de cobayo es diferente a las reportadas para humano, rata y ratón, siendo en el momento del envío al GenBank la primera secuencia parcial de este gen reportada para cobayo ( número de acceso al GenBank: JF_309109.1) (figura 1 y figura 2). Por lo tanto, la identificación y la expresión del MITF-H en corazón completo y en los cardiomiocitos aislados, que son una preparación libre de otros tipos de células (42-44), permiten deducir que la isoforma H del MITF se expresa de manera específica en estas células cardiacas.
Poco se sabe de la función del MITF en el tejido cardiaco. En este trabajo se encontró experimentalmente disminución relativa de la expresión del MITF inducida por un ARN de interferencia específico en el corazón de cobayos durante un periodo de 30 días, lo que se asoció con una diferencia significativa en los niveles relativos de expresión, principalmente del producto de la transcripción del MITF y de su proteína (figura 3). Esto sugeriría que en tejido de corazón, la regulación del MITF es mayor después de la transcripción que antes de esta. Además, estos cambios se asociaron con un incremento en el tamaño y el peso (figura 4A y B) y en el diámetro de las fibras cardiacas, así como en una reducción relativa del número de fibras cardiacas (figura 5D), lo que sugiere que el MITF podría estar involucrado en la regulación del crecimiento cardiaco y, especialmente, en la hipertrofia cardiaca. Estos resultados coinciden con los resultados del trabajo publicado por Tshori, et al., en el 2006 (32). Por lo tanto, los hallazgos experimentales disponibles sugieren, en conjunto, que el MITF-H está involucrado directa o indirectamente en la regulación del crecimiento en tamaño de las células cardiacas.
El c recimiento hipertrófico es indicativo de muchas alteraciones cardiacas, incluida la enfermedad isquémica, la hipertensión arterial, la insuficiencia cardíaca y la enfermedad valvular, entre otras (2,3) . A nivel celular, la hipertrofia de los cardiomiocitos se caracteriza por un incremento en el tamaño de la célula, por aumento de la síntesis de proteínas y por la reorganización de las sarcómeras; a nivel molecular, estos cambios en el fenotipo celular se acompañan de alteración en la expresión de los genes estructurales y los factores de transcripción que regulan las vías de señalización encargadas de la supervivencia y la viabilidad de las células cardiacas (40-44). Sin embargo, y a pesar de que existe suficiente evidencia clínica, el conocimiento sobre los eventos moleculares que desencadenan la hipertrofia cardiaca aún es muy incipiente.
Aunque el mecanismo celular y molecular del control del crecimiento y de la hipertrofia cardiaca asociado a la mutación del MITF en corazones de ratón (32) y a la disminución de su expresión en corazones de cobayo no se conoce, es posible que tal mecanismo sea diferente según el tipo de modelo experimental. Los análisis de expresión del MITF-H empleando los ADNc obtenidos de células sometidas a lesión por isquemia simulada, muestran que la expresión del ARNm del MITF-H disminuye con respecto al grupo de células sometidas a protección por preacondicionamiento isquémico (figura 6), lo que indica que el MITF puede estar implicado en la regulación de la viabilidad en respuesta a la isquemia inducida experimentalmente. Este resultado permite sugerir que, en el caso del modelo presentado en este trabajo, la hipertrofia se podría presentar asociada a una respuesta adaptativa alterada como consecuencia de la pérdida del número de cardiomiocitos y de la masa contráctil del miocardio, mientras que, en el caso de la hipertrofia cardiaca en el modelo de ratón (32), ésta podría estar asociada a una respuesta adaptativa alterada frente a la presión arterial inducida.
Considerando que la regulación funcional de la transcripción es fundamental para el desarrollo y la función del corazón, un mejor entendimiento del mecanismo de regulación y de los genes diana del factor de transcripción MITF en tejido cardiaco es necesario para avanzar en el conocimiento de la patogenia de las enfermedades cardiovasculares asociadas a la hipertrofia cardiaca más frecuentes, como la hipertensión arterial, la insuficiencia cardiaca y el infarto agudo de miocardio, entre otras.
En concordancia con esta propuesta derivada de los resultados experimentales, existen datos sobre los cambios en los perfiles de expresión génica de los ventrículos derecho e izquierdo de corazones normales y con insuficiencia cardiaca, la cual muestra que en el ventrículo izquierdo de corazones con insuficiencia cardiaca los genes implicados en la producción de energía están regulados nega tivamente y se encuentran en la contracción cardíaca, incluida la regulación de la longitud de los filamentos de actina, así como en la modulación del acoplamiento de excitación-contracción. Además, en corazones con insuficiencia cardiaca, se encuentra que los factores de transcripción fetales, como el MEF2 y el factor de respuesta al estrés, ATF4, presentan diferencias de expresión interventricular (51). Es importante resaltar que en nuestro estudio las células cardiacas aisladas se obtuvieron de cardiomiocitos ventriculares.
En conclusión, los resultados obtenidos en este estudio permiten sugerir que la isoforma H del MITF se expresa en cardiomiocitos aislados de cobayo y en corazón completo. Además, la expresión diferencial del MITF-H a nivel de ARNm y de la proteína en cardiomiocitos sometidos a condiciones de lesión y protección, sugiere que el MITF podría regular la expresión de genes relacionados con el crecimiento y la viabilidad de las células cardiacas de manera directa o indirecta . En consecuencia, la expresión y la actividad de esta isoforma pueden ser importantes en la regulación de la supervivencia de las células cardiacas, en la respuesta al estrés por isquemia y en condiciones asociadas con la hipertrofia cardiaca, ya sea por regulación específica de la isoforma H o por la regulación de genes diana del MITF-H involucrados en estas alteraciones cardiovasculares. Por último, se requieren investigaciones adicionales para esclarecer el papel desempeñado por el MITF-H en la fisiología del corazón y de la célula cardiaca, y en las enfermedades que lo pueden afectar.
A Jorge Rivera, del Grupo de Morfología Celular de la Subdirección de Investigación Científica y Tecnológica, Dirección de Investigación en Salud Pública, Instituto Nacional de Salud, por los cortes histológicos efectuados, así como al Instituto Nacional de Salud por el apoyo al trabajo de tesis de doctorado de María Luz Gunturiz.
Los autores declaran no tener conflicto de intereses.
Esta investigación, incluido el trabajo de tesis de doctorado de María Luz Gunturiz, fue financiada con recursos del Instituto Nacional de Salud.
Correspondencia: Luis Alberto Gómez, Grupo de Fisiología Molecular, Subdirección de Investigación Científica y Tecnológica, Dirección de Investigación en Salud Pública, Instituto Nacional de Salud, Avenida Calle 26 N° 51-20, Bogotá, D.C., Colombia Teléfono: 220 7700, extensiones 1483, 1419 y 1416 lgomez@ins.gov.co
1. Lim SS, Gaziano TA, Gakidou E, Reddy KS, Farzadfar F, Lozano R, et al. Prevention of cardiovascular disease in high-risk individuals in low-income and middle-income countries: Health effects and costs. Lancet. 2007;370:2054-62. http://dx.doi.org/10.1016/S0140-6736(07)61699-7 [ Links ]
2. Gómez LA. Enfermedades cardiovasculares: un problema de salud pública y un reto global. Biomédica. 2011;31:469-73. http://dx.doi:10.1590/S0120-41572011000400001 [ Links ]
3. Komuro I. Molecular mechanism of cardiac hypertrophy and development. Jpn Circ J. 2001;65:353-8. http://dx.doi.org/10.1253/jcj.65.353 [ Links ]
4. Alekseev AE, Gómez LA, Aleksandrova LA, Brady PA, Terzic A. Opening of cardiac sarcolemmal KATP channels by dinitrophenol separate from metabolic inhibition. J Membr Biol. 1997;157:203-14. http://dx.doi.org/10.1007/s002329900229 [ Links ]
5. Imokawa G, Yada Y, Mitsutoshi K. Signaling mechanisms of endothelin induced mitogenesis and melanogenesis in human melanocytes. Biochem J. 1996;314:305-12. [ Links ]
6. Díez J, López B, González A, Ardanaz N, Fortuño MA. Respuestas del miocardio al estrés biomecánico. Rev Esp Cardiol. 2001;54:507-15. [ Links ]
7. Cooper G 4th. Cardiocyte cytoskeleton in hypertrophied myocardium. Heart Failure Rev. 2000;5:187-201. http://dx.doi.org/10.1023/A:1009836918377 [ Links ]
8. Steingrimsson E. All for one, one for all: Alternative promoters and MITF. Pigment Cell Melanoma Res. 2008;21:412-4. http://dx.doi.org/10.1111/j.1755-148X.2008.00473.x [ Links ]
9. Shibahara S, Takeda K, Yasumoto K, Udono T, Watanabe K, Saito H, et al . Microphthalmia-associated transcription factor (MITF) : Multiplicity in structure, function, and regulation. J Investig Dermatol Symp Proc . 2001;6:99-104. http://dx.doi.org/10.1046/j.0022-202x.2001.00010.x [ Links ]
10. Hallsson JH, Favor J, Hodgkinson C, Glaser T, Lamoreux ML, Magnúsdóttir R, et al . Genomic, transcriptional and mutational analysis of the mouse microphthalmia locus. Genetics . 2000;155:291-300. [ Links ]
11. Saito H, Takeda K, Yasumoto K, Ohtani H, Watanabe K, Takahashi K, et al . Germ cell-specific expression of microphthalmia-associated transcription factor mRNA in mouse testis. J Biochem . 2003;134:143-50. http://dx.doi.org/10.1093/jb/mvg122 [ Links ]
12. Kumasaka M, Sato H, Sato S, Yajima I, Yamamoto H. Isolation and developmental expression of MITF in Xenopus laevis . Dev Dyn . 2004;230:107-13. http://dx.doi.org/10.1002/dvdy.20019 [ Links ]
13. Tsuchida S, Takizawa T, Abe K, Okamoto M, Tagawa M. Identification of microphthalmia-associated transcription factor isoforms in dogs. Vet J . 2009;182:283-93. http://dx. doi.org/10.1016/j.tvjl.2008.06.004 [ Links ]
14. Mansky KC, Sankar U, Han J, Ostrowski MC. Microphthalmia transcription factor is a target of the p38 MAPK pathway in response to receptor activator of NF-kappa B ligand signaling. J Biol Chem . 2002;277:11077-83. http://dx.doi.org/10.1074/jbc.M111696200 [ Links ]
15. Shibahara S, Yasumoto K, Amae S, Udono T, Watanabe K, Saito H, et al . Regulation of pigment cell-specific gene expression by MITF. Pigment Cell Res. 2000;13:98-102. http://dx.doi.org/10.1034/j.1600-0749.13.s8.18.x [ Links ]
16. Bemis LT, Chen R, Amato CM, Classen EH, Robinson SE, Coffey D, et al . MicroRNA-137 targets microphthalmia- associated transcription factor in melanoma cell lines. Cancer Res. 2008;68:1362-8. http://dx.doi.org/10.1158/0008-5472.CAN-07-2912 [ Links ]
17. Goswami S, Tarapore RS, Teslaa JJ, Grinblat Y, Setaluri V, Spiegelman VS. MicroRNA-340-mediated degradation of microphthalmia-associated transcription factor mRNA is inhibited by the coding region determinant-binding protein. J Biol Chem . 2010;285:20532-40. http://dx.doi.org/10.1074/jbc.M110.109298 [ Links ]
18. Murakami M, Iwata Y, Funaba M. Expression and transcriptional activity of alternative splice variants of MITF exon 6. Mol Cell Biochem. Mol Cell Biochem . 2007;303:251-7. http://dx.doi.org/10.1007/s11010-007-9474-x [ Links ]
19. Shahlaee AH, Brandal S, Lee YN, Jie C, Takemoto CM. Distinct and shared transcriptomes are regulated by microphthalmia-associated transcription factor isoforms in mast cells. J Immunol . 2007;178:378-88. http://dx.doi.org/10.4049/jimmunol.178.1.378 [ Links ]
20. Fuse N, Yasumoto K, Takeda K, Amae S, Yoshizawa M, Udono T, et al . Molecular cloning of cDNA encoding a novel microphthalmia-associated transcription factor isoform with a distinct amino-terminus. J Biochem . 1999;126:1043-51. [ Links ]
21. Hershey CL, Fisher DE. Genomic analysis of the microphthalmia locus and identification of the MITF-J/Mitf-J isoform. Gene . 2005;347:73-82. http://dx.doi.org/10.1016/j.gene.2004.12.002 [ Links ]
22. Bismuth K, Maric D, Arnheiter H. MITF and cell proliferation: The role of alternative splice forms. Pigment Cell Res . 2005;18:349-59. http://dx.doi.org/10.1111/j.1600-0749.2005.00249.x [ Links ]
23. Kuiper RP, Schepens M, Thijssen J, Schoenmakers EF, van Kessel AG. Regulation of the MiTF/TFE bHLH-LZ transcription factors through restricted spatial expression and alternative splicing of functional domains. Nucleic Acids Res . 2004;32:2315-22. http://dx.doi.org/10.1093/nar/gkh571 [ Links ]
24. Mansky KC, Sulzbacher S, Purdom G, Nelsen L, Hume DA, Rehli M, et al . The microphthalmia transcription factor and the related helix-loop-helix zipper factors TFE-3 and TFE-C collaborate to activate the tartrate-resistant acid phosphatase promoter. J Leukoc Biol . 2002;71:304-10. [ Links ]
25. McGill GG, Horstmann M, Widlund HR, Du J, Motyckova G, Nishimura EK, et al . Bcl2 regulation by the melanocyte master regulator MITF modulates lineage survival and melanoma cell viability. Cell. 2002;109:707-18. http://dx.doi.org/10.1016/S0092-8674(02)00762-6 [ Links ]
26. Loercher AE, Tank E, Delston R, William J. MITF links differentiation with cell cycle arrest in melanocytes by transcriptional activation of INK4A. J Cell Biol. 2005; 168 :35-40. http://dx.doi.org/10.1083/jcb.200410115 [ Links ]
27. Carreira S, Goodall J, Aksan I, La Rocca SA, Galibert MD, Denat L, et al . MITF cooperates with Rb1 and activates p21 Cip1 expression to regulate cell cycle progression . Nature. 2005;433:764-9. http://dx.doi.org/10.1038/nature03269 [ Links ]
28. Sestáková B, Ondrusová L, Vachtenheim J. Cell cycle inhibitor p21/ WAF1/ CIP1 as a cofactor of MITF expression in melanoma cells. Pigment Cell Melanoma Res. 2010;23:238-51. http://dx.doi.org/10.1111/j.1755-148X.2010.00670.x [ Links ]
29. Du J, Widlund HR, Horstmann MA, Ramaswamy S, Ross K, Huber WE, et al . Critical role of CDK2 for melanoma growth linked to its melanocyte-specific transcriptional regulation by MITF. Cancer Cell. 2004;6:565-76. http://dx.doi.org/10.1016/j.ccr.2004.10.014 [ Links ]
30. Lekmine F, Chang CK, Sethakorn N, Das Gupta T, Salti GI. Role of microphthalmia transcription factor (MITF) in melanoma differentiation. Biochem Biophys Res Commun. 2007;354:830-5. http://dx.doi.org/10.1016/j.bbrc.2007.01.075 [ Links ]
31. Bertolotto C, Bille K, Ortonne J, Ballotti R. Regulation of tyrosinase gene expression by cAMP in B16 melanoma cells involves two CATGTG motifs surrounding the TATA box: Implication of the microphtthalmia gene product. J Cell Biol. 1996;134:747-55. [ Links ]
32. Tshori S, Gilon D, Beeri R, Nechushtan H, Kaluzhny D, Pikarsky E, et al . Transcription factor MITF regulates cardiac growth and hypertrophy. J Clin Invest. 2006;116:2673-81. http://dx.doi.org/10.1172/JCI27643 [ Links ]
33. Tshori S, Sonnenblick A, Yannay-Cohen N, Kay G, Nechushtan H, Razin E. Microphthalmia transcription factor isoforms in mast cells and the heart. Molecular Cell Biol. 2007;27:3911-9. http://dx.doi.org/10.1128/MCB.01455-06 [ Links ]
34. Bharti K, Debbache J, Wang X, Arnheiter H. The basic- helix-loop-helix-leucine zipper gene Mitf : Analysis of alternative promoter choice and splicing. Methods Mol Biol. 2010;647:237-50. http://dx.doi.org/10.1007/978-1-60761-738-9_14 [ Links ]
35. Shizukuda Y, Buttrick PM. Subtype specific roles of beta-adrenergic receptors in apoptosis of adult rat ventricular myocytes. J Mol Cell Cardiology. 2002;34:823-31. http://dx.doi.org/10.1006/jmcc.2002.2020 [ Links ]
36. Muntz KH, Zhao M, Miller JC. Downregulation of myocardial beta-adrenergic receptors. Receptor subtype selectivity. Circ Res. 1994;74:369-75. http://dx.doi.org/10.1161/01.RES.74.3.369 [ Links ]
37. Cooke L, Muntz KH. Differences in beta adrenergic receptor agonist affinity between cardiac myocytes and coronary arterioles in canine heart. J Pharmacol Exp Ther. 1994;269:351-7. [ Links ]
38. Singal PK, Khaper N, Palace V, Kumar D. The role of oxidative stress in the genesis of heart disease. Cardiovasc Res. 1998;40:426-32. http://dx.doi.org/10.1016/S0008-6363(98)00244-2 [ Links ]
39. Devic E, Xiang Y, Gould D, Kobilka B. Beta-adrenergic receptor subtype-specific signaling in cardiac myocytes from beta(1) and beta(2) adrenoreceptor knockout mice. Mol Pharmacol. 2001;60:577-83. [ Links ]
40. Shizukuda Y, Reyland ME, Buttrick PM. Protein kinase C-delta modulates apoptosis induced by hyperglycemia in adult ventricular myocytes. Am J Physiol Heart Circ Physiology. 2002;282:H162534. http://dx.doi.org/10.1152/ajpheart.00783.2001 [ Links ]
41. Bernstein D, Fajardo G, Zhao M, Urashima T, Powers J, Berry G, et al . Differential cardioprotective/cardiotoxic effects mediated by beta-adrenergic receptor subtypes. Am J Physiol Heart Circ Physiol. 2005;289:H2441-9. http://dx.doi.org/10.1152/ajpheart.00005.2005 [ Links ]
42. Gómez LA, Alekseev AE, Aleksandrova LA, Brady PA, Terzic A. Use of the MTT assay in adult ventricular cardiomyocytes to assess viability: Effects of adenosine and potassium on cellular survival. J Mol Cell Cardiol 1997;29:1255-66. DOI: http://dx.doi.org/10.1006/jmcc.1996.0363 [ Links ]
43. Gómez LA , Gómez G, Fajardo G. Procesamiento y análisis de imágenes como herramienta para medir la actividad MTT reductasa en cardiomiocitos ventriculares aislados. Uso de imágenes para evaluar en tiempo real la viabilidad de células cardiacas. En: 75 maneras de generar conocimiento en Colombia, 1995-2005. Casos seleccionados por los programas nacionales de Ciencia, Tecnología e Innovación. Bogotá, D.C.: Colciencias; 2006. p. 26-7. [ Links ]
44. Gómez LA. Preacondicionamiento isquémico en cardio- miocitos ventriculares aislados. Identificación y expresión de algunos microRNAs asociados. Rev Acad Colomb Cienc. 2013;37:433-47. [ Links ]
45. Liu F, Song YK, Liu D. Hydrodynamics-based transfection in animals by systemic administration of plasmid DNA. Gene Ther. 1999;6:1258-66. [ Links ]
46. Gómez LA. Avances científicos y tecnológicos. Genoma humano y salud pública. En: Malagón-Londoño G, Moncayo, editores. Salud pública-perspectivas. Segunda edición. Bogotá, D.C.: Editorial Médica Panamericana; 2011. p. 615- 30. [ Links ]
47. Gómez LA. Premios Nobel en Fisiología o Medicina y Química, año 2006. Una nueva dimensión del ARN en la regulación de la expresión. Biomédica 2006;26:475-84. [ Links ]
48. Chomczynski P, Sacchi N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Anal Biochem. 1987;162:156-9. http://dx.doi.org/10.1016/0003-2697(87)90021-2 [ Links ]
49. Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W, et al . Gapped BLAST and PSI-BLAST: A new generation of protein database search programs. Nucleic Acids Res. 1997;25:3389-402. [ Links ]
50. Schefe JH, Lehmann KE, Buschmann IR, Unger T, Funke-Kaiser H. Quantitative real-time RT-PCR data analysis: Current concepts and the novel gene expression's CT difference formula. J Mol Med. 2006;84:901-10. http://dx.doi.org/10.1007/s00109-006-0097-6 [ Links ]
51. Gao Z, Xu H, DiSilvestre D, Halperin V, Tunin R, Tian Y, et al . Transcriptomic profiling of the canine tachycardia-induced heart failure model: Global comparison to human and murine heart failure. J Mol Cel Cardiol. 2006;40:76-86. http://dx.doi.org/10.1016/j.yjmcc.2005.08.002 [ Links ]













