Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157
Biomédica vol.34 supl.1 Bogotá Apr. 2014
https://doi.org/10.7705/biomedica.v34i0.1657
ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1657
1 Línea de Epidemiología Molecular Bacteriana, Grupo de Microbiología Molecular, Escuela de Microbiología, Universidad de Antioquia, Medellín, Colombia
2 Grupo de Investigación en Microbiología Básica y Aplicada, MICROBA, Escuela de Microbiología, Universidad de Antioquia, Medellín, Colombia
3 Grupo Investigador de Problemas en Enfermedades Infecciosas, GRIPE, Facultad de Medicina, Universidad de Antioquia, Medellín, Colombia
4 Unidad de Bacteriología y Micobacterias, Corporación para Investigaciones Biológicas (CIB), Medellín, Colombia
5 Escuela de Ciencias de la Salud, Universidad Pontificia Bolivariana, Medellín, Colombia
Contribución de los autores:
Judy Natalia Jiménez y Ana M. Ocampo: realización de los experimentos, análisis de los datos.
Todos los autores participaron en el diseño de los experimentos, la recolección de los datos y la elaboración del manuscrito.Recibido: 17/05/13; aceptado: 23/08/13
Introducción. Parte del éxito de Staphylococcus aureus resistente a la meticilina (SARM) como patógeno se debe a la rápida diseminación de linajes pandémicos con perfiles variables de virulencia y sensibilidad antimicrobiana. En Colombia se han identificado clones asociados al hospital como el pediátrico (CC5-ST5-SCC mec IV), el brasilero (CC8-ST239-SCC mec III) y el chileno/cordobés (CC5-ST5-SCC mec I). Asimismo, se describió el USA300 (CC8-ST8-SCC mec IV), tradicionalmente asociado a la comunidad, causante de infecciones hospitalarias .
Objetivo. Describir el comportamiento en el tiempo de los clones de SARM provenientes de un hospital universitario de Medellín en aislamientos recolectados con una década de diferencia.
Materiales y métodos. Se analizaron 398 aislamientos de SARM, 67 recolectados en 1994 y 331 recolectados entre 2008 y 2010. La identificación y la sensibilidad a la meticilina se confirmaron mediante los genes nuc y mec A. La caracterización molecular incluyó la tipificación de spa , SCC mec , la electroforesis en gel de campo pulsado ( Pulsed Field Gel Electrophoresis, PFGE), y la tipificación por secuenciación de locus múltiples ( Multilocus Sequence Typing , MLST).
Resultados. Al analizar los aislamientos de SARM de 1994 se encontró que pertenecían a un único linaje, el CC5-SCC mec IV, mientras que los aislamientos de 2008 a 2010 presentaron dos linajes dominantes: el CC8-SCC mec IVc, con cepas de los tipos spa t008 y t1610, estrechamente relacionadas con el clon USA 300, y el CC5-SCC mec I, con las de tipo spa t149, relacionadas con el clon chileno; no se detectaron cepas del linaje encontrado en 1994.
Conclusiones. En este estudio se demuestra una dinámica en el tiempo de las cepas de S. aureus , y se señala la importancia de la vigilancia local y la difusión de los resultados, sobre todo en países como el nuestro, donde SARM es prevalente y la comprensión de su epidemiología es limitada.
Palabras clave: Staphylococcus aureus resistente a meticilina, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1657
Changes over time in the distribution of dominant clonal complexes of methicillin-resistant Staphylococcus aureus in Medellín, Colombia
Introduction: Part of the success of methicillin-resistant Staphylococcus aureus (MRSA) as a pathogen responds to the rapid spread of pandemic lineages with diverse virulence and antimicrobial susceptibility profiles. In Colombia, several healthcare-associated MRSA (HA-MRSA) clones have been found, including the pediatric clone (CC5-ST5-SCC mec IV), the Brazilian clone (CC8-ST239-SCC mec III), and the Chilean/Cordobés clone (CC5-ST5-SCC mec I). Moreover, the community-associated MRSA (CA-MRSA) clone USA300 has been reported as causing hospital-acquired infections.
Objective: To describe the changes over time in the distribution of MRSA clones from a university hospital in Medellín collected at two time points a decade apart.
Materials and methods: A total of 398 MRSA strains were analyzed. Of these, 67 strains were collected in 1994, while the remaining 331 strains were collected between 2008 and 2010. Species identification and methicillin resistance were confirmed by detection of nuc and mec A genes, respectively. Molecular characterization included spa typing, SCC mec typing, PFGE and MLST.
Results: Analysis of the MRSA strains collected in 1994 revealed that they belonged to a single clone, the CC5-SCC mec IV, whereas among the isolates from 2008-2010, two dominant clones were identified: CC8-SCC mec IVc, which included spa types t008 and t1610 and is closely related to the USA 300 clone, and CC5-SCC mec I ( spa type t149), related to the Chilean clone. The ST5-SCC mec IV clone from 1994 was not detected.
Conclusions: This study identifies temporal dynamics in MRSA clone diversity, and highlights the importance of local surveillance and dissemination of results, especially in countries like Colombia where MRSA is prevalent and knowledge regarding its epidemiology is still insufficient.
Key words: Methicillin resistant Staphylococcus aureus , Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1657
Staphylococcus aureus resistente a la meticilina (SARM) es un patógeno de gran importancia en salud pública debido a la rápida diseminación y diversificación de linajes pandémicos que presentan perfiles variables de virulencia y sensibilidad antimicrobiana . Entre los clones más importantes asociados al ambiente hospitalario (SARM-AH) se encuentran el clon arcaico (CC8-ST250-SCC mec I), el clon ibérico (CC8-ST247-SCC mec I), el brasilero (CC8-ST239-SCC mec III) , el pediátrico (CC5-ST5-SCC mec IV), el Nueva York/Japón (CC5-ST5-SCC mec II) y el EMRSA- 15 (CC22-ST22-SCC mec IV). En estos clones se han observado cambios en el tiempo entre países, regiones, e incluso dentro de una misma institución hospitalaria (1,2).
Asimismo, cinco clones de SARM asociados a la comunidad (SARM-AC), se han diseminado por todo el mundo: el ST1 reportado en Asia, Europa y Estados Unidos, el ST8, en Europa y en Estados Unidos, el ST30, en Australia, Europa y Suramérica, el ST59, en Asia y Estados Unidos y el ST80, en Asia, Europa y Medio Oriente (2,3).
Recientemente, en el ámbito hospitalario, cepas asociadas a SARM-AC han comenzado a reemplazar las cepas tradicionales de SARM-AH, especialmente en Estados Unidos y Taiwán, donde la prevalencia de SARM-AC es alta (4-7).
Por otra parte, en Suramérica los linajes encontrados con mayor frecuencia son el clon pediátrico (CC5-ST5-SCC mec IV), el clon chileno/cordobés (CC5-ST5-SCC mec I), el clon Oceanía- South West Pacific (CC30-ST30-SCC mec IV), el clon (CC8-ST239-SCC mec III) y el clon Nueva York/Japón (CC5-ST5-SCC mec II) (8). En Colombia se han identificado clones de SARM-AH como el pediátrico, el brasilero y el chileno/cordobés, principalmente en hospitales de tercer nivel en Bogotá (9-11). Asimismo, el clon de SARM-AC USA300 (CC8-ST8-SCC me cIV) se ha descrito como causante de infecciones hospitalarias (12-14).
Considerando la naturaleza cambiante de los clones de SARM y la importancia de la vigilancia local, en este trabajo nos propusimos describir el comportamiento en el tiempo de los clones de SARM recolectados con más de una década de diferencia en un hospital universitario de alto nivel de complejidad localizado en Medellín, en el cual SARM es prevalente.
Materiales y métodos
Población de estudio
El estudio se llevó a cabo en un hospital universitario de alto nivel de complejidad de 700 camas ubicado en Medellín. Se incluyeron aislamientos de SARM obtenidos de pacientes hospitalizados con infección, y recolectados en dos periodos diferentes: 67 aislamientos recolectados de forma prospectiva, obtenidos durante el año 1994, y 331 aislamientos recolectados de forma prospectiva y obtenidos entre febrero de 2008 y junio de 2010. Estos últimos aislamientos hacían parte de un estudio reportado previamente (14). En ambos periodos de estudio se incluyó una muestra por paciente correspondiente al primer aislamiento clínico de SARM obtenido durante el proceso infeccioso.
Consideraciones éticas
El protocolo de investigación fue aprobado por el Comité de Bioética para Investigación en Humanos de la Sede de Investigación Universitaria de la Universidad de Antioquia (CBEIH-SIU), así como por el comité de ética de la institución hospitalaria.
Identificación de Staphylococcus aureus y determinación de la sensibilidad a meticilina
La identificación de S. aureus se realizó morfológica y bioquímicamente de la siguiente manera: a las colonias con morfología típica de Staphylococcus se les practicaron las pruebas de catalasa y de coagulasa en tubo para confirmar la presencia de S. aureus (15). La evaluación de la sensibilidad a meticilina (oxacilina) se hizo en el equipo automatizado Vitek 2 ® (bioMérieux, Marcy l'Etoile, France), de acuerdo con los estándares del Clinical Laboratory and Standards Institute (CLSI) (16).
Confirmación molecular de la especie, de la resistencia a la meticilina y tipificación del casete cromosómico mec (SCCmec)
La presencia de los genes específicos de especie nuc y femA , así como del gen mecA que determina la resistencia a la meticilina, se determinó empleando protocolos de reacción en cadena de la polimerasa (PCR) previamente descritos (17,18). Los tipos y subtipos del SCC mec se determinaron mediante la estrategia de PCR descrita por Kondo, et al . (19). Las siguientes son las cepas SARM empleadas como controles positivos, las cuales fueron donadas por Teruyo Ito (Universidad de Juntendo, Japón): NCTC10442 (SCC mec I), N315 (SCC mec II), 85/2082 (SCC mec III), JCSC4744 (SCC mec IVa), JCSC2172 (SCC mec IVb), 81/108 (SCC mec IVc), KSC4469 (SCC mec IVd), JCSC4796 (SCC mec IVg), y WIS (SCC mec V).
Tipificación de la región polimorfa de la proteína A (tipo spa) y tipificación por secuenciación de locus múltiples
La región polimorfa X de la proteína A ( spa ) se amplificó y secuenció en el total de las muestras siguiendo la metodología de Shopsin, et al. (20). El análisis de los tipos de secuencias de spa se realizó utilizando el software eGenomics (20,21). Posteriormente, los tipos spa se asignaron de acuerdo con el sitio web Ridom (http://www.spaserver.ridom.de/) desarrollado por Ridom GmbH © bajo la curaduría de SeqNet.org (http://www.SeqNet.org/) (22). Además, en un subconjunto de aislamientos se aplicó la tipificación por secuenciación de locus múltiple ( Multilocus Sequence Typing, MLST) empleando la metodología descrita por Enright, et al . (23). Los alelos y los tipos de secuencia se asignaron por medio de la base de datos del sitio web de MLST (http://saureus.mlst.net/), mientras que los complejos 'clonales' se determinaron mediante el algoritmo eBURST (24).
Electroforesis en gel de campo pulsado
Se empleó el método descrito por Mulvey, et al . (25), el cual utiliza la enzima S m a I. La electroforesis en gel de campo pulsado ( Pulsed Field Gel Electrophoresis, PFGE) se realizó en el sistema CHEF-DRIII ™ (Bio-Rad, Hercules, California, USA). Para normalizar los fragmentos de ADN y permitir la comparación entre los diferentes geles, se empleó la cepa de referencia S. aureus NCT C 8325 . La asignación de las bandas se hizo de forma automática y luego se ajustó manualmente; para el análisis se consideraron bandas entre 36 y 600 kb. El análisis de conglomerado se realizó empleando el coeficiente de Dice en el software Bionumerics, versión 6.0 (Applied Maths, Sint- Martens-Latem, Bélgica). Los dendrogramas se generaron mediante el promedio aritmético de los grupos de pares no ponderados ( Unweighted Pair Group Method with Arithmetic Mean, UPGMA) con 1 % de tolerancia y 0,5 % de optimización. Los puntos de corte empleados para el coeficiente de similitud fueron de 80 y 95% para definir tipos y subtipos, respectivamente (25).
Análisis estadístico
Las frecuencias de los linajes encontrados por año se compararon usando la prueba de ji al cuadrado o la prueba exacta de Fisher y estableciendo valores de p=0,05 como estadísticamente significativos. Los análisis se llevaron a cabo mediante el paquete estadístico SPSS ®, versión 15.0.
Resultados
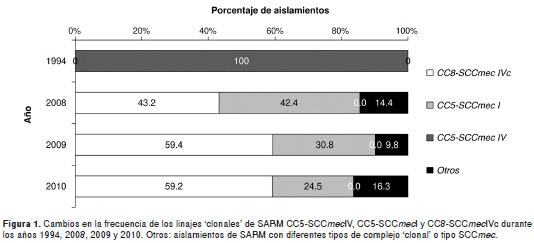
Al analizar los aislamientos recolectados durante el año 1994 se encontró que 100 % de las cepas (67) pertenecían al CC5 y presentaban el tipo spa t045 (eGenomics 47, TMDMGMK), asimismo, todos portaban un SCC mec tipo IV, pero el subtipo no pudo determinarse (figura 1).
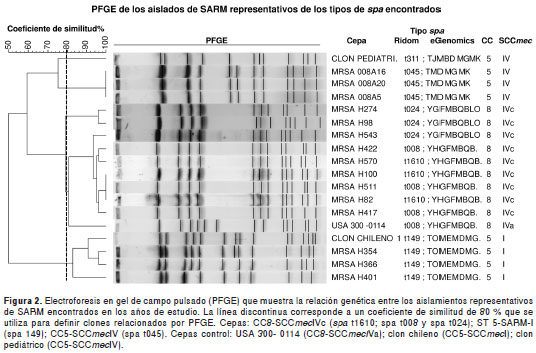
En los análisis de la PFGE se encontró que estos aislamientos presentaban un coeficiente de similitud de 75,4 % con el clon pediátrico (CC5 -ST8-SCC mec IV, actualmente clasificado como VI ) (26) (figura 2).
Al analizar los aislamientos SARM de los años 2008 a 2010, se encontraron 10 complejos 'clonales' de MLST; sin embargo, 96 % de las cepas pertenecía a los complejos 'clonales' CC5 (ST5) y CC8 (ST8) (37,4 y 58,6 %, respectivamente). Asimismo, los tipos spa más comunes fueron los siguientes: spa t1610 (eGenomics 814, YHGFMBQBBLO, 25,4 %), t149 (eGenomics 442, TO2MEMDMGMGMK, 20,8 %), t008 (eGenomics 1, YHGFMBQBLO, 13 %), y t024 (eGenomics 363, YGFMBQBLO, 9,7 %). Los aislamientos con spa t1610, t008, y t024, pertenecientes al complejo 'clonal' 8 (ST8), albergaban con gran frecuencia el SCC mec IVc (99,2 %), mientras que los aislamientos con tipo spa t149 del complejo 'clonal' 5 (ST5) portaban el SCC mec tipo I exclusivamente.
Al comparar las frecuencias de estos linajes entre los años de estudio se encontraron cambios en su distribución . Los aislamientos del CC8 que portaban el SCCmec IVc (tipos spa t1610, t008, y t024, principalmente) incrementaron su frecuencia de 43,2 % en el 2008 a 59,2 % en el 2010 (p<0,05). Por el contrario, los aislamientos del ST5 que portaban el SCC mec de tipo I, disminuyeron su frecuencia de 42,4 % en el 2008 a 24,5 % en el 2010 (p<0,05) (figura 1). Los resultados de la electroforesis en gel de campo pulsado evidenciaron que los tipos spa t1610 y t008 estaban estrechamente relacionados con el clon SARM-AC USA300-0114 (CC8-ST8- SCC mec IVa), con un coeficiente de similitud del 80,65 %, mientras que con los aislamientos del tipo spa t024 fue de 78 % (figura 2).
De acuerdo con lo esperado, los aislamientos pertenecientes al tipo spa t149 estaban estrechamente relacionados (coeficiente de similitud de 83 %) con el clon chileno (CC5-ST5-SCC mec I).
Ninguna de las cepas recolectadas del 2008 al 2010 mostró relación con las obtenidas en 1994 ni con el clon pediátrico (ST5-SARM-IV, actualmente clasificado como VI).
Discusión
La realización de este trabajo permitió evidenciar el cambio en el tiempo de los clones de SARM responsables de las infecciones en un hospital universitario de alto nivel de complejidad de Colombia. En este estudio se muestra la expansión de cepas del CC8-SCC mec IVc, el desplazamiento de cepas del CC5-SCC mec I y la desaparición de las cepas del CC5-SCC mec IV. Igualmente, se evidencian diferencias con relación a lo reportado previamente en el país y en Suramérica.
El primer estudio publicado en el país sobre caracterización molecular de SARM se realizó entre los años 1996 y 1998 como parte de una estrategia global de vigilancia de la resistencia con muestras de hospitales de Bogotá y Manizales (9). En dicho trabajo, la tipificación molecular mostró que todos los aislamientos compartían propiedades idénticas de un solo clon denominado "clon pediátrico", que había sido previamente descrito en los años 90 en Europa, Estados Unidos y Suramérica. Sin embargo, los aislamientos de Colombia diferían del clon pediátrico por su amplia resistencia a múltiples medicamentos y por haberse aislado de pacientes de todas las edades. En 2005, Cruz, et al . (10), evaluaron 200 muestras de SARM recolectadas entre 1996 y 2003 que provenían en su mayoría de hospitales de Bogotá y Cali (48 y 45 %, respectivamente); los autores no detectaron el clon pediátrico, solo encontraron el denominado clon chileno. Este fue el primer reporte de tal clon en Colombia, lo cual, según los autores, indicaba un cambio en la población genética de SARM en el país. Álvarez, et al. (27), presentaron en el 2006 el primer reporte de SARM-AC en Colombia; posteriormente, Reyes, et al., en 2009 (12) y Álvarez, et al ., en 2010 (13) reportaron el clon USA 300 (CC8-ST8-SCC mec IV) como causante de infecciones hospitalarias y Jiménez, et al ., en 2012 (14) documentaron el predominio de cepas de SARM del CC8 con SCC mec IVc como causantes de infecciones en hospitales de Medellín.
Los resultados de este trabajo evidencian hallazgos importantes con relación a las cepas previamente reportadas en el país. Las cepas recolectadas en 1994 presentaron un coeficiente de similitud de 75 % con el clon pediátrico (CC5-ST5-SCC mec -IV), el cual había sido reportado con alta prevalencia entre 1996 y 1998 (9), y que no fue detectado en las cepas recolectadas entre los años 2008 y 2010. Por otra parte, el clon chileno (CC5-ST5-SCC mec I) ocupó el segundo lugar en frecuencia entre los años 2008 y 2010, a diferencia de lo informado en estudios realizados en hospitales, principalmente de Bogotá, entre 2001 y 2003 y entre 2006 y 2007, donde fue el clon dominante (10,11).
Los resultados de este trabajo concuerdan con los reportes actuales que muestran un desplaza-miento de las cepas de SARM que portan el SCC mec tipo IVc, tradicionalmente asociadas a la comunidad, hacia el ambiente hospitalario, habiéndose convertido en las principales respon-sables de las infecciones asociadas a la atención en salud ocasionadas por S. aureus (7,12-13,28,29) . Es interesante anotar que estas cepas difieren del prototipo de cepas USA 300-0114 en que albergan el SCC mec IVc en lugar del IVa, y, por el contrario, comparten esta característica con la USA 300, variante latinoamericana (USA 300-LV), recientemente descrita (30).
El predominio de las cepas con SCC mec IV puede deberse a que presentan varias ventajas: i) un SCC mec de menor tamaño, que les permite transmitirse más fácilmente y tener una velocidad de replicación mayor; ii) un menor número de genes de resistencia a antibióticos comparado con el de las cepas que portan el SCC mec tipo I, II o III, lo cual constituye una ventaja para el costo de la eficacia biológica, y iii) una tasa de crecimiento mayor, que significa una ventaja frente a otras bacterias en cuanto a la colonización exitosa (31,32).
Los linajes encontrados con mayor frecuencia en el estudio mostraron diferencias con los linajes de SARM reportados con mayor prevalencia en Suramérica, como el CC5-ST5-SCC mec II, el CC5-ST5-SCC mec IV, el CC5-ST5-SCC mec I, el CC30-ST30-SCC mec IV (Oceanía South West Pacific) y el CC8-ST239-SCC mec III (8).
La combinación de diferentes metodologías de tipificación molecular como la MLST, la PFGE y la tipificación de spa y del SCC mec , permitieron contar con un mayor poder de discriminación y, de esta manera, alcanzar una mejor comprensión del comportamiento de las cepas de SARM-AH (33).
En conclusión, los cambios constantes en la epidemiología de las infecciones ocasionadas por S. aureus reafirman que este microorganismo sigue siendo una importante amenaza para la salud pública humana, de allí la importancia de la vigilancia local y la difusión de los resultados, sobre todo en países en desarrollo como el nuestro, donde la comprensión de la epidemiología y la dinámica de la transmisión de S. aureus todavía es limitada.
Los autores declaran no tener conflicto de intereses.
Esta investigación fue financiada por el Depar tamento Administrativo de Ciencia, Tecnología e Innovación, Colciencias, proyecto 1115-459- 21442, y por el Comité para el Desarrollo de la Investigación, CODI, Universidad de Antioquia, del proyecto CIMB-032-11. Judy Natalia Jiménez recibió apoyo financiero de Colciencias para su formación doctoral.
Correspondencia: Judy Natalia Jiménez, Universidad de Antioquia, Calle 67 N° 53-108, bloque 5, oficina 135, Medellín, Colombia Teléfonos: (574) 219 5497 y 219 5485; fax: 219 5498 judynatalia@yahoo.com
1. Aires de Sousa M, de Lencastre H. Bridges from hospitals to the laboratory: Genetic portraits of methicillin- resistant Staphylococcus aureus clones. Fems Immunol Med Microbiol. 2004;40:101-11. http://dx.doi.org/10.1016/S0928-8244(03)00370-5 [ Links ]
2. Deurenberg RH, Stobberingh EE. The evolution of Staphylococcus aureus . Infect Genet Evol. 2008;8:747-63. http://dx.doi.org/10.1016/j.meegid.2008.07.007 [ Links ]
3. Chambers HF, Deleo FR. Waves of resistance: Staphylococcus aureus in the antibiotic era. Nat Rev Microbiol. 2009;7:629-41. 1 http://dx.doi.org/10.1038/nrmicro2200 [ Links ]
4. Huang YH, Tseng SP, Hu JM, Tsai JC, Hsueh PR, Teng LJ. Clonal spread of SCC mec type IV methicillin-resistant Staphylococcus aureus between community and hospital. Clin Microbiol Infect. 2007;13:717-24. http://dx.doi.org/10. 1111/j.1469-0691.2007.01718.x [ Links ]
5. Klevens RM, Morrison MA, Nadle J, Petit S, Gershman K, Ray S, et al . Invasive methicillin-resistant S taphylococcus aureus infections in the United States. JAMA. 2007;298:1763-71. http://dx.doi.org/10.1001/jama.298.15.1763 [ Links ]
6. Moran Gj, Amii RN, Abrahamian FM, Talan DA. Methicillin-resistant Staphylococcus aureus in community-acquired skin infections. Emerg Infect Dis. 2005;11:928-30. http://dx.doi.org/10.3201/eid1106.040641 [ Links ]
7. Popovich KJ, Weinstein RA, Hota B. Are community- associated methicillin-resistant Staphylococcus aureus (MRSA) strains replacing traditional nosocomial MRSA strains? Clin Infect Dis. 2008;46:787-94. http://dx.doi. org/10.1086/528716 [ Links ]
8. Rodríguez-Noriega E, Seas C, Guzmán-Blanco M, Mejía C, Álvarez C, Bavestrello L, et al . Evolution of methicillin- resistant Staphylococcus aureus clones in Latin America. Int J Infect Dis. 2010;14:e560-6. http://dx.doi.org/10.1016/j.ijid.2009.08.018 [ Links ]
9. Gomes AR, Sanches IS, Aires de Sousa M, Castañeda E, De Lencastre H. Molecular epidemiology of methicillin- resistant Staphylococcus aureus in colombian hospitals: Dominance of a single unique multidrug-resistant clone. Microb Drug Resist. 2001;7:23-32. http://dx.doi.org/10.1089/107662901750152729 [ Links ]
10. Cruz C, Moreno J, Renzoni A, Hidalgo M, Reyes J, Schrenzel J, et al . Tracking methicillin-resistant Staphylococcus aureus clones in Colombian hospitals over 7 years (1996-2003): Emergence of a new dominant clone. Int J Antimicrob Agents. 2005;26:457-62. http://dx.doi.org/10.1016/j.ijantimicag.2005.08.013 [ Links ]
11. Yomayusa N, Álvarez CA, Hernández PA, Ibáñez M, Sossa MP, Suárez IC, et al . Las infecciones por Staphylococcus aureus resistente a meticilina son un problema de salud pública. Revista Medica Sanitas. 2009;12:8-16. [ Links ]
12. Reyes J, Rincón S, Díaz L, Panesso D, Contreras GA, Zurita J, et al . Dissemination of methicillin-resistant Staphylococcus aureus USA300 sequence type 8 lineage in Latin America. Clin Infect Dis. 2009;49:1861-7. http://dx.doi.org/10.1086/648426 [ Links ]
13. Álvarez CA, Yomayusa N, Leal AL, Moreno J, Méndez-Álvarez S, Ibañez M, et al . Nosocomial infections caused by community-associated methicillin-resistant Staphylococcus aureus in Colombia. Am J Infect Control. 2010;38:315-8. http://dx.doi.org/10.1016/j.ajic.2009.05.013 [ Links ]
14. Jiménez JN, Ocampo AM, Vanegas JM, Rodríguez EA, Mediavilla JR, Chen L, et al . CC8 MRSA strains harboring scc mec type ivc are predominant in Colombian hospitals. Plos One. 2012;7:e38576. http://dx.doi.org/10.1371/journal.pone.0038576 [ Links ]
15. Becker K, von Eiff C. Staphylococcus , micrococcus and other catalase-positive cocci. In: Versalovic J, Carroll KC, Funke G, Jorgensen JH, Lanchy ML, Warnock DW, editors. Manual of clinical microbiology. Washington, D.C.: ASM Press; 2011. p. 308-30. [ Links ]
16. Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing: nineteenth informational supplement. CLSI Document m100-s19. Wayne, PA: Clinical and Laboratory Standards Institute; 2009 . [ Links ]
17. Brakstad OG, Aasbakk K, Maeland JA. Detection of Staphylococcus aureus by polymerase chain reaction amplification of the nuc gene. J Clin Microbiol. 1992;30:1654-60. [ Links ]
18. Mehrotra M, Wang G, Johnson WM. Multiplex PCR for detection of genes for Staphylococcus aureus enterotoxins, exfoliative toxins, toxic shock syndrome toxin 1, and methicillin resistance. J Clin Microbiol. 2000;38:1032-5. [ Links ]
19. Kondo Y, Ito T, Ma XX, Watanabe S, Kreiswirth BN, Etienne J, et al. Combination of multiplex PCRs for staphylococcal cassette chromosome mec type assignment: Rapid identification system for mec, ccr, and major differences in junkyard regions. Antimicrob Agents Chemother. 2007;51:264-74. http://dx.doi.org/10.1128/AAC.00165-06 [ Links ]
20. Shopsin B, Gómez M, Montgomery SO, Smith DH, Waddington M, Dodge DE, et al. Evaluation of protein A gene polymorphic region DNA sequencing for typing of Staphylococcus aureus strains. J Clin Microbiol. 1999; 37:3556-63. [ Links ]
21. Mathema B, Mediavilla J, Kreiswirth BN. Sequence analysis of the variable number tandem repeat in Staphylococcus aureus protein A gene: spa typing. Methods Mol Biol. 2008;431:285-305. [ Links ]
22. Harmsen D, Claus H, Witte W, Rothgänger J, Claus H, Turnwald D, et al . Typing of methicillin-resistant Staphylococcus aureus in a university hospital setting by using novel software for spa repeat determination and database management. J Clin Microbiol. 2003;41:5442-8. http://dx.doi.org/10.1128/JCM.41.12.5442-5448.2003 [ Links ]
23. Enright MC, Day NP, Davies CE, Peacock SJ, Spratt BG. Multilocus sequence typing for characterization of methicillin-resistant and methicillin-susceptible clones of Staphylococcus aureus . J Clin Microbiol. 2000;38:1008-15. [ Links ]
24. Feil EJ, Li BC, Aanensen DM, Hanage WP, Spratt BG. eBURST: Inferring patterns of evolutionary descent among clusters of related bacterial genotypes from multilocus sequence typing data. J Bacteriol. 2004;186:1518-30. [ Links ]
25. Mulvey MR, Chui L, Ismail J, Louie L, Murphy C, Chang N, et al . Development of a Canadian standardized protocol for subtyping methicillin-resistant Staphylococcus aureus using pulsed-field gel electrophoresis. J Clin Microbiol. 2001;39:3481-5. http://dx.doi.org/10.1128/JCM.39.10.3481-3485.2001 [ Links ]
26. Oliveira DC, Milheiriço C, de Lencastre H. Redefining a structural variant of staphylococcal cassette chromosome mec, SCCmec type VI. Antimicrob Agents Chemother. 2006;50:3457-9. http://dx.doi.org/10.1128/AAC.00629-06 [ Links ]
27. Álvarez CA, Barrientes OJ, Leal AL, Contreras GA, Barrero L, Rincón S, et al . Community-associated methicillin-resistant Staphylococcus aureus , Colombia. Emerg Infect Dis. 2006;12:2000-1. http://dx.doi.org/10.3201/eid1212.060814 [ Links ]
28. Donnio PY, Preney L, Gautier-Lerestif AL, Avril JL, Lafforgue N. Changes in staphylococcal cassette chromosome type and antibiotic resistance profile in methicillin-resistant Staphylococcus aureus isolates from a French hospital over an 11 year period. J Antimicrob Chemother. 2004;53:808-13. http://dx.doi.org/10.1093/jac/dkh185 [ Links ]
29. Denis O, Deplano A, De Ryck R, Nonhoff C, Struelens MJ. Emergence and spread of gentamicin-susceptible strains of methicillin-resistant Staphylococcus aureus in Belgian hospitals. Microb Drug Resist. 2003;9:61-71. http://dx.doi.org/10.1089/107662903764736355 [ Links ]
30. Nimmo GR. USA300 abroad: Global spread of a virulent strain of community-associated methicillin-resistant Staphylococcus aureus . Clin Microbiol Infect. 2012;18:725-34. http://dx.doi.org/10.1111/j.1469-0691.2012.03822.x [ Links ]
31. D'Agata EM, Webb GF, Horn MA, Moellering RC Jr, Ruan S. Modeling the invasion of community-acquired methicillin-resistant Staphylococcus aureus into hospitals. Clin Infect Dis. 2009;48:274-84. http://dx.doi.org/10.1086/595844 [ Links ]
32. Okuma K, Iwakawa K, Turnidge JD, Grubb WB, Bell JM, O'Brien FG, et al . Dissemination of new methicillin- resistant Staphylococcus aureus clones in the community. J Clin Microbiol. 2002;40:4289-94. [ Links ]
33. Larsen AR, Goering R, Stegger M, Lindsay JA, Gould KA, Hinds J, et al . Two distinct clones of methicillin-resistant Staphylococcus aureus (MRSA) with the same USA300 pulsed-field gel electrophoresis profile: a potential pitfall for identification of USA300 community-associated MRSA. J Clin Microbiol. 2009;47:3765-8. http://dx.doi.org/10.1128/JCM.00934-09 [ Links ]













