Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157
Biomédica vol.34 supl.1 Bogotá Apr. 2014
https://doi.org/10.7705/biomedica.v34i0.1642
ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1642
1 Centro Internacional de Entrenamiento e Investigaciones Médicas, CIDEIM, Cali, Colombia
2 Grupo de Epidemiología y Salud Poblacional, Escuela de Salud Pública, Facultad de Salud, Universidad del Valle, Cali, Colombia
Contribución de los autores:
Robinson Pacheco: realización del estudio, recolección de la información clínica y epidemiológica, realización de los análisis estadísticos y redacción del manuscrito.
Lyda Osorio: participación en el diseño del estudio, supervisión de la recolección de información y del análisis estadístico.
Adriana M. Correa: coordinación de las actividades de laboratorio, desarrollo de la técnica molecular de PFGE.
María Virginia Villegas: participación en el diseño y coordinación general del estudio.
Todos los autores participaron en la revisión y corrección del manuscrito.Recibido: 05/05/13; aceptado: 17/10/13
Introducción. Las enzimas carbapenemasas de tipo KPC tienen gran capacidad de diseminación, son causantes de epidemias y se asocian a mayor mortalidad y estancia hospitalaria. En Colombia se han venido reportando cada vez más desde 2007, pero se desconoce la prevalencia hospitalaria.
Objetivo. Estimar la prevalencia hospitalaria del gen bla KPC .
Materiales y métodos. Se evaluó la presencia del gen bla KPC y su 'clonalidad' en aislamientos de enterobacterias y Pseudomonas aeruginosa de pacientes hospitalizados.
Resultados. De los 424 aislamientos evaluados durante el periodo de estudio, 273 cumplieron con criterios de elegibilidad, 31,1 % fue positivo para el gen bla KPC y, al ajustar por 'clonalidad', la positividad fue de 12,8 %. El gen bla KPC se encontró con mayor frecuencia en Klebsiella pneumoniae seguido de P. aeruginosa y otras enterobacterias. A pesar de que la unidad de cuidados intensivos aportó el mayor número de aislamientos, no se encontró un patrón más prevalente del gen bla KPC en las ellas que en las otras salas. El aparato respiratorio fue el sitio anatómico de origen con la mayor prevalencia . No se presentó estacionalidad en la frecuencia de los aislamientos portadores del gen bla KPC.
Conclusión. Este estudio reveló la alta prevalencia del gen bla KPC en diferentes microorganismos aislados en varias instituciones hospitalarias del país. La extraordinaria capacidad de propagación del gen bla KPC , las dificultades del diagnóstico y la limitada disponibilidad de antibióticos plantean la apremiante necesidad de fortalecer los sistemas de vigilancia epidemiológica y ajustar oportunamente las políticas institucionales de uso racional de antibióticos con el fin de contener su diseminación a otras instituciones de salud del país.
Palabras clave: bacterias Gram negativas, prevalencia, farmacorresistencia bacteriana, infección hospitalaria, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1642
Prevalence of Gram-negative bacteria harboring bla KPC gene in Colombian hospitals
Introduction: KPC enzymes are carbapenemases with a great capability to disseminate and to cause epidemics. They are frequently associated with higher mortality rates and prolonged hospital stay. In Colombia, they have been progressively reported since 2007; however, its prevalence in hospitals is not known.
Objective: To estimate the prevalence of bla KPC gene in hospitals.
Methods and materials: The presence of bla KPC gene and its clonality were evaluated in clinical isolates of Enterobacteriacea and Pseudomonas aeruginosa in hospitalized patients.
Results: Of the 424 isolates tested during the study period, 273 met eligibility criteria, and 31.1% were positive for bla KPC gene; after clonality adjustment, positivity was 12.8%. The bla KPC gene was more frequent in Klebsiella pneumonia, followed by P. aeruginosa and other Enterobacteriacea . Although intensive care units (ICU) provided the majority of the isolates, the bla KPC pattern was not more prevalent in ICUs than in other wards. The respiratory tract was the anatomic source with the highest prevalence. No seasonality was observed associated with the frequency of isolation of microorganisms carrying bla KPC gene.
Conclusion: This study revealed a high prevalence of bla KPC gene in microorganisms isolated from different hospitals in Colombia. The extraordinary ability of bla KPC gene to spread, the difficulties for its diagnosis and the limited antibiotics available for its treatment pose the urgent need to strengthen epidemiological surveillance systems, and to timely adjust institutional policies for rational use of antibiotics in order to limit its dissemination to other institutions in the country.
Key words: Gram-negative bacteria, prevalence; drug resistance, bacterial; cross infection, Colombia.
doi:http://dx.doi.org/10.7705/biomedica.v34i0.1642
Las bacterias multirresistentes causan cerca de 60 % de todas las infecciones asociadas a la atención en salud en los Estados Unidos (1,2). Esta cifra es superior en los países de bajos y medianos ingresos, y su impacto clínico y económico se magnifica en las unidades de cuidados intensivos, convirtiéndose en una amenaza para la seguridad del paciente (3). Durante la última década, se ha reportado a nivel mundial un aumento sostenido en el porcentaje de bacterias Gram negativas que son resistentes a todas las familias de antibióticos y especialmente a los carbapenem (1,4,5). Para que una bacteria Gram negativa exprese un fenotipo resistente a los carbapenem, requiere de la combinación de, al menos, dos mecanismos de resistencia, siendo la más frecuente la producción de enzimas y el cierre de porinas, lo cual, además, le confiere a la bacteria resistencia a otros betalactámicos (6-8).
Las enzimas más relevantes en la resistencia a los carbapenem son las betalactamasas de tipo AmpC y las carbapenemasas (7,9,10), entre las que se encuentran las KPC, enzimas principalmente codificadas por el gen bla KPC, el cual generalmente se localiza en plásmidos. La localización en dicho elemento genético móvil le confiere a las bacterias portadoras la capacidad de compartir esta información genética con otras especies y familias de bacterias (11), facilitando su diseminación 'clonal' (12) y geográfica (13).
Las KPC hidrolizan eficientemente penicilinas, cefalosporinas, monobactámicos y, menos eficientemente, las cefamicinas y los carbapenem (14,15). Desde su primera detección en Klebsiella pneumoniae en 1996 (16), las KPC se han reportado como causantes de epidemias dentro y fuera de Estados Unidos (17-23). En Colombia, el primer reporte de una KPC se hizo en K. pneumoniae en 2006 (14) y en 2007 se reportó por primera vez en el mundo en Pseudomonas aeruginosa (17). Desde entonces y hasta el 2010, se reportó en varias ciudades de Colombia; de igual manera, se ha detectado su diseminación a casi todas las enterobacterias y a P. aeruginosa (14,17). A pesar de los múltiples reportes aparecidos en el país, se desconoce la prevalencia de bacterias Gram negativas portadoras de KPC en las instituciones hospitalarias del país.
Una de las razones para este desconocimiento es la limitación que tienen los laboratorios de microbiología para su detección rutinaria, ya que estas bacterias pueden expresar fenotipos variables y mostrarse resistentes a las cefalosporinas pero sensibles a los carbapenem.
Para su confirmación microbiológica se ha utilizado el test de Hodge modificado (24), que ha mostrado excelente sensibilidad y especificidad para K. pneumoniae , pero no para los demás microorganismos. Para la confirmación definitiva de KPC es necesario el uso de herramientas moleculares como la reacción en cadena de la polimerasa ( Polymerase Chain Reaction , PCR), la cual es costosa y no está disponible en todos los laboratorios de microbiología clínica de los hospitales del país.
En este estudio se determinó la prevalencia de bacterias Gram negativas portadoras del gen bla KPC en seis instituciones hospitalarias de alta complejidad en Colombia. El conocimiento de esta información contribuirá a tomar decisiones terapéuticas adecuadas acordes con la capacidad microbiología de cada una de las instituciones participantes, y a generar conocimiento sobre la magnitud de la diseminación de las KPC en el entorno hospitalario del país.
Materiales y métodos
Diseño y población de estudio
Se realizó un estudio observacional de corte transversal entre el 1° de julio de 2009 y el 30 junio de 2010 en seis instituciones hospitalarias públicas y privadas de alta complejidad en cinco ciudades de Colombia. Todas las instituciones participantes contaban con un comité de infecciones activo y con un sistema automatizado de identificación microbiológica.
Se incluyeron en el estudio todos los aislamientos de P. aeruginosa resistentes a los carbapenem (imipenem y meropenem) y las enterobacterias resistentes al cefepime o a los carbapenem (imipenem, meropenem y ertapenem), según los criterios establecidos por el Clinical and Laboratory Standards Institute (CLSI) de 2008 (25). Los aislamientos provenían de pacientes de cualquier edad y sexo, sin importar la sala de hospitalización o el diagnóstico de ingreso.
Se excluyeron todos los aislamientos provenientes de urgencias y consulta externa o de cualquier servicio que no fuera hospitalización, así como aquellos en los que la identificación del microorganismo en el laboratorio de referencia (CIDEIM) no coincidiera con la reportada por el laboratorio de microbiología de las instituciones participantes ni con los aislamientos contaminados.
La confidencialidad de las instituciones se conservó mediante un código alfanumérico. Este estudio fue aprobado por los comités de ética en investigación en humanos del CIDEIM, de la Universidad del Valle y de cada una de las instituciones participantes.
Procedimientos
Entre los procesos de rutina, los laboratorios de microbiología de las instituciones participantes identificaron los microorganismos de interés que cumplieran con los criterios de elegibilidad. De cada cultivo se realizó un repique puro en un medio de transporte con carbón activado (AIMES); posteriormente estos medios inoculados fueron almacenados a temperatura de laboratorio (25 °C) y remitidos semanalmente al CIDEIM. El embalaje y transporte se realizó según las recomendaciones de la IATA.
En el CIDEIM se verificó que los aislamientos cumplieran con los criterios de elegibilidad y se reconfirmó la identificación de familia y especie utilizando Vitek 2 ™ (bioMerieux, France). La confirmación del perfil de sensibilidad a los antibióticos se realizó por el método de microdilución en caldo (Sensititre panels ™ ) (TREK Diagnostic Systems, USA). Cuando el perfil de sensibilidad reportado por el CIDEIM no coincidió con el reportado por las instituciones, se utilizó E-test ™, siguiendo las recomendaciones del fabricante (BioMerieux, France) para resolver dichas incongruencias.
La presencia del gen bla KPC se determinó por medio de PCR según el protocolo de Yigit , et al. (16). Se preparó una suspensión de una colonia en agua ultrapura y se usó 1 µ l como plantilla para la reacción de PCR. La reacción final de 25 µ l contenía 10 pmol de cada iniciador, 10 mM de dNTP, 1,5 µ M MgCl 2, y 2,5 U de ADN polimerasa taq preparada en tampón de reacción 5X. Los parámetros utilizados para la reacción fueron: desnaturalización a 95 °C por cinco minutos y 30 ciclos de desnaturalización (95 °C por 1 minuto), alineación (52 °C por 1 minuto), extensión (72 °C por 1 minuto). El producto de PCR fue visualizado utilizando un gel de agarosa al 1 % y el producto final (1.100 pb) con Sybr safe ™ (Invitrogen, Life Techonxologies, USA) bajo luz UV, utilizando para ello el sistema Gel-doc ™ (Bio-Rad Laboratories, USA).
La relación 'clonal' de los aislamientos portadores del gen bla KPC se estableció usando electroforesis de campos pulsados ( Pulsed Field Gel Electro-phoresis , PFGE). Para los moldes de agarosa de bajo punto de fusión (2 %), se utilizó una suspensión celular de tampón CSB (100 mM Tris, 100 mM EDTA); los moldes fueron sometidos a restricción con Xba I y el ADN digerido se corrió durante dos horas en un gel de agarosa al 1 %, usando un Chef Mapper ™ (BioRad Laboratories, USA) a 14 ºC y 6 v/cm con pulsos de 2,16 s y 63,8 s por 19 horas. Para la visualización de los geles se utilizó Sybr safe ™ (Invitrogen, Life Techonologies, USA).
Para el análisis de los resultados obtenidos por PFGE se utilizó el software Fingerprinting II ™ (Bio-Rad Laboratories, USA) y el coeficiente de Dice para el análisis de similitud. Se consideraron cepas 'clonalmente' relacionadas aquellas que compartían más de 75 % de similitud (26); la comparación de las cepas se realizó a través del algoritmo UPGMA ( Unweighted Pair Group Method using Arithmetic Averages ) (27).
Análisis
El manejo de los datos y los análisis estadísticos se realizaron con el software Stata 10 ™ (Stata Corp, College Station, TX, USA). La cobertura del muestreo se determinó como el número de aislamientos recibidos en el CIDEIM sobre el total de aislamientos elegibles por cada institución participante según los registros microbiológicos analizados con el software WHONET 5,4 (28). Este análisis tuvo en cuenta solo el primer aislamiento de cada paciente.
La prevalencia de microorganismos portadores del gen bla KPC, se calculó como una proporción con su correspondiente intervalo de confianza (IC) de 95 %, utilizando como numerador el número de aislamientos con resultado positivo para el gen bla KPC y como denominador el número de aislamientos que fueron evaluados en el CIDEIM. Esta proporción se discriminó por microorganismo, institución, salas de hospita-lización, y en todos los casos se reportaron los resultados crudos y ajustados por 'clonalidad', tomando como un solo aislamiento a todos los integrantes del mismo clon.
Resultados
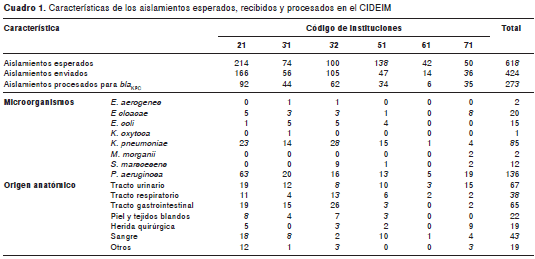
De los 618 aislamientos esperados según los registros microbiológicos de las instituciones en WHONET (figura 1), los cuales correspondían a 381 enterobacterias y 237 P. aeruginosa, se recibieron en el CIDEIM 424 (68,7 %) aislamientos, de los cuales, 242 (57 %) correspondieron a enterobacterias y 183 (43 %) a P. aeruginosa. El aporte de aislamientos por institución osciló entre 3,3 y 39,1 % (cuadro 1) con una mediana de 12,1 %; de estas, 216 (50,8 %) provenían de las unidades de cuidados intensivos, 37 (8,7 %), de unidades de cuidados intermedios y 172 (40,4 %) de salas de hospitalización general.
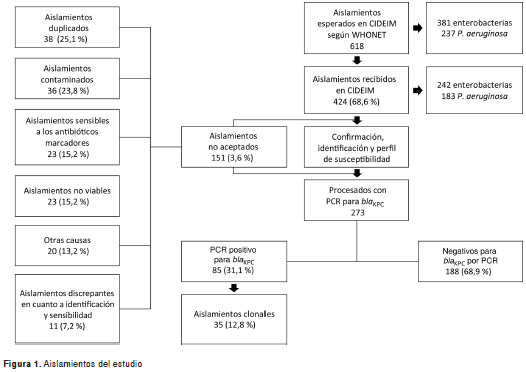
El cumplimiento de las instituciones con el envío de aislamientos se vio afectado por cambios en el personal del laboratorio, por el horario nocturno y festivo y por el descarte accidental de los aislamientos. Se excluyó 34,8 % (n=151) de los aislamientos recibidos: 47 (31,1 %) de P. aeruginosa y 104 (68,8 %) de enterobacterias, 38 (25,1 %) por ser aislamientos que se enviaron por duplicado, 36 (23,8 %) por ser aislamientos contaminados, 23 (15,2 %) por ser sensibles a los antibióticos marcadores, 23 (15,5 %) porque no fueron viables, 20 (13,2 %) por otras causas y 11 (7,2 %) por discrepancias en la identificación de la especie o el perfil de sensibilidad.
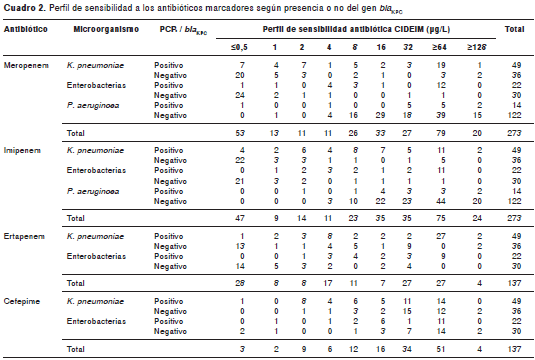
Finalmente se aceptaron y procesaron 273 aislamientos cuyo fenotipo frente a los carba-penem o al cefepime varió desde muy sensibles (=0,5 µg/L: 28 aislamientos) hasta muy resistentes (=125 µg/L: 14 aislamientos) (cuadro 2). La distribución por grupo de microorganismo fue: 136 (49,8 %) de P. aeruginosa , 85 (31,1 %) de K. pneumoniae y 52 (19 %) de otras enterobacterias. P. aeruginosa y K. pneumoniae fueron los microorganismos más frecuentes, con excepción de una institución que aportó un mayor número de aislamientos de Enterobacter. Del grupo de las enterobacterias, Enterobacter aerogenes y Morganella morganii fueron los únicos microorganismos a los cuales no se les encontró el gen bla KPC , mientras que todos los aislamientos de Serratia marcescens fueron positivos para este gen . De los 19 aislamientos de K. pneumoniae portadores del gen bla KPC que fueron sensibles a al cefepime (CIM=8 µg/L), 11 expresaron resistencia al ertapenem (nueve aislamientos con CIM=32 µg/L y dos con CIM de 8 µg/L); únicamente cinco de estos aislamientos presentaron resistencia única al ertapenem con CIM=32 µg/L; tres presentaron resistencia concomitante al imipinem con CIM de 16 µg/L; dos aislamientos al meropenem con CIM=32 µg/L y un aislamiento fue resistente a los tres carbapenem con CIM=32 µg/L.
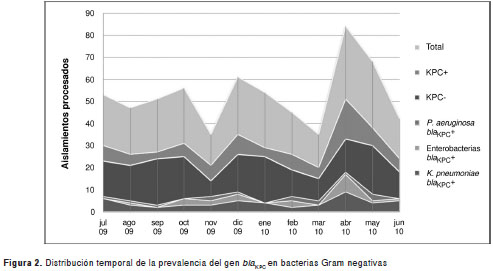
De los 273 aislamientos, 85 fueron positivos para el gen bla KPC, lo que indica una prevalencia global de 31,1 % (IC 95%, 26 - 37), mientras que por institución la prevalencia osciló entre 2 % (IC 95%, 0,05 - 12) y 55 % (IC 95%, 41 - 69) (cuadro 3). Al realizar el análisis de clonalidad por grupo de microorganismo, el número de aislamientos se ajustó para K. pneumoniae en 22 aislamientos, para otras enterobacterias en 10 aislamientos y para P. aeruginosa en cinco aislamientos. Los dendrogramas que muestran la 'clonalidad' por grupo de microorganismos se presentan en la figura 2. Con estos análisis se ajustó la prevalencia general por 'clonalidad' a 12,8 % (IC 95%, 10 - 19).
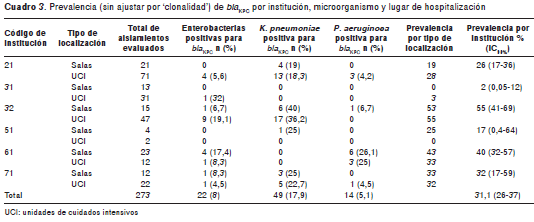
La mayor positividad (prevalencia) por microorganismo correspondió a K. pneumoniae con 19,1 %, seguido de P. aeruginosa con 16,5 % y otras enterobacterias con 15 %. No se observó un patrón en la prevalencia discriminada por salas y unidades de cuidados intensivos en las instituciones participantes (cuadro 3). De todos los aislamientos positivos para KPC, las unidades pediátricas y neonatales de cuidados intensivos representaron 19 %, mientras que las unidades especializadas de cuidados intensivos aportaron 11 % y las salas de cirugía y trauma, 8 %.
Los sistemas anatómicos en los que con mayor frecuencia se obtuvieron aislamientos positivos para KPC fueron el respiratorio con 11 (12,9 %) aislamientos, el sanguíneo con 14 (16,5 %), el urinario con 23 (27 %) y el gastrointestinal con 28 (32,9 %) aislamientos. En promedio, se aceptaron 23 aislamientos mensuales por institución y siete en promedio resultaron positivos para el gen bla KPC cada mes; abril fue el mes en el que se reportó el mayor número de aislamientos positivos con 18, mientras que enero y septiembre presentaron el menor número (figura 3).

Discusión
Los resultados de este estudio proveen una primera estimación sobre la magnitud y la distribución de microorganismos portadores del gen bla KPC en seis hospitales colombianos de alta complejidad; además, se demuestra la importancia de incorporar a los sistemas de vigilancia epidemiologia hospitalaria información molecular que les permita a las instituciones de salud hacer seguimiento a los cambios y tendencias de los mecanismos de resistencia bacteriana de mayor impacto clínico, con el objetivo de hacer ajustes oportunos a la política institucional de uso racional de antibióticos.
La prevalencia global de bla KPC fue de 31,1 % y en las instituciones osciló entre 2 y 55 %. Con el fin de no sobrestimar dicha prevalencia por la presencia de brotes de bacterias portadoras del bla KPC, se ajustó por 'clonalidad'; la prevalencia global en las instituciones bajo estudio en Colombia fue superior a la reportada en 24 % por Bratu , et al. , en un hospital de Nueva York durante un brote de K. pneumoniae, ocasión en la cual no se evaluó la 'clonalidad' de los microorganismos (15).
Tanto la prevalencia global de K. pneumoniae (17 %) como la ajustada (8 %) ubicaron a este grupo de microorganismos como el de mayor número de aislamientos positivos para el gen bla KPC, seguido por el de P. aeruginosa, cuya prevalencia global fue de 13 % y por el de las enterobacterias, cuya prevalencia ajustada fue de 1,8 %.
El elevado número de aislamientos positivos de P. aeruginosa sugiere una acelerada adquisición del gen bla KPC, sobre todo si se tiene en cuenta que el primer reporte de KPC en P. aeruginosa en el mundo se hizo en Colombia en 2007 en un solo hospital (16) y que sólo tres años después nuestro estudio lo reporta en cinco ciudades capitales, Cali, Medellín, Barranquilla, Ibagué y Pereira, distantes entre sí.
Algunos estudios moleculares previos sobre el entorno genético y la ubicación del gen bla KPC en el transposón Tn 4401 demuestran que por su alta inserción sin blanco específico en el cromosoma bacteriano, este muestra una gran diseminación entre bacterias de diferente especie y familia. Además, se ha informado que el Tn 4401 se ha diseminado entre países y regiones por medio de un clon muy exitoso, el ST 258 (12). La presencia de este clon en Colombia podría explicar, al igual que en otras regiones del mundo, la rápida diseminación del gen bla KPC en las instituciones del país (12,13,29). Este transposón está asociado a otros mecanismos de resistencia, por lo cual las bacterias que lo poseen son resistentes a múltiples clases de antibióticos.
Aunque Escherichia coli es la bacteria más frecuentemente aislada en las instituciones de salud del país, no fue la más prevalente como portadora del gen bla KPC en ninguna de las instituciones participantes; este hallazgo reafirma que K. pneumoniae tiene mayor afinidad por el transposón Tn 4401 , lo cual es un factor determinante de la dinámica de diseminación de las KPC.
En las unidades de cuidados intensivos se encuentran los pacientes con las condiciones clínicas más serias y con mayor riesgo de adquirir infecciones por bacterias multirresistentes, siendo, entonces, el servicio hospitalario con el mayor uso de antibióticos de amplio espectro como los carbapenem; sin embargo, y en contra de lo esperado, no se encontró un patrón más prevalente de KPC en las unidades de cuidados intensivos que en las salas de hospitalización general.
Este hallazgo sugiere que las bacterias Gram negativas portadoras del gen bla KPC podrían encontrarse en salas de hospitalización general debido al uso de antibióticos de menor espectro que los carbapenem; sin embargo, se requieren estudios longitudinales sobre el uso de antibióticos y sobre la epidemiologia molecular del bla KPC para identificar la dinámica de diseminación del gen en las instituciones de salud. Esto ayudaría a entender si la diseminación es un problema de presión selectiva frente a los antibióticos, o si se debe a fallas en las barreras de contacto y el lavado de manos, o a una combinación de todas las anteriores. Este conocimiento ayudaría a establecer prioridades en las acciones de control.
La prevalencia estimada refleja la situación de seis instituciones en ciudades del país, pero no es extrapolable a otras instituciones o ciudades cuyas condiciones de microbiología pueden ser diferentes de acuerdo con el tipo de pacientes que reciben y el patrón de uso de antibióticos. Sin embargo, los resultados indican que las KPC se diseminan rápidamente en las instituciones de diferentes regiones del país y su contención debe ser una prioridad. Los criterios de inclusión del estudio permitieron la participación y representación de pacientes de ambos sexos, de todas las edades y estratos socioeconómicos, de diversas condiciones clínicas y de diferentes regiones de la geografía colombiana. Con los criterios de exclusión se controló el posible impacto de sobrestimar o subestimar los resultados y con la duración de un año se controló la estacionalidad tanto del evento bajo estudio como de los eventos asociados. La captación de los aislamientos bacterianos sospechosos de portar el gen bla KPC a través de perfiles de resistencia al cefepime o cualquier carbapenem permitió la diversidad de aislamientos; además, el estudio de la relación 'clonal' de los aislamientos positivos por PFGE garantizó que los resultados obtenidos no estuvieran influenciados por brotes hospitalarios.
La principal limitación de este estudio radica en que solo se recibieron en el CIDEIM 44,8 % de los aislamientos esperados; sin embargo, no hay evidencia que demuestre que el envío de estos aislamientos estuviera sesgado y sobrestimara o subestimara los datos de prevalencia encontrados en el estudio.
En conclusión, este estudio permitió revelar la alta prevalencia del gen bla KPC en varias instituciones hospitalarias del país y en diferentes microorganismos. La extraordinaria capacidad de propagación del gen bla KPC , las dificultades del diagnóstico para hacer una identificación tem prana y la limitada disponibilidad de antibióticos plantean la apremiante necesidad de fortalecer los sistemas de vigilancia epidemiológica y las políticas institucionales de uso racional de antibióticos con el fin de contener la diseminación de bacterias portadoras de KPC a todas las instituciones de salud del país.
Agradecemos a todos los integrantes de los comités de infecciones y de los laboratorios de microbiología de las instituciones participantes por su valiosa colaboración en el envío de los aislamientos y por facilitar la información clínica y microbiológica. De igual manera, agradecemos a las bacteriólogas Elsa de la Cadena y Natalia Rosas y a las biólogas moleculares María Fernanda Mojica y Laura Rojas por su trabajo en la identificación y determinación de los perfiles moleculares de los aislamientos, así como a todo el personal del área de resistencia bacteriana del CIDEIM, y a Mauricio Pérez y Beatriz E. Ferro, por sus valiosos aportes en el desarrollo de este proyecto.
María Virginia Villegas recibe fondos de la industria farmacéutica para investigación. Los demás autores declaran no tener conflicto de intereses.
Esta investigación fue financiada por el Departamento Administrativo de Ciencia, Tecnología e Innovación - Colciencias (contrato 566-2008), y el CIDEIM.
Correspondencia: Robinson Pacheco, Centro Internacional de Entrenamiento e Investigaciones Médicas, CIDEIM, Carrera 125 N°19-225, Cali, Colombia Teléfono: (572) 555 2164; fax: (572) 555 2638 robinson.pacheco.73@gmail.com
1. Deshpande LM, Jones RN, Fritsche TR, Sader HS. Occurrence and characterization of carbapenemase- producing Enterobacteriaceae: Report from the SENTRY Antimicrobial Surveillance Program (2000-2004). Microb Drug Resist. 2006;12:223-30. http://dx.doi.org/10.1089/mdr.2006.12.223 [ Links ]
2. Jones RN. Global epidemiology of antimicrobial resistance among community-acquired and nosocomial pathogens: A five-year summary from the SENTRY Antimicrobial Surveillance Program (1997-2001). Semin Respir Crit Care Med. 2003;24:121-34. http://dx.doi.org/1010.1055/s-2003-37923 [ Links ]
3. Walsh TR. Clinically significant carbapenemases: An update. Curr Opin Infect Dis. 2008;21:367-71. http://dx.doi.org/10.1097/QCO.0b013e328303670b [ Links ]
4. Villegas MV, Blanco MG, Sifuentes-Osornio J, Rossi F. Increasing prevalence of extended-spectrum-beta- lactamase among Gram-negative bacilli in Latin America-2008 update from the Study for Monitoring Antimicrobial Resistance Trends (SMART). Braz J Infect Dis. 2011;15:34-9. http://dx.doi.org/10.1590/S1413-86702011000100007 [ Links ]
5. Briceño DF, Correa A, Valencia C, Torres JA, Pacheco R, Montealegre MC, et al . Actualización de la resistencia a los antimicrobianos de bacilos Gram negativos aislados en hospitales nivel III de Colombia, años 2006, 2007 y 2008. Biomedica. 2010;30:371-81. [ Links ]
6. Tafur JD, Torres JA, VIllegas MV. Mecanismos de resistencia a los antibióticos en bacterias Gram negativas. Infectio. 2008;12:217-26. [ Links ]
7. Bush K, Jacoby GA. Updated functional classification of beta-lactamases. Antimicrob Agents Chemother. 2010;54: 969-76. http://dx.doi.org/10.1128/AAC.01009-09 [ Links ]
8. Cuzon G, Naas T, Truong H, Villegas MV, Wisell KT, Carmeli Y, et al . Worldwide diversity of Klebsiella pneumoniae that produce beta-lactamase bla KPC -2 gene. Emerg Infect Dis. 2010;16:1349-56. http://dx.doi.org/10.3201/eid1609.091389 [ Links ]
9. Suárez CJ, Kattan JN, Guzmán AM, VIllegas MV. Mecanismos de resistencia a carbapenems en P. aeruginosa, Acinetobacter y Enterobacteriaceae y estrategias para su prevención y control. Infectio. 2006;10:85-93. [ Links ]
10. Maya JJ, Ruiz SJ, Blanco VM, Gotuzzo E, Guzmán-Blanco M, Labarca J, et al . Currents status of carbapenemases in Latin America. Expert Rev Anti Infect Ther. 2013;11:657-67. http://dx.doi.org/10.1586/14787210.2013.811924 [ Links ]
11. Gootz TD, Lescoe MK, Dib-Hajj F, Dougherty BA, He W, Della-Latta P, et al . Genetic organization of transposase regions surrounding bla KPC carbapenemase genes on plasmids from Klebsiella strains isolated in a New York City hospital. Antimicrob Agents Chemother. 2009;53:1998-2004. http://dx.doi.org/10.1128/AAC.01355-08 [ Links ]
12. Naas T, Cuzon G, Villegas MV, Lartigue MF, Quinn JP, Nordmann P. Genetic structures at the origin of acquisition of the beta-lactamase bla KPC gene. Antimicrob Agents Chemother. 2008;52:1257-63 . http://dx.doi.org/10.1128/AAC.01451-07 [ Links ]
13. Kitchel B, Rasheed JK, Patel JB, Srinivasan A, Navon-Venezia S, Carmeli Y, et al . Molecular epidemiology of KPC-producing Klebsiella pneumoniae isolates in the United States: Clonal expansion of multilocus sequence type 258. Antimicrob Agents Chemother. 2009;53:3365-70. http://dx.doi.org/10.1128/AAC.00126-09 [ Links ]
14. Villegas MV, Lolans K, Correa A, Suárez CJ, López JA, Vallejo M, et al . First detection of the plasmid-mediated class A carbapenemase KPC-2 in clinical isolates of Klebsiella pneumoniae from South America. Antimicrob Agents Chemother. 2006;50:2880-2. http://dx.doi.org/1010.1128/AAC.00186-06 [ Links ]
15. Livermore DM. The impact of carbapenemases on antimicrobial development and therapy. Curr Opin Investig Drugs. 2002;3:218-24. [ Links ]
16. Yigit H, Queenan AM, Anderson GJ, Domenech-Sánchez A, Biddle JW, Steward CD, et al . Novel carbapenem-hydrolyzing beta-lactamase, KPC-1, from a carbapenem-resistant strain of Klebsiella pneumoniae . Antimicrob Agents Chemother. 2001;45:1151-61. http://dx.doi.org/10.1128/ AAC.45.4.1151-1161.2001 [ Links ]
17. Villegas MV, Lolans K, Correa A, Kattan JN, López JA, Quinn JP. First identification of Pseudomonas aeruginosa isolates producing a KPC-type carbapenem-hydrolyzing beta-lactamase. Antimicrob Agents Chemother. 2007;51:1553-5. http://dx.doi.org/10.1128/AAC.01405-06 [ Links ]
18. Bratu S, Landman D, Haag R, Recco R, Eramo A, Alam M, et al . Rapid spread of carbapenem-resistant Klebsiella pneumoniae in New York City: A new threat to our antibiotic armamentarium. Arch Intern Med. 2005;27:1430-5. http://dx.doi.org/10.1001/archinte.165.12.1430 [ Links ]
19. Bratu S, Mooty M, Nichani S, Landman D, Gullans C, Pettinato B, et al . Emergence of KPC-possessing Klebsiella pneumoniae in Brooklyn, New York: Epidemiology and recommendations for detection. Antimicrob Agents Chemother. 2005;49:3018-20. http://dx.doi.org/10.1128/AAC.49.7.3018-3020.2005 [ Links ]
20. Nordmann P, Cuzon G, Naas T. The real threat of Klebsiella pneumoniae carbapenemase-producing bacteria. Lancet Infect Dis. 2009;9:228-36. http://dx.doi.org/10.1016/S1473-3099(09)70054-4 [ Links ]
21. Livermore DM, Hope R, Brick G, Lillie M, Reynolds R. BSAC Working Parties on Resistance Surveillance. Non- susceptibility trends among Enterobacteriaceae from bacteraemias in the UK and Ireland, 2001-06. J Antimicrob Chemother. 2008;62 (Suppl 2):ii41-54. http://dx.doi.org/10.1093/jac/dkn351 [ Links ]
22. Dortet L, Radu I, Gautier V, Blot F, Chachaty E, Arlet G. Intercontinental travels of patients and dissemination of plasmid-mediated carbapenemase KPC-3 associated with OXA-9 and TEM-1. J Antimicrob Chemother. 2008;61:455-7. http://dx.doi.org/10.1093/jac/dkm455 [ Links ]
23. Naas T, Nordmann P, Vedel G, Poyart C. Plasmid-mediated carbapenem-hydrolyzing beta-lactamase KPC in a Klebsiella pneumoniae isolate from France. Antimicrob Agents Chemother. 2005;49:4423-4. http://dx.doi.org/10.1128/AAC.49.10.4423-4424.2005 [ Links ]
24. Lee K, Kim CK, Yong D, Jeong SH, Yum JH, Seo YH, et al . Improved performance of Modified Hodge test with Mac Conkey agar 250 for screening of carbapenemase – producing Gram-negative bacilli. J Microbiol Methods. 2010;32:149-52. http://dx.doi.org/10.1016/j.mimet.2010.08.010 [ Links ]
25. Clinical Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing, eighteenth informational supplement. Wayne: CLSI; 2008. [ Links ]
26. Dice LR. Measures of the amount of ecological association between species. Ecology. 1945;26:297-302. [ Links ]
27. Tenover FC, Arbeit RD, Goering RV. How to select and interpret molecular strain typing methods for epidemiological studies of bacterial infections: A review for healthcare epidemiologists. Molecular Typing Working Group of the Society for Healthcare Epidemiology of America. Infect Control Hosp Epidemiol. 1997;18:426-39. [ Links ]
28. Stelling JM, O'Brien TF. Surveillance of antimicrobial resistance: the WHONET program. Clin Infect Dis. 1997;24:157- 68. http://dx.doi.org/10.1093/clinids/24.Supplement_1.S15 [ Links ]
29. Mojica MF, Correa A, Vargas DA, Maya JJ, Montealegre MC, Rojas LJ, et al . Molecular correlates of the spread of KPC-producing Enterobacteriacea in Colombia. Int J Antimicrob Agents. 2012;40:277–9. http://dx.doi.org/10.1016/j.ijantimicag.2012.05.006 [ Links ]













