Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157
Biomédica vol.34 supl.1 Bogotá Apr. 2014
https://doi.org/10.7705/biomedica.v34i0.1649
ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1649
1 Grupo de Investigación en Genética Humana Aplicada, Facultad de Ciencias de la Salud, Universidad del Cauca, Popayán, Colombia
2 Fundación InnovaGen, Popayán, Colombia
3 Grupo de Enfermedades Infecciosas, Departamento de Microbiología, Facultad de Ciencias, Pontificia Universidad Javeriana, Bogotá, D.C., Colombia
Contribución de los autores:
Claudia Patricia Acosta: coordinación general de actividades y supervisión metodológica.
Fabián Andrés Hurtado: ejecución de pruebas moleculares.
Alba Alicia Trespalacios: análisis de resultados y análisis de secuencias.
Todos los autores participaron en la escritura del manuscrito.Recibido: 31/05/13; aceptado: 27/11/13
Introducción. La terapia antibiótica combinada para la erradicación de Helicobacter pylori debería basarse en los patrones locales de resistencia.
Objetivo. Determinar la resistencia de H. pylori a claritromicina en una población del departamento del Cauca mediante la identificación de mutaciones en el gen 23S r RNA en ADN obtenido de biopsias gástricas.
Materiales y métodos. Se incluyeron en el estudio 162 pacientes con dispepsia funcional. El gen 23S rRNA se amplificó por PCR y el patrón de mutaciones se identificó por secuenciación directa.
Resultados. La frecuencia de resistencia a claritromicina fue de 4 %. La mutación A2143G del gen se encontró en cuatro pacientes (2,46 %) y la mutación A2142G, en tres pacientes (1,85 %).
Conclusiones. El estudio encontró que el genotipo más frecuente en los especímenes positivos para H. pylori fue 2143G, seguido por A2142G. La prevalencia observada de resistencia de H. pylori fue baja; por lo tanto, se considera que el tratamiento con claritromicina es una opción válida para la erradicación de H. pylori en la población objeto de estudio.
Palabras clave: Helicobacter pylori , claritromicina, mutación, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1649
Determination of single nucleotide mutations in the 23S rRNA gene of Helicobacter pylori related to clarithromycin resistance in a population from Cauca, Colombia
Introduction: Antibiotic combination therapy for the eradication of Helicobacter pylori should be based on local resistance patterns.
Objective: To d etermine the resistance of H. pylori to clarithromycin in a population from Cauca province, through the identification of mutations in the 23S rRNA gene in DNA from gastric biopsies.
Materials and methods: A total of 162 patients with functional dyspepsia were included in the study. The 23S rRNA gene and the DNA from 162 gastric specimens were amplified by PCR, and the mutation pattern was identified by direct sequencing.
Results: The frequency of clarithromycin resistance was 4%. A2143G mutation was found in four patients (2.46%) and A2142G mutation was found in three patients (1.85%).
Conclusions: Our study shows that the most frequent genotype in H. pylori -positive specimens was A2143G, followed by A2142G. The observed resistance prevalence of H. pylori was low; thus, we consider that clarithromycin treatment is a valid option for H. pylori eradication in the study population.
Key words: Clarithromycin, sequencing, 23S rRNA gene, mutation, Helicobacter pylori , Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1649
Helicobacter pylori es una bacteria Gram negativa que coloniza la mucosa gástrica de los seres humanos e infecta a más de la mitad de la población mundial. Es la principal causa de gastritis crónica, úlceras pépticas gastroduodenales y adenocarcinoma gástrico (1). Asimismo, produce linfomas gástricos de tipo MALT ( Mucosa-Associated Lymphoid Tissue ) (2,3), anemia ferropénica (4), y se relaciona, por lo menos, con 50 % de los casos de púrpura trombocitopénica inmune (5).
La erradicación de la infección con tratamiento antibiótico es lo recomendado para los pacientes con úlceras gástricas, gastritis atrófica, linfomas de tipo MALT de bajo grado, anemias inexplicadas por deficiencia de hierro y púrpura trombocitopénica idiopática (6). El tratamiento triple estándar, que incluye un inhibidor de la bomba de protones, claritromicina y amoxicilina, ha sido el tratamiento universal recomendado para el manejo de la infección (6).
Sin embargo, la resistencia bacteriana representa hoy un verdadero reto en la práctica clínica debido a la reducción de la eficacia de dicha terapia en los últimos años, en especial por el incremento de la resistencia de H. pylori a la claritromicina (7). La frecuencia de esta resistencia varía de un país a otro y está relacionada con el uso previo del antibiótico en infecciones respiratorias. La prevalencia promedio de la resistencia en Europa es de 11,1 %, con diferencias entre los países, pues es alta en España (49,2 %) y baja en Suecia (1,5 %) y Holanda (0,8 %); en Asia, la prevalencia promedio alcanza 18,9 %, siendo muy alta en Japón (40,7 %) y baja en Malasia (2,1 %); en América, el valor promedio de la prevalencia es de 29,3 %, con prevalencias altas en países suramericanos y una prevalencia menor en Norteamérica (8). En Colombia, dos estudios realizados por Álvarez, et al., reportan una resistencia a claritromicina de 2,2 y 3,8 % en la región del eje cafetero (9,10), mientras que en Bogotá oscila entre 15 y 17,7 % (11,12).
La resistencia a claritromicina ocurre por mutaciones en el dominio V de la peptidiltransferasa en la molécula 23S rRNA , el cual se ubica entre la subunidad ribosómica 50 S y la 70S (13,14) . La unión de la claritromicina en el dominio V en cepas sensibles interfiere con la elongación de proteínas, bloqueando efectivamente su síntesis. El mecanismo de resistencia fue descubierto por primera vez por Versalovic, et al. , quienes demostraron que ciertas mutaciones puntuales (adenina . ® . guanina, adenina . ® . citocina) en las posiciones 2142 (A2142G, A2142C) y 2143 (A2143G) (15,16) están asociadas con la pérdida de unión de la claritromicina a los ribosomas.
Helicobacter pylori contiene dos genes 23S rRNA , que en la mayoría de las cepas resistentes están mutados; el fenotipo bacteriano con un gen mutado y el otro silvestre no es muy frecuente, aunque también se ha reportado (7). Las mutaciones en las posiciones 2142 y 2143 se correlacionan muy bien con la resistencia fenotípica, lo cual es una ventaja, ya que las pruebas fenotípicas de sensibilidad que usualmente se determinan por cultivo requieren entre 10 y 14 días, razón por la cual esta prueba no se realiza de manera rutinaria a nivel clínico.
Debido a la buena correlación entre la presencia de mutaciones en el gen 23S rRNA y la resistencia fenotípica de H. pylori , cada día cobran más importancia las pruebas genotípicas para la determinación de la resistencia a la claritromicina dado el bajo número de mutaciones involucradas. La prueba de referencia de las pruebas de biología molecular para la detección de mutaciones es la secuenciación, una técnica sencilla en la que se amplifica por PCR convencional el gen de interés para después determinar la presencia de mutaciones por secuenciación directa y análisis bioinformático (1,7). Esta es una estrategia que se ha utilizado con éxito en la determinación de mutaciones relacionadas con resistencia a la claritromicina, en especial en áreas geográficas en donde los cultivos de la bacteria no están disponibles.
Según las recomendaciones del Consenso de Maastricht IV (6), el uso de las terapias de erradicación de la infección debe estar respaldado por estudios de prevalencia de la resistencia a los antimicrobianos en cada región, y se recomienda el uso empírico de las terapias en áreas en donde la prevalencia de la resistencia al antimicrobiano sea baja (6).
En Colombia, el estudio de sensibilidad a los antimicrobianos para H. pylori no se hace rutinaria mente y los pocos trabajos realizados muestran niveles diferentes de prevalencia de la resistencia. Por lo anterior, el objetivo principal de este trabajo fue determinar la resistencia de H. pylori a la claritromicina en una población del departamento del Cauca por medio de la identificación de las mutaciones en el gen 23S rRNA del ADN obtenido de biopsias gástricas.
Materiales y métodos
Población de estudio
Se incluyeron de manera prospectiva, entre marzo de 2011 y diciembre de 2012, 162 pacientes con dispepsia funcional remitidos para practicarles endoscopia digestiva alta a las unidades de gastroenterología de los hospitales Universitario San José y Susana López de Valencia, y a la Unidad de Endoscopia ENDOVIDEO 2000 de Popayán.
Se incluyeron pacientes mayores de 18 años nacidos en el departamento del Cauca que hubieran presentado síntomas de dispepsia y no hubieran recibido tratamiento de erradicación de H. pylori ni antibióticos o sales de bismuto durante el último año. Los pacientes que aceptaron participar en el estudio firmaron el consentimiento informado después de una explicación clara del estudio según lo dispuesto en la Resolución No. 008430 de 1993 del Ministerio de Salud. La investigación fue aprobada por los comités de ética de la Universidad del Cauca y la Pontificia Universidad Javeriana. Además, los participantes respondieron un cuestionario estandarizado sobre las variables sociodemográficas y clínicas incluidas en el estudio.
De cada paciente se tomaron dos biopsias del antro (una de la curva mayor y otra de la curva menor), dos del cuerpo (una de la curva mayor y otra de la curva menor) y una de la incisura angular según las guías internacionales para el estudio del cáncer gástrico (17). El análisis histopatológico se hizo de acuerdo con los sistemas de clasificación validados internacionalmente (18). Posteriormente, se tomó una muestra del antro, una de incisura angular y una del cuerpo para la extracción del ADN de H. pylori .
Extracción de ADN y amplificación por PCR
Las muestras de biopsias gástricas se colocaron y transportaron en medio de cultivo de caldo Brucella con 20 % de glicerol. El ADN genómico bacteriano se extrajo con el Wizard Genomic DNA Purification Kit ® (Promega, Madison, WI, USA), siguiendo las recomendaciones del fabricante. El ADN fue reconstituido en 100 µl de solución de rehidratación y almacenado a -30 °C hasta su uso. La calidad del ADN se determinó por medio de la relación de absorbancia A260/A280, usando un espectrofotómetro Thermo Scientific NanoDrop 2000 ™ .
Para determinar la presencia de H . pylori se tipificó el gen vacA en las muestras mediante una prueba PCR múltiple, empleando el estuche Qiagen Multiplex PCR ® . Las secuencias de los cebadores fueron las siguientes: vacA s F 5' - ATGGAAATACAACAAACACAC – 3'; R 5' - CTGCTTGAATGCGCCAAAC – 3'; vacA m F 5' - CAA TCT GTC CAA TCA AGC GAG – 3'; R 5' - GCG TCT AAA TAA TTC CAA GG – 3'. La reacción incluyó una mezcla de 2 µl de ADN genómico bacteriano, 1x de Qiagen Master Mix ®, 0,3 pmol/µl de cada cebador, dos µl de agua libre de RNAsas en un volumen de 12 µl de reacción. Los productos de amplificación se evidenciaron en geles de agarosa al 1,5 % por tinción con bromuro de etidio bajo luz ultravioleta, teniendo como referencia las bandas de amplificación de las cepas NCTC 11637 (genotipo: cagA, vacA s1/m1) y 3062 (genotipo: cagA negativo, vacA s2/m2) de H. pylori .
Un par de iniciadores se utilizaron para amplificar un fragmento de la peptidiltransferasa de la subunidad 23S rRNA en las muestras positivas para H . pylori . La secuencia de los iniciadores se basó en la secuencia publicada del gen 23S rRNA de H. pylori (número de acceso en GenBank, U27270). El juego de iniciadores amplifica un fragmento de 425 pb en donde se ubican las mutaciones de las posiciones 2142, 2143, 2144. Se usaron los iniciadores previamente reportados por Occhialini, et al., para la amplificación de esta región: K1 (5'-CCA CAG CGA TGT GGT CTC AG-3', complementaria a la posición 2191 a 2210) y K2 (5'CTC CAT AAG AGC CAA AGC CC-3', complementaria a la posición 2596 a 2615) (19).
La amplificación del ADN por PCR se realizó utilizando una mezcla de 2,4 unidades de ADN polimerasa Taq recombinante (Invitrogen™ Brasil), 6 µl de ADN genómico bacteriano, 0,8 pmol/µl de cada cebador (K1 y K2), 1,2X de tampón de reacción proporcionado por el fabricante (Invitrogen™ Brasil), 5 mmol/l de MgCl 2, 0,8 mmol/l de cada deoxinucleótido trifosfato (dNTP) (Amersham Pharmacia Biotech, Inc.) en un volumen final de 60 µl de reacción.
A continuación las muestras fueron llevadas a un termociclador (MyCycler Termal Cycler ™, BIORAD) bajo las siguientes condiciones: desnaturalización inicial durante 5 minutos a 95 °C, 35 ciclos de desnaturalización a 95 °C durante 30 segundos, hibridación a 54 °C por 30 segundos y extensión a 72 °C por 30 segundos, y extensión final a 72 °C durante 10 minutos.
Los productos de amplificación de 425 pb se evidenciaron en geles de agarosa al 2 % por tinción con bromuro de etidio bajo luz ultravioleta y por comparación con la banda de amplificación de la cepa NCTC 11637 de H. pylori . Como control de calidad, los productos de PCR ( 425 pb) se purificaron para eliminar restos de cebadores, de dNTP y de enzimas que pudiesen causar ruido de fondo en las secuencias. Este procedimiento y la reacción de secuenciación se realizaron en Macrogen, Corea. Las secuencias se analizaron usando la herramienta BLAST ( Basic Local Alignment Search Tool, http://blast.ncbi.nlm.nih.gov ) para comparar las secuencias de nucleótidos de los productos de PCR de la región amplificada con la secuencia reportada en el GenBank HPU27270, la cual es la secuencia publicada del gen 23S rRNA (20). La numeración de la posición de los nucleótidos siguió el sistema propuesto por Taylor, et al. (20).
Resultados
Características sociodemográficas de la población
A los 162 pacientes incluidos en el estudio se les diagnosticó la infección por H. pylori mediante estudio histopatológico. La edad media de los pacientes fue de 53 años (±13,5); 54 % (88/162) correspondía a mujeres y 46 %(74/162), a hombres; 56 % (91/162) provenía del área rural; 68 % (109/162) tenía un nivel educativo menor o igual a primaria, 22 % (36/162), un nivel educativo de secundaria, 3 % (5/162), nivel técnico de educación y 7 % (12/162) tenía nivel de formación universitario.
Las mujeres presentaron 57 % de las cepas con mutaciones en el gen 23S rRNA de H. pylori . Se presentó un mayor número de cepas con mutaciones a partir de los 51 años. La mayoría de las cepas con mutaciones se presentaron en pacientes del área rural. Dado el número de los pacientes que presentaban mutación, no se realizó un análisis estadístico comparativo entre los grupos (cuadro 1).
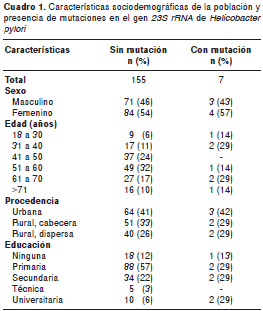
Frecuencia de mutaciones relacionadas con resistencia de Helicobacter pylori a la claritromicina
El análisis de la secuencia del fragmento de 425 pb del gen 23S rRNA obtenido de las muestras gástricas mostró que 4,3 % (7/162) de estas poseían mutaciones relacionadas con resistencia de H. pylori a la claritromicina, en tanto que 95,6 % (155/162) de las muestras no presentó ninguna de estas mutaciones relacionadas con la resistencia de H. pylori a la claritromicina.
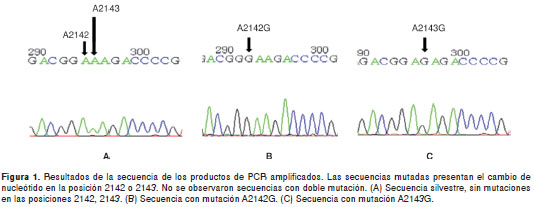
Entre las secuencias con mutación, la más frecuente fue la A2143G, identificada en cuatro de las siete muestras, seguida de la A2142G, presente en tres de las siete muestras. No se encontró ninguna secuencia con mutación en la posición 2144. Debido a la presencia de dos copias del gen 23S rRNA en el genoma de H. pylori , se realizó el análisis de los cromatogramas de todas las secuencias, encontrándose un patrón de cepas con mutación en los dos genes. En la figura 1 se observan los diferentes patrones de secuencias encontradas en el estudio.
Discusión
El tratamiento estándar de erradicación de la infección por H. pylori usualmente utiliza la combinación de un inhibidor de la bomba de protones y dos antibióticos, usualmente claritromicina y amoxicilina. La resistencia de la bacteria a claritromicina es la principal causa de falla terapéutica cuando se utiliza este esquema de tratamiento.
El primer hallazgo importante de la presente investigación fue la baja resistencia genotípica de H. pylori a la claritromicina en la población en estudio, con una frecuencia de 4,3 %. Según las recomendaciones de la Agencia Europea de Medicamentos, cuando el rango de prevalencia de la resistencia local a un antibiótico oscila entre 0 y 10 %, la región se considera de baja prevalencia de resistencia al antibiótico. Además, de acuerdo con el consenso de Maastricht IV, en regiones de baja prevalencia de resistencia a la claritromicina, los esquemas de tratamiento que contienen este antibiótico están recomendados como tratamientos empíricos de primera línea por estudios con un nivel de evidencia 1a y un grado de recomendación A. Por lo tanto, la prevalencia de 4,3 % de resistencia a la claritromicina en la población de estudio indica que el tratamiento triple estándar puede utilizarse de fo rma empírica en la erradicación de la infección.
Los resultados de este trabajo muestran que la resistencia obtenida es similar a la reportada por dos trabajos publicados por Álvarez, et al., en la región del eje cafetero (Pereira y Armenia), en donde la prevalencia de la resistencia a la claritromicina fue de 2,2 y 3,8 %, respectivamente (9,10). Sin embargo, difieren notablemente de los reportes de resistencia encontrados en otras regiones de Colombia como Bogotá, en donde la resistencia a la claritromicina reportada por Henao, et al., fue de 15 % y la obtenida por Trespalacios, et al., de 17,7 % (11,12); resultados similares encontraron Figueroa, et al., en la población de Tumaco, con una prevalencia de 19,8 % (21). Estas diferencias entre poblaciones de un mismo país indican la importancia de hacerle seguimiento localmente a la frecuencia de la resistencia, ya que existen variaciones geográficas en la prevalencia de la resistencia, lo que significa que los resultados obtenidos en una región no pueden extrapolarse a otras.
La resistencia de H. pylori a la claritromicina se explica en la gran mayoría de los casos por mutaciones en las posiciones 2142 y 2143 del dominio V de la peptidil-trasnferasa de la subunidad 23S rRNA (13,14); estas mutaciones se han correlacionado muy bien con los estudios fenotípicos de resistencia, en los cuales las concentraciones inhibitorias mínimas por dilución en agar iguales o superiores a 1 µ g/ml han revelado la presencia de dichas mutaciones.
Por esta razón, hoy en día las pruebas genotípicas pueden utilizarse de manera rutinaria para la evaluación de resistencia de H. pylori en ADN obtenido directamente de biopsias gástricas y sus resultados son comparables con las pruebas de dilución en agar y de epsilometría (E-test ® ) (13). En este estudio, la mutación A2143G fue la más frecuente en los aislamientos de H. pylori (4/7), seguida de A 2142G (3/7). La frecuencia de H. pylori con mutaciones resistentes a la claritromicina varía entre áreas geográficas: en Estados Unidos, entre 48 y 53 % porta la mutación A2142G; en Europa se ha reportado entre 23 y 33 % para A2142G, 44 y 67 % para A2143G y para A2142C, de 2 a 10 %. Algunos estudios en Japón demuestran que más de 90 % de las mutaciones tiene el genotipo A2143G y en China indican que 100 % de ellas es A2143G (19,22).
En el presente estudio se registró un comportamiento similar al observado en Asia, con una alta frecuencia de la mutación A2143G, hallazgo que también coincide con los trabajos realizados al sur de Colombia, en donde la mutación A2143G fue la más frecuente entre los aislamientos (10). La caracterización de los mecanismos de resistencia en cada país puede facilitar el desarrollo de métodos rápidos de detección de la resistencia que les permitan a los clínicos seleccionar el esquema de tratamiento y controlar adecuadamente la infección. El método de secuenciación directa utilizado en este estudio se considera el de referencia en las pruebas para determinar mutaciones y, por lo tanto, puede utilizarse para seguir la situación de resistencia en una región, en especial cuando los métodos fenotípicos no están disponibles.
Según el consenso de Maastricht IV, si el cultivo y las pruebas de sensibilidad, como la de epsilometría o la dilución en agar, no están disponibles, las pruebas moleculares pueden utilizarse para determinar la resistencia de H. pylori en biopsias gástricas o muestras de materia fecal. Estas pruebas han sido desarrolladas recientemente y son tan sensibles que pueden detectar organismos resistentes aun cuando constituyan una muy pequeña proporción entre un grupo grande de bacterias sensibles (6,23-25).
En conclusión, este es el primer estudio que se realiza en el departamento del Cauca para evaluar la resistencia de H. pylori a la claritromicina en pacientes con dispepsia funcional. La baja frecuencia de mutaciones en el gen 23S rRNA permite recomendar su uso de manera empírica en las terapias de primera línea. Además, es necesario realizar estudios clínicos para evaluar el impacto de la resistencia en la falla terapéutica en esta población y adelantar estudios de prevalencia de la resistencia a los otros antimicrobianos utilizados en las terapias de erradicación de la infección.
Los autores expresan sus agradecimientos a los pacientes por su participación en el estudio y al personal de los hospitales Universitario San José y Susana López de Valencia, así como a la Unidad de Endoscopia ENDOVIDEO 2000 de Popayán por su valiosa asistencia. Además, agradecen a la Universidad del Cauca y a la Pontificia Universidad Javeriana de Bogotá por el apoyo en la ejecución de este proyecto.
Los autores declaran no tener ningún tipo de conflicto de intereses.
Este estudio fue financiado por el Programa de Salud (código 110351929123) y el Programa de Jóvenes Investigadores (convenio No. 769 de 2012) de Colciencias.
Correspondencia: Claudia P. Acosta, Laboratorio de Genética Humana, Facultad de Ciencias de la Salud, Universidad del Cauca, Calle 5 N° 4-70, Popayán, Colombia Telefax: (572) 820 9872 cpacosta@unicauca.edu.co
1. Rimbara E, Fischbach LA, Graham DY. Optimal therapy for Helicobacter pylori infections. Nat Rev Gastroenterol Hepatol. 2011;8:79-88. http://dx.doi.org/10.1038/nrgastro. 2010.210 [ Links ]
2. Kusters JG, van Vliet AH, Kuipers EJ. Pathogenesis of Helicobacter pylori infection. Clin Microbiol Rev. 2006; 19:449-90. http://dx.doi.org/10.1128/CMR.00054-05 [ Links ]
3. Sierra F, Gutiérrez O, Otero, O. Campylobacter pylori en úlcera duodenal, gastritis crónica y dispepsia no ulcerosa. Acta Med Colomb . 1990;15:74-83. [ Links ]
4. Malfertheiner P, Megraud F, O'Morain C, Bazzoli F, El-Omar E, Graham D, et al. Current concepts in the management of Helicobacter pylori infection: The Maastricht III Consensus Report. Gut . 2007;56:772-81. http://dx.doi.org/10.1136/gut.2006.101634 [ Links ]
5. Graham DY, Fischbach L. Helicobacter pylori treatment in the era of increasing antibiotic resistance. Gut . 2010;59:1143- 53. http://dx.doi.org/10.1136/gut.2009.192757 [ Links ]
6. Malfertheiner P, Megraud F, O'Morain C, Atherton J, Axon ATR, Bazzoli F, et al. Management of Helicobacter pylori infection: The Maastricht IV/ Florence Consensus Report. Gut. 2012;61:646-64. http://dx.doi.org/10.1136/gutjnl-2012-302084 [ Links ]
7. Megraud F. Helicobacter pylori and antibiotic resistance. Gut . 2007;56:1502. http://dx.doi.org/10.1136/gut.2007.132514 [ Links ]
8. De Francesco V, Giorgio F, Hassan C, Manes G, Vannella L, Panella C, et al . Worldwide H. pylori antibiotic resistance: A systematic review. J Gastrointestin Liver Dis. 2010;19:409- 14. [ Links ]
9. Álvarez A, Moncayo JI, Santacruz JJ, Corredor LF, Reinosa E, Martínez JW, et al . Antimicrobial susceptibility of Helicobacter pylori strains isolated in Colombia. Rev Med Chil. 2009;137;1309-1314. http://dx.doi.org/S0034-98872009001000005. [ Links ]
10. Álvarez A, Moncayo JI, Santacruz JJ, Santacoloma M, Corredor LF, Reinosa E. Antimicrobial susceptibility and mutations involved in clarithromycin resistance in Helicobacter pylori isolates from patients in the western central region of Colombia. Antimicrob Agents Chemother. 2009;53:4022-4. http://dx.doi.org/10.1128/AAC.00145-09 [ Links ]
11. Henao SC, Quiroga A, Martínez JD, Otero W. Resistencia primaria a la claritromicina en aislamientos de Helicobacter pylori . Rev Colomb Gastroenterol. 2009;24:110-4. [ Links ]
12. Trespalacios AA, Otero W, Mercado MM. Resistencia de Helicobacter pylori a metronidazol, claritromicina y amoxicilina en pacientes colombianos. Rev Col Gastroenterol. 2010; 21:31-8. [ Links ]
13. Megraud F. Resistance of Helicobacter pylori to antibiotics. Aliment Pharmacol Ther. 1997;11(Suppl.1):43-53. http://dx.doi.org/10.1046/j.1365-2036.11.s1.11.x [ Links ]
14. Megraud F, Occhialini A, Doermann HP. Resistance of Helicobacter pylori to macrolides and nitroimidazole compounds. The current situation. J Physiol Pharmacol. 1997;48(Suppl.4):25-38. [ Links ]
15. Versalovic J, Shortridge D, Kibler K, Griffy MV, Beyer J, Flamm RK, et al . Mutations in 23S rRNA are associated with clarithromycin resistance in Helicobacter pylori. Antimicrob Agents Chemother.1996;40:477-80. [ Links ]
16. Versalovic J, Osato MS, Spakovsky K, Dore MP, Reddy R, Stone GG, et al. Point mutations in the 23S rRNA gene of Helicobacter pylori associated with different levels of clarithromycin resistance. J Antimicrob Chemother. 1997;40:283-6. http://dx.doi.org/10.1093/jac/40.2.283 [ Links ]
17. Kajitani T. The general rules for the gastric cancer study in surgery and pathology. Jpn J Surg. 1981;11:127-39. http://dx.doi.org/10.1007/BF02468884 [ Links ]
18. Dixon MF, Genta RM, Yardley JH, Correa P. Classification and grading of gastritis: The updated Sydney system. Am J Surg Pathol. 1996;20:1161. [ Links ]
19. Kim KS, Kang JO, Eun CS, Han DS, Choi TY. Mutations in the 23S rRNA gene of Helicobacter pylori associated with clarithromycin resistance. J Korean Med Sci. 2002;17:599-603. [ Links ]
20. Taylor DE, Ge Z, Purych D, Lo T, Hiratsuka K. Cloning and sequence analysis of two copies of a 23S rRNA gene from Helicobacter pylori and association of clarithromycin resistance with 23S rRNA mutations. Antimicrob Agents Chemother. 1997;41:2621-8. [ Links ]
21. Figueroa M, Cortés A, Pazos A, Bravo LE. Sensibilidad in vitro a amoxicilina y claritromicina de Helicobacter pylori obtenido de biopsias gástricas de pacientes en zona de bajo riesgo para cáncer gástrico. Biomédica. 2012;32:32-42. http://dx.doi.org/10.1590/S0120-41572012000100005 [ Links ]
22. Kim JJ, Reddy R, Lee M, Kim JG, El-Zaatari FA, Osato MS, et al . Analysis of metronidazole, clarithromycin and tetracycline resistance of Helicobacter pylori isolates from Korea. J Antimicrob Chemother. 2001;47:459-61. http://dx.doi.org/10.1093/jac/47.4.459 [ Links ]
23. Otero W, Trespalacios AA, Otero E. Helicobacter pylori : Tratamiento actual un importante reto en gastroenterología. Rev Colomb Gastroenterol. 2009;24:279-92. [ Links ]
24. Occhialini A, Urdaci M, Doucet-Populaire F, Bébéar CM, Lamouliatte H, Mégraud F. Macrolide resistance in Helicobacter pylori : Rapid detection of point mutations and assays of macrolide binding to ribosomes Antimicrob Agents Chemother. 1997;41:2724-8. [ Links ]
25. Alarcón T, Domingo D, López-Brea M. Antibiotic resistance problems with Helicobacter pylori . Int J Antimicrob Agents. 1999;12:19-26 . http://dx.doi.org/10.1016/S0924-8579(99)00051-5 [ Links ]













