Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157
Biomédica vol.34 supl.1 Bogotá abr. 2014
https://doi.org/10.7705/biomedica.v34i0.1696
COMUNICACIÓN BREVE
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1696
1 Grupo de Resistencia Bacteriana de Bogotá (GREBO), Bogotá, D.C., Colombia
2 Hospital Universitario San Ignacio, Bogotá, D.C., Colombia
3 Hospital El Tunal, Bogotá, D.C., Colombia
4 Instituto Nacional de Cancerología, Bogotá, D.C., Colombia
5 Instituto de Genética Molecular, Universidad El Bosque, Bogotá, D.C., Colombia
Contribución de los autores:
Edna Rodríguez: caracterización fenotípica de los aislamientos, determinación de relación 'clonal' y análisis de resultados.
Sandra Yamile Saavedra: caracterización genética de los aislamientos.
Aura Lucía Leal: obtención de aislamientos y análisis de resultados.
Narda Olarte: obtención de aislamientos y datos demográficos, análisis de resultados, elaboración del manuscrito.
Carlos Álvarez, Alberto Valderrama y Sonia Isabel Cuervo: obtención de aislamientos y datos demográficos, y análisis de resultados.
Javier Escobar: tipificación molecular de aislamientos.
Todos los autores participaron en la escritura del manuscrito.
Recibido: 05/06/13; aceptado: 17/01/14
Introducción. Uno de los principales problemas de salud pública a nivel mundial son las infecciones producidas por enterobacterias resistentes a los carbapenémicos, entre las cuales Klebsiella pneumoniae es uno de los patógenos que con mayor frecuencia causa infecciones en el ámbito hospitalario.
Objetivo. El objetivo del estudio fue describir la diseminación de aislamientos clínicos de K. pneumoniae productores de la enzima KPC-3 recuperados en hospitales de Bogotá.
Materiales y métodos. Se analizaron 82 aislamientos de K. pneumoniae resistentes a antibióticos carbapenémicos recuperados entre el 2008 y el 2010 en 10 hospitales, a los cuales se les realizaron pruebas de detección fenotípica de enzimas por difusión de disco y microdilución, y de detección genotípica por PCR. La determinación de perfiles de sensibilidad frente a 13 antimicrobianos se realizó por métodos automatizados y manuales. La relación genética de los aislamientos se obtuvo por la técnica de PFGE.
Resultados. Este estudio presenta el panorama del comportamiento de las enterobacterias resistentes a los carbapenémicos diseminadas en el curso de tres años en 10 hospitales de la ciudad, con características de resistencia a múltiples familias de antibióticos y pertenecientes a varios grupos de clones, cada uno con diferentes subtipos.
Conclusiones. La diseminación de aislamientos clínicos de K. pneumoniae productores de enzima KPC-3 en Bogotá plantea la necesidad de fortalecer las acciones de vigilancia epidemiológica frente a este tipo de microorganismos y el desarrollo prioritario de actividades específicas de prevención y control de infecciones.
Palabras clave: Klebsiella pneumoniae , farmacorresistencia bacteriana, salud pública, hospitales, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1696
The spread of KPC-3 Klebsiella pneumoniae in hospitals in Bogotá over a three-year period (2008-2010)
Introduction : One of the major worldwide public health problems today are the infections caused by carbapenem-resistant Enterobacteriaceae (CRE), among which carbapenem-resistant Klebsiella pneumoniae (CRKP), constitutes one of the most common pathogens causing nosocomial infection.
Objective: This study was aimed at describing the dissemination of KPC-3 enzyme-producing Klebsiella pneumoniae in clinical isolates from hospitals in Bogotá.
Materials and methods: Eighty-two CRKP isolates collected from 10 hospitals in Bogotá from 2008-2010 were analysed; disk diffusion and microdilution were used for phenotypic detection of enzymes and PCR for genotyping. Automated and manual methods were used for determining profiles for antimicrobial susceptibility testing (AST) with 13 agents. PFGE was used for obtaining the isolates' genetic relationship.
Results: This study gives an overview of CRKP patterns in 10 hospitals in Bogota which were found to present resistance to multiple antibiotic families. The CRKPs were grouped in different clones, each having different subtypes, and were spread in the 10 hospitals over the three-year period (2008-2010).
Conclusions: The dissemination of KPC-3-producing Klebsiella pneumoniae nosocomial isolates in Bogota highlights the need for strengthening epidemiological surveillance against this type of microorganism and the development of specific priority activities for preventing and controlling such infection.
Key words: Klebsiella pneumonia ; drug resistance, bacterial; public health , hospitals, Colombia.
doi: http://dx.doi.org/10.7705/biomedica.v34i0.1696
La resistencia a los antimicrobianos plantea una amenaza cada vez mayor para la salud pública. En el mundo, las cepas resistentes de algunos agentes infecciosos están repercutiendo de forma devastadora en el control de enfermedades. Uno de los principales problemas que han centrado la atención del mundo académico y científico, de las autoridades sanitarias y de la comunidad en general son las infecciones producidas por enterobacterias resistentes a los carbapenémicos o enterobacterias productoras de carbapenemasas, las cuales están emergiendo como importantes agentes patógenos en los centros de atención en salud (1).
Actualmente, Klebsiella pneumoniae resistente a carbapenémicos es una de las especies de enterobacterias resistentes a estos medicamentos más comúnmente aisladas en Estados Unidos, y su importancia radica en que es resistente a casi todos los agentes antimicrobianos disponibles (2). La Organización Panamericana de la Salud (OPS) resalta la importancia de la detección de este mecanismo de resistencia que incrementa de manera importante la morbilidad y la mortalidad en los pacientes afectados y, particularmente, de los infectados por K. pneumoniae , microorganismo que puede ser causante de 20 a 30 % de los casos de neumonías asociadas a la atención en salud en la región y se encuentra entre los tres primeros patógenos aislados en bacteriemias hospitalarias causadas por microorganismos Gram negativos (3). Asimismo, se le han atribuido tasas de mortalidad de más de 50 % (1,4,5).
La clasificación de las betalactamasas se basa tradicionalmente en sus características funcionales o estructura primaria; teniendo en cuenta la secuencia de proteínas se establecen cuatro grupos, A, B, C, y D (6), clasificación en la que se encuentran las carbapenemasas, enzimas capaces de hidrolizar la mayor parte de betalactámicos, incluidos los carbapenémicos; las de clase B o metalo-betalactamasas, por ejemplo, VIM ( Verona Integron-encoded Metallo-betalactamase) o IMP (imipenemasa), que no presentan actividad frente a aztreonam y cuya acción es inhibida con EDTA ( ethylene-diamine-tetra-acetic acid ); las de clase D, que tienen actividad frente a oxacilinas, siendo la OXA-48 la más frecuentemente reportada, y, por último, las de clase A, que suelen ser sensibles a la acción del ácido clavulánico y presentan una menor actividad frente a meropenem que a imipenem, y de las cuales la KPC ( Klebsiella pneumoniae carbapenemase ) es la más amplia mente diseminada en todo el mundo en sus variantes KPC-2 y KPC-3 (7).
La prevalencia actual de los aislamientos produc tores de carbapenemasas todavía se desconoce debido a que en muchos países no se han establecido protocolos para su detección. En Colombia, la magnitud del problema ha sido evidenciada por distintas redes de investigación, que han reportado la presencia de este tipo de enzimas circulando en diferentes hospitales (8) (Saavedra SY, Álvarez CA, Cuervo SI, Olarte N, Escobar JA, Leal AL, et al . Dissemination of a clone KPC-3-producing Klebsiella pneumoniae in hospitals, Bogota, Colombia. 20 th European Congress of Clinical Microbiology and Infectious Diseases . Vienna, Austria, April 10-13, 2010. Clin Microbiol Infect. 2010;16(S3):S361; Saavedra SY, Alvarez CA, Saavedra C, Cuervo SI, Escobar JA, Ovalle MV, et al . Diseminación de aislamientos de Klebsiella pneumoniae productores de KPC en hospitales de Colombia. VII Encuentro Nacional de Investigación en Enfermedades Infecciosas. Infectio. 2010:14(Supl.1):79 ).
El objetivo de este estudio fue describir la diseminación de aislamientos clínicos de K. pneumoniae productores de la enzima KPC-3 recuperados en 10 hospitales de alta complejidad pertenecientes al Grupo de Resistencia Bacteriana de Bogotá (GREBO) durante el periodo de enero de 2008 a diciembre de 2010.
Materiales y métodos
Muestra
En el estudio se utilizaron 82 aislamientos de K. pneumoniae recuperados entre enero de 2008 y diciembre de 2010 en 10 hospitales de tercer nivel de complejidad pertenecientes a la red GREBO de Bogotá, ocho de naturaleza pública y dos privados, con un promedio de egresos anuales superior a 17.000 durante el periodo de estudio y con un número promedio de 250 camas distribuidas en las unidades de cuidados intensivos de adultos y neonatos y en los servicios especializados. Los aislamientos fueron remitidos para ser procesados en el laboratorio de la Universidad Nacional de Colombia con caracterización previa del perfil de sensibilidad establecida mediante los métodos automatizados disponibles en cada institución.
Determinación de perfiles de sensibilidad antimicrobiana
La determinación se realizó para los antibióticos ampicilina y sulbactam, aztreonam, ceftriaxona, ceftazidima, cefotaxima, cefepime, amikacina, gentamicina, trimetoprim-sulfametoxasol, tigeciclina, imipenem y meropenem con los paneles o las tarjetas de los equipos automatizados o semi- automatizados disponibles en cada institución, los cuales incluían Microscan ® (Siemens), Phoenix ® (Beckton Dickinson) y Vitek ® (bioMérieux).
Para determinar la concentración inhibitoria mínima (CIM) del imipenem y el meropenem se utilizó la técnica del E-test ® utilizando la cepa de Escherichia coli ATCC 25922 como control de referencia y siguiendo las recomendaciones del Clinical and Laboratory Standards Institute (CLSI) (9). Para la tigeciclina, los aislamientos se evaluaron con el método de microdilución en caldo (Sensititre ® panels; TREK Diagnostic Systems, Westlake, OH); la interpretación de los resultados se hizo utilizando los puntos de corte establecidos por la Food and Drugs Administration (FDA). Con el mismo método se evaluó la sensibilidad a la colistina y la polimixina en los aislamientos de cada grupo 'clonal' y la interpretación de los resultados se hizo utilizando los puntos de corte establecidos por el European Committee on Antimicrobial Susceptibility Testing (EUCAST).
A todos los aislamientos se les practicó la prueba de difusión con disco (Kirby-Bauer) frente a los mismos antibióticos probados por microdilución. La determinación de la presencia de enzimas del tipo de las betalactamasas de espectro extendido (BLEE) se realizó por prueba confirmatoria de doble disco con ácido clavulánico. La producción de carbapenemasas se detectó inicialmente usando la prueba modificada de Hodge y la prueba de sinergia con meropenem y ácido fenil-borónico como tamización de la expresión de carbapenemasas clase A.
Detección de betalactamasas BLEE y carbapenemasas
La extracción de ADN de los aislamientos se hizo por ebullición; todos los aislamientos se evaluaron para carbapenemasas ( bla KPC) (10), betalactamasas AmpC de plásmidos (11) y betalactamasas BLEE (genes blaTEM , blaSHV y blaCTX-M) (12). En los aislamientos positivos para CTX-M se confirmó el filogrupo por PCR (13). El tipo de carbapenemasa KPC se determinó por ensayo de restricción para diferenciar KPC-2 y KPC-3 (14) y por secuenciación directa del producto de amplificación, el cual fue analizado con el programa BLAST (Basic Local Alignment Search Tool) (http://www.ncbi.nim.gov.co/BLAST).
Electroforesis en gel de campo pulsado
Esta prueba se hizo en los 82 aislamientos seleccionados utilizando la enzima de restricción Xba I con el equipo CHEF-DR ® IIII (Bio-Rad). La electroforesis se corrió con un tiempo inicial de 2,2 segundos, un tiempo final de 63,8 segundos, y una corriente de seis voltios durante 18 horas en tampón TBE (tris-borate-EDTA, pH 8,4) a 14 °C.
Los resultados se analizaron con el método de UPGMA ( Unweighted Pair Group Method using Arithmetic Average ) y el coeficiente de Dice del programa GelCompar, versión 4.0 (BioRad). Los patrones de PFGE que se agruparon con una similitud de más de 75 % se consideraron como pertenecientes al mismo grupo 'clonal'.
Resultados
Durante el periodo de estudio, los 82 aislamientos habían sido recuperados y caracterizados como resistentes a carbapenémicos en los hospitales que los remitieron al GREBO. Los aislamientos se recuperaron de los siguientes tipos de muestra: 32,9 % de orina, 25,6 % de sangre y el restante 41,5 % de cultivos de punta de catéter, aspirado traqueal, herida quirúrgica, hueso, lavado bronquial, líquidos corporales estériles y secreciones peritoneales.
Todos presentaron resistencia a la combinación de ampicilina y sulbactam, a aztreonam, a cefepime, a ceftriaxona, a ciprofloxacina y a gentamicina; 28 % (23/82) fue detectado por los sistemas automatizados como positivo para BLEE; sin embargo, al practicar la prueba confirmatoria se encontraron 18 aislamientos adicionales 41/82 (50 %). Todos los aislamientos fueron positivos en la prueba de Hodge y en la de sinergia con ácido borónico.
En cerca de la mitad de los resultados generados para amikacina, se presentaron discrepancias entre la determinación con los sistemas automatizados y la prueba manual, mostrando un porcentaje de resistencia de 48,7 % (40/82) y de 83 % (68/82), respectivamente. Todos los aislamientos presentaron sensibilidad a tigeciclina.
Los resultados se analizaron con los puntos de corte establecidos por el CLSI, 2010, para interpretar los resultados de sensibilidad antimicrobiana frente a carbapenémicos, y se halló que los paneles con imipenem y meropenem utilizados por los hospitales remitentes de los aislamientos permitieron clasificar 87,8 % de ellos como resistentes, y el restante 12,2 % en la categoría de sensible utilizando equipo automatizado.
La determinación de la CIM para carbapenémicos con la técnica de E-test mostró una CIM mayor de 32 µg/ml para meropenem en 73,8 % de los aislamientos, mientras que para imipenem se presentó una CIM de 8 µg/ml en 19 % y una mayor de 256 µg/ml en 29,4 % de los aislamientos.
Por otra parte, se probaron los aislamientos con ertapenem por difusión, identificándolos como resistentes.
En el cuadro 1 se puede observar la comparación de los resultados obtenidos con los puntos de corte del CLSI, 2012, lo que evidenció que todos los aislamientos resultaron resistentes con las técnicas empleadas. Los antibióticos colistina y polimixina se evaluaron en un representante de cada clon, obteniéndose una CIM menor de 2 µg/ml para cada antibiótico, lo que los ubicó en la clasificación de sensibles.
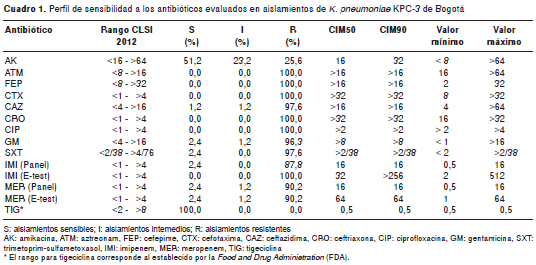
En todos los aislamientos se observó amplificación con el iniciador para KPC, confirmándose posteriormente la variante KPC-3 por ensayo de restricción en la totalidad de los aislamientos; a 19 % de ellos se le realizó ensayo de secuenciación. Todos los aislamientos fueron positivos para bla TEM, 96 % fue positivo para bla SHV y tres aislamientos fueron positivos para CTX-M (dos aislamientos del filogrupo CTX-M-1 y un aislamiento del CTX-M-2). Ningún aislamiento amplificó con los iniciadores para AmpC de plásmidos.
En el ensayo de subtipificación por PFGE de los 82 aislamientos, se encontraron tres grupos 'clonales' diferentes denominados por las primeras letras del alfabeto, A, B y C (figura 1); los grupos 'clonales' A y B mostraron una relación entre ellos de más de 75 %; el grupo A contenía 39 aislamientos distribuidos en 18 subtipos diferentes; el grupo B contenía 25 aislamientos con 14 subtipos, y, por último, el grupo C, con 13 aislamientos y nueve subtipos, se relacionó en más de 70 % con A y B; los subtipos de cada grupo se encontraron a lo largo de los tres años de estudio y en diferentes hospitales. Los aislamientos que no se agruparon en ningún clon correspondían a tres hospitales diferentes y se aislaron en 22008 y 2010.

Discusión
Desde el primer reporte de KPC y su posterior diseminación a otros países a mediados de los años noventa (15,16) son numerosos los informes sobre microorganismos que adquirieron elementos genéticos que codifican para estas enzimas, lo cual ha permitido observar y describir su variado comportamiento frente a los antibióticos utilizados para su contención y control.
En Colombia este fenómeno se reportó desde el 2006, cuando se encontró la variante KPC-2 (8), aunque también se ha presentado la variante KPC-3 en aislamientos relacionados con brotes (17) (Saavedra SY, Álvarez CA, Cuervo SI, Olarte N, Escobar JA, Leal AL, et al . Dissemination of a clone KPC-3-producing Klebsiella pneumoniae in hospitals, Bogotá, Colombia. 20 th European Congress of Clinical Microbiology and Infectious Diseases . Vienna, Austria, April 10-13, 2010. Clin Microbiol Infect. 2010;16(S3):S361; Saavedra SY, Álvarez CA, Saavedra C, Cuervo SI, Escobar JA, Ovalle MV, et al . Diseminación de aislamientos de Klebsiella pneumoniae productores de KPC en hospitales de Colombia. VII Encuentro Nacional de Investigación en Enfermedades Infecciosas. Infectio. 2010:14(Supl.1):79).
Nuestros resultados proporcionan un panorama del comportamiento de las KPC en Bogotá, que se resume en los siguientes tres aspectos.
1) Se evidenció la presencia de clones que estuvieron circulando durante los tres años de estudio en 10 hospitales de Bogotá, lo que demuestra el éxito de este tipo de mecanismos de resistencia para mantenerse en el tiempo y diseminarse.
2) Las CIM elevadas encontradas en nuestros aislamientos contrastan con lo reportado en otros microorganismos portadores de KPC-3 que se caracterizan por presentar resistencia de bajo nivel, por lo menos, frente a los carbapenémicos (18), situación que tiene un impacto clínico, especial mente en lo referente al uso de tratamientos combinados con antibióticos que han demostrado actividad frente a productores de KPC, como los aminoglucósidos (19), aunque en nuestra evaluación la gentamicina, por ejemplo, presentó resistencia en más de 96 % de los aislamientos.
Considerando lo anterior, se hace necesario que los laboratorios de microbiología garanticen sus técnicas actuales y trabajen en su mejoramiento continuo, con el fin de generar resultados que reflejen con mayor certeza datos como los de la CIM, ya que es una de las variables en las que se fundamenta el tratamiento contra estos microorganismos.
Otros antibióticos, como la colistina y la tigeciclina, se utilizan para el tratamiento contra microorganismos portadores de carbapenemasas (20,21), y nuestros aislamientos presentaron sensibilidad in vitro frente a estos dos agentes. Sin embargo, actualmente ya hay reportes de aislamientos portadores de KPC resistentes a estos antibióticos en Estados Unidos (22), Europa (23,24) Asia (25) y, más recientemente, en Suramérica (26).
3) Por último, la variante KPC-3 resultó ser el mecanismo de resistencia a carbapenémicos más frecuente entre los aislamientos estudiados, situación que no es usual en países de América pero sí en Europa y Asia, donde se ha asociado principalmente con pacientes que viajaron a zonas endémicas (27-30).
Los sistemas de determinación de la sensibilidad antimicrobiana, ya fueran los métodos automatizados o los manuales, mostraron diferencias en la detección de enzimas de tipo BLEE y en la determinación de la resistencia a amikacina y a carbapenémicos, lo cual sugiere que el antibiograma debe analizarse en un contexto global en el que se tengan en cuenta todas las variables que puedan influenciar el resultado. Aquí se demuestra que la combinación de las dos técnicas incrementó la detección de este tipo de mecanismos de resistencia.
Este trabajo colaborativo permitió identificar que las instituciones participantes cuentan con la capacidad técnica para llevar a cabo las caracterizaciones fenotípicas de identificación y establecer los perfiles de sensibilidad, así como algunas pruebas de tamización de enzimas. Se debe trabajar para introducir pruebas confirmatorias de los patrones de sensibilidad y contar con el apoyo de laboratorios de referencia en estudios de biología molecular que pueden aportar de forma significativa al análisis, la tipificación, la caracterización de brotes, la detección de clones epidémicos y la diseminación local de microorganismos resistentes.
La diseminación de aislamientos clínicos de K. pneumoniae productores de enzima KPC-3 en Bogotá subraya la necesidad de fortalecer las acciones de vigilancia epidemiológica frente a estos microorganismos y el desarrollo prioritario de actividades específicas de prevención y control de infecciones.
Frente al surgimiento y diseminación de este tipo de resistencia con tan importante impacto clínico, es fundamental establecer políticas institucionales que permitan su contención. Entre ellas se debe incluir, en primer lugar, el desarrollo y fortalecimiento de programas de vigilancia de bacterias resistentes en los hospitales, que incluyan aspectos como la estandarización de técnicas para la detección y la confirmación de estos perfiles en los laboratorios de microbiología, así como la optimización de los procesos y flujos de información con los laboratorios de referencia. En los sistemas de vigilancia institucional se deben definir los microorganismos prioritarios con su marcador de resistencia, por ejemplo, SARM, Klebsiella KPC, Acinetobacter resistente a carbapenémicos, o Clostriduim difficile, de acuerdo con la epidemiologia local.
En segundo lugar, deben desarrollarse y fortalecerse los programas de control de infecciones causadas por microorganismos multirresistentes que estén orientados a la prevención de la diseminación y, asimismo, implementar estrategias basadas en la información científica disponible que sean costo-efectivas, como la higiene de las manos, las precauciones de aislamiento, la educación, la desinfección de áreas, los programas de gerencia de antibióticos, la retroalimentación institucional e interinstitucional y la búsqueda de alternativas o mecanismos orientados al cambio de cultura con relación a la aplicación permanente de medidas de prevención que cuenten con mayor respuesta y con el compromiso administrativo tanto de las instituciones como de los entes de verificación y control, de manera que este problema sea visto como prioridad de salud pública en nuestro país.
Nuestro reconocimiento a los hospitales pertene cientes al GREBO.
Los autores de este manuscrito declaran que no hay conflicto de intereses.
Este proyecto se financió con recursos del GREBO.
Correspondencia: Edna Catering Rodríguez, Grupo de Resistencia Bacteriana de Bogotá (GREBO), Calle 31 Nº 33-38, 2º piso, Bogotá, D.C., Colombia Teléfono: 244 1508 edcrodriguezca@unal.edu.co
1. Schwaber MJ, Carmeli Y. Carbapenem-resistant Enterobacteriaceae: A potential threat. JAMA. 2008;24: 2911-3. http://dx.doi.org/10.1001/jama.2008.896 [ Links ]
2. Lledo W, Hernández M, López E, Molinari OL, Soto RQ, Hernández E, et al. Guidance for control of infections with carbapenem-resistant or carbapenemase-producing Enterobacteriaceae in acute care facilities. MMWR. 2009; 58:256-60. [ Links ]
3. Lizaso D, Aguilera C. Epidemiología y factores de riesgo de mortalidad de las bacteriemias intrahospitalarias por bacilos gramnegativos. Rev Chil Infectol. 2008;5:368-73. http://dx.doi.org/10.4067/S0716-10182008000500010 [ Links ]
4. Borer A, Saidel-Odes L, Riesenberg K, Eskira S, Peled N, Nativ R, et al. Attributable mortality rate for carbapenem-resistant Klebsiella pneumoniae bacteremia. Infect Control Hosp Epidemiol. 2009;10: 972-6. http://dx.doi.org/10.1086/605922 [ Links ]
5. Patel G, Huprikar S, Factor SH, Jenkins SG, Calfee DP. Outcomes of carbapenem-resistant Klebsiella pneumoniae infection and the impact of antimicrobial and adjunctive therapies. Infect Control Hosp Epidemiol. 2008;12:1099- 1106. http://dx.doi.org/10.1086/592412 [ Links ]
6. Bush K, Jacoby GA. Updated functional classification of beta- lactamases. Antimicrob Agents Chemother. 2010;54:969- 76. http://dx.doi.org/10.1128/AAC.01009-09 [ Links ]
7. Muñoz-Price LS, Poirel L, Bonomo RA, Schwaber MJ, Daikos GL, Cormican M, et al. Clinical epidemiology of the global expansion of Klebsiella pneumoniae carbapenemases. Lancet Infect Dis. 2013;13:785-96. http://dx.doi.org/10.1016/S1473-3099(13)70190-7 [ Links ]
8. Villegas MV, Lolans K, Correa A, Suárez CJ, López JA, Vallejo M, et al. First detection of the plasmid-mediated class A carbapenemase KPC-2 in clinical isolates of Klebsiella pneumoniae from South America. Antimicrob Agents Chemother. 2006;50:2880-2. http://dx.doi.org/10.1128/AAC.00186-06 [ Links ]
9. Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing: Nineteenth information supplement. M100 S-19. Wayne, PA: CLSI; 2009. [ Links ]
10. Bradford PA, Bratu S, Urban C, Visalli M, Mariano N, Landman D, et al. Emergence of carbapenem-resistant Klebsiella species possessing the class A carbapenem- hydrolyzing KPC-2 and inhibitor-resistant TEM-30 beta- lactamases in New York City. Clin Infect Dis. 2004;39:55-60. http://dx.doi.org/10.1086/421495 [ Links ]
11. Pérez-Pérez FJ, Hanson ND. Detection of plasmid-mediated AmpC beta-lactamase genes in clinical isolates by using multiplex PCR. J Clin Microbiol. 2002;40:2153-62. http://dx.doi.org/10.1128/JCM.40.6.2153-2162.2002 [ Links ]
12. Monstein HJ, Ostholm-Balkhed A, Nilsson MV, Nilsson M, Dornbusch K, Nilsson LE. Multiplex PCR amplification assay for the detection of blaSHV, blaTEM and blaCTX-M genes in Enterobacteriaceae. APMIS. 2007;115:1400-8. http://dx.doi.org/10.1111/j.1600-0463.2007.00722.x [ Links ]
13. Woodford N, Fagan EJ, Ellington MJ. Multiplex PCR for rapid detection of genes encoding CTX-M extended- spectrum (beta)-lactamases. J Antimicrob Chemother. 2006; 57:154-5. http://dx.doi.org/10.1093/jac/dki412 [ Links ]
14. Cole JM, Schuetz AN, Hill CE, Nolte FS. Development and evaluation of a real-time PCR assay for detection of Klebsiella pneumoniae carbapenemase genes. J Clin Microbiol. 2009;47:322-6. http://dx.doi.org/10.1128/JCM.01550-08 [ Links ]
15. Yigit H, Queenan AM, Anderson GJ, Domenech-Sánchez A, Biddle JW, Steward CD, et al. Novel carbapenem- hydrolyzing beta-lactamase, KPC-1, from a carbapenem- resistant strain of Klebsiella pneumoniae. Antimicrob Agents Chemother. 2001;45:1151-61. http://dx.doi.org/10.1128/AAC.45.4.1151-1161.2001 [ Links ]
16. Samra Z, Ofir O, Lishtzinsky Y, Madar-Shapiro L, Bishara J. Outbreak of carbapenem-resistant Klebsiella pneumoniae producing KPC-3 in a tertiary medical centre in Israel. Int J Antimicrob Agents. 2007;30:525-9. http://dx.doi.org/10.1016/j.ijantimicag.2007.07.024 [ Links ]
17. López JA, Correa A, Navon-Venezia S, Correa AL, Torres JA, Briceño DF, et al. Intercontinental spread from Israel to Colombia of a KPC-3-producing Klebsiella pneumoniae strain. Clin Microbiol Infect. 2011;17:52-6. http://dx.doi.org/10.1111/j.1469-0691.2010.03209.x [ Links ]
18. Mojica MF, Correa A, Vargas DA, Maya JJ, Montealegre MC, Rojas LJ, et al. Molecular correlates of the spread of KPC-producing Enterobacteriaceae in Colombia. Int J Antimicrob Agents. 2012;40:277-9. http://dx.doi.org/10.1016/j.ijantimicag.2012.05.006 [ Links ]
19. Navon-Venezia S, Leavitt A, Schwaber MJ, Rasheed JK, Srinivasan A, Patel JB, et al. First report on a hyperepidemic clone of KPC-3-producing Klebsiella pneumoniae in Israel genetically related to a strain causing outbreaks in the United States. Antimicrob Agents Chemother. 2009;53:818-20. http://dx.doi.org/10.1128/AAC.00987-08 [ Links ]
20. Guzmán-Blanco M, Labarca JA, Villegas MV, Gotuzzo E. Extended spectrum beta-lactamase producers among nosocomial Enterobacteriaceae in Latin America. Braz J Infect Dis. 2014. http://dx.doi.org/10.1016/j.bjid.2013.10.005 [ Links ]
21. Petrosillo N, Giannella M, Antonelli M, Antonini M, Barsic B, Belancic L, et al. Colistin-glycopeptide combination in critically ill patients with Gram negative infection: The clinical experience. Antimicrob Agents Chemother. 2013. http://dx.doi.org/10.1128/AAC.00871-13 [ Links ]
22. Bratu S, Tolaney P, Karumudi U, Quale J, Mooty M, Nichani S, et al. Carbapenemase-producing Klebsiella pneumoniae in Brooklyn, NY: Molecular epidemiology and in vitro activity of polymyxin B and other agents. J Antimicrob Chemother. 2005;56:128-32. http://dx.doi.org/10.1093/jac/dki175 [ Links ]
23. Zarkotou O, Pournaras S, Voulgari E, Chrysos G, Prekates A, Voutsinas D, et al. Risk factors and outcomes associated with acquisition of colistin-resistant KPC- producing Klebsiella pneumoniae : A matched case-control study. J Clin Microbiol. 2010;48:2271-4. http://dx.doi.org/10.1128/JCM.02301-09 [ Links ]
24. Toth A, Damjanova I, Puskas E, Jánvári L, Farkas M, Dobák A, et al. Emergence of a colistin-resistant KPC-2-producing Klebsiella pneumoniae ST258 clone in Hungary. Eur J Clin Microbiol Infect Dis. 2010;29:765-69. http://dx.doi.org/10.1007/s10096-010-0921-3 [ Links ]
25. Suh JY, Son JS, Chung DR, Peck KR, Ko KS, Song JH. Nonclonal emergence of colistin-resistant Klebsiella pneumoniae isolates from blood samples in South Korea. Antimicrob Agents Chemother. 2010;54:560-2. http://dx.doi.org/10.1128/AAC.00762-09 [ Links ]
26. Nastro M, Carranza N, Aprigliano F, Saposnik E, Barberis C, García S, et al. Emergence of colistin-resistant Klebsiella pneumoniae. Microbiological and epidemio- logical characterization of the isolates producing and non- producing KPC-type carbapenemase. Rev Argent Microbiol. 2013;45:185-90. [ Links ]
27. Robustillo A, Díaz-Agero C, Sánchez T, Ruiz-Garbajosa P, Pita MJ, Monge V. Emergence and outbreak of carbapenemase-producing KPC-3 Klebsiella pneumoniae in Spain, September 2009 to February 2010: Control measures. Euro Surveill. 2012;17:20086. [ Links ]
28. Migliavacca R, Nucleo E, Asticcioli S, Casari E, Bracco S, Sironi MC. Multifocal diffusion of a KPC-3 producing ST512 K. pneumoniae clone in Northern Italy. New Microbiol. 2013;36:109-10. [ Links ]
29. Cuzon G, Naas T, Demachy MC, Nordmann P . Nosocomial outbreak of Klebsiella pneumoniae harbouring bla (KPC-3) in France subsequent to a patient transfer from Italy. Int J Antimicrob Agents. 2012;39:448-9. http://dx.doi.org/10.1016/j.ijantimicag.2012.01.008 [ Links ]
30. Miriagou V, Cornaglia G, Edelstein M, Galani I, Giske CG, Gniadkowski M. Acquired carbapenemases in Gram-negative bacterial pathogens: Detection and surveillance issues. Clin Microbiol Infect. 2010;16:112-22. http://dx.doi.org/10.1111/j.1469-0691.2009.03116.x [ Links ]













