Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Biomédica
versão impressa ISSN 0120-4157
Biomédica vol.35 no.4 Bogotá out./dez. 2015
https://doi.org/10.7705/biomedica.v35i4.2610
ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v35i4.2610
1 Línea de Epidemiología Molecular Bacteriana, Grupo de Microbiología Molecular y Grupo de Investigación en Microbiología Básica y Aplicada (MICROBA), Escuela de Microbiología, Universidad de Antioquia, Medellín, Colombia
2 IPS Universitaria, Clínica León XIII, Medellín, Colombia
Contribución de los autores:
Astrid Vanessa Cienfuegos y Judy Natalia Jiménez: diseño de los experimentos y análisis de los datos
Patricia María Sierra: recopilación de los datos
Ana María Ocampo y Carlos Andrés Vargas: participación en todas las etapas del estudio
Todos los autores participaron en la escritura del manuscrito.
Recibido: 19/11/14; aceptado: 25/05/15
Introducción. La resistencia a los carbapenémicos constituye una seria amenaza para la salud pública a nivel mundial, ya que estos antibióticos son una de las últimas opciones terapéuticas contra las bacterias multirresistentes. La caracterización molecular de los brotes causados por bacterias resistentes aporta información relevante para el diseño de estrategias de control de infecciones.
Objetivo. Describir las características moleculares de un brote de Klebsiella pneumoniae resistente a carbapenémicos ocurrido en un hospital de alto nivel de complejidad de Medellín entre 2010 y 2011.
Materiales y métodos. A partir de una colección de cepas del brote ocurrido en la institución hospitalaria, se recuperaron 84 aislamientos de 32 pacientes infectados y 52 colonizados. La identificación y la sensibilidad de los aislamientos se establecieron mediante el sistema Vitek2 ® . La detección de carbapenemasas se hizo mediante el test de Hodge modificado y usando la reacción en cadena de la polimerasa. La relación genética entre los aislamientos se evaluó mediante electroforesis en gel de campo pulsado y tipificación de secuencias de locus múltiple.
Resultados. Todos los aislamientos analizados fueron multirresistentes; el análisis molecular reveló que todos eran portadores del gen bla KPC-3 . El análisis genético mostró que los aislamientos de pacientes infectados y colonizados (58/64 aislamientos) estaban estrechamente relacionados (>80 %) y pertenecían al linaje ST258.
Conclusión. Mediante el empleo de técnicas de tipificación molecular fue posible confirmar un brote ocasionado por K. pneumoniae ST258 portador del bla KPC-3 con un perfil de multirresistencia, el cual había sido asociado a uno anterior ocurrido en otro hospital de Medellín. El ST258 es un clon de alto riesgo presente a nivel mundial, lo que debe alertar sobre la posible diseminación de resistencia en el país. El empleo de herramientas moleculares en la vigilancia epidemiológica, es útil para evaluar la diseminación de microorganismos de interés en salud pública.
Palabras clave: Klebsiella pneumoniae , farmacorresistencia microbiana , brotes de enfermedades, carbapenémicos, betalactamasas, epidemiología molecular, tipificación molecular.
doi: http://dx.doi.org/10.7705/biomedica.v35i4.2610
Molecular characterization of an outbreak of carbapenem-resistant Klebsiella pneumoniae in a tertiary care hospital in Medellín, Colombia
Introduction: Resistance to carbapenems is considered to represent a serious threat to public health at the global level, since these antibiotics are one of the last therapeutic options for the treatment of multidrug-resistant bacteria. Molecular characterization of outbreaks due to resistant bacteria provides information that can be used in the design of infection control strategies.
Objective: To describe the molecular characteristics of an outbreak of carbapenem-resistant Klebsiella pneumoniae that occurred in a tertiary care hospital in Medellín in 2010-2011.
Materials and methods: Eighty-four isolates were obtained from a collection of strains associated with the hospital outbreak, of which 32 were from patients infected at that time and 52 were carriers. Identification and susceptibility of the isolates was performed using Vitek2 ® . Carbapenemases were detected using a modified Hodge test and polymerase chain reaction. Genetic relationships between the isolates were evaluated using pulsed field gel electrophoresis and multiple locus sequence typing.
Results: All the isolates analyzed were multidrug resistant; molecular analysis revealed that all harbored bla KPC-3 . The genetic analysis showed that 58/64 of the isolates from both infected and colonized patients were closely related (Dice similarity index >80%) and belonged to the ST258 lineage.
Conclusion: Using molecular typing techniques it was possible to confirm the occurrence of an outbreak caused by K. pneumoniae ST258, a carrier of bla KPC-3 with a multidrug-resistant profile which had been associated with a previous outbreak in another hospital in the city of Medellín. ST258 is a high risk clone at the global level, demonstrating the potential for dissemination of resistance in this country. Implementation of molecular tools in support of epidemiological surveillance is useful for evaluating the spread of microorganisms of public health significance.
Key words: Klebsiella pneumonia , drug resistance , microbial; carbapenems, beta-lactamases, disease outbreaks, molecular epidemiology, molecular typing.
doi: http://dx.doi.org/10.7705/biomedica.v35i4.2610
La resistencia a carbapenémicos en bacilos Gram negativos es un problema creciente a nivel mundial. Se ha reportado que la frecuencia de aislamientos de Klebsiella pneumoniae resistente a carbapenémicos en Colombia (25,4 %) casi triplica los porcentajes encontrados en otros países, como Argentina, Brasil, México y Chile (4, 4,9, 7 y 8,9 %, respectivamente) (1). El principal mecanismo de resistencia a este grupo de antibióticos en bacilos Gram negativos, ha sido la presencia de carbapenemasas, del tipo de Klebsiella pneumoniae productor de carbapenemasas (KPC) (2-5). Esta enzima se reportó por primera vez en Carolina del Norte, Estados Unidos, en 1996, y actualmente se ha registrado en más de 20 países alrededor del mundo (6-9).
El primer reporte de K. pneumoniae productor de carbapenemasas en Colombia y Suramérica ocurrió en el año 2005, en dos aislamientos recolectados en dos hospitales diferentes de Medellín (10). Posteriormente, se presentó el primer reporte de un brote causado por esta bacteria en el país, entre 2007 y 2008, también en un hospital de Medellín. Durante este brote se encontraron dos clones principales, el primero portador del bla KPC-3 y el segundo portador del bla KPC-2 (11).
Varios estudios posteriores en el país han demostrado la diseminación del gen bla KPC en enterobacterias como Enterobacter cloacae , Serratia marcescens , Escherichia coli , Klebsiella ornithinolytica , Klebsiella oxytoca y en bacilos Gram negativos no fermentadores, como Pseudomonas aeruginosa (12,13). En estos aislamientos el bla KPC-2 ha sido la variante predominante, mientras que el bla KPC-3 se ha restringido a clones particulares de K. pneumoniae (12,13).
Dichos hallazgos han llevado a que hoy Colombia se considere como un país endémico para organismos productores de carbapenemasas (6). Resulta interesante que su diseminación global se haya relacionado principalmente con el linaje genético ST258 y sus variantes cercanas, incluidas en el grupo clonal GC258 (9). El GC258 es de gran importancia epidemiológica a nivel mundial, puesto que se ha asociado a más del 70 % de los brotes causados por K. pneumoniae productora de carbapenemasas en Estados Unidos y a cerca del 90 % de las infecciones en Israel, y es el principal clon asociado a la aparición y diseminación de K. pneumoniae productor de carbapenemasas en Europa (8,9,14). Este clon también se ha descrito en Colombia asociado a un brote en una institución hospitalaria de Medellín, durante 2007 y 2008 (11).
Debido a la creciente diseminación de microorganismos resistentes en el ámbito hospitalario, es necesario fortalecer la capacidad de vigilancia epidemiológica de los laboratorios para la detección y el control de la resistencia mediante métodos de biología molecular (15), los cuales mejoran el diagnóstico fenotípico dada su mayor sensibilidad en la detección de los mecanismos de resistencia y mayor poder de resolución para establecer las relaciones genéticas entre los microorganismos. La caracterización molecular permite obtener información detallada sobre el comportamiento, la evolución y la diseminación de microorganismos de importancia en salud pública (16,17), con el objeto de diseñar estrategias que permitan el control de la resistencia a los antibióticos.
En este marco, el objetivo de este trabajo fue la caracterización molecular de un brote de K. pneumoniae resistente a carbapenémicos ocurrido en un hospital de alto nivel de complejidad de Medellín entre 2010 y 2011.
Descripción del brote
El primer caso de K. pneumoniae resistente a carbapenémicos en la institución hospitalaria, se presentó en julio de 2010 en un paciente de 62 años con una infección del sitio operatorio. En el transcurso del mes de julio y en los meses siguientes, se registraron nuevos casos (figura 1). Las tasas de infección por K. pneumoniae resistente a carbapenémicos en 1.000 días de estancia hospitalaria fueron de 0,12 en 2010 y de 0,07 en 2011. La institución implementó la búsqueda activa de portadores y se encontraron tasas de colonización en 1.000 días de estancia, de 0,20 en 2010 y de 0,32 en 2011.
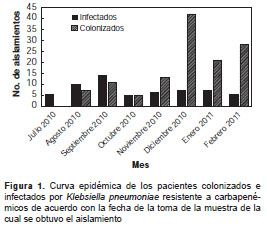
A partir de los primeros tres casos de julio, el comité de control y prevención de infecciones de la institución hospitalaria tomó medidas inmediatas, con el fin de detener la cadena de transmisión del agente causal, conocer y modificar los factores de riesgo, y así, aplicar una intervención adecuada. La estrategia se basó en el establecimiento de cohortes entre los pacientes y el personal, el reforzamiento de la higiene de manos y la adopción de precauciones extremas de aislamiento por contacto, cuyo cumplimiento se supervisó cuidadosamente, la búsqueda activa de colonizados en áreas de alto riesgo (unidades de cuidados intensivos y de cuidados intermedios, de hematología, nefrología, trasplantes y urgencias), y la intensificación de la educación general y por competencias en las áreas afectadas.
Materiales y métodos
Tipo de estudio
Se hizo un estudio transversal.
Población de estudio
Esta incluyó los pacientes involucrados en el brote ocurrido en una unidad de cuidados intensivos dotada con 32 camas y en otras salas de hospitalización de una institución de alto nivel de complejidad de Medellín durante 2010 y 2011. El brote afectó a 59 pacientes infectados y 127 colonizados por K. pneumoniae resistente a carbapenémicos caracterizados de acuerdo con los criterios de los Centers for Disease Control and Prevention (18).
Recolección de aislamientos del brote e información clínico-epidemiológica
Todos los aislamientos obtenidos de los pacientes infectados y colonizados afectados por el brote, se sometieron a criogenia y se almacenaron. Sin embargo, solo fue posible recuperar en cultivo 84 de los 186 aislamientos de K. pneumoniae resistentes a carbapenémicos para la tipificación molecular. Además, se analizó la información clínico-epidemiológica de los pacientes, incluidas las características demográficas y los factores de riesgo descritos en la literatura científica para la adquisición de dicho microorganismo.
Identificación bacteriana y pruebas de sensibilidad
La identificación de los aislamientos y las pruebas de sensibilidad se hicieron de acuerdo con los protocolos estandarizados para el diagnóstico de K. pneumoniae en la institución hospitalaria y utilizando el sistema Vitek ® (bioMérieux Clinical Diagnostics).
Se analizaron los perfiles de resistencia, los cuales incluyeron las cefalosporinas de amplio espectro (ceftriaxona, ceftazidima, cefepime), los carbapenémicos (ertapenem, imipenem, meropenem), los aminoglucósidos, (amikacina, gentamicina), las quinolonas (ácido nalidíxico y ciprofloxacina) y la tigeciclina.
Detección de carbapenemasas
La confirmación fenotípica de la producción de carbapenemasas se hizo mediante el test de Hodge, según las recomendaciones del Clinical and Laboratory Standards Institute (CLSI) (19),
y mediante la amplificación de los genes bla NDM, bla VIM, bla IMP y bla OXA-48 por reacción en cadena de la polimerasa múltiple convencional (PCR) (20,21); además, se empleó PCR en tiempo real para la detección simultánea de las variantes del gen bla KPC (22).
Tipificación molecular mediante electroforesis en gel de campo pulsado
La preparación del ADN se hizo según un protocolo previamente descrito (23), empleando la enzima de restricción Xba I. Para el corrido electroforético se utilizó el sistema Chef-DR III (Bio-Rad ® ). El grado de relación de los aislamientos se analizó mediante el programa BioNumerics, versión 6.0 (Applied Maths), usando el coeficiente de similitud de Dice, y el método de Unweighted Pairs Geometric-Matched Analysis (UPGMA) para la construcción de dendrogramas con una tolerancia del 1 %. Los patrones agrupados con una similitud de más del 80 % según el coeficiente de Dice, se consideraron estrechamente relacionados o como pertenecientes al mismo grupo clonal.
Tipificación por secuenciación de locus múltiples
La tipificación ( Multilocus Sequence Typing , MLST) se hizo en aislamientos seleccionados del grupo más representativo generado mediante la PFGE. El procedimiento se ajustó al protocolo descrito por Diancourt, et al . (24). Para la comparación y la asignación de alelos y de tipo de secuencia, se empleó la base de datos centralizada de MLST (http://www.pasteur.fr/recherche/genopole/PF8/mlst/Kpneumoniae.html).
Plan de análisis
Se empleó la distribución de frecuencias absolutas y relativas para el análisis de variables cualitativas y, para las cuantitativas, medidas de resumen. Posteriormente, se compararon las características de los pacientes infectados y colonizados mediante una prueba de ji al cuadrado de independencia o prueba exacta de Fisher. El análisis de las variables se hizo con el programa estadístico SPSS ®, versión 21,0 (25).
Consideraciones éticas
Este proyecto recibió el aval del Comité de Bioética para Investigación en Humanos de la Sede de Investigación Universitaria de la Universidad de Antioquia (CBE-SIU), según consta en el acta de aprobación Nº 11-35-359.
Resultados
Características demográficas y clínicas de los pacientes
En el cuadro 1 se encuentran las principales características clínicas de los 84 pacientes analizados: 32 infectados (38,1 %) y 52 colonizados (61,9 %).
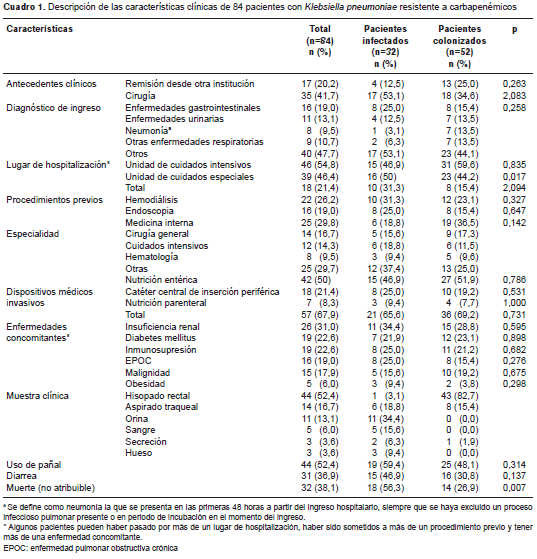
La mayoría eran hombres (56 %), de edad avanzada (mediana de 66 años; rango intercuartílico de 55 a 78), con diversas enfermedades subyacentes (65,6 % de los infectados y 69,2 % de los colonizados), entre las cuales la insuficiencia renal (31 %) y la diabetes mellitus (23 %) fueron las más frecuentes. La mayor parte de los pacientes estuvieron hospitalizados en la unidad de cuidados intensivos (46,9 % de los infectados y 59,6 % de los colonizados).
Se observó la asociación entre la infección o colonización y las características clínicas y epidemiológicas evaluadas. En el grupo de los infectados se observó el uso de pañal y la presencia de diarrea con mayor frecuencia (59,4 % Vs . 46,9 %). Las infecciones más frecuentes fueron la infección urinaria (40,6 %), la infección del sitio operatorio (15,6 %) y la neumonía adquirida en el hospital (21,9 %). También, se presentaron infecciones de la vía biliar e infecciones abdominales, pero con menor frecuencia (6,3 y 9,4 %, respectivamente).
Los pacientes colonizados habían sido remitidos desde otros hospitales con mayor frecuencia que los infectados (25,0 Vs. 12,5 %), mientras que el antecedente de cirugía durante el período de hospitalización fue más común en los infectados que en los colonizados (53,1 % Vs. 34,6 %). Con respecto al tratamiento, la gentamicina (65,2 %) y la tigeciclina (62,4 %) fueron los agentes usados con mayor frecuencia, seguidos por la colistina (31,3 %) y la rifampicina (18,8 %). La mortalidad por cualquier causa en los pacientes analizados fue de 38,1 %.
Perfiles de resistencia y pruebas de sensibilidad
Todos los aislamientos fueron multirresistentes, es decir, presentaron resistencia a, por lo menos, tres clases de agentes antimicrobianos (26). Se obtuvieron ocho patrones de resistencia, entre los cuales se encontraron dos patrones predominantes que agruparon el 66 % (n=49) y el 23 % de los aislamientos (n=17), respectivamente. Al comparar la distribución de los perfiles de los aislamientos de los pacientes
infectados y los colonizados, no se observaron diferencias. El perfil de resistencia que agrupó la mayor cantidad de cepas correspondió al de ceftriaxona, ceftazidime, cefepime, ertapenem, imipenem, meropenem, aztreonam, amikacina, ácido nalidíxico y ciprofloxacina, con el 63,6 % de los aislamientos infectados (n=14) y el 67,3 % de los colonizados (n=35) (figura 2).

Todos los aislamientos evaluados fueron positivos en el test de Hodge modificado, lo cual indicó la presencia de carbapenemasas.
Caracterización molecular de las carbapenemasas y tipificación molecular
El análisis molecular reveló que el 100 % de los aislamientos eran portadores de bla KPC-3 . No se detectaron las otras carbapenemasas evaluadas.
La tipificación molecular mediante PFGE reveló la presencia de cuatro pulsotipos. Se obtuvo un pulsotipo dominante que agrupó al 94 % de los aislamientos (52 de 55 evaluados) con una similitud de más de 80 %, y en el que se encontraron tres grupos de aislamientos con una similitud de más de 85 %, dos de los cuales incluyeron solo aislamientos provenientes de pacientes colonizados y el tercero, solo aislamientos provenientes de pacientes infectados (figura 3).
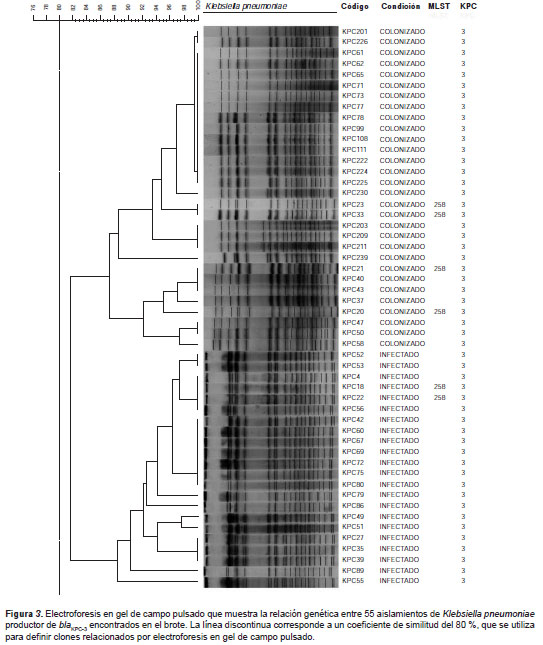
Además, la tipificación mediante MLSTde 20 aislamientos, reveló que todas las cepas pertenecían al linaje ST258.
Discusión
En el estudio se confirmó mediante técnicas de tipificación molecular un brote de K. pneumoniae ST258 portador de bla KPC-3 con un perfil de multirresistencia (resistencia a todos los betalactámicos: ceftriaxona, ceftazidima, cefepima, ertapenem, imipenem, meropenem y aztreonam, y a amikacina y ciprofloxacina). Aunque la carbapenemasa se ha encontrado en varios clones de K. pneumoniae (27), así como en otras especies de bacterias Gram negativas (4,5,28), una proporción significativa de la carga global de esta carbapenemasa se ha asociado conla diseminación clonal del linaje ST258 (4,6,29). Se han encontrado cepas ST258 de K. pneumoniae productoras de carbapenemasas en hospitales en todo el mundo, por lo que se cree que la cepa ha desempeñado un papel importante en la difusión de estas enzimas (29).
Por otro lado, aunque se han reportado diferentes variantes de la carbapenemasa de K. pneumoniae , la mayoría de las cepas ST258 producen KPC-3 (3). La multirresistencia en estas cepas se ha asociado a la presencia de un transposón del tipo Tn3, el Tn4401, que además de contener los genes bla KPC puede contener genes que codifican enzimas modificadoras de aminoglucósidos y genes Qnr que le brindan resistencia a las quinolonas (30,31). La capacidad de inserción de este transposón en diversos plásmidos de bacterias Gram negativas, explica en gran medida el potencial de transmisión entre diferentes especies y la diseminación geográfica de estos agentes determinantes de resistencia. Vale la pena reiterar que, a pesar de la estrecha relación genética entre los aislamientos de pacientes infectados y colonizados, fue posible distinguir tres grupos con gran similitud (85 %), uno de los cuales correspondió a los aislamientos de los pacientes infectados.
En Medellín se reportó por primera vez en Colombia un brote ocurrido en otra institución causado por cepas con caraterísticas similares (la ST258 portadora del bla KPC-3 multirresistente); el caso índice fue un paciente que viajó desde Israel, país en el cual este clon se ha asociado a múltiples brotes (11). Asimismo, varios estudios de vigilancia llevados a cabo en Israel demostraron que la prevalencia del clon ST258 sigue en aumento, pues pasó de 65 % en el 2008 hasta el 80 % en el 2013 (32).
Por otra parte, en Latinoamérica también se ha reportado la diseminación del clon de K. pneumoniae productor de carbapenemasas pertenecientes al ST258 en Argentina, Brasil y Uruguay (33-35). Estos hallazgos sugieren que la diseminación de estas cepas en las instituciones hospitalarias puede ocurrir, entre otras causas, debido al movimiento frecuente de pacientes y de personal de salud entre los hospitales, por lo que los pacientes colonizados podrían tener un papel importante. La transferencia entre unidades hospitalarias se ha descrito como un factor de riesgo para la presencia de K. pneumoniae resistente a carbapenémicos (36).
Con respecto a las características clínicas, se encontró que el comportamiento fue similar al de otros brotes descritos en la literatura científica, en los cuales la mayor parte de los casos se presentó en la unidad de cuidados intensivos, entre hombres mayores y con enfermedades subyacentes, y la mortalidad no atribuible a K. pneumoniae resistente a carbapeménicos fue superior al 30 %. Se observó la asociación entre la infección o colonización y las características clínicas y epidemiológicas, lo que es explicable teniendo en cuenta que los aislamientos de los pacientes infectados y de los colonizados estaban estrechamente relacionados (80 % de similitud).
La caracterización molecular de los microorganismos resistentes a antibióticos permite fortalecer los proyectos de vigilancia establecidos en las instituciones hospitalarias, y orientar la elección del tratamiento antimicrobiano y las medidas para el control de infecciones. En este sentido, el apoyo a la vigilancia mediante las técnicas de biología molecular es de gran importancia para contener la diseminación de los microorganismos resistentes y evitar amenazas a la seguridad del paciente, así como el aumento de la morbilidad y la mortalidad y de los costos de la atención hospitalaria (16,17).
En este estudio, la utilización de la PCR, y métodos de tipificación como la PFGE y la MLST, permitió la detección de genes específicos asociados con la resistencia, así como el establecimiento del linaje genético y la relación clonal de los aislamientos. Estos resultados evidencian que la implementación de herramientas moleculares para apoyar la vigilancia epidemiológica es útil en la evaluación de la diseminación de microorganismos de interés en salud pública.
Los autores declaran que no existen relaciones económicas ni personales de ninguna índole que pudieran influenciar su juicio respecto a los resultados de la presente investigación. Todos los autores tuvieron acceso irrestricto a los datos y no existió participación de agentes externos en el diseño del estudio, ni en la recopilación, análisis e interpretación de la información.
Estudio financiado por Colciencias, código 111554531404, y la Asociación Colombiana de Infectología, ACIN, Capítulo Antioquia, convocatoria de apoyo a proyectos de investigación, 2012.
Correspondencia: Judy Natalia Jiménez, Escuela de Microbiología, Universidad de Antioquia, Calle 67 N° 53-108, bloque 5, oficina 438, Medellín, Colombia Teléfonos: (574) 219 5485; fax: (574) 219 1007 nataliajiudea@gmail.com
1. Fernández-Canigia L, Dowzicky MJ. Susceptibility of important Gram-negative pathogens to tigecycline and other antibiotics in Latin America between 2004 and 2010. Ann Clin Microbiol Antimicrob. 2012;11:29. http://dx.doi.org/10.1186/1476-0711-11-29 [ Links ]
2. Aloush V, Navon-Venezia S, Seigman-Igra Y, Cabili S, Carmeli Y. Multidrug-resistant Pseudomonas aeruginosa : Risk factors and clinical impact. Antimicrob Agents Chemother. 2006;50:43-8. http://dx.doi.org/10.1128/AAC.50.1.43-48.2006 [ Links ]
3. Arnold RS, Thom KA, Sharma S, Phillips M, Kristie Johnson J, Morgan DJ. Emergence of Klebsiella pneumoniae carbapenemase-producing bacteria. South Med J. 2011;104:40-5. http://dx.doi.org/10.1097/SMJ.0b013e3181fd7d5a [ Links ]
4. Chen LF, Anderson DJ, Paterson DL. Overview of the epidemiology and the threat of Klebsiella pneumoniae carbapenemases (KPC) resistance. Infect Drug Resist. 2012;5:133-41. http://dx.doi.org/10.2147/IDR.S26613 [ Links ]
5. Nordmann P, Cuzon G, Naas T. The real threat of Klebsiella pneumoniae carbapenemase-producing bacteria. Lancet Infect Dis. 2009;9:228-36. http://dx.doi.org/10.1016/S1473-3099(09)70054-4 [ Links ]
6. Muñoz-Price LS, Poirel L, Bonomo RA, Schwaber MJ, Daikos GL, Cormican M, et al . Clinical epidemiology of the global expansion of Klebsiella pneumoniae carbapenemases. Lancet Infect Dis. 2013;13:785-96. http://dx.doi.org/10.1016/S1473-3099(13)70190-7 [ Links ]
7. Yigit H, Queenan AM, Anderson GJ, Domenech-Sánchez A, Biddle JW, Steward CD, et al . Novel carbapenem-hydrolyzing beta-lactamase, KPC-1, from a carbapenem-resistant strain of Klebsiella pneumoniae . Antimicrob Agents Chemother. 2001;45:1151-61. http://dx.doi.org/10.1128/AAC.45.4.1151-1161.2001 [ Links ]
8. Canton R, Akova M, Carmeli Y, Giske CG, Glupczynski Y, Gniadkowski M, et al . Rapid evolution and spread of carbapenemases among Enterobacteriaceae in Europe. Clin Microbiol Infect. 2012;18:413-31. http://dx.doi.org/10.1111/j.1469-0691.2012.03821.x [ Links ]
9. Chen L, Mathema B, Chavda KD, DeLeo FR, Bonomo RA, Kreiswirth BN. Carbapenemase-producing Klebsiella pneumoniae : Molecular and genetic decoding. Trends Microbiol. 2014;22:686-96. http://dx.doi.org/10.1016/j.tim.2014.09.003 [ Links ]
10. Villegas MV, Lolans K, Correa A, Suárez CJ, López JA, Vallejo M, et al . First detection of the plasmid-mediated class A carbapenemase KPC-2 in clinical isolates of Klebsiella pneumoniae from South America. Antimicrob Agents Chemother. 2006;50:2880-2. http://dx.doi.org/10.1128/AAC.00186-06 [ Links ]
11. López JA, Correa A, Navon-Venezia S, Correa AL, Torres JA, Briceño DF, et al . Intercontinental spread from Israel to Colombia of a KPC-3-producing Klebsiella pneumoniae strain. Clin Microbiol Infect. 2011;17:52-6. http://dx.doi.org/10.1111/j.1469-0691.2010.03209.x [ Links ]
12. Mojica MF, Correa A, Vargas DA, Maya JJ, Montealegre MC, Rojas LJ, et al . Molecular correlates of the spread of KPC-producing Enterobacteriaceae in Colombia. Int J Antimicrob Agents. 2012;40:277-9. http://dx.doi.org/10. 1016/j.ijantimicag.2012.05.006 [ Links ]
13. Vanegas JM, Cienfuegos AV, Ocampo AM, López L, del Corral H, Roncancio G, et al . Similar frequencies of Pseudomonas aeruginosa isolates producing KPC and VIM carbapenemases in diverse genetic clones at tertiary-care hospitals in Medellin, Colombia. J Clin Microbiol. 2014;52:3978-86. http://dx.doi.org/10.1128/JCM.01879-14 [ Links ]
14. Deleo FR, Chen L, Porcella SF, Martens CA, Kobayashi SD, Porter AR, et al . Molecular dissection of the evolution of carbapenem-resistant multilocus sequence type 258 Klebsiella pneumoniae . Proc Natl Acad Sci USA. 2014;111:4988-93. http://dx.doi.org/10.1073/pnas.1321364111 [ Links ]
15. Leung E, Weil DE, Raviglione M, Nakatani H, World Health Organization World Health Day Antimicrobial Resistance Technical Working Group. The WHO policy package to combat antimicrobial resistance. Bull World Health Organ. 2011;89:390-2. http://dx.doi.org/10.2471/BLT.11.088435 [ Links ]
16. Miriagou V, Cornaglia G, Edelstein M, Galani I, Giske CG, Gniadkowski M, et al . Acquired carbapenemases in Gram-negative bacterial pathogens: Detection and surveillance issues. Clin Microbiol Infect. 2010;16:112-22. http://dx.doi.org/10.1111/j.1469-0691.2009.03116.x [ Links ]
17. Carmeli Y, Akova M, Cornaglia G, Daikos GL, Garau J, Harbarth S, et al . Controlling the spread of carbapenemase-producing Gram-negatives: Therapeutic approach and infection control. Clin Microbiol Infect. 2010;16:102-11. http://dx.doi.org/10.1111/j.1469-0691.2009.03115.x [ Links ]
18. Horan TC, Andrus M, Dudeck MA. CDC/NHSN surveillance definition of health care-associated infection and criteria for specific types of infections in the acute care setting. Am J Infect Control. 2008;36:309-32. http://dx.doi.org/10.1016/j.ajic.2008.03.002 [ Links ]
19. Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing: Twenty-second informational supplement. CLSI document M100-S22. Wayne, PA: Clinical and Laboratory Standards Institute; 2012. [ Links ]
20. Ellington MJ, Kistler J, Livermore DM, Woodford N. Multiplex PCR for rapid detection of genes encoding acquired metallo-beta-lactamases. J Antimicrob Chemother. 2007;59:321-2. http://dx.doi.org/10.1093/jac/dkl481 [ Links ]
21. Poirel L, Walsh TR, Cuvillier V, Nordmann P. Multiplex PCR for detection of acquired carbapenemase genes. Diagn Microbiol Infect Dis. 2011;70:119-23. http://dx.doi.org/10.1016/j.diagmicrobio.2010.12.002 [ Links ]
22. Chen L, Mediavilla JR, Endimiani A, Rosenthal ME, Zhao Y, Bonomo RA, et al . Multiplex real-time PCR assay for detection and classification of Klebsiella pneumoniae carbapenemase gene (bla KPC) variants. J Clin Microbiol. 2011;49:579-85. http://dx.doi.org/10.1128/JCM.01588-10 [ Links ]
23. Durmaz R, Otlu B, Koksal F, Hosoglu S, Ozturk R, Ersoy Y, et al . The optimization of a rapid pulsed-field gel electrophoresis protocol for the typing of Acinetobacter baumannii , Escherichia coli and Klebsiella spp. Jpn J Infect Dis. 2009;62:372-7. [ Links ]
24. Diancourt L, Passet V, Verhoef J, Grimont PA, Brisse S. Multilocus sequence typing of Klebsiella pneumoniae nosocomial isolates. J Clin Microbiol. 2005;43:4178-82. http://dx.doi.org/10.1128/JCM.43.8.4178-4182.2005 [ Links ]
25. IBM. SPSS Statistics for Windows, version 21.0. Armonk, NY: IBM Corp.; 2012. [ Links ]
26. Paterson DL, Doi Y. A step closer to extreme drug resistance (XDR) in Gram-negative bacilli. Clin Infect Dis. 2007;45:1179-81. http://dx.doi.org/10.1086/522287 [ Links ]
27. Ruiz-Garbajosa P, Curiao T, Tato M, Gijón D, Pintado V, Valverde A, et al . Multiclonal dispersal of KPC genes following the emergence of non-ST258 KPC-producing Klebsiella pneumoniae clones in Madrid, Spain. J Antimicrob Chemother. 2013;68:2487-92. http://dx.doi.org/10.1093/jac/dkt237 [ Links ]
28. Villegas MV, Lolans K, Correa A, Kattan JN, López JA, Quinn JP, et al . First identification of Pseudomonas aeruginosa isolates producing a KPC-type carbapenem-hydrolyzing beta-lactamase. Antimicrob Agents Chemother. 2007;51:1553-5. http://dx.doi.org/10.1128/AAC.01405-06 [ Links ]
29. Kitchel B, Rasheed JK, Patel JB, Srinivasan A, Navon-Venezia S, Carmeli Y, et al . Molecular epidemiology of KPC-producing Klebsiella pneumoniae isolates in the United States: Clonal expansion of multilocus sequence type 258. Antimicrob Agents Chemother. 2009;53:3365-70. http://dx.doi.org/10.1128/AAC.00126-09 [ Links ]
30. Endimiani A, Hujer AM, Pérez F, Bethel CR, Hujer KM, Kroeger J, et al . Characterization of blaKPC -containing Klebsiella pneumoniae isolates detected in different institutions in the Eastern USA. J Antimicrob Chemother. 2009;63:427-37. http://dx.doi.org/10.1093/jac/dkn547 [ Links ]
31. Naas T, Cuzón G, Villegas MV, Lartigue MF, Quinn JP, Nordmann P. Genetic structures at the origin of acquisition of the beta-lactamase blaKPC gene. Antimicrob Agents Chemother. 2008;52:1257-63. http://dx.doi.org/10.1128/AAC.01451-07 [ Links ]
32. Adler A, Hussein O, Ben-David D, Masarwa S, Navon-Venezia S, Schwaber MJ, et al . Persistence of Klebsiella pneumoniae ST258 as the predominant clone of carbapenemase-producing Enterobacteriaceae in post-acute-care hospitals in Israel, 2008-13. J Antimicrob Chemother. 2015;70:89-92. http://dx.doi.org/10.1093/jac/dku333 [ Links ]
33. Andrade LN, Curiao T, Ferreira JC, Longo JM, Climaco EC, Martínez R, et al . Dissemination of blaKPC-2 by the spread of Klebsiella pneumoniae clonal complex 258 clones (ST258, ST11, ST437) and plasmids (IncFII, IncN, IncL/M) among Enterobacteriaceae species in Brazil. Antimicrob Agents Chemother. 2011;55:3579-83. http://dx.doi.org/10.1128/AAC.01783-10 [ Links ]
34. Gómez SA, Pasteran FG, Faccone D, Tijet N, Rapoport M, Lucero C, et al . Clonal dissemination of Klebsiella pneumoniae ST258 harbouring KPC-2 in Argentina. Clin Microbiol Infect. 2011;17:1520-4. http://dx.doi.org/10.1111/j.1469-0691.2011.03600.x [ Links ]
35. Márquez C, Ingold A, Echeverría N, Acevedo A, Vignoli R, García-Fulgueiras V, et al. Emergence of KPC-producing Klebsiella pneumoniae in Uruguay: Infection control and molecular characterization. New Microbes New Infect. 2014;2:58-63. http://dx.doi.org/10.1002/nmi2.40 [ Links ]
36. Gregory CJ, Llata E, Stine N, Gould C, Santiago LM, Vázquez GJ, et al. Outbreak of carbapenem-resistant Klebsiella pneumoniae in Puerto Rico associated with a novel carbapenemase variant. Infect Control Hosp Epidemiol. 2010;31:476-84. http://dx.doi.org/10.1086/651670 [ Links ]













