Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157
Biomédica vol.36 no.1 Bogotá Jan./Mar. 2016
https://doi.org/10.7705/biomedica.v36i1.2702
ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v36i1.2702
1 Laboratorio de Genética Humana, Centro de Medicina Experimental, Instituto Venezolano de Investigaciones Científicas, Caracas, Venezuela
2 Departamento de Bioanálisis, Universidad de Oriente, Cumaná, Venezuela
Contribución de los autores: Greta Rodríguez-Arroyo: concepción y diseño del estudio, extracción del ADN, asignación de genotipos, análisis estadístico, escritura y corrección del manuscrito
Irene Paradisi: asesoría en el diseño, selección de las variables adecuadas para el estudio de los polimorfismos y para las técnicas requeridas, entrenamiento, estandarización y control de calidad para la asignación de genotipos, y supervisión general del grupo de investigación
Merlyn Vívenes-Lugo: supervisión y ejecución de la toma de muestras en el trabajo de campo, recolección de los datos y extracción del ADN
Dinorah Castro-Guerra y Álvaro Rodríguez-Larralde: concepción y diseño del estudio, obtención del financiamiento y supervisión general del grupo de investigación
Álvaro Rodríguez-Larralde: asesoría y orientación en el análisis estadístico
Todos los autores participaron en el análisis e interpretación de los resultados, la discusión crítica y la corrección del manuscrito.
Recibido: 11/02/15; aceptado: 30/07/15
Introducción. La prevalencia del sobrepeso, la obesidad y algunas enfermedades crónicas no transmisibles ha aumentado; sus causas pueden ser genéticas, epigenéticas o ambientales, por lo cual es importante evaluar la variabilidad en estas interacciones.
Objetivo. Analizar las relaciones entre nueve polimorfismos de nucleótido simple de los genes LEP (rs2167270), LDLR (rs885765, rs688, rs5925, rs55903358, rs5742911) y APOA4 (rs5095, rs675, rs5110), y los fenotipos asociados al sobrepeso, la obesidad y otras enfermedades concomitantes.
Materiales y métodos. Se evaluaron parámetros clínicos y antropométricos en 144 sujetos del estado Sucre, Venezuela, 76 hombres y 68 mujeres, con medias de edad de 29,93±8,29 y 32,49±11,15 años, respectivamente. Se hizo la genotipificación de los polimorfismos seleccionados mediante enzimas de restricción; se estudiaron las asociaciones entre genotipo y riesgo, y se compararon los promedios de las medidas antropométricas y bioquímicas previamente ajustadas a variables biológicas y ambientales.
Resultados. Según el índice de masa corporal (IMC), el 38,9 % de los individuos tenía sobrepeso (25≤IMC≤29,9 kg/m2) y el 20,1 %, obesidad (IMC≥30 kg/m2) . Las frecuencias genotípicas y alélicas de los grupos con un índice de masa corporal normal y uno alto (sobrepeso y obesidad) resultaron similares. Solo se encontró asociación entre el genotipo ancestral A/A del rs5742911 y el riesgo alto por los niveles de la lipoproteína de alta densidad o colesterol HDL (OR=2,944, IC95% 1,446-5,996; p=0,003). La diferencia entre los promedios corregidos de colesterol HDL para los genotipos del rs5742911 resultó significativa (p=0,005) (A/A: 41,50±14,81 mg/dl; A/G: 45,00±12,07 mg/dl; G/G: 47,17±9,43 mg/dl).
Conclusión. En la mayoría de las variantes genéticas estudiadas, se registró la asociación con el sobrepeso y la obesidad de los genotipos ancestrales, aunque sin ser significativa. El polimorfismo rs5742911 podría resultar útil como indicador del riesgo de enfermedades crónicas.
Palabras clave: polimorfismo de nucleótido simple, sobrepeso, obesidad, índice de masa corporal, dislipidemias, factores de riesgo, enfermedad crónica, adulto, Venezuela
doi: http://dx.doi.org/10.7705/biomedica.v36i1.2702
LEP, LDLR and APOA4 gene polymorphisms and their relationship with the risk of overweight, obesity and chronic diseases in adults of the State of Sucre, Venezuela
Introduction: Overweight, obesity and some chronic diseases have become more prevalent recently. It is well known that their causes may be genetic, epigenetic, environmental, or a mixture of these.
Objective: To analyze the relationship between nine single nucleotide polymorphisms of genes LEP (rs2167270) , LDLR (rs885765, rs688, rs5925, rs55903358, rs5742911) and APOA4 (rs5095, rs675, rs5110) with obesity-related phenotypes and other comorbidities.
Material and methods: We recruited 144 adults (76 males and 68 females, with average ages of 29.93±8.29 and 32.49±11.15 years, respectively) in the State of Sucre, Venezuela. Clinical and anthropometric parameters were obtained. Genotype-risk associations were studied. We then compared the averages registered for anthropometric and biochemical variables previously adjusted for biological and environmental factors.
Results: According to the body mass index, 38.9% of the individuals in the sample were overweight (25≤BMI≤29.9 kg/m2) and 20.1% were obese (BMI≥30 kg/m2). Genotype and allele frequencies did not differ statistically for groups with normal and high body mass index (overweight plus obesity). The association between LDLR rs5742911 ancestral genotype A/A and high risk condition related to HDL-cholesterol was the only one found to be significant (OR=2.944, 95% CI: 1.446-5.996; p=0.003). The difference in adjusted mean HDL-cholesterol for LDLR rs5742911 genotypes was statistically significant (p=0.005) (A/A: 41.50±14.81 mg/dL; A/G: 45.00±12.07 mg/dL; G/G: 47.17±9.43 mg/dL).
Conclusions: For most of the genetic variants studied, there was an association with the presence of overweight and obesity among ancestral genotype carriers, although this was not statistically significant. The rs5742911 polymorphism may be useful as an indicator of a risk of chronic diseases.
Key words: Polymorphism, single nucleotide; overweight, obesity, body mass index, dyslipidemias, risk factors, chronic disease, adult, Venezuela.
doi: http://dx.doi.org/10.7705/biomedica.v36i1.2702
Se prevé que para el 2020, las enfermedades crónicas no transmisibles del adulto, como las cardiovasculares, la hipertensión arterial sistémica, la diabetes, y la obesidad, entre otras, representarán casi las tres cuartas partes del total de la mortalidad en el ámbito mundial (1,2). En particular, la frecuencia de la obesidad ha aumentado en los países desarrollados y en desarrollo, hecho inquietante por el riesgo que supone para otras enfermedades concomitantes (3). Según datos suministrados por el Instituto Nacional de Nutrición en el 2013, en Venezuela el porcentaje de malnutrición por exceso era de 54,95 %, del cual 29,52 % correspondía al sobrepeso y, 25,43 %, a la obesidad (4), con base en mediciones del índice de masa corporal (IMC, kg/m2) en una muestra representativa de sujetos entre los 18 y 40 años de todo el territorio nacional ( n=10.151). En el estado Sucre este porcentaje de malnutrición por exceso era de 59,03 %, con 36,34 % de sobrepeso y 22,69 % de obesidad, lo que la convierte en una de las regiones del país donde se presenta con mayor frecuencia este tipo de malnutrición (4).
Aun cuando se reconoce la contribución del medio ambiente en el desarrollo del fenotipo obeso, en los últimos años se ha puesto de manifiesto la significativa contribución de los genes en la variación de la masa corporal. Los estudios genéticos han aumentado la comprensión de los mecanismos moleculares involucrados en la regulación del peso corporal, y han revelado que la obesidad debida a trastornos monogénicos es poco frecuente. Por tal motivo, el principal reto de la investigación en este campo es la detección de las variantes genéticas que puedan explicar las formas más comunes de la obesidad humana (5,6).
Entre los genes asociados con fenotipos caracterizados por el sobrepeso y la obesidad, se encuentran el gen de la proteína leptina ( LEP ), la cual actúa como señal de circulación que reduce la ingestión de alimentos e incrementa el gasto de energía mediante la unión a su receptor, lo que activa la vía de la melanocortina hipotalámica y controla los depósitos de grasa corporal, así como el gen del receptor de las lipoproteínas de baja densidad ( LDLR ), que intervienen en el metabolismo de las lipoproteínas, y el gen de la apolipoproteína A-IV (APOA4), que participa en el transporte de las lipoproteínas y es muy importante para el control integral del consumo de alimentos, el metabolismo y la homeostasis de la glucosa (7-11).
Se han descrito varios polimorfismos mononucleotídicos ( Single Nucleotide Polymorphism , SNP ) en los genes mencionados: en el gen LEP , el rs2167270 se ha asociado con la obesidad (12,13 ), el índice de masa corporal (14), la preferencia por el dulce (15), los niveles de actividad física (16) y el control epigenético de factores perinatales, como la obesidad materna y el crecimiento del infante (17).
En el LDLR , los polim orfis mos rs885765, rs688, rs5925, rs55903358 y rs5742911, entre otros, se han utilizado ampliamente en los estudios sobre la asociación de haplotipos con la hipercolesterolemia familiar. Estas cinco variantes se han asociado también con fenotipos relacionados con el sobrepeso y la obesidad, en cinco etnias de amerindios brasileños (18). Por su parte, Zee, et al. , estudiaron la relación del rs688 con la obesidad en pacientes obesos e hipertensos, y encontraron dicha asociación solo en el grupo de los obes os hipertensos (19).
Las variantes rs675 y rs5110 del gen APOA4 son mutaciones con cambio de sentido, es decir, implican cambios de aminoácido en la proteína (Tre367Ser y Gln380His, respectivamente) y, conjuntamente con el rs5095, se ha estudiado su su asociación con lípidos y lipoproteínas (20-23), con el peso corporal, la dieta, los parámetros de la composición y la grasa corporal, así como con el síndrome metabólico (24-28).
En este contexto, el objetivo de la presente investigación fue analizar las relaciones existentes entre los genotipos de nueve polimorfismos y algunos fenotipos asociados al sobrepeso, la obesidad y el riesgo de padecer enfermedades crónicas no transmisibles, en un grupo de individuos originarios del estado Sucre, como un estudio preliminar de orientación exploratoria y descriptiva.
Materiales y métodos
Grupo de estudio
El diseño de investigación partió de la selección de los sujetos sin haberlos clasificados previamente según peso normal, sobrepeso u obesidad, con la finalidad de comparar el fenotipo medido cuantitativamente. Para determinar el tamaño de la muestra, se hicieron simulaciones estadísticas para cada marcador, utilizando las distribuciones genotípicas, así como valores de promedios y desviaciones estándar para el índice de masa corporal (IMC) y el índice cintura-cadera registrados en estudios publicados (15,18,27), ya que dichos valores no se conocían para la población en estudio. Mediante estas pruebas se estableció que una muestra de, al menos, 120 individuos permitiría detectar diferencias significativas entre los promedios de distintos genotipos para estas variables, a pesar de que tal tamaño de muestra no fuese representativo de la población. Esta limitación será discutida más adelante.
A partir de una muestra intencional no probabi lística, se estudiaron 144 individuos (76 hombres y 68 mujeres), con edades comprendidas entre los 18 y los 65 años y provenientes del estado Sucre (región nororiental de Venezuela). Las muestras se recolectaron de personas asistentes al Hospital Universitario "Antonio Patricio de Alcalá", ciudad de Cumaná, y la Escuela de Bioanálisis de la Universidad de Oriente (UDO), núcleo Sucre. Entre los asistentes se contó con donantes voluntarios de sangre, empleados del hospital y estudiantes universitarios, entre otros, provenientes de la población general urbana de Cumaná; se trataba de individuos mestizos cuya composición genética es el resultado de una mezcla de aportes amerindios, europeos y africanos (29- 32). Los sujetos participantes debían cumplir con los siguientes criterios de inclusión: cuatro abuelos nacidos en el estado Sucre, no presentar relaciones de consanguinidad próxima entre sí, no padecer enfermedades agudas en el momento del estudio, ni tener trastornos de hiperglucemia (glucemia=110 mg/dl) o diabetes. Cada participante firmó el consentimiento informado aprobado por la Comisión de Bioética del Instituto Venezolano de Investigaciones Científicas (IVIC).
Pruebas bioquímicas y genéticas
A cada sujeto se le tomaron 10 ml de sangre por venopunción en el antebrazo después de un ayuno de 12 a 14 horas, con el fin de hacer pruebas bioquímicas, el análisis hematológico completo y la extracción del ADN (para estas dos últimas se utilizó como anticoagulante el EDTA-Na 4 al 15 %).
Se determinaron la glucemia y el perfil lipídico, empleando los reactivos y protocolos de Bayer Corporation (2000) (33) con el equipo Analizador Express 550®. La medición de la glucemia se incluyó en una segunda fase del trabajo de campo, razón por la cual no se contó con este valor para todos los participantes. El análisis hematológico se hizo en un contador automatizado (contador Coulter ).
El aislamiento del ADN se hizo mediante el método de extracción salina de Lahiri y Nurnberger (34), estandarizado en el Laboratorio de Genética Humana del Instituto Venezolano de Investigaciones Científicas. Para la identificación de los polimorfismos genéticos se utilizó la técnica de reacción en cadena de la polimerasa (PCR). En el cuadro 1 se muestran los oligómeros y las enzimas de restricción utilizadas para cada polimorfismo de nucleótido simple estudiado. Se empleó el método descrito por Saiki, et al. (35), con modificaciones para una mezcla de PCR de 15 µl de volumen final. Las digestiones con enzimas de restricción se ajustaron a las especificaciones técnicas del fabricante, para un volumen final de 10 µl, utilizando la cantidad mínima requerida de enzima durante un tiempo de incubación de 16 horas. La asignación de los genotipos se efectuó mediante electroforesis en geles de poliacrilamida 29: 1 al 8, 10 y 12 % según los tamaños de los fragmentos. El revelado se hizo por tinción con nitrato de plata (36). El error técnico en la asignación de genotipos se descartó usando siempre muestras de control con genotipos conocidos (control positivo) y un control sin enzima (control negativo), en cada digestión. También, se hicieron controles de calidad aleatorios de un porcentaje de muestras (10-15 %) para cada SNP, con el fin de replicar resultados luego de la asignación definitiva. No se hizo secuenciación.
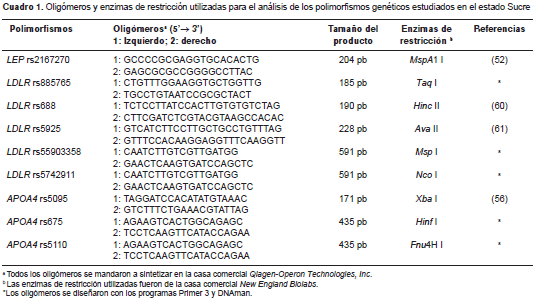
Fenotipos asociados al sobrepeso, la obesidad y el riesgo de enfermedades crónicas no transmisibles
Se tomaron las medidas antropométricas de peso, talla y circunferencias de cintura y de cadera según los protocolos del Programa Biológico Internacional (37), utilizando una balanza clínica Detecto-Medic® y una cinta métrica.
Se calculó el IMC y se clasificó a cada sujeto de acuerdo con los criterios de los National Institutes of Health (NIH) (38) y la Organización Mundial de la Salud (OMS) (1), con base en las categorías de peso normal (18,5≤IMC≤24,9 kg/m2), sobrepeso (25≤IMC≤29,9 kg/m2) y obesidad (IMC = 30 kg/m2). Para clasificar el riesgo de padecer enfermedades crónicas, se utilizaron el índice cintura-cadera y la circunferencia de cintura (39,40). Los criterios fueron los siguientes: niveles de riesgo alto para los hombres, índice de cintura-cadera mayor o igual a 1,0 y circunferencia de cintura mayor de 102 cm, y para las mujeres, mayor o igual a 0,85 y mayor de 88 cm, respectivamente (30). Como indicador de la adiposidad corporal se calculó el índice de conicidad (41,42), que aún no cuenta con puntos de corte para los niveles de riesgo.
Las categorías de clasificación para definir las dislipidemias fueron las del Adult Treatment Panel III (ATP III) (43,44) y el Consenso Venezolano de Lípidos (45), que definen el riesgo alto para el colesterol total como igual o mayor de 200 mg/dl, para triglicéridos, como igual o mayor de 150 mg/dl, para el colesterol de lipoproteínas de alta densidad (colesterol HDL), como menor de 40 mg/dl, y para el colesterol de lipoproteínas de baja densidad (colesterol LDL), como igual o mayor de 130 mg/dl.
Características biodemográficas, hábitos y estilo de vida
Los participantes respondieron un cuestionario con información sobre sus características bio-demográficas (edad, sexo, lugar de nacimiento), sus hábitos y estilo de vida (tabaquismo, consumo de alcohol y actividad física), sus antecedentes médicos personales y familiares (cardiovasculares, hipertensión, diabetes, dislipidemias), y su estrato socioeconómico, para su clasificación según el método de Graffar-Méndez-Castellano (46).
Análisis estadístico
Se utilizó el programa Statistical Package for the Social Sciences (SPSS) ®, versión 21,0 (47). Se calcularon las frecuencias para las variables biodemográficas, hábitos, estilo de vida, IMC y riesgo de enfermedades crónicas según los indicadores antropométricos y bioquímicos. Se obtuvieron medias y desviaciones estándar para las variables cuantitativas hematológicas, bioquímicas y antropométricas discriminadas por sexo. Se evaluaron las diferencias entre uno y otro sexo para dichas variables mediante la prueba t de Student después de la transformación logarítmica de cada una en caso de no cumplirse las asunciones de ´parametricidad´ (sic.) (normalidad).
Se determinaron las frecuencias genotípicas y alélicas para cada polimorfismo por frecuencias absolutas (48) en todos los sujetos y en los grupos clasificados según el valor del IMC (normal: <25 kg/m2; alto: sobrepeso y obesidad: IMC=25 kg/m2). Se calculó el equilibrio de Hardy-Weinberg en cada grupo y para cada polimorfismo mediante la prueba de ji al cuadrado con el programa MAXLIK y con base en el método de máxima verosimilitud descrito por Reed y Schull (49). Luego, se evaluó la asociación entre genotipos y alelos según IMC o sexo, utilizando la prueba de ji al cuadrado o el estadístico exacto de Fisher según el número de individuos en cada genotipo.
También, se construyeron tablas de contingencia para probar la asociación entre los genotipos de cada polimorfismo (ancestral con relación a portadores del alelo variante) y las categorías de riesgo con base en el índice cintura-cadera, la circunferencia de la cintura, el colesterol total, los triglicéridos, el colesterol HDL, y el colesterol LDL (riesgo bajo Vs . riesgo alto), mediante la prueba de ji al cuadrado, el test exacto de Fisher y las relaciones probabilísticas de riesgo ( odds ratio, OR ) con sus correspondientes intervalos de confianza.
Mediante el análisis de la varianza (ANOVA), se determinó la diferencia entre los promedios de las variables bioquímicas y antropométricas para los distintos genotipos de los nueve polimorfismos analizados por separado. Cada una de estas variables se controló previamente frente a la edad, el sexo, el tabaquismo y el consumo de alcohol, los hábitos de caminata y de ejercicio, y los antecedentes de enfermedades en la familia, usando modelos multivariados (regresión lineal múltiple). Cuando los análisis de varianza evidenciaron la existencia de diferencias significativas, se efectuaron pruebas post hoc , como la prueba de comparaciones múltiples de Bonferroni, para determinar entre cuáles genotipos había diferencias estadísticamente significativas. En los casos para los cuales no se encontró homogeneidad de varianzas, se calcularon las pruebas estadísticas de Games-Howell, de Welch y de Brown-Forsythe. También, se hicieron prue bas no paramétricas (Kruskal-Wallis, mediana y Jonckheere-Terpstra, o U de Mann-Whitney), utilizando para los cálculos los residuos de las regresiones (variables corregidas).
La corrección de Bonferroni para múltiples comparaciones (50), se aplicó en todas las pruebas estadísticas con el fin de minimizar la probabilidad de cometer un error de tipo I (51).
Resultados
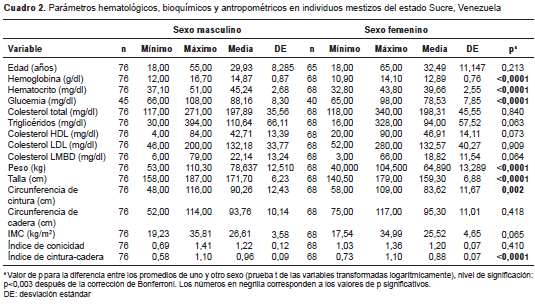
La caracterización de los individuos según las variables hematológicas, bioquímicas y antropométricas, se muestra en el cuadro 2.
Según se refleja en el cuadro 3, se trató de un grupo mayoritariamente joven, perteneciente a los estratos socioeconómicos pobre y medio. El 90,7 % afirmó no tener antecedentes personales de enfermedad, pero sí familiares (51,4 %), pues uno o varios parientes (principalmente padres, abuelos y tíos) presentaban una o más afecciones. Los sujetos en su mayoría informaron ser "no fumadores" (74,8 %); en cuanto al alcohol, en la categoría "no tomador" se registró el 66,9 % de los sujetos, porcentaje que incluía a quienes lo consumían de forma ocasional, llamados "bebedores sociales" (47,5 %), y quienes nunca lo consumían (19,4 %). El 62,8 % de los participantes indicó que sí hacía caminatas, y más de la mitad respondió que no practicaba ningún deporte (61,2 %).
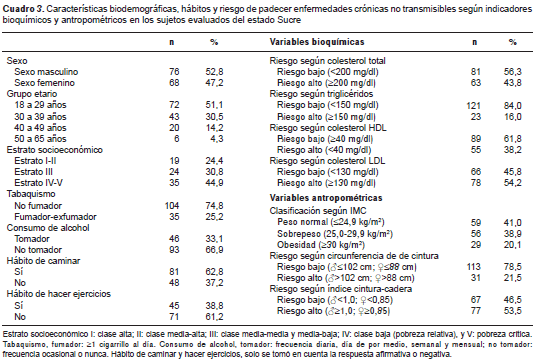
En cuanto al IMC, el 41 % de los individuos se clasificó como normal; solo cinco mujeres presentaron valores menores de 18,5 kg/m2, lo que corresponde a bajo peso, pero, en concordancia con los objetivos del trabajo, estas participantes se incluyeron en el grupo con IMC normal. El 38,9 % tuvo sobrepeso y el 20,1 % presentó obesidad. Es decir que según este indicador del estado nutricional, más de la mitad de los sujetos evaluados (59 %) padecía malnutrición por exceso. En cuanto a los índices relacionados con la distribución de la grasa corporal, según el índice cintura-cadera, 53,5 % estaba en riesgo alto de padecer enfermedades crónicas y, según la circunferencia de cintura, el 21,5 %. Según la clasificación del perfil lipídico, los individuos evaluados estaban en riesgo alto de padecer enfermedades crónicas no transmisibles: 54,2 % por el colesterol LDL; 43,8 % por el colesterol total, 38,2 % por el colesterol HDL, y 16 % por los triglicéridos.
La distribución de las frecuencias genotípicas masculinas y femeninas para cada uno de los nueve polimorfismos estudiados, no resultaron significativamente diferentes (no se presentan los datos), razón por la cual no se discriminó por sexo en el análisis.
Ocho de los polimorfismos estudiados se encontraron en equilibrio de Hardy-Weinberg para todos los subgrupos y grupos totales (no se presentan los datos); el único subgrupo en el que no se halló equilibrio fue el de los individuos con IMC normal para el polimorfismo LEP rs2167270 ( c 2 =4,875, un grado de libertad). Es probable que esta falta de equilibrio por déficit de heterocigotos G/A y exceso de homocigotos A/A, se deba a que el tamaño de la muestra era relativamente pequeño (n=59) y a un error propio del muestreo (sesgo involuntario), luego de haberse descartado el error técnico en la asignación de genotipos.
Las frecuencias genotípicas y alélicas de los nueve polimorfismos se comportaron de forma similar entre los grupos con IMC normal e IMC alto (cuadro 4); en consecuencia, la asociación de los genotipos o alelos con algún fenotipo específico de IMC, no resultó estadísticamente significativa.
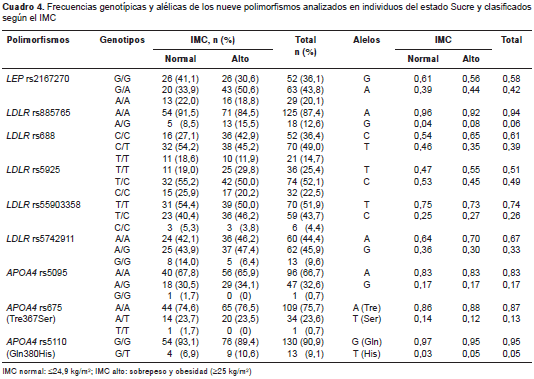
En cambio, la evaluación de la asociación entre los genotipos de cada variante y alguna de las categorías de riesgo debido al índice cintura-cadera, la circunferencia de la cintura, el colesterol total, los triglicéridos, el colesterol HDL o el colesterol LDL, reveló un resultado estadísticamente significativo luego de la corrección de Bonferroni para el polimorfismo rs5742911 del gen LDLR , en el cual se encontró asociación entre los homocigotos del alelo ancestral (A/A) y la categoría de riesgo alto para el colesterol HDL, con un OR de 2,944 (IC 95% 1,446-5,996) (cuadro 5).

En el cuadro 6 se muestran las variables inde pendientes que resultaron significativas en la regresión múltiple hecha para corregir las medidas antropométricas y bioquímicas.

En lo concerniente al ANOVA y las pruebas no paramétricas, después de aplicar la corrección de Bonferroni solo uno de los resultados se mantuvo estadísticamente significativo: el correspondiente a la diferencia entre el colesterol HDL del genotipo rs5742911 del gen LDLR (cuadro 7), con promedios de 41,50±14,81 mg/dl en los homocigotos A/A, de 45,00±12,07 mg/dl en los heterocigotos A/G, y de 47,17±9,43 mg/dl en los homocigotos G/G.
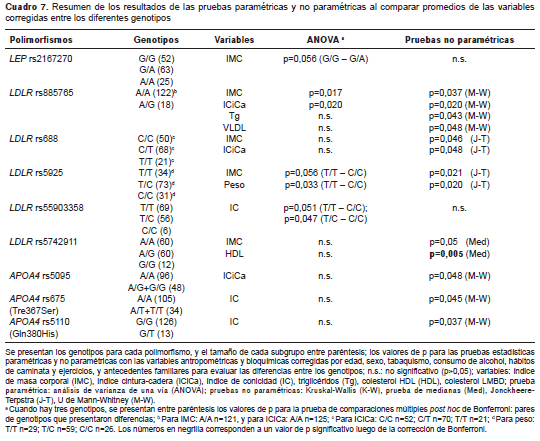
Aunque no llegaron a ser significativos, vale la pena destacar algunos resultados. En cuanto al IMC corregido, en el rs2167270 del gen LEP el promedio de los homocigotos del alelo ancestral (G/G) fue de 25,04±4,20 kg/m2, el de los homocigotos A/A fue de 25,87±4,08 kg/m2, y el de los heterocigotos G/A fue de 27,02±4,08 kg/m2. Para cuatro de las cinco variantes del gen LDLR estudiadas, también se hallaron diferencias en el IMC corregido; los polimorfismos rs688, rs5925 y rs5742911 presentaron mayores promedios para los genotipos ancestrales, con valores de 26,86±4,12 kg/m2 (C/C), 27,32±3,99 kg/m2 (T/T), y 26,07±4,41 kg/m2 (A/A), respectivamente, en contraste con los genotipos homocigotos del alelo variante cuyos promedios estuvieron entre 23 y 25 kg/m2.
Otras variables antropométricas, como el índice cintura-cadera, el peso y el índice de conicidad, para estos polimorfismos del LDLR también tuvieron mayores promedios en los genotipos homocigotos ancestrales. Solo el rs885765 exhibió promedios más altos entre los portadores del alelo variante (A/G), tanto para los indicadores antropométricos de IMC y el índice de cintura-cadera, como de triglicéridos y de colesterol LMBD. Los tres SNP del gen APOA4 presentaron mayores promedios entre los homocigotos ancestrales para las variables antropométricas del índice cintura-cadera (rs5095) y el índice de conicidad (rs675 y rs5110).
Discusión
Las variables antropométricas de peso, talla, circunferencia de cintura e índice cintura-cadera mostraron el dimorfismo sexual esperado para sujetos adultos. La hemoglobina, el hematocrito y la glucemia presentaron medias significativamente diferentes entre uno y otro sexo, en tanto que las variables del perfil lipídico no presentaron diferencias significativas entre hombres y mujeres, y se encontraron dentro de los límites de la normalidad, excepto los promedios para el colesterol LDL, que resultaron ligeramente altos. De la misma forma, la media del IMC se ubicó en la categoría de sobrepeso. Como era de esperarse, la edad y el sexo influyeron sobre la mayoría de las variables cuantitativas estudiadas. La presencia o ausencia de antecedentes familiares de enfermedad tuvo un efecto importante en el ajuste de las medidas originales, tanto bioquímicas como antropométricas.
Las cifras de sobrepeso y obesidad para el periodo 2008-2010 en el estado Sucre, estuvieron entre las más altas de Venezuela (4); los resultados del presente estudio coinciden con esta alta frecuencia de malnutrición por exceso, lo que, aunado a la presencia de hipercolesterolemia en la mitad del grupo evaluado, sugiere que los habitantes de esta región podrían considerarse vulnerables a las enfermedades crónicas o en riesgo de complicaciones de otras enfermedades concomitantes como las cardiovasculares, la hipertensión arterial sistémica y la diabetes, entre otras (3).
En la búsqueda de asociaciones entre las variantes genéticas estudiadas con los fenotipos de sobrepeso, obesidad y riesgo de padecer enfermedades crónicas no transmisibles, se encontró que en el polimorfismo rs2167270 de LEP no hubo asociación de los genotipos y alelos con el IMC (cuadro 4), lo cual también se ha descrito en estudios en Finlandia (52) e Italia (53). Sin embargo, cuando se analizaron los promedios corregidos de las variables antropométricas y bioquímicas según los genotipos, se pudo observar que el IMC de los sujetos heterocigotos G/A resultó más elevado que en los homocigotos de cada tipo, tendencia que ya se ha reportado en un estudio previo (12), pero solamente en mujeres obesas, probablemente debido al desequilibrio de ligamiento de este SNP con otros loci de LEP ( 12,13). Las mutaciones de las regiones codificantes en este gen son muy poco frecuentes y producen una obesidad extrema de inicio temprano; la mayoría de los polimorfismos encontrados en la región 5´ no traducida ( como rs2167270), se encuentran en desequilibrio de ligamiento con otras variantes que podrían tener efectos directos sobre el fenotipo, pero el grado de desequilibrio puede variar entre poblaciones o tener mayor intensidad a medida que la cantidad de grasa corporal aumenta (12).
En la presente investigación solo se halló significación estadística para el rs5742911 del gen LDLR en la asociación del genotipo ancestral con el alto riesgo debido al colesterol HDL, y para las diferencias en los promedios corregidos de esta misma variable bioquímica; los valores para el colesterol HDL entre los genotipos formaron un gradiente, en el cual los homocigotos ancestrales A/A presentaron el promedio más bajo, seguidos de los heterocigotos A/G, en tanto que el promedio más alto fue el los homocigotos del tipo de la variante G/G, comportamiento que podría sugerir un efecto de dosis, es decir, una mayor tendencia a la disminución de los niveles de colesterol HDL en la medida en que se presenta el alelo A de forma heterocigota u homocigota. En este sentido, los hallazgos de este estudio sugieren que este polimorfismo genético puede resultar útil como indicador del riesgo de enfermedades crónicas no transmisibles.
En el polimorfismo rs688 del gen LDLR, la mayor frecuencia del alelo ancestral C entre los individuos con IMC alto (cuadro 4), aunque no estadísticamente significativa, coincide con los resultados de un estudio en australianos hipertensos (19). Otra investigación reportó estos hallazgos para el rs688, y encontró asociación para tres variantes más del LDLR (rs5925, rs55903358 y rs5742911) en amerindios brasileños (18). Las diferencias de los resultados de los grupos amerindios y los individuos del presente estudio, puede deberse a que los sujetos del estado Sucre provienen de una población mestiza con distintos grados de aporte europeo, amerindio y africano (29-32), mientras los sujetos estudiados por Mattevi, et al. (18), pertenecen a cinco poblados indígenas de Brasil con muy bajo o ningún grado de mestizaje con criollos brasileños.
El efecto funcional para estos SNP de LDLR no se ha determinado con exactitud en la obesidad. Tratándose de cambios en el intrón (rs885765) o en regiones no traducidas del gen (rs55903358 y rs5742911), es posible que su papel sea regulador; por su parte, los cambios dentro de los exones (rs688 y rs5925) son sinónimos; sin embargo, hay estudios que revelan su influencia sobre la modulación del proceso de corte y empalme del gen, produciendo diferencias en los niveles del colesterol LDL (54,55).
Para el gen APOA4, ninguno de los alelos o genotipos de los tres polimorfismos estudiados demostraron asociación con alguna de las categorías de IMC (cuadro 4), resultado que se ha reportado previamente (56), o de las otras clasificaciones para el riesgo de padecer enfermedades crónicas no transmisibles. Sin embargo, el análisis de los promedios corregidos sugiere que, para los indicadores de la distribución de grasa corporal (índice cintura-cadera e índice de conicidad), los valores más altos se hallaron entre los sujetos con los genotipos ancestrales. El rs5110 (Gln380His) exhibió promedios de 1,22±0,104 para el índice de conicidad en el genotipo ancestral G/G (Gln/Gln) y de 1,19±0,077 para los heterocigotos G/T (Gln/His), lo que concuerda con la hipótesis del perfil anti-aterogénico protector asociado con la presencia del alelo variante His380, que es muy poco frecuente, planteada por Fisher, et al ., en un amplio estudio de cohorte (24). La apolipoproteína A-IV con His380 adopta una conformación más estrechamente plegada, con mayor contenido de hélices alfa y aparentemente tiene una mayor afinidad por los fosfolípidos, lo cual podría permitirle una penetración más profunda en la monocapa de estos, y explicaría la mayor activación de la lecitina colesterol acil-transferasa (24).
Los resultados de este estudio, aun cuando son preliminares, llevan a plantear si este grupo mestizo del estado Sucre es un ejemplo de la hipótesis de Neel sobre el "genotipo ahorrador", la cual plantea que en la historia evolutiva del hombre se favoreció la selección de variantes genéticas excepcionalmente eficientes para el consumo y utilización del alimento y para el ahorro de energía, debido a las condiciones hostiles del medio ambiente en esos momentos, y que, al adoptar las condiciones del denominado estilo de vida occidental, dicho sistema, que favoreció la supervivencia en tiempos de hambruna, aumentó la vulnerabilidad ante trastornos metabólicos como la diabetes (57). En el estudio, en siete de los nueve polimorfismos evaluados (rs688, rs5925, rs55903358, rs5742911, rs5095, rs675, rs5110), se observó una propensión al sobrepeso o la obesidad, a una mayor adiposidad abdominal y al riesgo de enfermedades crónicas no transmisibles en los genotipos ancestrales.
Las enfermedades complejas son el resultado de interacciones entre causas ambientales y genéticas; estas últimas se deben a polimorfismos comunes que tienen efectos pequeños y aditivos, lo cual representa una dificultad para detectar asociaciones apreciables y lograr suficiente reproducibilidad en distintas poblaciones (58,59).
Desde el punto de vista del componente ambiental, la frecuencia de sobrepeso, la obesidad y los niveles elevados de colesterol, seguramente también se asociaron con los hábitos alimentarios y un estilo de vida sedentario en los sujetos del estudio. La evaluación de la frecuencia del consumo de alimentos en 84 de los entrevistados, reveló hábitos de consumo diario de grasas, aceites, azúcar y sal en sus comidas (no se presentan los datos).
Las respuestas disponibles sobre actividad física se referían únicamente a la práctica de caminatas y ejercicios, pero no a su frecuencia e intensidad, debido a la resistencia de los participantes a responder dichas preguntas, lo que implica que esta posible relación con los hábitos y el estilo de vida debe considerarse en futuros estudios.
La principal limitación de la presente investigación fue el número de individuos evaluados, así como el tipo de muestreo (no probabilístico), pues este restringe la posibilidad de hacer inferencias precisas sobre la población del estado Sucre. Estas debilidades del estudio se debieron al costo económico y a las restricciones logísticas para hacer un muestreo aleatorio con un tamaño representativo. Sin embargo, el estudio constituyó una primera aproximación a la situación del problema planteado y permitió obtener resultados importantes (como el polimorfismo rs5742911 del gen LDLR ), a pesar del tamaño de la muestra.
Por primera vez se caracterizaron en individuos venezolanos originarios del estado Sucre estos polimorfismos previamente publicados en la literatura científica, utilizando el criterio genético de los cuatro abuelos nacidos en el estado para seleccionar a los sujetos de la muestra, lo cual es de suma relevancia para garantizar la mayor homogeneidad genética posible. Es importante aumentar, en lo posible, el tamaño de la muestra y continuar analizando variantes genéticas invo lucradas en las principales vías metabólicas relacionadas con el aumento o disminución de la grasa corporal, con el fin de complementar el conocimiento para el diagnóstico y el tratamiento de grupos específicos de riesgo.
Agradecemos a todos los participantes del estudio, al personal del Banco de Sangre, del Laboratorio General y del Servicio de Nutrición y Dietética del Hospital Universitario "Antonio Patricio de Alcalá", ciudad de Cumaná, así como a los colaboradores voluntarios y pasantes de la Escuela de Bioanálisis de la Universidad de Oriente, núcleo de Sucre. También, hacemos extensivo nuestro agradecimiento a todo el equipo que labora en el Laboratorio de Genética Humana del Instituto Venezolano de Investigaciones Científicas.
Los autores declaran que no existe ningún tipo de conflicto de intereses.
El financiamiento de este proyecto fue otorgado por el Instituto Venezolano de Investigaciones Científicas.
Correspondencia: Greta Rodríguez-Arroyo, Laboratorio de Genética Humana, Centro de Medicina Experimental, Instituto Venezolano de Investigaciones Científicas, Carretera Panamericana, Km 11, Altos de Pipe, Caracas, Venezuela Teléfono: (58) 212 504 1491. Fax: (58) 212 504 1086 gretamrb@gmail.com y gmrodrig@ivic.gob.ve
1. World Health Organization. Diet, nutrition and the prevention of chronic diseases. WHO Technical Report Series 916. WHO/FAO Expert Consultation. Geneva: World Health Organization ; 2003. p. 1-149. [ Links ]
2. World Health Organization. Global health risks: Mortality and burden of disease attributable to selected major risks. Geneva: World Health Organization; 2009. p. 1-62. [ Links ]
3. Friedman JM. Obesity in the new millennium. Nature. 2000;404:632-4. http://dx.doi.org/10.1038/35007504 [ Links ]
4. Instituto Nacional de Nutrición. Sobrepeso y obesidad en Venezuela (prevalencia y factores condicionantes). Colección Lecciones Institucionales. Instituto Nacional de Nutrición. Caracas: Fondo Editorial Gente de Maíz; 2013. p. 1-148. [ Links ]
5. Warden CH, Fisler JS. Obesity: From animal models to human genetics to practical applications. Prog Mol Biol Transl Sci. 2010;94:373-89 . http://dx.doi.org/10.1016/S1877-1173(10)94013-1 [ Links ]
6. Urano T, Inoue S. Recent genetic discoveries in osteo porosis, sarcopenia and obesity. Endocr J. 2015;62:475-84. http://dx.doi.org/10.1507/endocrj.EJ15-0154 [ Links ]
7. Yu Z, Han S, Cao X, Zhu C, Wang X, Guo X. Genetic polymorphisms in adipokine genes and a systematic review and meta-analysis. Obesity. (Silver Spring). 2012;20:396- 406. http://dx.doi.org/10.1038/oby.2011.148 [ Links ]
8. Kohan AB, Wang F, Lo CM, Liu M, Tso P. ApoA-IV: Current and emerging roles in intestinal lipid metabolism, glucose homeostasis, and satiety. Am J Physiol Gastrointest Liver Physiol. 2015;308:G472-81. http://dx.doi.org/10.1152/ajpgi.00098.2014 [ Links ]
9. Wang F, Kohan AB, Lo CM, Liu M, Howles P, Tso P. Apolipoprotein A-IV: A protein intimately involved in metabolism. J Lipid Res. 2015;56:1403-18. http://dx.doi.org/10.1194/jlr.R052753 [ Links ]
10. Pérusse L, Rankinen T, Zuberi A, Chagnon YC, Weisnagel SJ, Argyropoulos G, et al . The human obesity gene map: The 2004 update. Obes Res. 2005;13:381-490. http://dx.doi.org/10.1038/oby.2005.50 [ Links ]
11. Rankinen T, Zuberi A, Chagnon YC, Weisnagel SJ, Argyropoulos G, Walts B, et al . The human obesity gene map: The 2005 update. Obesity (Silver Spring). 2006;14:529- 644. http://dx.doi.org/10.1038/oby.2006.71 [ Links ]
12. Li WD, Reed DR, Lee JH, Xu W, Kilker RL, Sodam BR, et al . Sequence variants in the 5´ flanking region of the leptin gene are associated with obesity in women. Ann Hum Genet. 1999;63:227-34. http://dx.doi.org/10.1046/j.1469-1809.1999.6330227.x [ Links ]
13. Jiang Y, Wilk JB, Borecki I, Williamson S, DeStefano AL, Xu G, et al . Common variants in the 5´ region of the leptin gene are associated with body mass index in men from the National Heart, Lung, and Blood Institute Family Heart Study. Am J Hum Genet. 2004;75:220-30. http://dx.doi.org/10.1086/422699 [ Links ]
14. Hart Sailors ML, Folsom AR, Ballantyne CM, Hoelscher DM, Jackson AS, Linda Kao WH, et al . Genetic variation and decreased risk for obesity in the Atherosclerosis Risk in Communities Study. Diabetes Obes Metab. 2007;9:548-57. http://dx.doi.org/10.1111/j.1463-1326.2006.00637.x [ Links ]
15. Mizuta E, Kokubo Y, Yamanaka I, Miyamoto Y, Okayama A, Yoshimasa Y, et al . Leptin gene and leptin receptor gene polymorphisms are associated with sweet preference and obesity. Hypertens Res. 2008;31:1069-77. http://dx.doi.org/10.1291/hypres.31.1069 [ Links ]
16. Walsh S, Haddad CJ, Kostek MA, Angelopoulos TJ, Clarkson PM, Gordon PM, et al . Leptin and leptin receptor genetic variants associate with habitual physical activity and the arm body composition response to resistance training. Gene. 2012;510:66-70. http://dx.doi.org/10.1016/j.gene.2012.08.020 [ Links ]
17. Lesseur C, Armstrong DA, Paquette AG, Koestler DC, Padbury JF, Marsit CJ. Tissue-specific leptin promoter DNA methylation is associated with maternal and infant perinatal factors. Mol Cell Endocrinol. 2013;381:160-7. http://dx.doi.org/10.1016/j.mce.2013.07.024 [ Links ]
18. Mattevi VS, Coimbra CE Jr, Santos RV, Salzano FM, Hutz MH. Association of the low-density lipoprotein receptor gene with obesity in Native American populations. Hum Genet. 2000;106:546-52. http://dx.doi.org/10.1007/s004390000299 [ Links ]
19. Zee RYL, Schrader AP, Robinson BG, Griffiths LR, Morris BJ. Association of HincII RFLP of low density lipoprotein receptor gene with obesity in essential hypertensives. Clin Genet. 1995;47:118-21. http://dx.doi.org/10.1111/j.1399-0004.1995.tb03942.x [ Links ]
20. Menzel HJ, Boerwinkle E, Schrangl-Will S, Utermann G. Human apolipoprotein A-IV polymorphism: Frequency and effect on lipid and lipoprotein levels. Hum Genet. 1988;79:368-72. [ Links ]
21. von Eckardstein A, Funke H, Chirazi A, Chen- Haudenschild C, Schulte H, Schonfeld R, et al . Sex-specific effects of the glutamine/histidine polymorphism in apo A - IV on HDL metabolism. Arterioscler Thromb. 1994;14 :1114-20. 1 http://dx.doi.org/0.1161/01.ATV.14.7.1114 [ Links ]
22. Wong WM, Hawe E, Li LK, Miller GJ, Nicaud V, Pennacchio LA, et al . Apolipoprotein AIV gene variant S347 is associated with increased risk of coronary heart disease and lower plasma apolipoprotein AIV levels. Circ Res. 2003;92:969-75. http://dx.doi.org/10.1161/01.RES.0000069688.94567.7A [ Links ]
23. Dallongeville J, Delcroix AG, Wagner A, Ducimetière P, Ruidavets JB, Arveiler D, et al . The APOA4 Thr347- >Ser347 polymorphism is not a major risk factor of obesity. Obes Res. 2005;13:2132-8. http://dx.doi.org/10.1038/oby.2005.264 [ Links ]
24. Fisher RM, Burke H, Nicaud V, Ehnholm C, Humphries SE. Effect of variation in the apo A-IV gene on body mass index and fasting and postprandial lipids in the European Atherosclerosis Research Study II. J Lipid Res. 1999;40: 287-94. [ Links ]
25. Lefevre M, Lovejoy JC, DeFelice SM, Keener JW, Bray GA, Ryan DH, et al . Common apolipoprotein A-IV variants are associated with differences in body mass index levels and percentage body fat. Int J Obes Relat Metab Disord. 2000;24:945-53. [ Links ]
26. Ordovás JM, Schaefer EJ. Genetic determinants of plasma lipid response to dietary intervention: The role of the APOA1/C3/A4 gene cluster and the APOE gene. Br J Nutr. 2000;83:S127-S36. http://dx.doi.org/10.1017/S0007114500001069 [ Links ]
27. Fiegenbaum M, Hutz MH. Further evidence for the association between obesity-related traits and the apolipo- protein A-IV gene. Int J Obes Relat Metab Disord. 2003;27: 484-90. http://dx.doi.org/10.1038/sj.ijo.0802256 [ Links ]
28. Feitosa MF, An P, Ordovás JM, Ketkar S, Hopkins PN, Straka RJ, et al . Association of gene variants with lipid levels in response to fenofibrate is influenced by metabolic syndrome status. Atherosclerosis. 2011;215:435-9. http://dx.doi.org/10.1016/j.atherosclerosis.2011.01.011 [ Links ]
29. Rodríguez-Larralde A, Castro-de Guerra D, González- Coira M, Morales J. Frecuencia génica y porcentaje de mezcla en diferentes áreas geográficas de Venezuela, de acuerdo a los grupos Rh y ABO. Interciencia. 2001;26: 8- 12. [ Links ]
30. Vívenes-de Lugo M, Rodríguez-Larralde A, Castro- de Guerra D. Beta-globin gene cluster haplotypes as an evidence of African gene flow to the northeastern coast of Venezuela. Am J Hum Biol. 2003;15:29-37. http://dx.doi.org/10.1002/ajhb.10120 [ Links ]
31. Vívenes-de Lugo M, Rodríguez-Larralde A, Guerrero B, Castro-de Guerra D. Ethnic-geographic variation of the val34leu polymorph ism of coagulation factor XIII and its distribution in American admixed populations. Internet J Biol Anthropol. 2008;2. http://dx.doi.org/10.5580/12bd [ Links ]
32. Castro-de Guerra D, Figuera-Pérez C, Izaguirre MH, Arroyo-Barahona E, Rodríguez-Larralde A, Vívenes- de Lugo M. Gender differences in ancestral contribution and admixture in Venezuelan populations. Hum Biol. 2011;83: 345-61. http://dx.doi.org/10.3378/027.083.0302 [ Links ]
33. Bauer J. Análisis clínico. Métodos e interpretación. 9ª edición. Barcelona: Editorial Reverté; 1986. p. 1-1302. [ Links ]
34. Lahiri DK, Nurnberger JI Jr. A rapid non-enzymatic method for the preparation of HMW DNA from blood for RFLP studies. Nucleic Acids Res. 1991;19:5444. http://dx.doi.org/10.1093/nar/19.19.5444 [ Links ]
35. Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, et al . Primer directed enzymatic amplification of DNA with a thermostable DNA polimerase. Science. 1988;239:487-91. http://dx.doi.org/10.1126/science.2448875 [ Links ]
36. Brandt B, Greger V, Yandell D, Passarge E, Horsthemke B. A simple and nonradioactive method for detecting the Rb1.20 DNA polymorphism in the retinoblastoma gene. Am J Hum Genet. 1992;51:1450-1. [ Links ]
37. Weiner JS, Lourie JA. Practical Human Biology. 2nd edition. London: Academic Press; 1981. p. 439. [ Links ]
38. World Health Organization. Obesity: Preventing and managing the global epidemic: Report of a WHO consulta-tion on obesity. WHO technical report series, 894. Geneva: WHO; 2000. p. 1-252. [ Links ]
39. Bray GA. Classification and evaluation of the obesities. Med Clin North Am. 1989;73:161-84. [ Links ]
40. Aronne LJ. Classification of obesity and assessment of obesity-related health risks. Obes Res. 2002;10:105S-15. [ Links ]
41. Valdéz R. A simple model-based index of abdominal adiposity. J Clin Epidemiol. 1991;44:955-6. http://dx.doi. org/10.1016/0895-4356(91)90059-I [ Links ]
42. Valdéz R, Seidell JC, Ahn YI, Weiss KM. A new index of abdominal adiposity as an indicator of risk for cardio- vascular disease. A cross-population study. Int J Obes Relat Metab Disord. 1993;17:77-82. [ Links ]
43. Grundy SM, Cleeman JI, Merz CN, Brewer HB Jr, Clark LT, Hunninghake DB, et al . Implications of recent clinical trials for the National Cholesterol Education Program Adult Treatment Panel III guidelines. Circulation. 2004;110:227-39. http://dx.doi.org/10.1161/01.CIR.0000133317.49796.0E [ Links ]
44. National Cholesterol Education Program (NCEP) Expert Panel on Detection, Evaluation, and Treatment of High Blood Cholesterol in Adults (Adult Treatment Panel III). Third Report of the National Cholesterol Education Program (NCEP) Expert Panel on Detection, Evaluation, and Treatment of High Blood Cholesterol in Adults (Adult Treatment Panel III) final report. Circulation. 2002;106: 3143-421. [ Links ]
45. Consenso Venezolano de Lípidos. International Lipid Information Bureau. Capítulo Venezuela. Caracas: Park Davis; 2000. p. 1-39. [ Links ]
46. Méndez-Castellano H, Méndez MC. Sociedad y estrati- ficación: método Graffar-Méndez-Castellano. Caracas: Editorial Fundacredesa; 1994. p. 1-206. [ Links ]
47. IBM Corp. Released 2012. IBM SPSS Statistics for Windows, Version 21.0. Armonk, NY: IBM Corp; 2012. [ Links ]
48. Cavalli-Sforza LL, Bodmer WF. The genetics of human populations. New York: Dover Publications; 1999. p. 1-965. [ Links ]
49. Re ed TE, Schull WJ. A general maximum likelihood method estimation program. Am J Hum Genet. 1968;20:579-80. [ Links ]
50. Bland JM, Altman DG. Multiple significance tests: The Bonferroni method. BMJ. 1995;310: 170. http://dx.doi.org/10.1136/bmj.310.6973.170 [ Links ]
51. Balding DJ. A tutorial on statistical methods for population association studies. Nat Rev Genet. 2006;7:781-91. http://dx.doi.org/10.1038/nrg1916 [ Links ]
52. Karvonen MK, Pesonen U, Heinonen P, Laakso M, Rissanen A, Naukkarinen H, et al . Identification of new sequence variants in the leptin gene. J Clin Endocrinol Metab. 1998;83:3239-42. [ Links ]
53. Lucantoni R, Ponti E, Berselli ME, Savia G, Minocci A, Calò G, et al . The A19G polymorphism in the 5´ untranslated region of the human obese gene does not affect leptin levels in severely obese patients. J Clin Endocrinol Metab. 2000;85:3589-91. http://dx.doi.org/10.1210/jcem.85.10.6860 [ Links ]
54. Lee JD, Hsiao KM, Wang TC, Lee TH, Kuo YW, Huang YC, et al . Mutual effect of rs688 and rs5925 in regulating low-density lipoprotein receptor splicing. DNA Cell Biol. 2014;33:869-75. http://dx.doi.org/10.1089/dna.2014.2577 [ Links ]
55. Medina MW, Gao F, Naidoo D, Rudel LL, Temel RE, McDaniel AL, et al . Coordinately regulated alternative splicing of genes involved in cholesterol biosynthesis and uptake. PLoS One. 2011;6:e19420. http://dx.doi.org/10.1371/journal.pone.0019420 [ Links ]
56. Zaiou M, Visvikis S, Gueguen R, Parra HJ, Fruchart JC, Siest G. DNA polymorphisms of human apolipoprotein A-IV gene: Frequency and effects on lipid, lipoprotein and apolipoprotein levels in a French population. Clin Genet. 1994;46:248-54. http://dx.doi.org/10.1111/j.1399-0004.1994.tb04235.x [ Links ]
57. Neel JV. Diabetes mellitus: A ´thrifty´ genotype rendered detrimental by ´progress´? Am J Hum Genet. 1962;14: 353- 62. [ Links ]
58. Chacín M, Rojas J, Pineda C, Rodríguez D, Núñez-Pacheco M, Márquez-Gómez M, et al . Predisposición humana a la obesidad, síndrome metabólico y diabetes: el genotipo ahorrador y la incorporación de los diabetogenes al genoma humano desde la Antropología Biológica. Diabetes Internacional. 2011;III:36-49. [ Links ]
59. Mattevi VS, Zembrzuski VM, Hutz MH. Association analysis of genes involved in the leptin-signaling pathway with obesity in Brazil. Int J Obes. 2002;26:1179-85. http://dx.doi.org/10.1038/sj.ijo.0802067 [ Links ]
60. Leitersdorf E, Hobbs HH. Human LDL receptor gene: HincII polymorphism detected by gene amplification. Nucleic Acids Res. 1988;16:7215. http://dx.doi.org/10.1093/nar/16.14.7215 [ Links ]
61. Ahn YI, Kamboh MI, Aston CE, Ferrell RE, Hamman RF. Role of common genetic polymorphisms in the LDL receptor gene in affecting plasma cholesterol levels in the general population. Arterioscler Thromb. 1994;14:663-70. http://dx.doi.org/10.1161/01.ATV.14.5.663 [ Links ]













